文章信息
- 王雪, 郭传影, 王侠, 曹生亚
- Wang Xue, Guo Chuanying, Wang Xia, Cao Shengya
- 基于TCGA数据集分析CSTF2基因在肝细胞癌中的表达和临床意义
- Expression and clinical signifi cance of CSTF2 gene in hepatocellular carcinoma based on TCGA dataset
- 实用肿瘤杂志, 2021, 36(6): 550-555
- Journal of Practical Oncology, 2021, 36(6): 550-555
-
通信作者
- 曹生亚, E-mail: 532970282@qq.com
-
文章历史
- 收稿日期:2021-03-08
肝细胞癌是常见的消化道恶性肿瘤,是原发性肝癌的主要病理类型[1-2]。中国是肝细胞癌高发的国家,多数患者在确诊时即为中晚期[3]。据报道我国肝细胞癌患者的5年生存率仅为14%,术后5年复发率高达70%[4]。肝细胞癌发生和发展密切相关的分子标志物可以用于患者的临床决策和预后判断。近年来,基因测序技术的发展为探究肝细胞癌的分子发病机制和寻找新型预后分子标志物开辟新途径。
裂解刺激因子亚单位2(cleavage stimulation factor subunit 2,CSTF2)基因负责编码一种核蛋白,在其N末端区域含有核糖核蛋白(ribonucleoprotein, RNP)型RNA结合域。该蛋白是CSTF的一个成员,对mRNA切割和多聚腺苷酸化至关重要[5]。据报道,CSTF2基因在小鼠和大鼠的雄性生殖细胞中表达升高[6]。新近研究表明,CSTF2在肿瘤的发生和发展中也起到了重要的作用。CSTF2被报道在肺癌和膀胱癌中表达异常升高,可作为潜在的新型分子靶标[7-8]。然而,目前国内外尚无研究报道CSTF2在肝细胞癌中的临床意义和作用机制。因此,本研究通过生物信息学方法探究CSTF2基因在肝细胞癌中的临床价值和预后意义,旨在为肝细胞癌提供新的分子标志物。
1 资料与方法 1.1 数据的下载与分析在癌症基因图谱(The Cancer Genome Atlas,TCGA)数据库上检索并下载肝细胞癌的原始转录组测序数据(level 3,HTSeq-FPKM)和患者的临床信息。其中包括肝细胞癌组织样本374例,正常组织样本50例。使用R 4.0.2软件提取CSTF2基因的表达量,使用Mann-Whitney U检验分析两组之间基因表达水平的差异。在分析患者临床病理特征时,删除资料缺失、不全的和生存时间为0的样本。在后续分析中,根据CSTF2在肝细胞癌患者组织中表达量的中位值(3.86),将肝细胞癌患者分为高表达组和低表达组,分析CSTF2基因表达与患者临床特征的关系。
1.2 CSTF2基因表达与患者预后关系的分析根据TCGA数据库中患者的生存随访数据,结合CSTF2基因表达,使用Kaplan-Meier法分析高表达组和低表达组患者总体生存率的差异。使用单因素和多因素Cox回归分析研究CSTF2在肝细胞癌中的预后价值。使用UALCAN在线数据库(http://ualcan.path.uab.edu/analysis.html)、GEPIA在线数据库(http://gepia.cancer-pku.cn)和OncoLnc数据库(http://www.oncolnc.org)验证CSTF2基因表达与患者总体生存率的关系。
1.3 CSTF2基因富集分析使用基因集富集分析(gene set enrichment analysis,GSEA)的GSEA 4.0.2分析软件研究CSTF2在肝细胞癌中的分子作用机制。分析的通路来自Molecular Signature Database(Msig-DB)数据库中的c2. cp. kegg. v7. 0. symbols. gmt数据集。用缺省加权富集法进行富集分析,随机组合1 000次。P < 0.05和错误发现率(false discovery rate,FDR) < 0.05的基因集作为显著富集的基因集。利用STRING数据库(https://string-db.org)构建与CSTF2蛋白互相作用的调节网络。
1.4 统计学分析利用R 4.0.2和IBM SPSS Statistics 25. 0软件进行统计学分析。非参数检验(Mann-Whitney U)用于分析CSTF2基因在肝细胞癌组织和正常组织中的表达差异。χ2检验用于分析CSTF2基因表达与患者临床病理特征的关系。Kaplan-Meier法和Cox回归分析用于分析CSTF2基因表达与患者预后的关系。以P < 0.05为差异具有统计学意义。
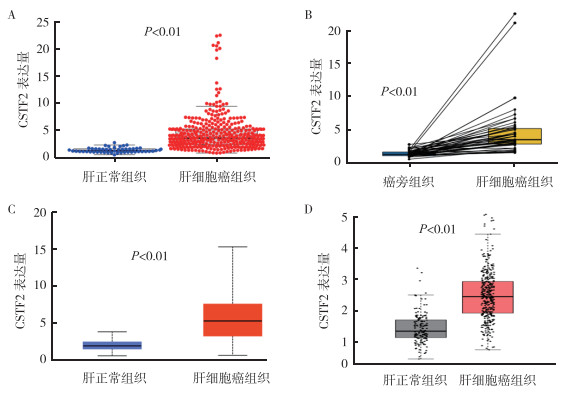
2 结果 2.1 CSTF2基因在肝细胞癌组织中高表达在TCGA数据库中,与正常组织比较,肝细胞癌组织中CSTF2基因表达水平升高(P < 0.01,图 1A)。根据TCGA数据库样本的命名规则,保留癌组织和相对应的癌旁组织分析发现,CSTF2基因表达水平高于相应的癌旁组织(P < 0.01,图 1B)。此外,在UALCAN数据库和GEPIA数据库中验证分析结果也显示,CSTF2基因在肝细胞癌组织中高表达(均P < 0.01,图 1C~1D)。
|
| 注 A:TCGA数库中CSTF2基因在肝细胞癌组织和肝正常组织中的表达情况;B:TCGA数据库中CSTF2基因在肝细胞癌组织和相应癌旁组织中的表达情况;C:UALCAN数据库中CSTF2基因在肝细胞癌组织和肝正常组织中的表达情况;D:GEPIA数据库CSTF2基因在肝细胞癌组织和肝正常组织中的表达情况;CSTF2:裂解刺激因子亚单位2(cleavage stimulation factor subunit 2) 图 1 CSTF2基因在肝细胞癌组织中的表达情况 Fig.1 Expression of CSTF2 gene in hepatocellular carcinoma |
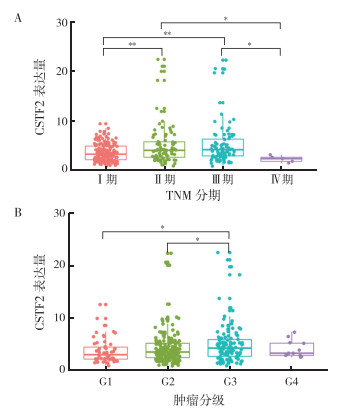
在TCGA数据库中,CSTF2基因低表达组和高表达组患者在TNM分期(P=0.024)和T分期(P=0.008)方面比较,差异均具有统计学意义(表 1)。Ⅱ期和Ⅲ期患者CSTF2基因表达均高于Ⅰ期(均P < 0.05),均低于Ⅳ期患者(均P < 0.05,图 2A)。同时,肿瘤分级G3级患者的CSTF2基因表达高于G1级和G2级的患者(均P < 0.05,图 2B)。
| 临床特征 | 低表达组 | 高表达组 | χ2值 | P值 |
| 年龄 | 0.218 | 0.640 | ||
| ≤60岁 | 64 | 67 | ||
| >60岁 | 54 | 50 | ||
| 性别 | 2.693 | 0.101 | ||
| 男性 | 75 | 86 | ||
| 女性 | 43 | 31 | ||
| 肿瘤分级 | 0.204 | 0.651 | ||
| G1+G2 | 78 | 74 | ||
| G3+G4 | 40 | 43 | ||
| TNM分期 | 5.119 | 0.024 | ||
| Ⅰ+Ⅱ期 | 87 | 70 | ||
| Ⅲ+Ⅳ期 | 31 | 47 | ||
| T分期 | 7.050 | 0.008 | ||
| T1+T2 | 90 | 67 | ||
| T3+T4 | 28 | 50 | ||
| M分期 | 0.667 | 0.414 | ||
| M0 | 114 | 115 | ||
| M1 | 4 | 2 | ||
| N分期 | 1.000 | 0.317 | ||
| N0 | 115 | 116 | ||
| N1 | 3 | 1 |
|
| 注 A:不同TNM分期患者CSTF2基因表达比较;B:不同肿瘤分级患者CSTF2基因表达比较;*P < 0.05;**P < 0.01;CSTF2:裂解刺激因子亚单位2(cleavage stimulation factor subunit 2) 图 2 CSTF2基因表达与肝细胞癌患者的肿瘤分级和TNM分期的关系 Fig.2 Relationship between CSTF2 gene expression and tumor grade and TNM stage in patients with hepatocellular carcinoma |
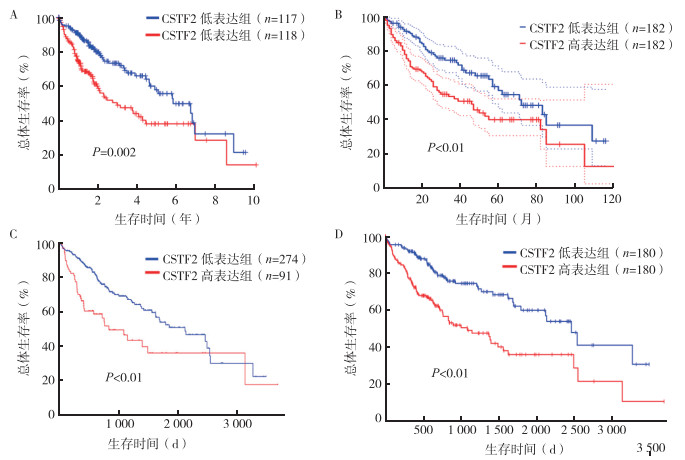
在TCGA数据库中,Kaplan‐Meier法分析发现,CSTF2基因高表达组患者的总体生存率低于低表达组(P=0.002,图 3A)。在GEPIA、UALCAN和OncoLnc数据库中,高表达组患者总体生存率也均低于低表达组(均P < 0.01,图 3B~3D)。
|
| 注 A:TCGA数据库中CSTF2基因高表达组和低表达组患者总体生存曲线;B:GEPIA数据库中CSTF2基因高表达组和低表达组患者总体生存曲线;C:UALCAN数据库中CSTF2基因高表达组和低表达组患者总体生存曲线;D:OncoLnc数据库中CSTF2基因高表达组和低表达组患者总体生存曲线;CSTF2:裂解刺激因子亚单位2(cleavage stimulation factor subunit 2) 图 3 各数据库中CSTF2基因高表达组和低表达组患者总体生存曲线比较 Fig.3 Comparison of survival curves of patients with high and low CSTF2 gene expression in each database |
单因素Cox回归分析显示,TNM分期(HR=1.865,95%CI:1.456~2.388,P < 0.01)、T分期(HR=1.804,95%CI:1.434~2.270,P < 0.01)和CSTF2基因表达(HR=1.120,95%CI:1.065~1.178,P < 0.01)均为影响患者预后的因素(表 2)。而多因素Cox回归分析显示,CSTF2基因表达为肝细胞癌患者的独立预后因素(HR=1.107,95%CI:1.045~1.172,P=0.001;表 2)。
| 因素 | 单因素分析 | 多因素分析 | |||||
| HR值 | 95%CI | P值 | HR值 | 95%CI | P值 | ||
| 年龄 | 1.005 | 0.987~1.023 | 0.591 | 1.011 | 0.991~1.031 | 0.287 | |
| 性别 | 0.780 | 0.487~1.249 | 0.301 | 0.898 | 0.527~1.531 | 0.692 | |
| 肿瘤分级 | 1.017 | 0.746~1.387 | 0.914 | 1.132 | 0.808~1.585 | 0.471 | |
| TNM分期 | 1.865 | 1.456~2.388 | < 0.01 | 1.108 | 0.415~2.959 | 0.837 | |
| T分期 | 1.804 | 1.434~2.270 | < 0.01 | 1.507 | 0.622~3.648 | 0.363 | |
| M分期 | 3.850 | 1.207~12.281 | 0.023 | 1.775 | 0.461~6.831 | 0.404 | |
| N分期 | 2.022 | 0.494~8.276 | 0.328 | 1.999 | 0.293~13.639 | 0.479 | |
| CSTF2基因表达 | 1.120 | 1.065~1.178 | < 0.01 | 1.107 | 1.045~1.172 | 0.001 | |
| 注 CSTF2:裂解刺激因子亚单位2(cleavage stimulation factor subunit 2) | |||||||
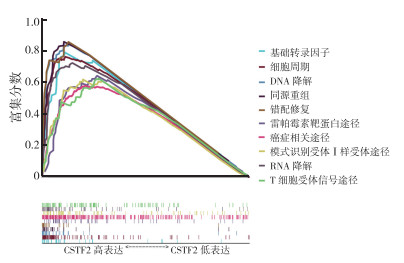
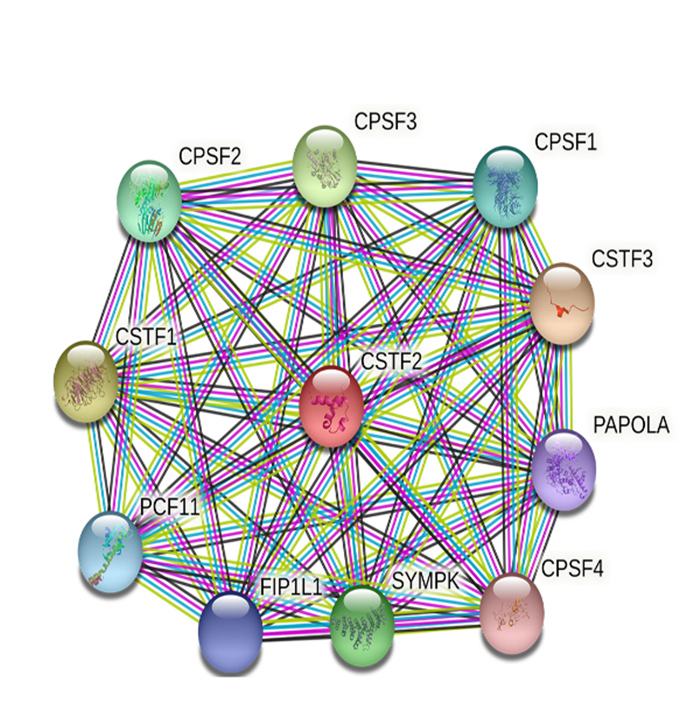
GSEA富集分析结果显示,CSTF2基因在肝细胞癌中主要参与基础转录因子、细胞周期、DNA降解、同源重组、错配修复、哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)信号途径、T细胞受体信号途径以及癌症相关途径等(图 4)。利用STRING数据构建与CSTF2蛋白相互作用的调控网络,结果显示共有10个蛋白质与CSTF2蛋白互相作用与调节,参与基因监控途径(图 5)。
|
| 图 4 GSEA富集结果分析 Fig.4 Analysis of GSEA enrichment results |
|
| 注 CSTF1~3:裂解刺激因子亚单位1~3(cleavage stimulation factor subunit 1~3);SYMPK:支架蛋白(Symplekin);CPSF1~4:切割和聚腺苷酸化特异性因子亚基1~4(cleavage and polyadenylation specificity factor subunit 1~4);PAPOLA:聚α聚合酶[poly(A)polymerase alpha];PCF11:前mRNA切割复合物2蛋白Pcf11(pre-mRNA cleavage complex 2 protein Pcf11);FIP1L1:前mRNA 3’-末端加工因子FIP1(pre-mRNA 3’-end-processing factor FIP1) 图 5 CSTF2蛋白互作网络 Fig.5 CSTF2 protein interaction network |
癌基因的激活在肿瘤的发病机制中起着至关重要的作用。据报道,mRNA的3’非翻译区(3’ untranslated region,3’UTR)的缩短是癌基因异常表达和(或)激活的重要机制,导致恶性肿瘤的发生和发展[9-10]。而在一些癌细胞和组织中,某些原癌基因的3’UTR缩短是由选择性切割和聚腺苷酸化引起的[11]。CSTF2是CSTF的成员。CSTF2基因编码一种含有557个氨基酸的蛋白质,其N末端具有RNP型核糖核酸结合域、一个富含脯氨酸和甘氨酸的长结合域和一个五肽重复区域,形成一个延伸的α-螺旋,组成聚腺苷酸化和前体mRNA 3’末端切割所需的CSTF多亚基复合物之一[12]。据报道,CSTF2在子宫颈癌细胞株HeLa细胞中可与其他2个裂解刺激因子成员在mRNA的聚腺苷酸化中发挥作用[13],提示CSTF2可能在肿瘤的发生和发展中起到重要作用。
有研究利用基因芯片技术对120例肺癌进行全基因组表达谱分析,筛选出异常表达的基因作为肺癌的治疗靶点[7]。结果发现,CSTF2高表达与患者的不良预后相关;CSTF2的外源性表达可以促进细胞的生长和侵袭,而使用小干扰RNA(small interfering RNA,siRNA)抑制CSTF2表达可以抑制肺癌细胞的生长。在三苯氧胺耐药的乳腺癌细胞中,高表达水平CSTF2可以介导具有更短的3′-UTR的RNA结合蛋白人抗原R(human antigen R,HuR)mRNA同工型的上调,从而导致较高的HuR蛋白表达,降低ERBB2的表达,减弱三苯氧胺乳腺癌细胞对他莫昔芬的反应[14]。有研究指出,CSTF2通过诱导RAC1 3’UTR的缩短,可以促进膀胱尿路上皮癌的发生和发展,可作为潜在的分子标志物[8]。这些研究表明,CSTF2可作为肿瘤治疗的潜在分子靶点。
然而,目前国内外尚无研究报道CSTF2在肝细胞癌中的临床意义和作用机制。本研究通过生物信息学分析发现,CSTF2在肝细胞癌组织中表达升高,并且与肿瘤患者的TNM分期相关。CSTF2的高表达可预示患者预后不良,是肝细胞癌潜在的分子预后标志物。机制分析表明,CSTF2参与基因调控和癌症相关信号途径,表明CSTF2在肝细胞癌中也具有重要的生物学意义。
综上所述,CSTF2在肝细胞癌中表达异常升高,与患者的恶性进展特征相关,有望成为肝细胞癌患者的独立预后因素和潜在分子治疗靶点。
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2020[J]. CA Cancer J Clin, 2020, 70(1): 7-30. DOI:10.3322/caac.21590 |
| [2] |
孙瑾瑜, 刘亨晶, 汪泳, 等. ABCB1与肝细胞癌多药耐药的研究进展[J]. 实用肿瘤杂志, 2020, 35(6): 574-578. |
| [3] |
白亮亮, 杨雨. 晚期肝细胞癌系统治疗进展与挑战[J]. 中国普外基础与临床杂志, 2021, 28(3): 287-291. |
| [4] |
Yang JD, Hainaut P, Gores GJ, et al. A global view of hepatocellular carcinoma: trends, risk, prevention and management[J]. Nat Rev Gastroenterol Hepatol, 2019, 16(10): 589-604. DOI:10.1038/s41575-019-0186-y |
| [5] |
Grozdanov PN, Masoumzadeh E, Kalscheuer VM, et al. A missense mutation in the CSTF2 gene that impairs the function of the RNA recognition motif and causes defects in 3' end processing is associated with intellectual disability in humans[J]. Nucl Acids Res, 2020, 48(17): 9804-9821. DOI:10.1093/nar/gkaa689 |
| [6] |
Dass B, Tardif S, Park JY, et al. Loss of polyadenylation protein tauCstF-64 causes spermatogenic defects and male infertility[J]. Proc Nat Acad Sci U S A, 2007, 104(51): 20374-20379. DOI:10.1073/pnas.0707589104 |
| [7] |
Aragaki M, Takahashi K, Akiyama H, et al. Characterization of a cleavage stimulation factor, 3' pre-RNA, subunit 2, 64 kDa (CSTF2) as a therapeutic target for lung cancer[J]. Clin Cancer Res, 2011, 17(18): 5889-5900. DOI:10.1158/1078-0432.CCR-11-0240 |
| [8] |
Chen X, Zhang JX, Luo JH, et al. CSTF2-induced shortening of the RAC13'UTR promotes the pathogenesis of urothelial carcinoma of the bladder[J]. Cancer Res, 2018, 78(20): 5848-5862. |
| [9] |
Mayr C, Bartel DP. Widespread shortening of 3'UTRs by alternative cleavage and polyadenylation activates oncogenes in cancer cells[J]. Cell, 2009, 138(4): 673-684. DOI:10.1016/j.cell.2009.06.016 |
| [10] |
Kataoka K, Shiraishi Y, Takeda Y, et al. Aberrant PD-L1 expression through 3'-UTR disruption in multiple cancers[J]. Nature, 2016, 534(7607): 402-406. DOI:10.1038/nature18294 |
| [11] |
Xia Z, Donehower LA, Cooper TA, et al. Dynamic analyses of alternative polyadenylation from RNA-seq reveal a 3'-UTR landscape across seven tumour types[J]. Nat Comm, 2014, 11(5): 5274-5298. |
| [12] |
Murthy KG, Manley JL. Characterization of the multisubunit cleavage-polyadenylation specificity factor from calf thymus[J]. J Biol Chem, 1992, 267(21): 14804-14811. DOI:10.1016/S0021-9258(18)42111-4 |
| [13] |
Deka P, Rajan PK, Perez-Canadillas JM, et al. Protein and RNA dynamics play key roles in determining the specific recognition of GU-rich polyadenylation regulatory elements by human Cstf-64 protein[J]. J Mol Biol, 2005, 347(4): 719-733. DOI:10.1016/j.jmb.2005.01.046 |
| [14] |
Tan S, Ding K, Chong QY, et al. Post-transcriptional regulation of ERBB2 by miR26a/b and HuR confers resistance to tamoxifen in estrogen receptor-positive breast cancer cells[J]. J Biol Chem, 2017, 292(33): 13551-13564. DOI:10.1074/jbc.M117.780973 |
 2021, Vol. 36
2021, Vol. 36


