文章信息
- 丁永锋, 陈艳妍, 郑玉龙, 蒋微琴, 卢骏, 王海勇, 滕理送, 徐农
- 原发灶不明肿瘤的诊治现状及研究进展
- 实用肿瘤杂志, 2020, 35(6): 567-573
基金项目
- 浙江省自然科学基金(LQ20H160043);国家卫生和计划生育委员会科研基金-浙江省医药卫生重大科技计划项目(KWJ-ZJ-1802);胃肠恶性肿瘤区域诊治中心建设课题(JBZX-201903)
-
作者简介
- 丁永锋(1991-),男,浙江绍兴人,主治医师,博士,从事肿瘤生物信息学及临床转化研究.
-
通信作者
- 徐农, E-mail:nongxu@zju.edu.cn
-
文章历史
- 收稿日期:2020-04-06
2. 浙江大学医学院附属第一医院肿瘤外科,浙江 杭州 310009
原发灶不明肿瘤(cancer of unknown primary site,CUP)是对一类异质性强的肿瘤的统称。该类肿瘤的定义是指经病理学检查证实为转移性癌,但目前通过详尽的临床信息和诊断手段仍无法明确其原发灶[1]。原发灶不明肿瘤的发病率占所有恶性肿瘤的3%~5%[1-2]。对于CUP的产生原因,目前并不十分清楚,可能与原发灶过小且生长缓慢或微小原发灶由于免疫等因素发生消退而转移灶继续生长等原因有关[1]。CUP的自然病程与原发灶明确的肿瘤不同,具有以下临床特征:侵袭性强、早期发生转移、短期内就表现出与转移部位相关的症状和体征以及转移方式的不可预知性(转移发生的位置与已知的原发肿瘤不同)。此外,约33%的CUP患者被诊断时在3个或更多器官已发生转移[1-3]。由于CUP原发灶不明的特性,多数患者接受局部治疗或经验性全身化疗,其中约80%的患者预后不良,平均总生存时间只有6~9个月[4-7]。目前针对此类肿瘤的诊断与治疗尚无充足的循证学依据。然而近年来随着技术的进步及肿瘤精准医疗的实施,国内外学者对该领域的关注和研究逐渐增多[7]。本文主要针对CUP的诊断和治疗的相关进展作一综述。
1 诊断 1.1 临床诊断对CUP的诊断,完整详尽的病史采集必不可少,包括退行性病变疾患和肿瘤手术史等既往史以及肿瘤相关家族史。体格检查应全面,包括Virchow淋巴结在内的浅表淋巴结、直肠指检、女性的乳房检查和男性的前列腺检查等[6-7]。
肿瘤标志物[如癌胚抗原、甲胎蛋白(alpha-fetoprotein, AFP)、糖类抗原-125(carbohydrate antigen 125,CA125)、CA19-9、CA72-4、CA15-3和前列腺特异性抗原(prostate-specific antigen, PSA)等]的升高可以对原发肿瘤的类型有提示作用,但多数血液中的肿瘤标志物的敏感度和特异度不高[8-9]。
胸腹部和盆腔CT检查是诊断CUP的首选影像检查。MRI检查对怀疑肝脏和乳腺来源的CUP有重要意义[6-7]。PET-CT检查对CUP的诊断具有较高的敏感度、特异度和定位作用[10]。
怀疑消化道肿瘤时,可选择胃镜和肠镜等检查,对可疑病灶进行活检。怀疑头颈部肿瘤,则行鼻咽镜和喉镜等检查[8-9]。
1.2 病理诊断组织病理学检查是目前CUP诊断最主要的手段。约60%的CUP为高-中分化腺癌,25%~30%为低分化腺癌或低分化癌,鳞癌、低分化肉瘤和神经内分泌肿瘤较少见[11]。对CUP的免疫组织化学(immunohistochemistry, IHC)检查包括以下主要步骤(图 1)[8-9]:(1)通过IHC检测角蛋白-AE1/AE3(cytokeratin AE1/AE3, CKAE1/AE3)、淋巴细胞共同抗原(common leukocyte antigen, CLA)、S100、人黑色素瘤抗原-45(human melanoma black-45, HMB-45)和波形蛋白(vimentin)等标志物初步判断组织学起源类型,以确定肿瘤属于癌、淋巴瘤、软组织肉瘤或黑色素瘤;(2)通过部分标志物缩小范围,确定肿瘤的亚型,如腺癌、鳞癌、神经内分泌肿瘤、生殖细胞肿瘤或间皮瘤等[细胞角蛋白-7(cytokeratin 7, CK7)和CK20的表达情况可以提示不同器官来源腺癌的诊断,p63和CK5/6在鳞癌中表达;突触素(synaptophsin)、嗜铬粒蛋白A(chromogranin A, CgA)和神经元特异性烯醇化酶(neuron-specific enolase, NSE)是神经内分泌肿瘤的标志物;AFP、人绒毛膜促性腺激素(human chorionic gonadotropin, HCG)和八聚体结合转录因子4(octamer-binding transcription factor-4, OCT4)是生殖细胞肿瘤的标志物];(3)通过更细致的免疫组织化学标志物组合确定原发位置。一些组织特异性的蛋白如PSA、前列腺酸性磷酸酶(prostate acid phosphatase, PAP)、甲状腺球蛋白(thyroglobulin, TG)和肾细胞癌标志物(renal cell carcinoma marker, RCC)有助于提示肿瘤原发灶;雌激素受体(estrogen receptor, ER)和孕激素受体(progesterone receptor, PR)阳性提示乳腺癌、卵巢癌和子宫内膜癌等,甲状腺转录因子-1(thyroid transcription factor-1, TTF-1)在肺腺癌和甲状腺癌中表达;肠上皮标志物尾型同源框转录因子-2(caudal-related homeobox 2, CDX2)结合CK7和CK20的表达情况可以提示胰腺、胆系和肠道肿瘤;Wilm瘤基因-1(Wilm’s tumor gene 1,WT-1)和间皮素(mesothelin)是间皮瘤的标志物。组织病理检查具有明显的局限性,光学显微镜检查结合IHC只能确定约30%CUP的组织起源[12]。
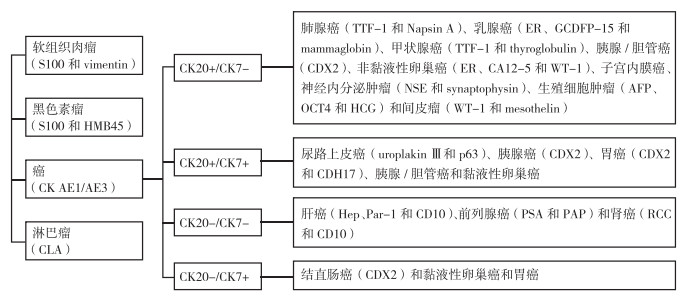
|
| 注 vimentin:波形蛋白;HMB-45:人黑色素瘤抗原-45(human melanoma black-45);CK AE1/AE3: 细胞角蛋白-AE1/AE3(cytokeratin AE1/AE3, );CLA:淋巴细胞共同抗原(common leukocyte antigen);CK20:细胞角蛋白-20(cytokeratin 20);TTF-1:甲状腺转录因子-1(thyroid transcription factor-1);Napsin A:天冬氨酸蛋白酶A;ER:雌激素受体(estrogen receptor);GCDFP-15:巨大囊肿病液体蛋白-15(gross cystic disease fluid protein-15);mammaglobin:乳球蛋白;thyroglobulin:甲状腺球蛋白;CDX2:尾型同源框转录因子-2(caudal-related homeobox 2);WT-1:Wilm瘤基因-1(Wilm’s tumor gene 1);NSE:神经元特异性烯醇化酶(neuron-specific enolase);synaptophsin:突触素;AFP:甲胎蛋白(alpha fetoprotein);OCT4:八聚体结合转录因子4(octamer-binding transcription factor-4);HCG:人绒毛膜促性腺激素(human chorionic gonadotropin);mesothelin:间皮素;uroplakin Ⅲ:尿溶蛋白Ⅲ;Hep:肝细胞特异标志物(hepatocyte);Par-1:蛋白酶激活受体-1(proteinase activated receptor-1);CD10:白细胞分化抗原-10(cluster of differentiation-10);PSA:前列腺特异性抗原(prostate-specific antigen);PAP:前列腺酸性磷酸酶(prostate acid phosphatase);RCC:肾细胞癌标志物(renal cell carcinoma marker) 图 1 CUP免疫组织化学分步诊断流程 |
既往研究表明,转移灶肿瘤的基因表达谱保留了更多原发部位组织的特征,而与转移部位的表达谱存在明显差异[13]。基于上述原理,基因表达谱分析技术(gene expression profiling, GEP)正被越来越多的研究者开发并应用于预测CUP的原发部位。GEP通过对获取的CUP组织进行实时反转录酶聚合酶链式反应(real-time polymerase chain reaction, RT-PCR)、基因芯片或二代测序(转录组测序)检测,进一步利用生物信息学方法提取基因特征表达分子作为判断原发灶的特异性“分子指纹”,并建立相应数学模型进行预测与判断的一种新型诊断技术[13-15]。有研究早期选取10个来自肺、结肠、胰腺、乳腺、前列腺和卵巢癌的组织特异表达的基因,并在260个原发灶已知的转移癌样本中进行RT-PCR检测,模型正确率达78%[14]。来自密歇根大学的研究组通过公开的肿瘤数据库收集来自26种不同组织类型的3 244个肿瘤样本的转录组测序(RNA Sequencing,RNAseq)表达数据,通过特征提取的方式选择每种肿瘤的特征分子构建模型,总体判断准确度达到90.5%,敏感度和特异度分别为92.0%和97.7%[15]。国内复旦大学团队利用支持向量机模型鉴定出154个分子特征可作为CUP原发灶判断依据,总体准确率能达到92%[16]。近期,Shen等[17]通过整合不同平台的公开测序数据,开发一套兼容性和稳定性较好的新型算法进行CUP的原发灶判断,并在实际临床样本的外部验证中,取得96.0%(24/25)的准确度。此外,笔者所在团队在特定临床情景下的CUP原发灶探究中也做了一些探索性工作[18-19]。临床上,肝胆胰系统中同时性多灶性肿瘤较为常见,但三者在癌组织病理分化差等情况下难以区分原发灶,尤其是胆囊癌和胰腺癌之间。本研究组通过肿瘤基因组图谱(The Cancer Genome Atlas,TCGA)数据库中肝胆胰系统肿瘤以及本中心临床样本的RNAseq测序,提取近1 000个特征分子并建立基于朴素贝叶斯算法的组织起源判断算法(naive Bayes algorithm for tissue origin diagnosis,TOD-Bayes),进行了肝胆胰系统中同时性多灶性肿瘤的组织起源判断[18]。结果显示,利用TOD-Bayes算法内部验证中能达到>95%的准确度,在外部验证中有94.4%的准确度。此外,利用TCGA肿瘤数据库和本中心样本,进行90基因表达谱在脑转移瘤原发灶判断上的研究,其判断准确度可达89%[19]。
1.3.2 表观组学分析表观遗传学是指在不改变DNA序列的情况下调控基因表达的遗传学变化,包括DNA甲基化、组蛋白修饰和染色体重塑等[20]。利用二代测序的甲基化分析技术,一项2012年的研究显示,通过对1 000余例肿瘤患者的甲基化图谱分析,鉴定并识别出24种肿瘤特异性的甲基化改变模式[21]。基于该模式构建的CUP诊断工具,在组织起源的判断上达87%的预测准确率,随之进行的肿瘤特异性治疗带来总生存的获益,中位总生存时间提高近8个月[22]。
1.3.3 液体活检液体活检对患者血液样本中来源于肿瘤的细胞、核酸或蛋白等物质进行分析。近年来液体活检是肿瘤诊疗的研究热点,已有大量基于液体活检的肿瘤标志物被发现并尝试应用于肿瘤早筛、治疗评估和复发监测等领域。液体活检技术在判断CUP的组织起源上也进行了不少应用研究[23]。对循环肿瘤细胞(circulating tumor cell)的分选结合CK7和CK20等标志物的免疫荧光染色可以协助CUP组织起源的判断[24]。对肿瘤来源的血小板(tumor-educated platelets)进行转录组学测序和机器学习,可识别出6种组织起源的肿瘤,在228例肿瘤患者中,该方法对组织起源的中位预测准确率达到73%[25]。一种基于cell-free DNA(cfDNA)和蛋白标志物的泛肿瘤早期筛查工具CancerSEEK在筛查阳性(即患有肿瘤)患者的组织起源判断上达到83%的准确度[26]。
2 治疗 2.1 预后良好亚型CUP的治疗约20%的CUP患者属于预后良好的类型,部分CUP具有与已知原发灶的肿瘤类似的特征,可采取相应疾病的治疗方案并获得类似的预后[7, 27],具体的亚型及对应的治疗方案见表 1。
| 亚型 | 首选治疗 |
| 腹腔转移性乳头状腺癌(女性患者) | Ⅲ~Ⅳ期卵巢癌:手术减瘤+紫杉醇/铂类联合化疗 |
| 腋窝淋巴结转移性腺癌(女性患者) | Ⅱ~Ⅲ期乳腺癌:腋窝淋巴结清扫+乳房切除+辅助放化疗±内分泌治疗±HER2靶向治疗 |
| 颈部淋巴结转移性鳞癌 | 局部进展期头颈部鳞癌:颈淋巴结清扫+局部放疗±辅助化疗 |
| 中线部位的低分化癌 | 生殖细胞肿瘤:以铂类为基础的联合化疗 |
| 低分化神经内分泌癌 | 以铂类为基础的联合化疗或紫杉醇/铂类联合化疗 |
| 具有结直肠癌特征的腺癌(CK20+/CK7-/CDX2+) | 转移性结直肠癌:化疗±靶向治疗±免疫治疗 |
| 骨转移腺癌伴IHC或血清PSA阳性(男性患者) | 转移性前列腺癌:内分泌治疗±化疗 |
| 腹股沟淋巴结转移性鳞癌 | 腹股沟淋巴结清扫+局部放疗±辅助化疗 |
| 单发、可切除转移瘤 | 手术切除±局部放疗 |
| 注CUP:原发灶不明肿瘤(cancer of unknown primary site); HER2:人表皮生长因子受体-2(human epidermal growth factor receptor 2);CK20:细胞角蛋白-20(cytokeratin 20);CK7:细胞角蛋白-7(cytokeratin 7);CDX2:尾型同源框转录因子-2(caudal-related homeobox 2);IHC:免疫组织化学(immunohistochemistry);PSA:前列腺特异性抗原(prostate-specific antigen)。 | |
腹腔转移性乳头状腺癌的女性患者应依据Ⅲ~Ⅳ期卵巢癌方案治疗。通过手术减瘤,术后行铂类联合紫杉醇化疗,中位反应率约80%,30%~40%可获得完全缓解(complete response, CR),中位生存时间为36个月[28]。
腋窝淋巴结转移性腺癌的女性患者可参照Ⅱ~Ⅲ期乳腺癌治疗,在检测ER、PR和人表皮生长因子受体-2(human epidermal growth factor receptor 2,HER2)免疫组织化学的基础上选择腋窝淋巴结清扫及同侧乳房切除(对此类患者保留乳房有较高的复发和转移风险),术后行辅助放化疗,根据Luminal分型进行内分泌治疗及HER2靶向治疗,5年总生存率约为72%[29]。
中线部位的低分化癌有较大的异质性,仅有少数具有非典型生殖细胞肿瘤的特征。首选依据不良预后的生殖细胞肿瘤治疗,即以铂类为基础的化疗,反应率为35%~65%,中位生存时间为12个月[30]。
原发灶不明的神经内分泌肿瘤的自然病程和预后类似于高级别肺部和胃肠道神经内分泌肿瘤,通常选用铂类为基础的化疗,报道的中位生存时间为15.5个月[31]。
具有结直肠癌免疫组织化学特征(CK20+/CK7-和CDX2+)的腺癌可依据转移性结直肠癌的适应证选用化疗、靶向治疗和(或)免疫治疗。骨转移腺癌伴血清PSA升高的男性患者按转移性前列腺癌治疗,首选抗雄激素内分泌治疗[8, 27]。
此外,对于腹股沟淋巴结转移性鳞癌以及单发、局限的转移瘤,可通过手术达到根治切除,术后视情况行局部放疗,患者可获得长期无瘤生存[11, 27]。
2.2 预后不良亚型CUP的治疗 2.2.1 经验性化疗目前的肿瘤治疗决策主体上仍是基于病理分类(基于原发器官)。对于预后不良亚型CUP患者的治疗,临床上往往采用经验性化疗,如以紫杉类、铂类和吉西他滨等为基础的单药或联合治疗方案,但缺乏足够的循证学依据[4]。
一项2009年的荟萃分析显示,CUP患者接受经验性化疗后中位生存时间为8.9个月,3年生存率为12%[5]。一项单臂研究发现,102例CUP患者接受卡铂联合多柔比星和依托泊苷的化疗方案,获得26.5%的客观缓解率,中位总生存时间和无进展生存时间分别为9个月和4个月[32]。有研究探究吉西他滨、卡铂和紫杉醇联合化疗在CUP患者中的疗效,客观缓解率为25%,中位总生存时间和无进展生存时间分别为9个月和6个月[33]。一项对紫杉醇/卡铂/依托泊苷联合方案和吉西他滨/伊立替康联合方案在CUP患者中的疗效和毒性比较研究发现,两组在中位总生存时间上相近,分别为7.4个月和8.5个月,但吉西他滨/伊立替康组患者的毒性反应相对更小[34]。
2.2.2 基因表达谱指导下的器官特异性化疗近年来已有一些报道探究了基因表达谱指导下的器官特异性化疗给CUP患者带来的获益程度。一项回顾性研究发现,32例基于92基因表达谱预测出可能为肠癌的CUP患者接受肠癌相关一线或二线治疗,取得27个月的中位生存时间,远超过CUP的平均总生存时间[35]。另一项前瞻性研究显示,194例CUP患者接受基于92基因表达谱检测的器官特异性化疗,另有396例CUP患者接受经验性化疗,前者的中位生存时间为12.5个月,后者只有9.1个月[36]。然而近期一项针对CUP患者的Ⅱ期随机对照临床试验结果显示,基因表达谱指导下的器官特异性化疗并未显示出优于经验性化疗的疗效[37]。值得注意的是,对已报道的4项针对CUP患者的经验性化疗和器官特异性化疗疗效对比的前瞻性研究进行的一项荟萃分析发现,接受器官特异性化疗的CUP患者有获益倾向,但差异尚未达到统计学意义(P=0.06)[38]。因此,未来需要更大样本的多中心随机对照研究或高质量的荟萃分析来进一步明确基因表达谱指导下的器官特异性化疗的临床价值。
2.2.3 靶向治疗近年来,靶向治疗的研究和应用取得突破性进展,如肺癌、黑色素瘤和乳腺癌等领域。随着CUP特征突变谱不断被揭示,靶向治疗有望成为CUP患者的治疗选择之一。多项大样本研究显示,肿瘤相关蛋白53(tumor associated protein 53, TP53)和鼠类肉瘤病毒癌基因(Kirsten rat sarcoma viral proto-oncogene, KRAS)突变在CUP患者中最常见,突变比例分别为38%~55%以及18%~22%[39-41]。一项针对442例CUP患者循环肿瘤DNA(circulating tumor DNA,ctDNA)的基因组学分析显示,80%的CUP患者检出ctDNA的基因变异,其中检测到的潜在治疗靶点可用于指导治疗[42]。目前探究CUP靶向治疗临床疗效的研究很少。一项小样本研究发现,11例接受靶向治疗的CUP患者中,有4例获得≥4个月的无进展生存时间[43]。目前的研究表明,多数患者靶向治疗的疗效与肿瘤的具体类型密切相关,如鼠类肉瘤滤过性毒菌致癌同源体B1(V-Raf murine sarcoma viral oncogene homolog B1, BRAF)突变抑制剂单药治疗在黑色素瘤中的疗效远好于结直肠癌[44]。因此,原发灶的预测对靶向治疗潜在获益的CUP人群的选择具有重要意义。
2.2.4 免疫治疗以免疫检查点抑制剂(immune checkpoint inhibitor, ICI)为代表的肿瘤免疫治疗正在颠覆目前临床上对晚期肿瘤治疗的认识,在某些瘤种,如恶性黑色素瘤,其疗效显著,部分患者甚至在停药后,仍然获得长期生存。对免疫治疗反应的预测指标目前仍无定论,在已知的肿瘤类型中包括肿瘤突变负荷(tumor mutation burden, TMB)、微卫星不稳定(microsatellite instability, MSI)、错配修复缺陷(deficient mismatch repair, dMMR)和程序性死亡受体配体1(programmed death ligand 1,PD-L1)表达等。Pembrolizumab已被美国食品药品监督管理局(food and drug administration,FDA)批准用于MSI-high和dMMR特征的实体肿瘤。一项研究报道在389例CUP患者中,约28%的病例具有TMB-high、MSI/dMMR或PD-L1高表达等与免疫治疗疗效相关的分子特征[40]。也有病例报道显示,在具有上述分子特征的CUP中使用ICI取得良好的治疗反应[42, 45]。因此,对预后不良型的CUP患者,免疫治疗将是未来的选择之一。一项正在进行中的Ⅱ期临床研究NCT03498521将对分子导向的CUP治疗(包括靶向治疗和免疫治疗)进行疗效评估,为CUP的免疫治疗及其疗效预测标志物提供更多循证医学依据。
2.2.5 基于人源肿瘤异种移植瘤(patient-derived xenograft,PDX)的个体化治疗PDX模型可以很好地再现原发肿瘤的组织学和分子病理学特征,因此是一种理想的个体化药敏模型,可用于晚期患者治疗药物筛选和新药临床前研究,结合二代测序进行药敏靶点研究和耐药机制研究等[46-48]。Miyake等[49]首次报道在CUP构建的原位PDX模型上发现优于经验性化疗的治疗方案。除了常规PDX外,培养周期更短的miniPDX[50]、反映肿瘤三维结构和肿瘤微环境的类器官模型(organoid)[51]在临床应用也有很大的潜力。对于预后不良亚型CUP,基于PDX、miniPDX和类器官等模型的个体化治疗将是未来的发展方向之一。
3 结语CUP的诊断与治疗目前仍然是临床上的难点。由于缺乏循证医学依据,目前主要依赖于经验性治疗,多数患者预后不良。基因表达谱分析、表观组学分析和液体活检等原发灶判断方法的研究为器官特异性治疗带来希望,已有早期临床试验初步验证其临床实用价值,但仍具有争议,尚缺乏大型随机前瞻性临床试验。靶向治疗和免疫治疗的发展也为CUP治疗带来新的思路,相关临床试验已陆续开展。此外,基于PDX/miniPDX/类器官模型指导下的个体化治疗也将成为CUP患者可能的选择。未来亟需更多有效的生物预测标志物对CUP患者合理分层,并提供更好的治疗选择,实现CUP精准化治疗,最终为CUP患者带来生存获益及生存质量的提高。
| [1] |
Pavlidis N, Briasoulis E, Hainsworth J, et al. Diagnostic and therapeutic management of cancer of an unknown primary[J]. Eur J Cancer, 2003, 39(14): 1990-2005. DOI:10.1016/S0959-8049(03)00547-1 |
| [2] |
Pavlidis N, Pentheroudakis G. Cancer of unknown primary site[J]. Lancet, 2012, 379(9824): 1428-1435. DOI:10.1016/S0140-6736(11)61178-1 |
| [3] |
Pavlidis N, Fizazi K. Carcinoma of unknown primary (CUP)[J]. Crit Rev Oncol/Hematol, 2009, 69(3): 271-278. DOI:10.1016/j.critrevonc.2008.09.005 |
| [4] |
Pavlidis N. Forty years experience of treating cancer of unknown primary[J]. Acta Oncologica, 2007, 46(5): 592-601. DOI:10.1080/02841860701243095 |
| [5] |
Greco FA, Pavlidis N. Treatment for patients with unknown primary carcinoma and unfavorable prognostic factors[J]. Semin Oncol, 2009, 36(1): 65-74. DOI:10.1053/j.seminoncol.2008.10.005 |
| [6] |
Pavlidis N, Khaled H, Gaafar R. A mini review on cancer of unknown primary site: A clinical puzzle for the oncologists[J]. J Adv Res, 2015, 6(3): 375-382. DOI:10.1016/j.jare.2014.11.007 |
| [7] |
王金艳, 仲悦娇, 陈凌翔, 等. 原发灶不明肿瘤的诊断与治疗[J]. 中华全科医学, 2018, 16(12): 2067-2071. |
| [8] |
Fizazi K, Greco FA, Pavlidis N, et al. Cancers of unknown primary site: ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up[J]. Ann Oncol, 2015, 26(Suppl 5): v133-138. |
| [9] |
Losa F, Soler G, Casado A, et al. SEOM clinical guideline on unknown primary cancer (2017)[J]. Clin Transl Oncol, 2018, 20(1): 89-96. DOI:10.1007/s12094-017-1807-y |
| [10] |
Wolpert F, Weller M, Berghoff AS, et al. Diagnostic value of 18F-fluordesoxyglucose positron emission tomography for patients with brain metastasis from unknown primary site[J]. Eur J Cancer, 2018, 96: 64-72. DOI:10.1016/j.ejca.2018.03.010 |
| [11] |
Pavlidis N, Pentheroudakis G. Cancer of unknown primary site[J]. The Lancet, 2012, 379(9824): 1428-1435. DOI:10.1016/S0140-6736(11)61178-1 |
| [12] |
Economopoulou P, Mountzios G, Pavlidis N, et al. Cancer of Unknown Primary origin in the genomic era: Elucidating the dark box of cancer[J]. Cancer Treat Rev, 2015, 41(7): 598-604. DOI:10.1016/j.ctrv.2015.05.010 |
| [13] |
Hainsworth JD, Greco FA. Gene expression profiling in patients with carcinoma of unknown primary site: from translational research to standard of care[J]. Virchows Archiv, 2014, 464(4): 393-402. DOI:10.1007/s00428-014-1545-2 |
| [14] |
Talantov D, Baden J, Jatkoe T, et al. A quantitative reverse transcriptase-polymerase chain reaction assay to identify metastatic carcinoma tissue of origin[J]. J Mol Diagn, 2006, 8(3): 320-329. DOI:10.2353/jmoldx.2006.050136 |
| [15] |
Wei IH, Shi Y, Jiang H, et al. RNA-Seq accurately identifies cancer biomarker signatures to distinguish tissue of origin[J]. Neoplasia, 2014, 16(11): 918-927. DOI:10.1016/j.neo.2014.09.007 |
| [16] |
Xu Q, Chen J, Ni S, et al. Pan-cancer transcriptome analysis reveals a gene expression signature for the identification of tumor tissue origin[J]. Modern Pathol, 2016, 29(6): 546-556. DOI:10.1038/modpathol.2016.60 |
| [17] |
Shen Y, Chu Q, Yin X, et al. TOD-CUP: a gene expression rank-based majority vote algorithm for tissue origin diagnosis of cancers of unknown primary[J]. Brief Bioinform, 2020, Epub ahead of print.
|
| [18] |
Jiang W, Shen Y, Ding Y, et al. A naive Bayes algorithm for tissue origin diagnosis (TOD-Bayes) of synchronous multifocal tumors in the hepatobiliary and pancreatic system[J]. Int J Cancer, 2018, 142(2): 357-368. DOI:10.1002/ijc.31054 |
| [19] |
Zheng Y, Ding Y, Wang Q, et al. 90-gene signature assay for tissue origin diagnosis of brain metastases[J]. J Transl Med, 2019, 17(1): 331. DOI:10.1186/s12967-019-2082-1 |
| [20] |
Portela A, Esteller M. Epigenetic modifications and human disease[J]. Nat Biotechnol, 2010, 28(10): 1057-1068. DOI:10.1038/nbt.1685 |
| [21] |
Fernandez AF, Assenov Y, Martin-Subero JI, et al. A DNA methylation fingerprint of 1628 human samples[J]. Genome Res, 2011, 22(2): 407-419. |
| [22] |
Moran S, Martínez-Cardús A, Sayols S, et al. Epigenetic profiling to classify cancer of unknown primary: a multicentre, retrospective analysis[J]. Lancet Oncol, 2016, 17(10): 1386-1395. DOI:10.1016/S1470-2045(16)30297-2 |
| [23] |
Conway AM, Mitchell C, Kilgour E, et al. Molecular characterisation and liquid biomarkers in Carcinoma of Unknown Primary (CUP): taking the 'U' out of 'CUP'[J]. Br J Cancer, 2019, 120(2): 141-153. DOI:10.1038/s41416-018-0332-2 |
| [24] |
Lu SH, Tsai WS, Chang YH, et al. Identifying cancer origin using circulating tumor cells[J]. Cancer Biol Ther, 2016, 17(4): 430-438. DOI:10.1080/15384047.2016.1141839 |
| [25] |
Best MG, Sol N, Kooi I, et al. RNA-Seq of tumor-educated platelets enables blood-based pan-cancer, multiclass, and molecular pathway cancer diagnostics[J]. Cancer Cell, 2015, 28(5): 666-676. DOI:10.1016/j.ccell.2015.09.018 |
| [26] |
Cohen JD, Li L, Wang Y, et al. Detection and localization of surgically resectable cancers with a multi-analyte blood test[J]. Science, 2018, 359(6378): 926-930. DOI:10.1126/science.aar3247 |
| [27] |
Hainsworth JD, Fizazi K. Treatment for patients with unknown primary cancer and favorable prognostic factors[J]. Semin Oncol, 2009, 36(1): 44-51. DOI:10.1053/j.seminoncol.2008.10.006 |
| [28] |
Pentheroudakis G, Pavlidis N. Serous papillary peritoneal carcinoma: unknown primary tumour, ovarian cancer counterpart or a distinct entity? A systematic review[J]. Crit Rev Oncol Hematol, 2010, 75(1): 27-42. DOI:10.1016/j.critrevonc.2009.10.003 |
| [29] |
Pentheroudakis G, Lazaridis G, Pavlidis N. Axillary nodal metastases from carcinoma of unknown primary (CUPAx): a systematic review of published evidence[J]. Breast Cancer Res Treat, 2010, 119(1): 1-11. DOI:10.1007/s10549-009-0554-3 |
| [30] |
Pentheroudakis G, Stoyianni A, Pavlidis N. Cancer of unknown primary patients with midline nodal distribution: midway between poor and favourable prognosis?[J]. Cancer Treat Rev, 2011, 37(2): 120-126. DOI:10.1016/j.ctrv.2010.06.003 |
| [31] |
Stoyianni A, Pentheroudakis G, Pavlidis N. Neuroendocrine carcinoma of unknown primary: a systematic review of the literature and a comparative study with other neuroendocrine tumors[J]. Cancer Treat Rev, 2011, 37(5): 358-365. DOI:10.1016/j.ctrv.2011.03.002 |
| [32] |
Piga A, Nortilli R, Cetto GL, et al. Carboplatin, doxorubicin and etoposide in the treatment of tumours of unknown primary site[J]. Br J Cancer, 2004, 90(10): 1898-1904. DOI:10.1038/sj.bjc.6601785 |
| [33] |
Greco FA, Burris HA, Litchy S, et al. Gemcitabine, carboplatin, and paclitaxel for patients with carcinoma of unknown primary site: a Minnie Pearl Cancer Research Network study[J]. J Clin Oncol, 2002, 20(6): 1651-1656. DOI:10.1200/JCO.2002.20.6.1651 |
| [34] |
Hainsworth JD, Spigel DR, Clark BL, et al. Paclitaxel/carboplatin/etoposide versus gemcitabine/irinotecan in the first-line treatment of patients with carcinoma of unknown primary site: a randomized, phase Ⅲ Sarah Cannon Oncology Research Consortium Trial[J]. Cancer J, 2010, 16(1): 70-75. DOI:10.1097/PPO.0b013e3181c6aa89 |
| [35] |
Hainsworth JD, Schnabel CA, Erlander MG, et al. A retrospective study of treatment outcomes in patients with carcinoma of unknown primary site and a colorectal cancer molecular profile[J]. Clin Colorect Cancer, 2012, 11(2): 112-118. DOI:10.1016/j.clcc.2011.08.001 |
| [36] |
Hainsworth JD, Rubin MS, Spigel DR, et al. Molecular gene expression profiling to predict the tissue of origin and direct site-specific therapy in patients with carcinoma of unknown primary site: a prospective trial of the Sarah Cannon research institute[J]. J Clin Oncol, 2013, 31(2): 217-223. DOI:10.1200/JCO.2012.43.3755 |
| [37] |
Hayashi H, Kurata T, Takiguchi Y, et al. Randomized phase Ⅱ trial comparing site-specific treatment based on gene expression profiling with carboplatin and paclitaxel for patients with cancer of unknown primary site[J]. J Clin Oncol, 2019, 37(7): 570-579. DOI:10.1200/JCO.18.00771 |
| [38] |
Rassy E, Bakouny Z, Choueiri TK, et al. The role of site-specific therapy for cancers of unknown of primary: A meta-analysis[J]. Eur J Cancer, 2020, 127: 118-122. DOI:10.1016/j.ejca.2019.12.016 |
| [39] |
Gatalica Z, Millis SZ, Vranic S, et al. Comprehensive tumor profiling identifies numerous biomarkers of drug response in cancers of unknown primary site: analysis of 1806 cases[J]. Oncotarget, 2014, 5(23): 12440-12447. DOI:10.18632/oncotarget.2574 |
| [40] |
Gatalica Z, Xiu J, Swensen J, et al. Comprehensive analysis of cancers of unknown primary for the biomarkers of response to immune checkpoint blockade therapy[J]. Eur J Cancer, 2018, 94: 179-186. DOI:10.1016/j.ejca.2018.02.021 |
| [41] |
Ross JS, Wang K, Gay L, et al. Comprehensive genomic profiling of carcinoma of unknown primary site: new routes to targeted therapies[J]. JAMA Oncol, 2015, 1(1): 40-49. DOI:10.1001/jamaoncol.2014.216 |
| [42] |
Kato S, Krishnamurthy N, Banks KC, et al. Utility of genomic analysis in circulating tumor DNA from patients with carcinoma of unknown primary[J]. Cancer Res, 2017, 77(16): 4238-4246. DOI:10.1158/0008-5472.CAN-17-0628 |
| [43] |
Subbiah IM, Tsimberidou A, Subbiah V, et al. Next generation sequencing of carcinoma of unknown primary reveals novel combinatorial strategies in a heterogeneous mutational landscape[J]. Oncoscience, 2017, 4(5/6): 47-56. |
| [44] |
Prahallad A, Sun C, Huang S, et al. Unresponsiveness of colon cancer to BRAF(V600E) inhibition through feedback activation of EGFR[J]. Nature, 2012, 483(7387): 100-103. DOI:10.1038/nature10868 |
| [45] |
Groschel S, Bommer M, Hutter B, et al. Integration of genomics and histology revises diagnosis and enables effective therapy of refractory cancer of unknown primary with PD-L1 amplification[J]. Cold Spring Harb Mol Case Stud, 2016, 2(6): a001180. DOI:10.1101/mcs.a001180 |
| [46] |
Siolas D, Hannon GJ. Patient-derived tumor xenografts: transforming clinical samples into mouse models[J]. Cancer Res, 2013, 73(17): 5315-5319. DOI:10.1158/0008-5472.CAN-13-1069 |
| [47] |
Gao H, Korn JM, Ferretti S, et al. High-throughput screening using patient-derived tumor xenografts to predict clinical trial drug response[J]. Nat Med, 2015, 21(11): 1318-1325. DOI:10.1038/nm.3954 |
| [48] |
Tentler JJ, Tan AC, Weekes CD, et al. Patient-derived tumour xenografts as models for oncology drug development[J]. Nat Rev Clin Oncol, 2012, 9(6): 338-350. DOI:10.1038/nrclinonc.2012.61 |
| [49] |
Miyake K, Kiyuna T, Miyake M, et al. Patient-derived orthotopic xenograft models for cancer of unknown primary precisely distinguish chemotherapy, and tumor-targeting S. typhimurium A1-R is superior to first-line chemotherapy[J]. Signal Transduct Target Ther, 2018, 3(1): 12. DOI:10.1038/s41392-018-0016-7 |
| [50] |
Zhao P, Chen H, Wen D, et al. Personalized treatment based on mini patient-derived xenografts and WES/RNA sequencing in a patient with metastatic duodenal adenocarcinoma[J]. Cancer Commun, 2018, 38(1): 54. DOI:10.1186/s40880-018-0323-y |
| [51] |
Gao D, Vela I, Sboner A, et al. Organoid cultures derived from patients with advanced prostate cancer[J]. Cell, 2014, 159(1): 176-187. DOI:10.1016/j.cell.2014.08.016 |
 2020, Vol. 35
2020, Vol. 35


