文章信息
- 刘兴华, 林泳煌, 陈东宇, 樊文龙, 王红心, 杨晓雨, 何玉清
- Liu Xinghua, Lin Yonghuang, Chen Dongyu, Fan Wenlong, Wang Hongxin, Yang Xiaoyu, He Yuqing
- 基于自噬相关lncRNA建立子宫颈癌预后风险评估模型
- Development of prognostic risk model for cervical cancer based on autophagy-related lncRNAs
- 实用肿瘤杂志, 2022, 37(6): 514-521
- Journal of Practical Oncology, 2022, 37(6): 514-521
基金项目
- 国家自然科学基金项目(81773312);广东省自然科学基金项目(2015A030313517)
-
通信作者
- 何玉清,E-mail:dr.hyq@hotmail.com
-
文章历史
- 收稿日期:2021-10-14
2. 广东医科大学寮步医院皮肤科, 广东 东莞 523400
2. Department of Dermatology, Liaobu Hospital, Guangdong Medical University, Dongguan 523400, China
全球范围内,子宫颈癌是仅次于乳腺癌的女性第二常见恶性肿瘤[1]。全球每年新增50多万例女性子宫颈癌患者,且有30多万例死亡[2]。发展中国家子宫颈癌的发病率较发达国家更高。全世界约85%的子宫颈癌死亡发生在欠发达或发展中国家,而且低收入和中等收入国家的死亡率较富裕国家高18倍[3]。目前,可以通过手术、化疗和一些靶向治疗来改善子宫颈癌的预后。然而其预后仍然很差,5年生存率仅为40%[4]。因此,对部分高危患者的早期识别可以提供个性化的疾病管理,有助于改善子宫颈癌患者的预后。
自噬是一种自我降解的过程,通常被认为是一种生存机制,在去除特定的细胞器、核糖体和蛋白质聚集物时可以是非选择性或选择性。自噬还促进细胞衰老和细胞表面抗原呈递,防止基因组不稳定和坏死,使其在预防癌症、神经退行性疾病、心肌病、糖尿病、肝病、自身免疫疾病和感染方面发挥着关键作用[5]。在肿瘤发展的不同阶段和微环境中,自噬在肿瘤中起着抑制或促进的动态作用[6]。一方面,自噬产生的各种代谢产物来源为肿瘤提供了代谢可塑性,使其能够在恶劣的微环境中茁壮成长[7]。另一方面,也已经有人提出,自噬可以通过限制基因组突变、促进癌基因诱导减弱和减少肿瘤诱导的炎性反应来抑制癌症的发展[8]。自噬在非小细胞肺癌治疗的各个阶段(放疗、化疗和靶向治疗)都有着抑制和促进的双重作用[9]。自噬的激活与抑制干预已经被提出可用于癌症治疗[10]。子宫颈癌中,自噬相关蛋白Beclin 1的表达与提高生存率有关。Beclin 1的表达抑制Caski细胞的增殖[11]。高危人乳头瘤病毒(high-risk human papillomavirus,HR-HPV)持续感染是导致子宫颈癌的主要原因,抑制自噬可促进HPV的生命周期和肿瘤进展[12]。因此,自噬在子宫颈癌中也发挥重要作用。
长链非编码RNA(long non-coding RNA,lncRNA)是一种非蛋白质编码转录本,长度 > 200个核苷酸[13]。lncRNA的失调与包括肿瘤在内的多种人类疾病相关[14]。lncRNA在各种肿瘤的进展、各种细胞的生物学和病理过程如增殖、凋亡、侵袭、分化和上皮-间充质转化(epithelial-mesenchymal transformation,EMT)以及炎性反应等中都发挥着关键作用[15-18]。lncRNA参与自噬的动态调节过程[19]。随着测序技术的发展,lncRNA可以很容易地在癌症患者的组织或血浆中检测到。因此,本研究利用癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库的表达数据构建自噬相关lncRNA的预后模型,试图寻找新的预测子宫颈癌患者预后的生物标志物。
1 资料与方法 1.1 样本和数据集提取2020年11月2日TCGA数据库(https://portal.gdc.cancer.gov/)获取RNA转录组数据和相应的临床资料。样本的入选标准: (1)确诊为子宫颈癌; (2)该样本具有完整的表达谱数据和临床信息。随访时间 < 30 d的样本因死于子宫颈癌以外原因的概率较高而被排除。根据纳入和排除标准,共纳入273个肿瘤样本,每个样本对应1例子宫颈癌患者。
1.2 自噬相关基因及lncRNA筛选2021年4月21日从人类自噬数据库(Human Autophagy Database,HADb;http://autophagy.lu/clustering/index.html)获取自噬相关基因列表。从TCGA数据库获得的总RNA表达谱中分离出lncRNA和自噬相关基因的表达矩阵。通过构建共表达网络筛选自噬相关lncRNA[20]。为了分析lncRNA与自噬相关基因的共表达情况,本研究采用Pearson相关性计算lncRNA与自噬相关基因之间的相关性。以相关系数r > 0.4和P < 0.001作为自噬相关lncRNA的纳入标准。
1.3 预后模型的构建采用R(4.0.2)软件的Survival软件包对自噬相关lncRNA进行单因素Cox分析,以P < 0.01为纳入标准判断自噬相关lncRNA与子宫颈癌预后的相关性。对单因素分析中具有统计学意义(P < 0.01)的自噬相关lncRNA进行多因素Cox分析,并基于Akaike Information Criterion(AIC)确定最优预测模型。通过纳入模型中lncRNA的表达水平和多元分析中加权线性组合的回归系数(β)确定每个样本的风险评分(risk score)。公式如下:Risk score=β1×exp(lncRNA1)+β2×exp(lncRNA2)+…+βN×exp(lncRNAN),其中β为模型中对应lncRNA的系数,exp为对应lncRNA的表达水平。
1.4 模型有效性验证样本风险评分在中位数以上的样本定义为高危组,在中位数及以下的样本定义为低危组。对高危组和低危组应用Kaplan-Meier法作生存分析。R软件中的pheatmap包被用来绘制在该预测模型下高危组和低危组的风险曲线、生存状态以及lncRNA表达热图。对预测模型及其他临床特征进行单因素和多因素分析。绘制该模型与其他临床特征的受试者工作特征(receiver operating characteristic,ROC)曲线,并比较曲线下面积(area under the curve,AUC),验证该模型的预测效果是否高于其他临床特征。
1.5 富集分析和主成分分析(principal component analysis,PCA)PCA分析验证模型的有效性。整理预后模型中自噬lncRNA的表达量及其相应的风险评分,通过基因集富集分析(gene set enrichment analysis,GSEA)来确定高危组与低危组之间存在的自噬表型差异。使用GSEA(version 4.1.0)软件进行GSEA分析。
1.6 统计学分析采用R软件(version 4.0.2)进行统计学分析。生存曲线采用Kaplan-Meier法生成,采用log-rank检验作生存比较。使用单因素和多因素Cox比例风险回归模型评估自噬相关lncRNA预后模型和临床病理特征的预后影响。采用ROC曲线评价预测模型的可靠性和准确性。以P < 0.05为差异具有统计学意义。
2 结果 2.1 样本特征273例子宫颈癌患者年龄20~88岁,平均年龄47.9岁。临床特征资料汇总见表 1。
| 临床特征 | 例数(%) |
| 年龄 | |
| < 60岁 | 217(79.5) |
| ≥60岁 | 56(20.5) |
| 肿瘤分级 | |
| G1 | 16(5.9) |
| G2 | 126(46.2) |
| G3 | 104(38.1) |
| G4 | 1(0.4) |
| GX | 24(8.8) |
| 未知 | 2(0.7) |
| FIGO分期 | |
| Ⅰ | 149(54.6) |
| Ⅱ | 60(22.0) |
| Ⅲ | 38(13.9) |
| Ⅳ | 20(7.3) |
| 未知 | 6(2.2) |
| T分期 | |
| T1 | 128(46.9) |
| T2 | 64(23.4) |
| T3 | 16(5.9) |
| T4 | 10(3.7) |
| Tis | 1(0.4) |
| TX | 16(5.9) |
| 未知 | 38(13.9) |
| N分期 | |
| N0 | 118(43.2) |
| N1 | 53(19.4) |
| NX | 64(23.4) |
| 未知 | 38(13.9) |
| M分期 | |
| M0 | 101(37.0) |
| M1 | 10(3.7) |
| MX | 122(44.7) |
| 未知 | 40(14.6) |
从TCGA数据库获取的子宫颈癌表达谱共鉴定出13 413个lncRNA。HADb数据库共获得232个自噬相关基因。构建共表达网络用以识别自噬相关的lncRNA。最终,577个lncRNA被鉴定为自噬相关lncRNA(均r > 0.4,P < 0.001)。
2.3 自噬相关lncRNA的预后模型构建为了获得对子宫颈癌患者具有预后价值的自噬相关lncRNA,对之前获得的577个自噬相关lncRNA进行单因素Cox分析。单因素分析显示,共有50个与自噬相关的lncRNA在子宫颈癌中具有预后价值(均P < 0.01,图 1)。对单因素分析中差异具有统计学意义(P < 0.01)的自噬相关lncRNA进行多因素Cox分析,并基于AIC确定最优预测模型。多因素Cox回归显示,共有11个与自噬相关的lncRNA是独立的预后因素(均P < 0.05,表 2)。在11个与自噬相关的lncRNA中,8个为保护因素(ERICH6-AS1、AC027020.2、AC008771.1、ATP2A1-AS1、AC099568.2、AC096992.2、ZNF674-AS1和LINC01943),其余为危险因素(HCG15、AC004540.2和KMT2E-AS1)。这11个与自噬相关的lncRNA被用来构建预后模型。风险评分的计算公式如下:Risk score=0.885 06×exp(HCG15)+0.739 15×exp(AC004540.2)+0.457 62×exp(KMT2E-AS1)+(-0.372 30)×exp(ERICH6-AS1)+(-0.466 97)×exp(AC027020.2)+(-0.503 51)×exp(AC008771.1)+(-0.506 15)×exp(ATP2A1-AS1)+(-0.615 57)×exp(AC099568.2)+(-0.822 96)×exp(AC096992.2)+(-0.967 28)×exp(ZNF674-AS1)+(-1.582 40)×exp(LINC01943).
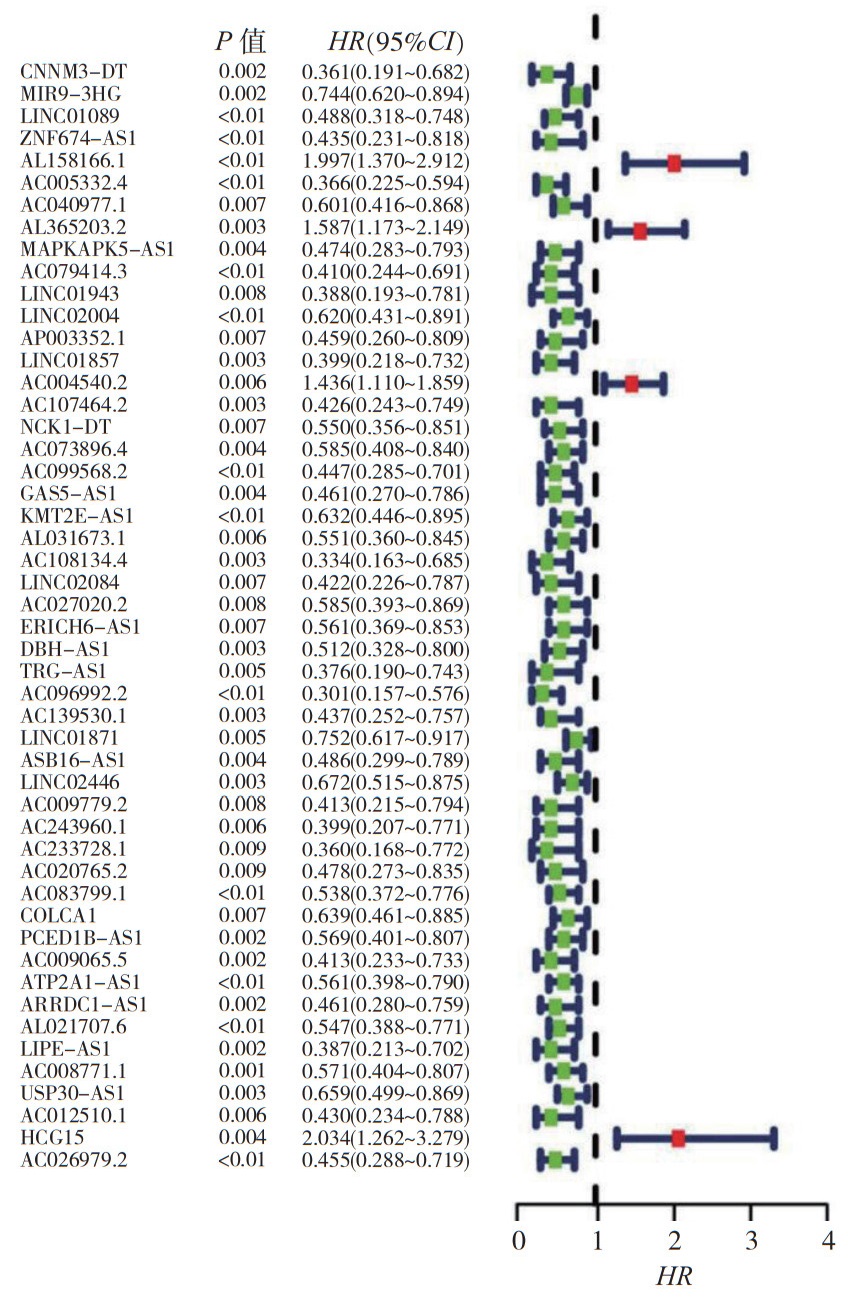
|
| 图 1 通过单因素Cox分析筛选50个与子宫颈癌预后相关的自噬相关lncRNA的森林图 Fig.1 Forest map of 50 autophagy-related lncRNAs associated with the prognosis of cervical cancer screened by univariate Cox analysis |
| lncRNA | 回归系数 | HR(95%CI) | P值 |
| ZNF674-AS1 | -0.967 | 0.380(0.166~0.868) | 0.022 |
| LINC01943 | -1.582 | 0.205(0.088~0.479) | < 0.01 |
| AC004540.2 | 0.739 | 2.094(1.585~2.767) | < 0.01 |
| AC099568.2 | -0.616 | 0.540(0.301~0.971) | 0.039 |
| KMT2E-AS1 | 0.458 | 1.580(1.040~2.402) | 0.032 |
| AC027020.2 | -0.467 | 0.627(0.403~0.974) | 0.038 |
| ERICH6-AS1 | -0.372 | 0.689(0.440~1.078) | 0.103 |
| AC096992.2 | -0.823 | 0.439(0.212~0.910) | 0.027 |
| ATP2A1-AS1 | -0.506 | 0.603(0.428~0.849) | 0.004 |
| AC008771.1 | -0.504 | 0.604(0.409~0.892) | 0.011 |
| HCG15 | 0.885 | 2.423(1.418~4.142) | 0.001 |
根据风险评分计算公式,计算每例患者的风险评分,根据风险评分中位数(1.072)将患者分为高危组和低危组。生存分析显示,低危组的预后优于高危组(P < 0.01,图 2)。根据所得到的模型,将低危组与高危组的患者按风险评分高低排列(图 3A)。随着患者风险评分的增加患者死亡例数增加,提示风险评分有利于评估患者的预后情况(图 3B)。纳入预后风险模型的自噬相关lncRNA在低危组与高危组中表达情况的热图见图 3C。单因素Cox回归分析显示,风险评分是预后指标(HR=1.106,95%CI:1.078~1.133,P < 0.01;图 4A)。多因素Cox回归分析也显示,风险评分是独立的预后因素(HR=1.103,95%CI:1.071~1.136,P < 0.01;图 4B)。ROC曲线显示,预后模型的AUC为0.836,预测效果高于其他临床指标(图 4C)。
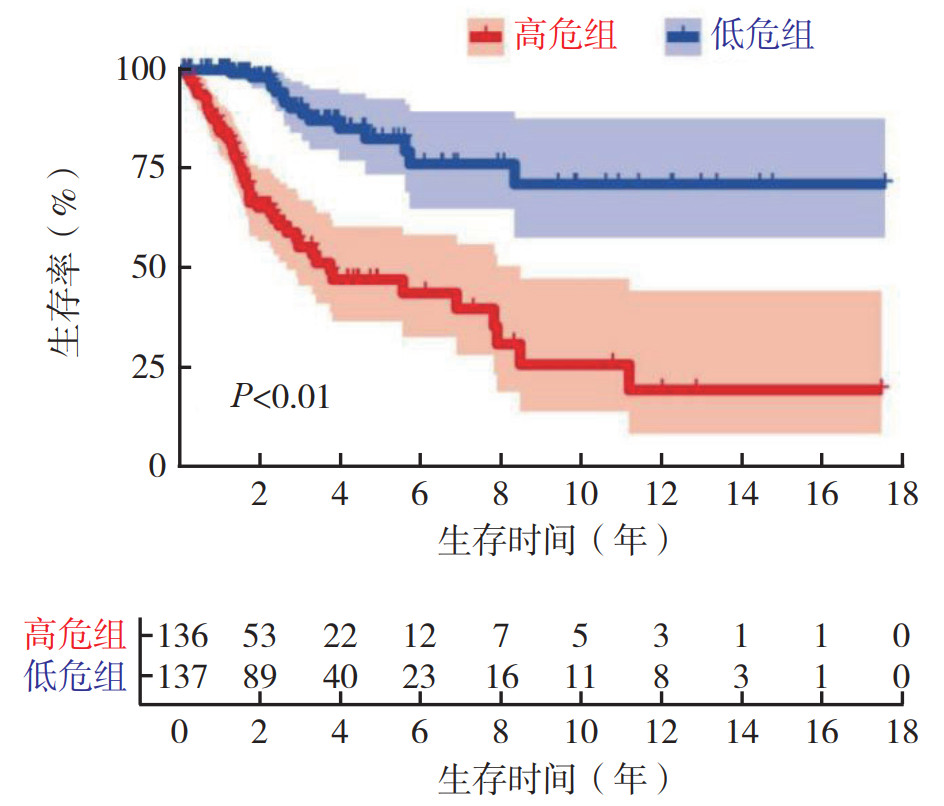
|
| 图 2 高危组和低危组子宫颈癌患者的Kaplan-Meier生存曲线 Fig.2 Kaplan-Meier survival curves of high-risk and lowrisk cervical cancer patients |
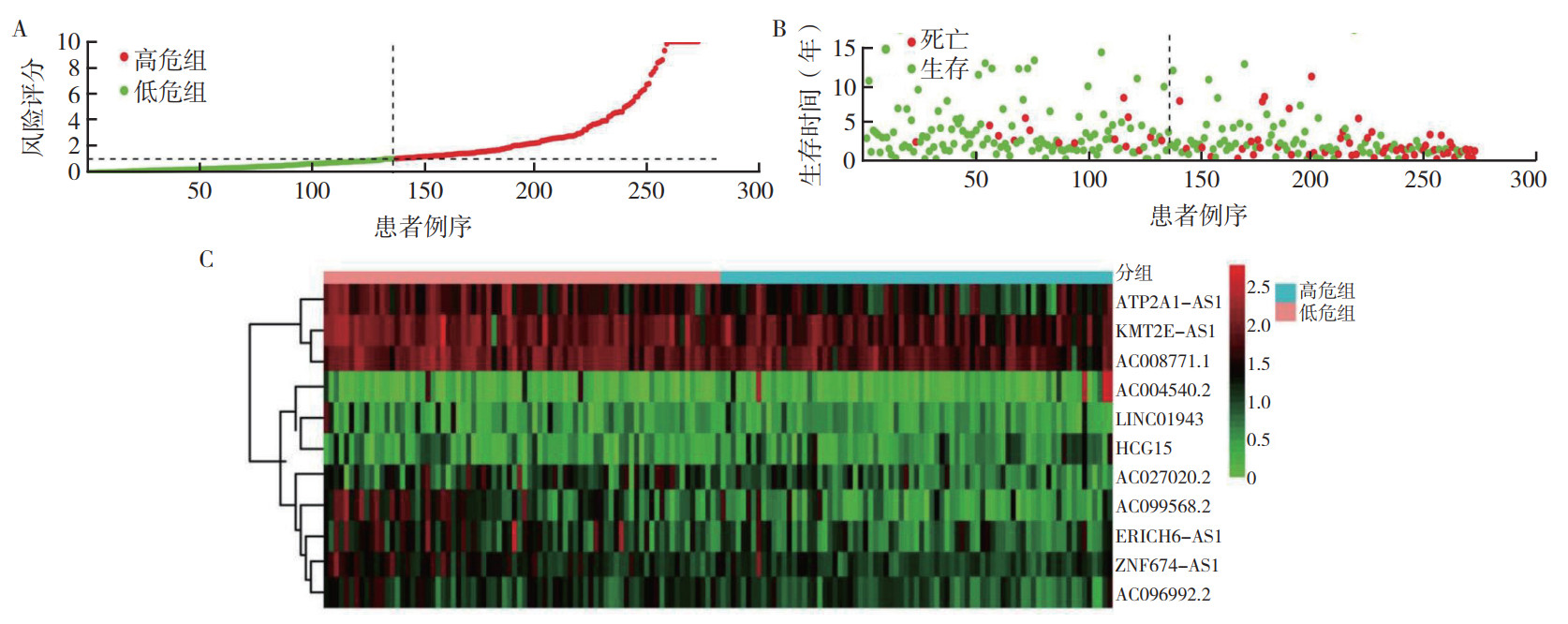
|
| 注 A:低危组与高危组子宫颈癌患者的风险评分;B:低危组与高危组子宫颈癌患者随访的生存状态;C:纳入预后风险模型中的11个自噬相关lncRNA表达热图 图 3 子宫颈癌自噬相关lncRNA预后模型 Fig.3 Prognostic model of autophagy-related lncRNAs for cervical cancer |
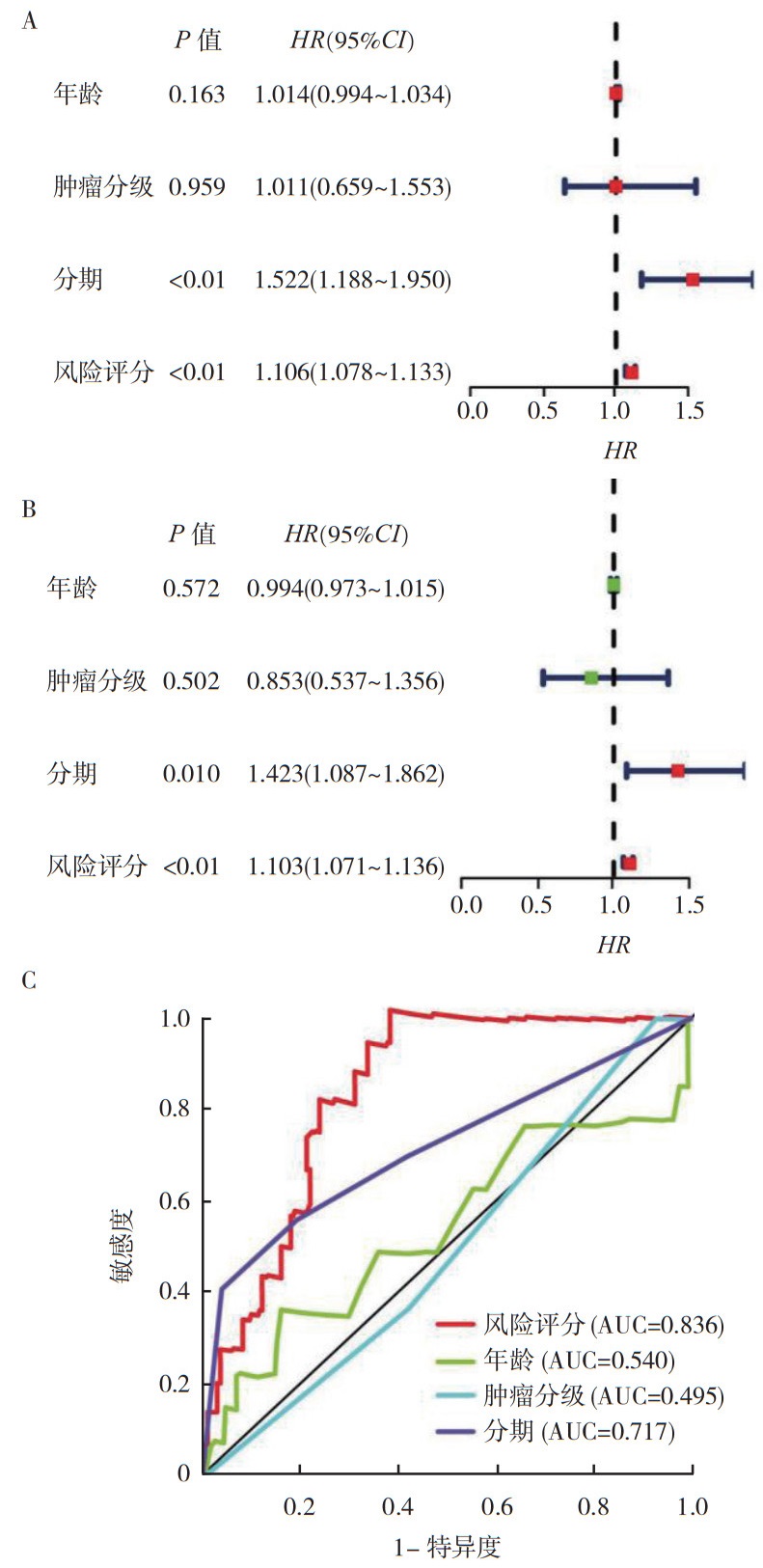
|
| 注 A:子宫颈癌的单因素Cox回归分析的森林图;B:子宫颈癌的多因素Cox回归分析的森林图;C:风险评分、年龄、肿瘤分级和分期的ROC曲线;AUC:曲线下面积(area under the curve) 图 4 基于自噬相关lncRNA的风险评分和其他临床指标对子宫颈癌患者预后的预测性能比较 Fig.4 Comparison of predictive performance of risk score based on autophagy-related lncRNAs and other clinical indicators for the prognosis of cervical cancer patients |
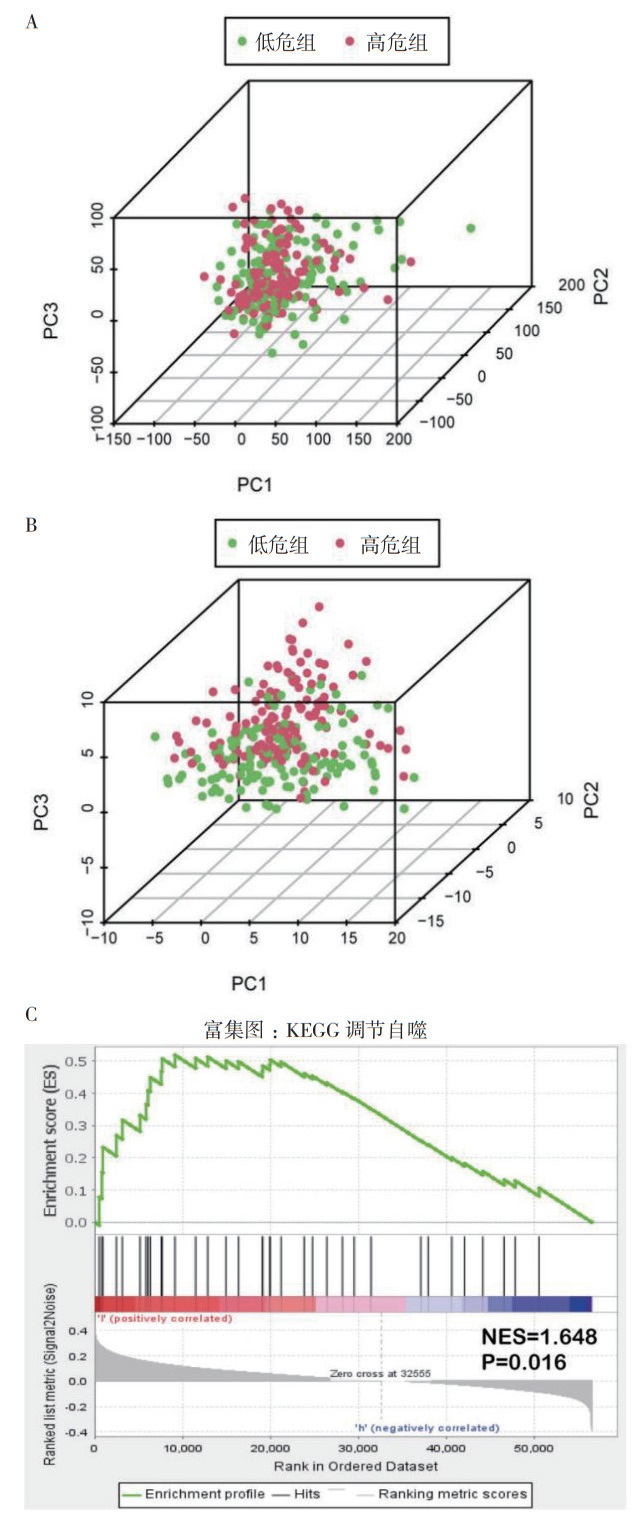
基于模型自噬相关lncRNA和全基因表达谱,采用PCA方法,探讨高危人群和低危人群的分布特征。对于整个转录组表达数据,PCA显示高危组和低危组是混合的(图 5A)。根据模型中自噬相关lncRNA的表达可以区分高危组和低危组,说明自噬相关基因的表达状态在高危组和低危组之间的分布差异具有统计学意义(P < 0.01,图 5B)。进一步分析GSEA发现,低危组与高危组在KEGG调节自噬方面有富集区别,低危组相关基因集富集(P < 0.05,图 5C)。
|
| 注 A:主成分分析显示,整个基因在低危组和高危组中是混合的;B:主成分分析显示,自噬相关基因可以更好区分低危组和高危组;C:GSEA富集分析显示,低危组和高危组的自噬状态不同;NES:标准化富集分数(normalized enrichment score) 图 5 自噬相关lncRNA预后模型的富集分析和主成分分析 Fig.5 Enrichment analysis and principal component analysis of the prognostic model of autophagy-related lncRNAs |
在过去的几十年里,子宫颈癌的发病率和死亡率一直呈上升趋势。尽管接受了化疗和手术等治疗,但晚期患者的5年生存率仍 < 40%。因此,识别有效的生物标志物对子宫颈癌患者的治疗是有益的。lncRNA的失调与包括肿瘤在内的多种人类疾病有关。在许多人类肿瘤中,lncRNA越来越显示出潜在的诊断和预后价值。有研究联合lncRNA Lnc-cCSC1构建了结直肠癌预后模型,预测行根治术后患者1、3和5年总生存率的AUC分别为0.796、0.817和0.831,优于传统的TMN分期系统[21]。在子宫颈癌方面,有研究构建了由6个lncRNA组成的预测子宫颈癌患者的预后模型,AUC达0.65[22]。由10个lncRNA组成的预后模型也可以很好地预测子宫颈癌患者的预后[23]。由于lncRNA也可以参与自噬的动态过程,本研究探索并建立一个以自噬相关lncRNA为指导的预后模型。一项胶质瘤研究通过构建自噬基因lncRNA共表达网络,构建的基于10个自噬相关lncRNA的模型可以有效预测患者的预后[24]。此外,自噬相关的lncRNA在结肠癌、膀胱尿路上皮癌和乳腺癌中已被报道构建预后模型,具有良好的预测效率[20, 24-25]。但在子宫颈癌中未见报道,因此本研究尝试构建自噬相关的lncRNA预后模型来预测子宫颈癌患者的预后。
本研究构建了包含11个与自噬相关的lncRNA(ERICH6-AS1、AC027020.2、AC008771.1、ATP2A1-AS1、AC099568.2、AC096992.2、ZNF674-AS1、LINC01943、HCG15、AC004540.2和KMT2E-AS1)的预后模型。生存分析显示,低危组与高危组预后差异具有统计学意义(P < 0.01)。单因素和多因素分析表明,风险评分可作为子宫颈癌预后的独立预测指标(P < 0.01)。ROC曲线显示,风险评分的AUC达0.836,高于其他临床特征,预测效果较好。
在肝细胞癌(hepatocellular carcinoma,HCC)中,采用实时定量反转录-聚合酶链反应(reverse transcription-polymerase chain reaction,RT-PCR)检测137例HCC患者肿瘤组织及癌旁正常组织中ZNF674-AS1的表达[26]。结果显示,ZNF674-AS1在肿瘤组织中的表达下调,且ZNF674-AS1低表达与HCC患者预后不良相关。ZNF674-AS1也是胶质瘤自噬相关lncRNA预测模型的组成部分。在胶质瘤中,HCG15在胶质瘤组织和细胞中高表达,敲除HCG15可抑制血管拟态(vascular mimicry,VM)的形成,这是颅内血液供应的途径之一;提示PABCC5/HCG15/ZNF331反馈回路在胶质瘤VM的形成中发挥重要作用[27]。除ZNF674-AS1和HCG15外,其余9个lncRNA相关研究未见报道。
本研究也有一些局限性:(1)数据来自公共数据库,且表达式数据类型是回顾性的;(2)没有独立的验证组来验证模型的有效性;(3)模型中的某些lncRNA分子机制及其在子宫颈癌中的作用尚不清楚,需要进一步研究。总而言之,本研究构建了一个包含11个自噬相关lncRNA的预后模型来评估子宫颈癌患者的预后。模型与子宫颈癌患者生存率相关,高危组预后较差。该模型是子宫颈癌患者重要的独立预后因素(P < 0.01)。因此,该模型有利于给子宫颈癌患者提供更有效的个体化治疗管理。
| [1] |
Vu M, Yu J, Awolude OA, et al. Cervical cancer worldwide[J]. Curr Probl Cancer, 2018, 42(5): 457-465. DOI:10.1016/j.currproblcancer.2018.06.003 |
| [2] |
Waggoner SE. Cervical cancer[J]. Lancet, 2003, 361(9376): 2217-2225. DOI:10.1016/S0140-6736(03)13778-6 |
| [3] |
Small W Jr, Bacon MA, Bajaj A, et al. Cervical cancer: a global health crisis[J]. Cancer, 2017, 123(13): 2404-2412. DOI:10.1002/cncr.30667 |
| [4] |
Zhang Y, Li X, Zhang J, et al. Natural killer T cell cytotoxic activity in cervical cancer is facilitated by the LINC00240/microRNA-124-3p/STAT3/MICA axis[J]. Cancer Lett, 2020, 474: 63-73. DOI:10.1016/j.canlet.2019.12.038 |
| [5] |
Glick D, Barth S, MacLeod KF. Autophagy: cellular and molecular mechanisms[J]. J Pathol, 2010, 221(1): 3-12. DOI:10.1002/path.2697 |
| [6] |
Li XH, He SK, Ma BY. Autophagy and autophagy-related proteins in cancer[J]. Mol Cancer, 2020, 19(1): 12. DOI:10.1186/s12943-020-1138-4 |
| [7] |
Kimmelman AC, White E. Autophagy and tumor metabolism[J]. Cell Metab, 2017, 25(5): 1037-1043. DOI:10.1016/j.cmet.2017.04.004 |
| [8] |
Chen HY, White E. Role of autophagy in cancer prevention[J]. Cancer Prev Res (Phila), 2011, 4(7): 973-983. DOI:10.1158/1940-6207.CAPR-10-0387 |
| [9] |
谭化姣, 廖琼. 自噬与非小细胞肺癌治疗研究进展[J]. 实用肿瘤杂志, 2021, 36(3): 209-214. DOI:10.13267/j.cnki.syzlzz.2021.044 |
| [10] |
Levy JMM, Towers CG, Thorburn A. Targeting autophagy in cancer[J]. Nat Rev Cancer, 2017, 17(9): 528-542. DOI:10.1038/nrc.2017.53 |
| [11] |
Orfanelli T, Jeong JM, Doulaveris G, et al. Involvement of autophagy in cervical, endometrial and ovarian cancer[J]. Int J Cancer, 2014, 135(3): 519-528. DOI:10.1002/ijc.28524 |
| [12] |
Mattoscio D, Medda A, Chiocca S. Human papillomavirus and autophagy[J]. Int J Mol Sci, 2018, 19(6): E1775. DOI:10.3390/ijms19061775 |
| [13] |
Song LL, Wang LL, Pan XL, et al. lncRNA OIP5-AS1 targets ROCK1 to promote cell proliferation and inhibit cell apoptosis through a mechanism involving miR-143-3p in cervical cancer[J]. Revista Brasileira De Pesquisas Med E Biol, 2020, 53(1): e8883. |
| [14] |
Liang HL, Zhang CY, Guan HY, et al. LncRNA DANCR promotes cervical cancer progression by upregulating ROCK1 via sponging miR-335-5p[J]. J Cell Physiol, 2019, 234(5): 7266-7278. DOI:10.1002/jcp.27484 |
| [15] |
Zhang L, Liu SK, Song LL, et al. SP1-induced up-regulation of lncRNA LUCAT1 promotes proliferation, migration and invasion of cervical cancer by sponging miR-181a[J]. Artif Cells Nanomed Biotechnol, 2019, 47(1): 556-564. |
| [16] |
Zeng J, Ma YX, Liu ZH, et al. LncRNA SNHG7 contributes to cell proliferation, invasion and prognosis of cervical cancer[J]. Eur Rev Med Pharmacol Sci, 2019, 23(21): 9277-9285. |
| [17] |
Zou RM, Chen XJ, Jin XJ, et al. Up-regulated BCAR4 contributes to proliferation and migration of cervical cancer cells[J]. Surg Oncol, 2018, 27(2): 306-313. DOI:10.1016/j.suronc.2018.05.013 |
| [18] |
Zhang L, Yao HR, Liu SK, et al. Long noncoding RNA ROR1-AS1 overexpression predicts poor prognosis and promotes metastasis by activating Wnt/β-catenin/EMT signaling cascade in cervical cancer[J]. Eur Rev Med Pharmacol Sci, 2020, 24(6): 2928-2937. |
| [19] |
Yao HH, Han B, Zhang Y, et al. Non-coding RNAs and autophagy[J]. Adv Exp Med Biol, 2019, 1206: 199-220. |
| [20] |
Zhou WG, Zhang SJ, Li HB, et al. Development of prognostic indicator based on autophagy-related lncRNA analysis in colon adenocarcinoma[J]. Biomed Res Int, 2020, 2020: 9807918. |
| [21] |
黄徐建, 李敬东, 杨嘉琳, 等. 联合长链非编码RNA Lnc-cCSC1构建结直肠癌临床预后个体化模型[J]. 实用肿瘤杂志, 2021, 36(5): 412-418. |
| [22] |
Cheng YP, Yang S, Shen Y, et al. The role of high-risk human papillomavirus-related long non-coding RNAs in the prognosis of cervical squamous cell carcinoma[J]. DNA Cell Biol, 2020, 39(4): 645-653. DOI:10.1089/dna.2019.5167 |
| [23] |
Shen L, Yu HC, Liu M, et al. A ten-long non-coding RNA signature for predicting prognosis of patients with cervical cancer[J]. Onco Targets Ther, 2018, 11: 6317-6326. DOI:10.2147/OTT.S175057 |
| [24] |
Luan FK, Chen WJ, Chen M, et al. An autophagy-related long non-coding RNA signature for glioma[J]. FEBS Open Bio, 2019, 9(4): 653-667. DOI:10.1002/2211-5463.12601 |
| [25] |
Li XY, Jin F, Li Y. A novel autophagy-related lncRNA prognostic risk model for breast cancer[J]. J Cell Mol Med, 2021, 25(1): 4-14. DOI:10.1111/jcmm.15980 |
| [26] |
Zhang LF, He TY, Yan YC, et al. Expression and clinical significance of the novel long noncoding RNA ZNF674-AS1 in human hepatocellular carcinoma[J]. Biomed Res Int, 2016, 2016: 3608914. |
| [27] |
Jing FK, Ruan XL, Liu XB, et al. The PABPC5/HCG15/ZNF331 feedback loop regulates vasculogenic mimicry of glioma via STAU1-mediated mRNA decay[J]. Mol Ther Oncolytics, 2020, 17: 216-231. DOI:10.1016/j.omto.2020.03.017 |
 2022, Vol. 37
2022, Vol. 37


