文章信息
- 赵春兰, 张会辰, 王维娜
- Zhao Chunlan, Zhang Huichen, Wang Weina
- UBTF在乳腺癌中的表达及其对乳腺癌细胞生物学行为的影响
- Expression of UBTF in breast cancer and its eff ect on biological behavior of breast cancer cells
- 实用肿瘤杂志, 2022, 37(5): 403-410
- Journal of Practical Oncology, 2022, 37(5): 403-410
基金项目
- 河北省科学技术研究与发展计划项目(17277752D)
-
通信作者
- 赵春兰, E-mail: zhaochunlanhebei@163.com
-
文章历史
- 收稿日期:2021-07-19
2. 河北大学附属医院病理科, 河北 保定 071000
2. Department of Pathology, Affi liated Hospital of Hebei University, Baoding 071000, China
全球女性恶性肿瘤相关死因中乳腺癌居首[1]。2018年全球癌症统计数据表明,乳腺癌新增病例占比11.6%,乳腺癌死亡占比6.6%[2]。乳腺癌依据雌激素受体(estrogen receptor,ER)、孕激素受体(progesterone receptor,PR)、人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)和Ki-67等指标,可分为5种分子亚型,即Luminal A、Luminal B、HER2过表达类型、三阴性乳腺癌(triple negative breast cancer, TNBC)和正常乳腺样型[3]。目前,乳腺癌的诊断和治疗技术得到很大的发展,但由于肿瘤的复发、转移和抗药性的分子机制仍未研究清楚,一些乳腺癌患者的预后仍不理想[4]。因此,寻找早期诊断和评估患者预后的分子靶标及其发病机制是提高乳腺癌患者生存率的重要手段。
上游结合转录因子(upstream binding transcription factor,UBTF)是人类基因UBTF编码的蛋白质[5]。UBTF与selectivity factor 1(SL1)因子[由TATA结合蛋白(TATA-binding protein,TBP)和3个转录辅因子TATA-box binding protein associated factor 1(TAF1)组成的四聚体]相互作用,是18S、5.8S和28S转录过程中核糖体RNA的必需转录因子[6]。UBTF是high mobility group(HMG)盒DNA结合蛋白家族的成员,在人体中有相对分子量为94 000和97 000的2种多肽链,具有DNA结合结构域和反式激活结构域的核仁磷酸化蛋白,在核糖体RNA转录中,可作为前启动复合物关键成分,介导RNA聚合酶Ⅰ(RNA polymerase Ⅰ,Pol Ⅰ)募集到rDNA启动子区域[7]。UBTF蛋白在染色质重塑和前rRNA加工中也可能起重要作用,其活性受磷酸化和乙酰化作用的调节[8]。UBTF是新型的癌基因,可影响肿瘤细胞的迁移、侵袭和增殖[9]。但有关UBTF在乳腺癌中的表达及生物学特性研究较少。因此,本研究首先检测UBTF在乳腺癌中的表达情况并分析其与患者临床病理特征的关系,构建UBTF敲低及过表达质粒并转染到乳腺癌细胞株MDA-MB-231中,检测其对MDA-MB-231细胞体外增殖、迁移和侵袭的作用,为UBTF促进乳腺癌的发生和发展的分子机制提供科学依据。
1 材料与方法 1.1 主要试剂DMEM培养液购自美国格兰德岛生物公司(Gibco)。胎牛血清购自德国Pan-biotech公司。TRIzol购自北京索莱宝科技有限公司。反转录试剂盒和qPCR-mix体系购自美国普洛麦格生物技术有限公司(Promega)。引物均由上海生工生物工程股份有限公司合成。
1.2 研究对象及标本来源收集保定市妇幼保健院乳腺科2018年1月至2020年1月住院的63例女性乳腺癌手术患者。患者年龄32~83岁,中位年龄60岁。所有乳腺癌患者术前均未接受放疗或化疗。从乳腺癌原发病灶及距原发病灶 > 5 cm的正常组织中取样。将切下的标本分成两部分使用:一部分立即放入RNAlater液中,在4℃的冰箱中孵育过夜,然后保存在-80℃的冰箱中,用于后续实验提取RNA;另一部分固定于4%甲醛溶液中,制备蜡块并切片,切片厚度为4 μm。HE染色和病理分析:癌组织标本由3位病理医师确诊为乳腺癌,且在正常组织中未见癌细胞浸润。所有标本均具有完整的病理诊断和临床资料。本研究实验方案由保定市妇幼保健院伦理委员会批准通过,所有受试者均已签署知情同意书。
1.3 细胞培养乳腺癌细胞株MDA-MB-231由河北医科大学第四医院惠赠,在含有10%胎牛血清的DMEM培养液中培养,置于37℃、5%CO2的培养箱中。
1.4 方法 1.4.1 qPCR法检测UBTFmRNA表达根据TRIzol试剂说明书,从MDA-MB-231细胞、乳腺癌和相应正常组织样品中提取总RNA。根据反转录试剂盒的说明书,将总RNA反转录为cDNA,并使用β-肌动蛋白(β-actin,ACTB)作为内参,用cDNA作为qPCR扩增的模板。qPCR引物如下:UBTF上游引物为5'-GCCAGATGCCACTACGAAGG-3',下游引物为5'-CCTCACTGATGTTCAGCTCT-3';ACTB上游引物为5'-ACCGAGCGCGGCTACAG-3',下游引物为5'-CTTAATGT CACGCACGATTTCC-3'。qPCR反应条件:95℃预变性10 min后;95℃变性15 s、60℃退火30 s、72℃延伸30 s,共35个循环;72℃延伸7 min,每个样本设3个复孔。根据每孔荧光信号达到阈值时经历的循环数记为该组织中UBTF和ACTB转录水平的CT值。采用相对定量法:△CT=CTUBTF-CTACTB,△△CT=△CT癌组织-△CT配对癌旁组织,以N=2-△△CT表示相对表达量,其数值表示癌组织相对于正常组织的相对倍数,实验重复3次取平均值。
1.4.2 UBTF敲低/过表达质粒构建通过上海吉玛基因公司设计并合成UBTF siRNA,命名为si-UBTF,另附si-NC作为对照。UBTF过表达载体由上海生工合成pcDNA3.1-UBTF质粒,抗性Ampicillin,pcDNA3.1质粒作为对照。将含有目的载体的甘油菌液储存于-80℃。
1.4.3 UBTF敲低/过表达质粒转染MDA-MB-231细胞选择对数生长期MDA-MB-231细胞进行胰蛋白酶消化并计数,将其均匀铺于6孔板上,细胞长至80%时转染。在2个EP管中分别加入无血清培养液和UBTF敲低/过表达质粒以及无血清培养液和转染试剂LipofectamineTM 2000[转染试剂LipofectamineTM 2000(体积)∶质粒(质量)=2∶1],在室温下孵育5 min。混合2个EP管中的液体,室温下孵育20 min。将200 μL转染试剂质粒混合物添加到si-UBTF组/pcDNA3.1-UBTF组的1孔中,另1孔加等量空质粒作为si-NC组/pcDNA3.1-NC组,另设置1孔为常规细胞培养组(MDA-MB-231组)。培养24~48 h后,从MDA-MB-231细胞中提取总RNA,并观察UBTF转染后的mRNA表达。实验重复3次取平均值。
1.4.4 MTS实验将各组MDA-MB-231细胞培养24 h后常规消化并悬浮于无血清培养液中,调整细胞浓度分别接种于96孔板(1 000个/孔),每组设置6个复孔。分别于细胞贴壁后0、24、48、72和96 h后,每孔加入MTS试剂20 μL(l 500 μg/mL),孵育2 h后用酶标仪测定492 nm处的吸光度(absorbance,A)值,代表细胞增殖水平。实验重复3次取平均值。
1.4.5 细胞集落形成实验将各组MDA-MB-231细胞培养24 h后,常规消化并悬浮于无血清培养液中,调整细胞浓度分别接种于6孔板(3 000个/孔),常规培养1周。4%多聚甲醛固定后结晶紫染色,显微镜下计数细胞集落形成数,> 50个细胞为1个细胞集落,分别计算各组细胞细胞集落形成率。实验重复3次取平均值。
1.4.6 划痕实验将各组MDA-MB-231细胞培养24 h后,常规消化悬浮于无血清培养液中,调整细胞浓度分别接种于6孔板(5×105个/孔)。待细胞完全融合时,用200 μL移液枪头垂直划痕。用PBS洗去划落细胞,并加入2 mL无血清培养液继续常规培养。分别于划痕后0、12和24 h在倒置显微镜下观察细胞的划痕间距,计算细胞迁移率,细胞迁移率=(划痕宽度0 h-划痕宽度12 h/24 h)/划痕宽度0 h×100%。实验重复3次取平均值。
1.4.7 Transwell侵袭实验将各组MDA-MB-231细胞常规消化悬浮于无血清培养液,调整细胞浓度接种于小室(1×105个/孔),小室上层加20 μL Matrigel胶,下层加600 μL含10%胎牛血清的完全培养液。常规培养24 h后取出小室,用棉签擦去基质胶和上室内的细胞,PBS洗2次,4%多聚甲醛固定后结晶紫染液染色,显微镜下计数5个随机视野内的细胞数,比较si-UBTF组/pcDNA3.1-UBTF组和MDA-MB-231组的差异。实验重复3次取平均值。
1.5 统计学分析采用SPSS21.0统计学软件进行数据分析。计量资料采用均数±标准差(x±s)表示,两组间比较采用Student's t检验,多组间均数比较采用单因素方差分析,进一步两两比较采用LSD-t检验。以P < 0.05为差异具有统计学意义。
2 结果 2.1 UBTF mRNA在乳腺癌组织中的表达乳腺癌组织中UBTF mRNA的表达量较正常组织增高,差异具有统计学意义(t=-5.246,P < 0.01;图 1)。TNM分期Ⅲ+Ⅳ期患者UBTF mRNA水平高于Ⅰ+Ⅱ期者,差异具有统计学意义(t=-2.224,P=0.030;表 1)。UBTF mRNA水平在患者年龄、组织分化程度、淋巴结转移、ER、PR、HER2和Ki-67表达方面比较,差异均无统计学意义(均P > 0.05)。
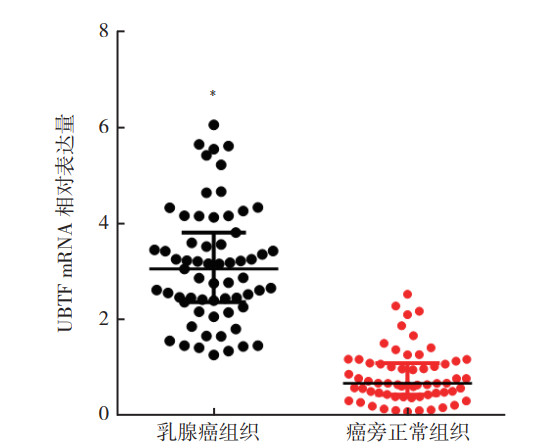
|
| 注 *P < 0.01 图 1 乳腺癌组织及癌旁正常组织中UBTF mRNA表达 Fig.1 Expression of UBTF mRNA in breast cancer tissues and adjacent normal tissues |
| 临床特征 | 例数 | UBTF mRNA | t值 | P值 |
| 年龄 | -0.684 | 0.497 | ||
| ≤60岁 | 28 | 2.597±1.879 | ||
| > 60岁 | 35 | 2.965±2.296 | ||
| 淋巴结转移 | 0.821 | 0.415 | ||
| 有 | 39 | 2.973±2.312 | ||
| 无 | 24 | 2.522±1.751 | ||
| 组织分化程度 | 0.163 | 0.871 | ||
| 中~高 | 36 | 2.839±1.944 | ||
| 低 | 27 | 2.751±2.356 | ||
| ER | 1.526 | 0.132 | ||
| 阳性 | 37 | 3.138±2.333 | ||
| 阴性 | 26 | 2.322±1.678 | ||
| PR | 1.998 | 0.050 | ||
| 阳性 | 31 | 3.329±2.612 | ||
| 阴性 | 32 | 2.290±1.332 | ||
| HER2 | 0.426 | 0.671 | ||
| 阳性 | 44 | 2.876±2.098 | ||
| 阴性 | 19 | 2.628±2.191 | ||
| Ki-67 | 0.514 | 0.609 | ||
| 阳性 | 38 | 2.913±2.376 | ||
| 阴性 | 25 | 2.613±1.667 | ||
| TNM分期 | -2.224 | 0.030 | ||
| Ⅰ+Ⅱ期 | 20 | 2.091±1.343 | ||
| Ⅲ+Ⅳ期 | 43 | 3.295±2.234 | ||
| 注 UBTF:上游结合转录因子(upstream binding transcription factor);ER:雌激素受体(estrogen receptor);PR:孕激素受体(progesterone receptor);HER2:人表皮生长因子受体2(human epidermal growth factor receptor 2) | ||||
UBTF敲低载体转染MDA-MB-231细胞48 h后,UBTF mRNA表达减低,差异具有统计学意义(F=588.974,P < 0.01;图 2)。MDA-MB-231组和si-NC组比较,差异无统计学意义(t=-1.162,P > 0.05)。
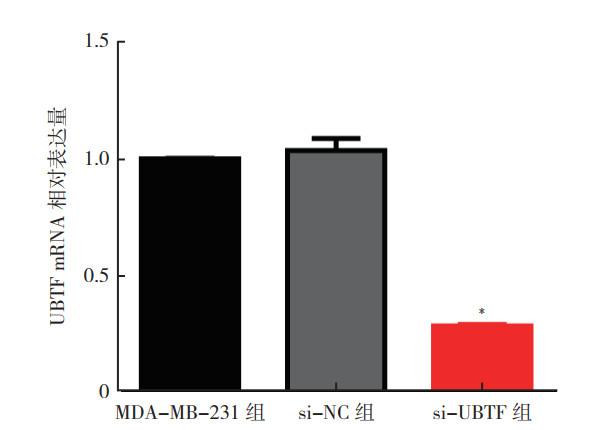
|
| 注 *P < 0.01 图 2 UBTF敲低载体转染MDA-MB-231细胞的转染效率 Fig.2 Transfection efficiency of MDA-MB-231 cells transfected with UBTF knockdown vector |
MTS实验结果显示,在MDA-MB-231细胞中敲低UBTF 96 h后细胞的增殖能力降低(F=94.607,P < 0.01;图 3)。细胞集落形成实验结果显示,si-UBTF组细胞集落形成数较MDA-MB-231组和si-NC组减少(F=145.309,P < 0.01,图 4)。MDA-MB-231组和si-NC组细胞集落形成数比较,差异无统计学意义(t=-0.500,P > 0.05)。
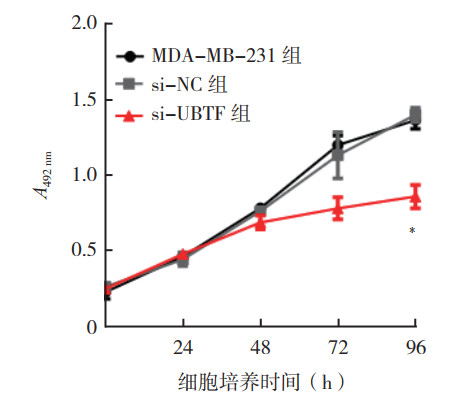
|
| 注 *P < 0.01 图 3 敲低UBTF对MDA-MB-231细胞增殖的影响 Fig.3 Effect of UBTF knockdown on the proliferation of MDA-MB-231 cells |
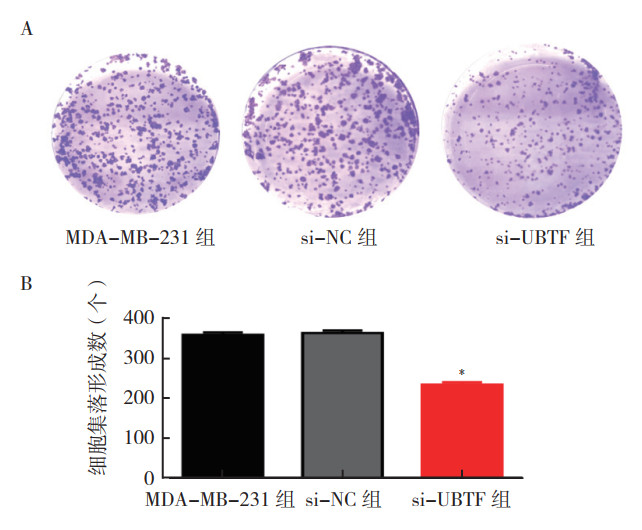
|
| 注 A:细胞集落形成实验;B:细胞集落形成柱状图;*P < 0.01 图 4 敲低UBTF对MDA-MB-231细胞集落形成的影响 Fig.4 Effect of UBTF knockdown on MDA-MB-231 cell colony formation |
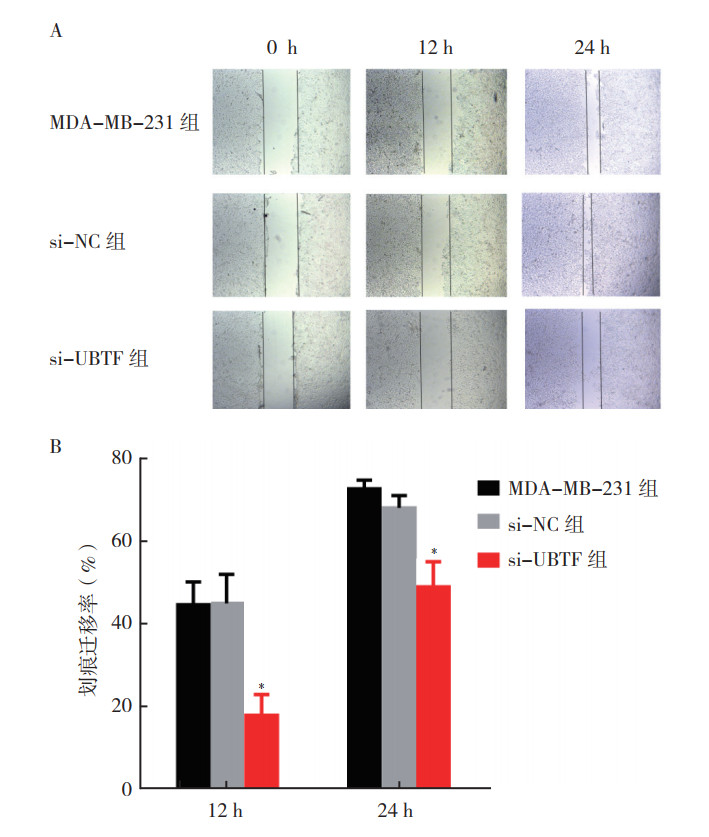
划痕实验结果显示,在MDA-MB-231细胞中敲低UBTF,si-UBTF组12 h划痕迁移率均较MDA-MB-231组和si-NC组降低,差异具有统计学意义(F=48.399,P < 0.01;图 5),MDA-MB-231组和si-NC组划痕迁移率比较,差异均无统计学意义(t=1.175,P=0.305)。si-UBTF组24 h划痕迁移率均较MDA-MB-231组和si-NC组降低,差异具有统计学意义(F=36.445,P < 0.01;图 5),MDA-MB-231组和si-NC组划痕迁移率比较,差异无统计学意义(t=0.697,P=0.488)。
|
| 注 A:细胞划痕实验;B:划痕迁移率柱状图;*P < 0.01 图 5 敲低UBTF对MDA-MB-231细胞迁移能力的影响 Fig.5 Effect of UBTF knockdown on the migration of MDA-MB-231 cells |
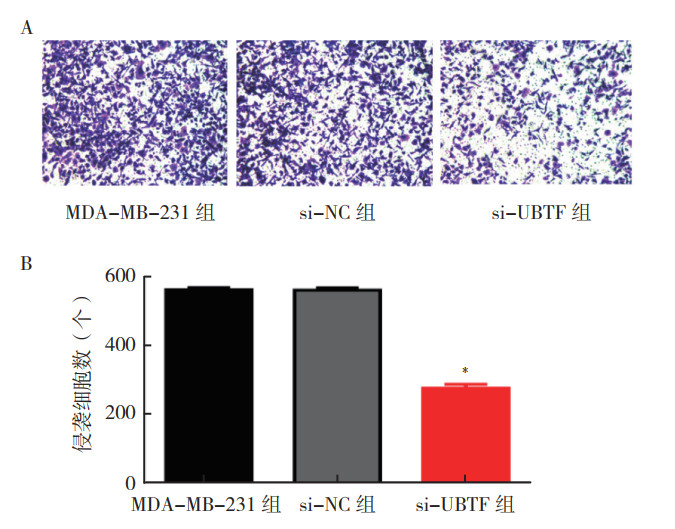
Transwell侵袭实验显示,敲低UBTF 24 h后,si-UBTF组侵袭细胞数与si-NC组和MDA-MB-231组比较减少,差异具有统计学意义(F=366.256,P < 0.01,图 6)。si-NC组和MDA-MB-231组比较,差异无统计学意义(t=0.176,P > 0.05)。
|
| 注 A:细胞侵袭实验;B:细胞侵袭实验柱状图;*P < 0.01 图 6 敲低UBTF对MDA-MB-231细胞侵袭能力的影响 Fig.6 Effect of UBTF knockdown on the invasion of MDA-MB-231 cells |
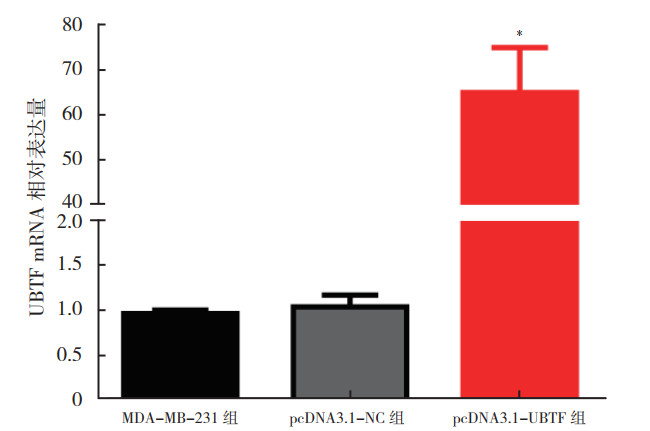
在MDA-MB-231细胞中过表达UBTF 48 h后,UBTF mRNA表达较MDA-MB-231组和pcDNA3.1-NC组上调(F=40.865,P < 0.01;图 7)。MDA-MB-231组和pcDNA3.1-NC组UBTF mRNA表达比较,差异无统计学意义(t=-0.542,P > 0.05)。
|
| 注 *P < 0.01 图 7 UBTF过表达载体转染MDA-MB-231细胞的转染效率 Fig.7 Transfection efficiency of MDA-MB-231 cells transfected with the UBTF overexpressing vector |
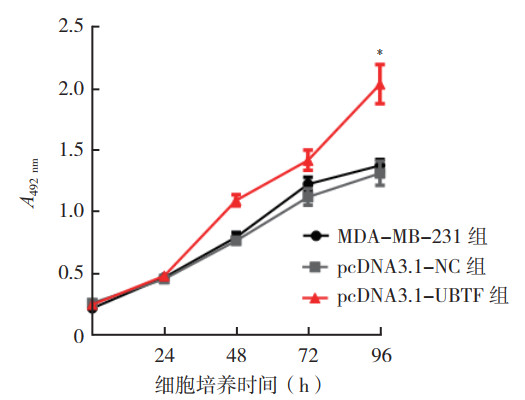
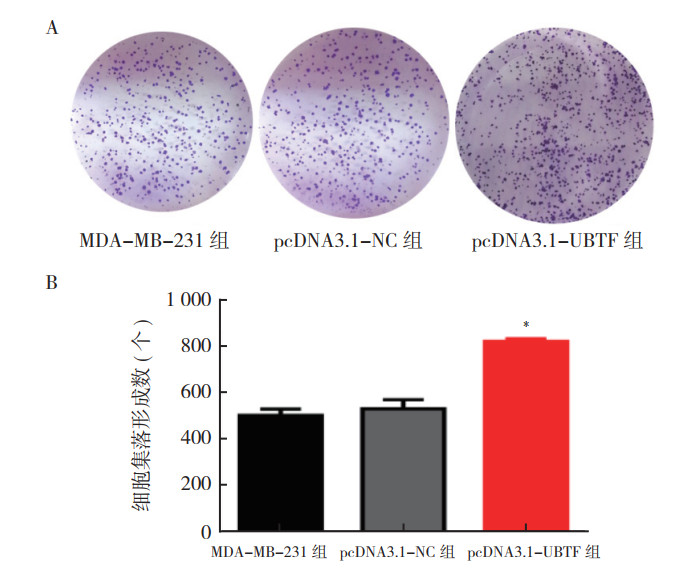
MTS实验结果显示,在MDA-MB-231细胞中过表达UBTF 96 h后,细胞增殖能力较MDA-MB-231组和pcDNA3.1-NC组增强(F=52.312,P < 0.01;图 8)。细胞集落形成实验结果显示,MDA-MB-231细胞过表达UBTF 7 d后细胞集落形成数量较MDA-MB-231组和pcDNA3.1-NC组增加(F=39.138,P < 0.01;图 9),MDA-MB-231组和pcDNA3.1-NC组细胞集落形成数量比较,差异无统计学意义(t=-0.587,P > 0.05)。
|
| 注 *P < 0.01 图 8 过表达UBTF对MDA-MB-231细胞增殖的影响 Fig.8 Effect of UBTF overexpression on the proliferation of MDA-MB-231 cells |
|
| 注 A:细胞集落形成图;B:细胞集落形成柱状图;*P < 0.01 图 9 过表达UBTF对MDA-MB-231细胞集落形成实验的影响 Fig.9 Effect of UBTF overexpression on the colony formation experiment of MDA-MB-231 cells |
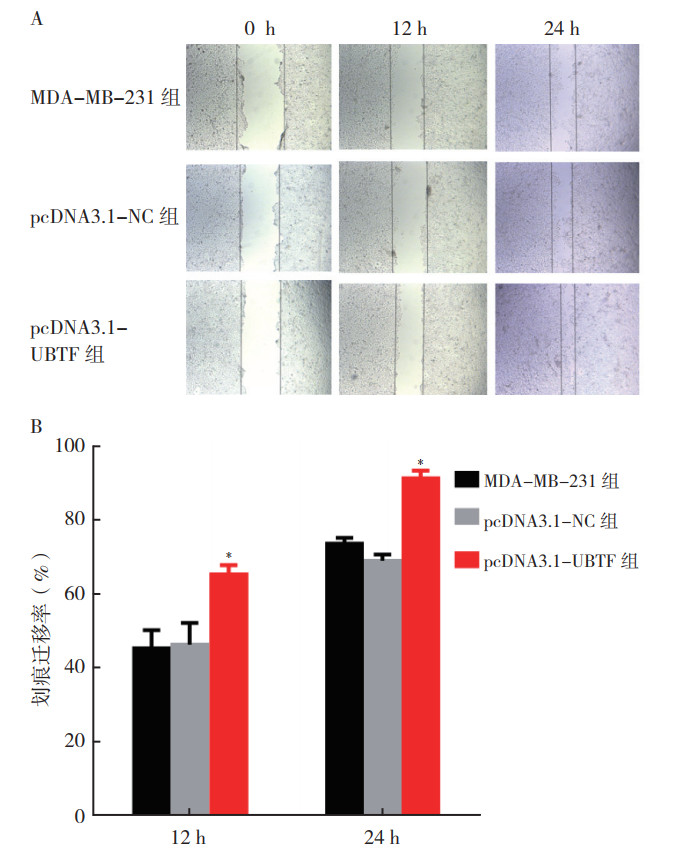
划痕实验结果显示,在MDA-MB-231细胞中过表达UBTF 12 h后,划痕迁移率较MDA-MB-231组和pcDNA3.1-NC组升高(F=27.805,P < 0.01;图 10);MDA-MB-231组和pcDNA3.1-NC组划痕迁移率比较,差异无统计学意义(t=-0.226,P=0.305)。在MDA-MB-231细胞中过表达UBTF 24 h后,划痕迁移率较MDA-MB-231组和pcDNA3.1-NC组升高(F=21.375,P < 0.01;图 10),MDA-MB-231组和pcDNA3.1-NC组划痕迁移率比较,差异无统计学意义(t=0.054,P=0.907)。
|
| 注 A:细胞划痕实验;B:划痕迁移率柱状图;*P < 0.01 图 10 过表达UBTF对MDA-MB-231细胞迁移能力的影响 Fig.10 Effect of UBTF overexpression on the migration of MDA-MB-231 cells |
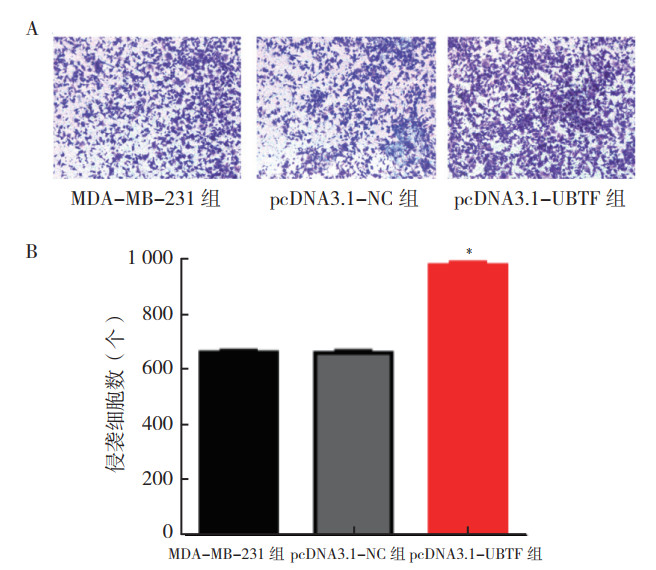
Transwell侵袭实验结果显示,在MDA-MB-231细胞中过表达UBTF 24 h后,通过人工基底膜的细胞数量较pcDNA3.1-NC组和MDA-MB-231组增加(F=533.580,P < 0.01;图 11),而pcDNA3.1-NC组和MDA-MB-231组比较,差异无统计学意义(t=0.176,P > 0.05)。
|
| 注 A:细胞侵袭实验;B:细胞侵袭实验柱状图;*P < 0.01 图 11 过表达UBTF对MDA-MB-231细胞侵袭能力的影响 Fig.11 Effect of UBTF overexpression on the invasion of MDA-MB-231 cells |
核仁转录与肿瘤的发生和发展密切相关,在肿瘤组织中常表现为核仁肥大和核仁功能的增强[10]。主要原因有2个:(1)为应对核糖体合成增多而产生的适应性变化;(2)细胞增殖具体调控机制的改变,癌基因可以直接作用于核仁转录系统,促进核仁转录,为肿瘤细胞增殖作准备[11]。
UBTF是直接与rDNA启动子结合的转录因子,并且是Pol Ⅰ转录中的重要辅助因子之一[12]。UBTF由氨基末端结构域、6个HMG Boxes和羧基末端尾巴组成的二聚体。其中,HMG Box 1、2和3具有与DNA结合的小凹槽结构。羧基末端也称为酸尾,富含丝氨酸,具有多个磷酸化位点,可与SLl结合。因此UBTF是核仁转录过程中必不可少的关键组成部分[13]。同时UBTF在多个肿瘤中高表达,包括肝癌、肺癌、胃癌和结直肠癌[14-17]。本研究首先检测发现,乳腺癌组织中UBTF mRNA水平高于癌旁组织。分析UBTF表达与临床病理数据之间的关系发现,UBTF表达与乳腺癌TNM分期有关。这表明UBTF基因可能在乳腺癌中发挥癌基因作用。
本研究成功构建了UBTF敲低载体并将其转染到人乳腺癌MDA-MB-231细胞中。通过qPCR检测UBTF在MDA-MB-231细胞中的表达。研究显示,用UBTF敲低载体质粒转染后,UBTF在细胞中的表达降低。通过MTS、细胞集落形成、划痕和transwell侵袭实验观察UBTF对体外乳腺癌生物学行为的影响。本研究结果显示,敲低UBTF后,MDA-MB-231细胞的增殖能力、侵袭能力和迁移能力均降低。本研究成功构建了pcDNA3.1-UBTF过表达载体并转染MDA-MB-231细胞。通过生物学功能实验发现过表达UBTF后,MDA-MB-231细胞转移和侵袭的能力增强。其可能机制为:UBTF是核仁转录过程中必不可少的关键组成部分,敲低UBTF可以导致肿瘤组织中核仁功能的减弱,减少适应性变化,最终导致肿瘤细胞生长和增殖受限。同时,UBTF可以在乳腺癌中发挥肿瘤促进因子的作用,UBTF可作为下游靶基因的转录因子而发挥作用,从而影响乳腺癌的增殖、迁移和侵袭。
总之,本研究表明,UBTF在乳腺癌中高表达,并且与乳腺癌的发生和发展密切相关。UBTF的下调可以在体外抑制MDA-MB-231细胞的增殖和侵袭。过表达UBTF后MDA-MB-231细胞转移和侵袭的能力增强,其生物学行为改变。本研究为乳腺癌的分子靶向治疗提供了一些新思路,但具体机制仍有待进一步研究。
| [1] |
Yu FL, Quan F, Xu JY, et al. Breast cancer prognosis signature: linking risk stratification to disease subtypes[J]. Brief Bioinform, 2019, 20(6): 2130-2140. DOI:10.1093/bib/bby073 |
| [2] |
郑思悦, 陈楚莹, 陈佳艺, 等. 乳腺癌的术前放疗: 差异化人群, 差异化目标[J]. 实用肿瘤杂志, 2021, 36(2): 99-104. |
| [3] |
Tsang JYS, Tse GM. Molecular classification of breast cancer[J]. Adv Anat Pathol, 2020, 27(1): 27-35. DOI:10.1097/PAP.0000000000000232 |
| [4] |
寇建锋, 刘永, 马鹏飞. PRICKLE1与ECT2在三阴性乳腺癌患者中表达的临床意义[J]. 实用肿瘤杂志, 2020, 35(6): 555-558. |
| [5] |
Toro C, Hori RT, Malicdan MCV, et al. A recurrent de novo missense mutation in UBTF causes developmental neuroregression[J]. Hum Mol Genet, 2018, 27(4): 691-705. DOI:10.1093/hmg/ddx435 |
| [6] |
Tchelidze P, Kaplan H, Terryn C, et al. Electron tomography reveals changes in spatial distribution of UBTF1 and UBTF2 isoforms within nucleolar components during rRNA synthesis inhibition[J]. J Struct Biol, 2019, 208(2): 191-204. DOI:10.1016/j.jsb.2019.08.014 |
| [7] |
Diesch J, Bywater MJ, Sanij E, et al. Changes in long-range rDNA-genomic interactions associate with altered RNA polymerase Ⅱ gene programs during malignant transformation[J]. Commun Biol, 2019, 2: 39. DOI:10.1038/s42003-019-0284-y |
| [8] |
Woolnough JL, Atwood BL, Liu Z, et al. The regulation of rRNA gene transcription during directed differentiation of human embryonic stem cells[J]. PLoS One, 2016, 11(6): e0157276. DOI:10.1371/journal.pone.0157276 |
| [9] |
Moss T, Mars JC, Tremblay MG, et al. The chromatin landscape of the ribosomal RNA genes in mouse and human[J]. Chromosome Res, 2019, 27(1/2): 31-40. |
| [10] |
Hamdane N, Tremblay MG, Dillinger S, et al. Disruption of the UBF gene induces aberrant somatic nucleolar bodies and disrupts embryo nucleolar precursor bodies[J]. Gene, 2017, 612: 5-11. DOI:10.1016/j.gene.2016.09.013 |
| [11] |
Hernández-Ortega K, Garcia-Esparcia P, Gil L, et al. Altered machinery of protein synthesis in Alzheimer's: from the nucleolus to the ribosome[J]. Brain Pathol, 2016, 26(5): 593-605. DOI:10.1111/bpa.12335 |
| [12] |
Mars JC, Sabourin-Felix M, Tremblay MG, et al. A deconvolution protocol for ChIP-seq reveals analogous enhancer structures on the mouse and human ribosomal RNA genes[J]. G3 (Bethesda), 2018, 8(1): 303-314. DOI:10.1534/g3.117.300225 |
| [13] |
Federico C, Gil L, Bruno F, et al. Phosphorylated nucleolar Tau protein is related to the neuronal in vitro differentiation[J]. Gene, 2018, 664: 1-11. DOI:10.1016/j.gene.2018.04.051 |
| [14] |
Xie ZC, Tang RX, Gao X, et al. A meta-analysis and bioinformatics exploration of the diagnostic value and molecular mechanism of miR-193a-5p in lung cancer[J]. Oncol Lett, 2018, 16(4): 4114-4128. |
| [15] |
Tsoi H, Lam KC, Dong Y, et al. Pre-45s rRNA promotes colon cancer and is associated with poor survival of CRC patients[J]. Oncogene, 2017, 36(44): 6109-6118. DOI:10.1038/onc.2017.86 |
| [16] |
Oh HR, An CH, Yoo NJ, et al. Frameshift mutations of TAF1C gene, a core component for transcription by RNA polymerase I, and its regional heterogeneity in gastric and colorectal cancers[J]. Pathology, 2015, 47(2): 101-104. DOI:10.1097/PAT.0000000000000212 |
| [17] |
Rajput P, Shukla SK, Kumar V. The HBx oncoprotein of hepatitis B virus potentiates cell transformation by inducing c-Myc-dependent expression of the RNA polymerase I transcription factor UBF[J]. Virol J, 2015, 12: 62. DOI:10.1186/s12985-015-0293-5 |
 2022, Vol. 37
2022, Vol. 37


