文章信息
- 裴珍珍, 王革生, 郑粲
- Pei Zhenzhen, Wang Gesheng, Zheng Can
- 头颈部鳞状细胞癌铁死亡相关lncRNA预后模型的建立与验证
- Establishment and validation of ferroptosis related lncRNA prognosis model in head and neck squamous cell carcinoma
- 实用肿瘤杂志, 2022, 37(6): 530-542
- Journal of Practical Oncology, 2022, 37(6): 530-542
基金项目
- 北京中医药大学基本科研业务重点攻关项目(2020-JYB-ZDGG-129)
-
通信作者
- 王革生,E-mail:wanggesh@sina.com
-
文章历史
- 收稿日期:2021-07-07
头颈部鳞状细胞癌(head and neck squamous cell carcinoma,HNSCC)是发生在头颈部区域鳞状上皮中的一种侵袭性恶性肿瘤,包括喉鳞状细胞癌(laryngeal squamous cell carcinoma,LSCC)、食管鳞状细胞癌(esophageal squamous cell carcinoma,ESCC)、鼻咽癌(nasopharyngeal carcinoma,NPC)、口腔鳞状细胞癌(oral squamous cell carcinoma,OSCC)和舌鳞状细胞癌(tongue squamous cell carcinomas,TSCC)[1-2]。全球癌症最新统计数据显示,头颈部恶性肿瘤发病率居全身恶性肿瘤发病的第6位[3],每年确诊HNSCC患者 > 55万例,因HNSCC死亡 > 38万例[4-5]。我国HNSCC发病率曾远低于欧美,但随着经济发展以及环境和生活方式的改变,全国HNSCC发病率和死亡率呈现上升趋势,每年发病高达6万多例[6]。HNSCC具有高增殖、区域淋巴结转移和预后差等特点。当前金标准治疗方案包括根治性手术切除、放疗、化疗和靶向治疗[7]。即使采用上述治疗方法在内的多模式治疗方案,仍有大约一半的患者会出现疾病复发或进展,患者5年生存率在过去的几十年里并没有显著提高[8]。迄今为止,由于缺乏强大的临床上有用的预测或预后性生物标志物,多数HNSCC患者确诊时已处于晚期,这使得肿瘤的远处转移和复发风险更高,预后严重不良[9-10]。
铁死亡是一种铁依赖性的脂质过氧化介导的程序性细胞死亡方式,不同于细胞凋亡、细胞坏死和自噬。许多种类的癌症对铁死亡敏感,因此这种机制是癌症治疗的一个有希望的因素。此外,诸多研究都观察到铁死亡和化疗耐药之间的微妙联系,铁死亡可以逆转化疗耐药或增强HNSCC细胞对化疗药物的敏感性[11-12]。在对HNSCC与铁死亡的研究中发现,治疗晚期、复发性或转移性HNSCC患者的主要障碍是癌细胞对化疗药物的内在或获得性耐药,尤其是顺铂。然而,已有研究指出,抑制胱氨酸/谷氨酸反向转运蛋白(system Xc-,xCT)在顺铂耐药的HNSCC中可以诱导铁死亡[12],逆转HNSCC细胞中的顺铂耐药性。
长链非编码RNA(long non-coding RNA,lncRNA)是一种无编码能力但长度 > 200个核苷酸的RNA。lncRNA参与许多生物过程,如细胞生长、抗凋亡、迁移和侵袭,在组蛋白修饰、染色质重塑、转录激活、转录干扰、核转运和细胞周期调节等过程中发挥重要的作用[13]。研究发现,lncRNA参与调节铁死亡。在肿瘤中,细胞质lncRNA P53RRA下调并与Ras-GTP酶激活蛋白SH3结构域结合蛋白1(Ras-GAP SH3 domain-binding protein, G3BP1)相互作用,将p53从G3BP1复合物中转移,导致p53滞留在细胞核中,导致细胞周期停滞、铁死亡和细胞凋亡[14]。LINC00618可以降低淋巴特异性解旋酶(lymphoid specific helicase,LSH)的表达,从而抑制铁死亡[15]。然而,目前关于铁死亡相关lncRNA与HNSCC关系的研究较少。铁死亡相关lncRNA是否与HNSCC患者的治疗或预后相关也仍不清楚。此外,诱导铁死亡有助于增强免疫疗法的抗肿瘤功效[16],且免疫检查点抑制剂在晚期HNSCC中的应用是HNSCC免疫治疗领域取得的突破性进展[17]。然而,目前针对铁死亡和免疫反应之间的综合调控关系,也鲜有研究。因此,本研究拟从癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库收集HNSCC患者的全部转录组数据和临床资料,并通过多种途径获得铁死亡相关基因、N6-腺苷酸甲基化(N6-methyladenosine,m6A)调节基因以及TCGA数据库中所有肿瘤的免疫细胞浸润评估资料,对所获资料进行编辑和整理后,借助R软件的分析程辑包,筛选与HNSCC预后相关的铁死亡lncRNA,构建预后模型,并进行一系列检测和验证,为HNSCC的早期诊断、治疗以及预后提供新的思路和参考。
1 资料与方法 1.1 资料获取 1.1.1 转录组数据与临床数据的获取2021年6月26日从TCGA数据库(https://portal.gdc.cancer.gov/)下载HNSCC患者肿瘤组织和癌旁组织的基因转录组高通量测序数据及临床资料。利用Perl脚本对下载的转录组数据和临床资料分别进行合并、id转换和信息提取,得到每个样本的基因表达矩阵和具体的临床信息。在此基础上,再次借助Perl进行转录组数据中lncRNA的区分与筛选。最终纳入121个样本数据,包括108例HNSCC患者组织标本数据,共注释18 729个lncRNA表达数据。临床数据包含527例HNSCC患者的年龄、性别、病理分期、生存时间和生存状态等临床信息,年龄19~90岁,(60.63±11.93)岁;男性385例,女性142例。
1.1.2 HNSCC的铁死亡相关基因的获取铁死亡相关基因从2个途径获得:(1)FerrDb数据库(http://www.zhounan.org/ferrdb)下载铁死亡相关基因(下载截止时间:2021年6月26日);(2)文献检索,截至2021年6月12日,以主题词“ferroptosis”和“铁死亡”分别在PubMed(https://pubmed.ncbi.nlm.nih.gov/)、中国知网(https://www.cnki.net/)和万方数据库(http://med.wanfangdata.com.cn/)检索与铁死亡相关的文献,获得与铁死亡相关的基因。将FerrDb数据库下载和文献阅读挖掘的铁死亡相关基因过滤去重,得到全部的铁死亡相关基因,以此为基础,使用R软件中的“Limma”包筛选出HNSCC组织和癌旁组织的铁死亡相关基因。利用相同的方法,获取m6A相关基因。
1.1.3 免疫细胞浸润数据获取2021年6月26日于肿瘤免疫数据库(http://timer.cistrome.org/)下载所有TCGA数据库中肿瘤免疫细胞浸润评估数据。
1.2 方法 1.2.1 HNSCC铁死亡基因与相关lncRNA的共表达分析使用R软件计算铁死亡基因和铁死亡相关lncRNA之间表达的相关性。若P < 0.001,两者之间存在共表达关系;相关系数cor > 0,两者为正调控关系,cor < 0,两者为负调控关系。借助Cytoscape软件(version: 3.8.2)将铁死亡基因与铁死亡基因相关的lncRNA之间的共表达关系构建可视化的共表达网络。
1.2.2 HNSCC的铁死亡基因差异分析应用R软件中的“Limma”包和edgeR算法对HNSCC的铁死亡基因数据集进行标准化处理和差异表达分析,若log2FC > 0,则该基因在肿瘤组是上调的,若log2FC < 0,则该基因在肿瘤组是下调的。同时,根据∣log2FC∣ > 1和错误发现率(false discovery rate,FDR) < 0.05作为标准进行过滤,找到显著的差异基因,然后利用STRING(https://www.string-db.org/)平台呈现显著差异基因相互作用关系。
1.2.3 HNSCC患者预后相关的铁死亡lncRNA分析通过上一步的研究确定HNSCC铁死亡相关的基因和铁死亡相关lncRNA,使用R软件中的“survival”包,以P < 0.05为标准筛选出与HNSCC预后相关的铁死亡lncRNA。其中,若HR > 1,该lncRNA为高风险的lncRNA;若HR < 1,该lncRNA为低风险的lncRNA。对预后相关的lncRNA进行可视化分析,绘制森林图。
1.2.4 差异表达基因的GO富集分析和KEGG富集分析利用R软件的“clusterprofiler”和“Limma”包对筛选出的显著差异基因进行GO和KEGG富集分析,以P < 0.05为差异具有统计学意义。借助“enrichplot”和“ggplot2”包进行结果可视化,绘制气泡图。
1.2.5 临床预后模型的建立与评价根据每个基因的标准化表达水平及其相应的回归系数计算每例患者的风险评分(risk score),风险评分=基因1表达量×相应的回归系数+…+基因n表达量×相应的回归系数。以风险评分的中位数为截断值, 将患者分为高风险组和低风险组。运用R语言的“survminer”包和Kaplan-Meier生存分析对高风险组和低风险组进行评价并绘制Kaplan-Meier生存曲线,评估高和低风险组样本的总生存率差异情况,以P < 0.05为差异具有统计学意义。同时使用“survivalROC”包绘制受试者操作特性(receiver operating characteristic,ROC)曲线,“timeROC”包对1年、3年及5年总生存率的ROC曲线下面积(area under the curve,AUC)值进行计算,评估预后模型的预测能力。本研究结合年龄、性别和病理分期等临床信息,采用多因素Cox回归对预后模型进行分析,以验证构建的模型的预测能力是否独立于其他临床因素,可以成为独立的预测因子。此外,通过使用R语言stdca软件包绘制决策曲线以评价模型在临床中的实际应用。
1.2.6 基因富集分析应用基因集富集分析(Gene Set Enrichment Analysis,GSEA)软件进行基因富集分析,排列检验的次数设置为1 000。以识别TCGA队列中高风险和低风险HNSCC患者之间的途径和相应的生物标志物的差异,定义富集分数(enrichment score,ES) > 0的通路或功能在高风险组是活跃的,ES < 0的通路在低风险组是活跃的。定义FDR < 0.05为显著富集的功能或通路,找不到显著富集结果,则可按照标准化P值(normal P value,NOMp-val) < 0.05进行过滤。
1.2.7 免疫相关性分析肿瘤免疫数据库(http://timer.cistrome.org/)下载所有TCGA数据库中的肿瘤免疫细胞浸润评估数据,为进一步了解高和低风险组之间总生存率的差异,表征预后特征的肿瘤免疫微环境,本研究比较高和低风险组之间的免疫评分、免疫细胞浸润情况、免疫功能和差异免疫检查点。
1.2.8 m6A差异分析选择m6A调节基因,借助“limma”包进行与转录组数据相关的m6A相关基因的差异分析,筛选出在高和低风险组有显著差异分析的m6A调节基因。
1.3 统计学分析基于R平台(Version:4.0.2)进行数据分析。lncRNA与铁死亡基因之间的相关性使用Pearson相关分析进行评估。使用LASSO回归降维处理,单因素和多因素Cox回归分析确定独立预后因子。通过ROC曲线评估预后模型的准确性。如无特殊说明,以P < 0.05为差异具有统计学意义,且所有P值均为双尾分布。
2 结果 2.1 HNSCC测序数据和临床资料从TCGA数据库中下载、整理及分析546例HNSCC患者的数据,其中包含108例HNSCC样本和13例癌旁正常组织样本;临床数据最终符合纳入标准的HNSCC患者527例,其中男性死亡患者151例,未死亡234例;女性死亡患者72例,未死亡70例。
2.2 HNSCC的铁死亡相关基因与lncRNA共表达与差异分析HNSCC铁死亡相关基因246个,铁死亡相关lncRNA 1 328个;185个铁死亡基因与1 328个lncRNA之间相互存在共表达关系,其中5 474对存在正调控关系,90对存在负调控关系(图 1A)。差异分析中,共发现差异表达基因1 850个,其中表达上调基因760个,表达下调基因1 090个;具有显著差异的铁死亡基因64个,47个基因在肿瘤组是上调的,17个是下调的;利用STRING平台呈现显著差异基因相互作用关系,取可信度0.900进行呈现(图 1B)。
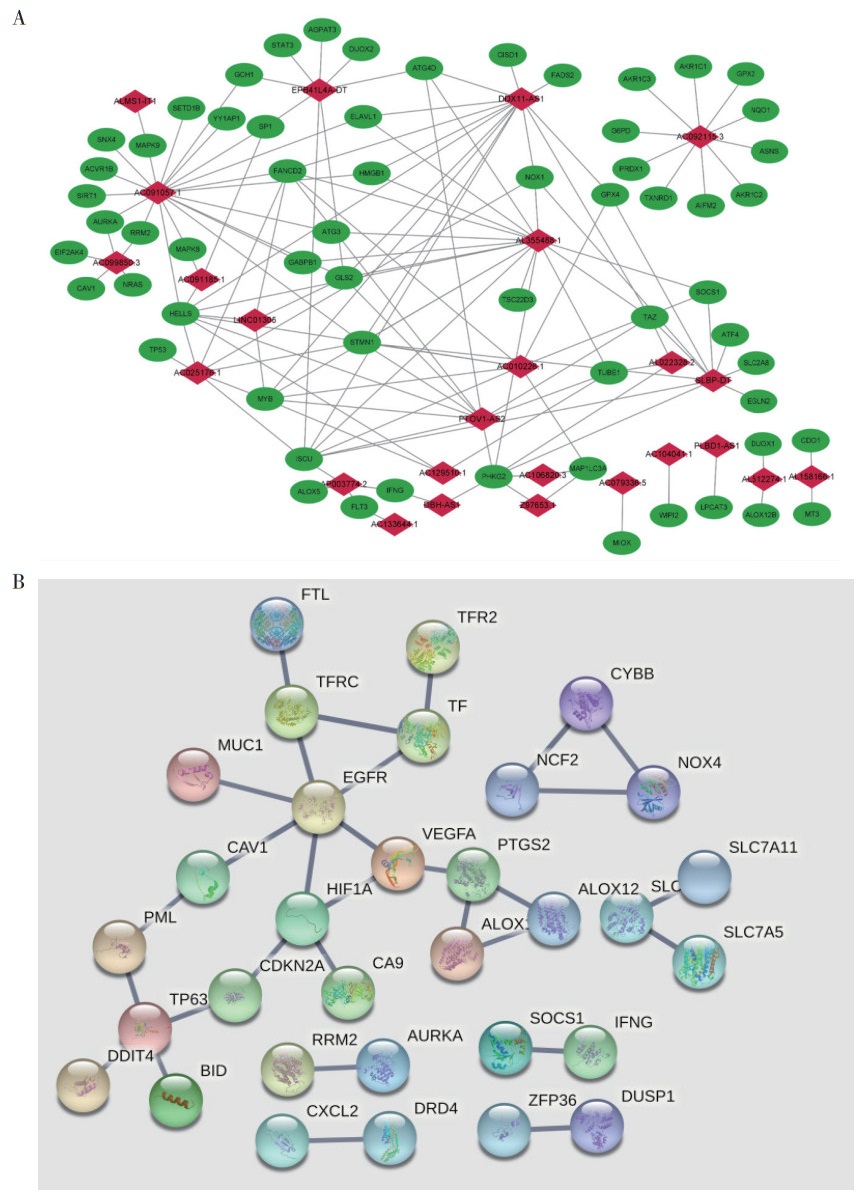
|
| 注 A:铁死亡基因与相关lncRNA的共表达网络图,红色表示铁死亡相关lncRNA,绿色表示铁死亡基因;B:具有显著差异的铁死亡相关基因的蛋白-蛋白相互作用(protein-protein interaction,PPI)关系图 图 1 HNSCC的铁死亡相关基因与lncRNA共表达与差异分析 Fig.1 Coexpression and differential analysis of ferroptosis related genes and lncRNAs in HNSCC |
通过GO富集分析和KEGG通路富集分析筛选的差异表达基因的生物学功能,GO富集分析中包括生物学过程(biological process,BP)、细胞组成(cell composition,CC)和分子功能(molec-ular function,MF),见图 2。在BP中差异基因主要富集于氧化应激反应、活性氧代谢和超氧化物代谢。在CC中差异基因主要富集于质膜、细胞基膜、顶质膜和氧化还原酶复合物。在MF中差异基因主要富集于有机阴离子跨膜转运蛋白活性、作用于烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate,NADPH)氧化还原酶活性和三价铁结合。KEGG分析表明,差异基因主要富集于癌症中微小RNA、铁死亡、坏死性凋亡、哺乳动物雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)和癌症中的中枢碳代谢等信号通路。
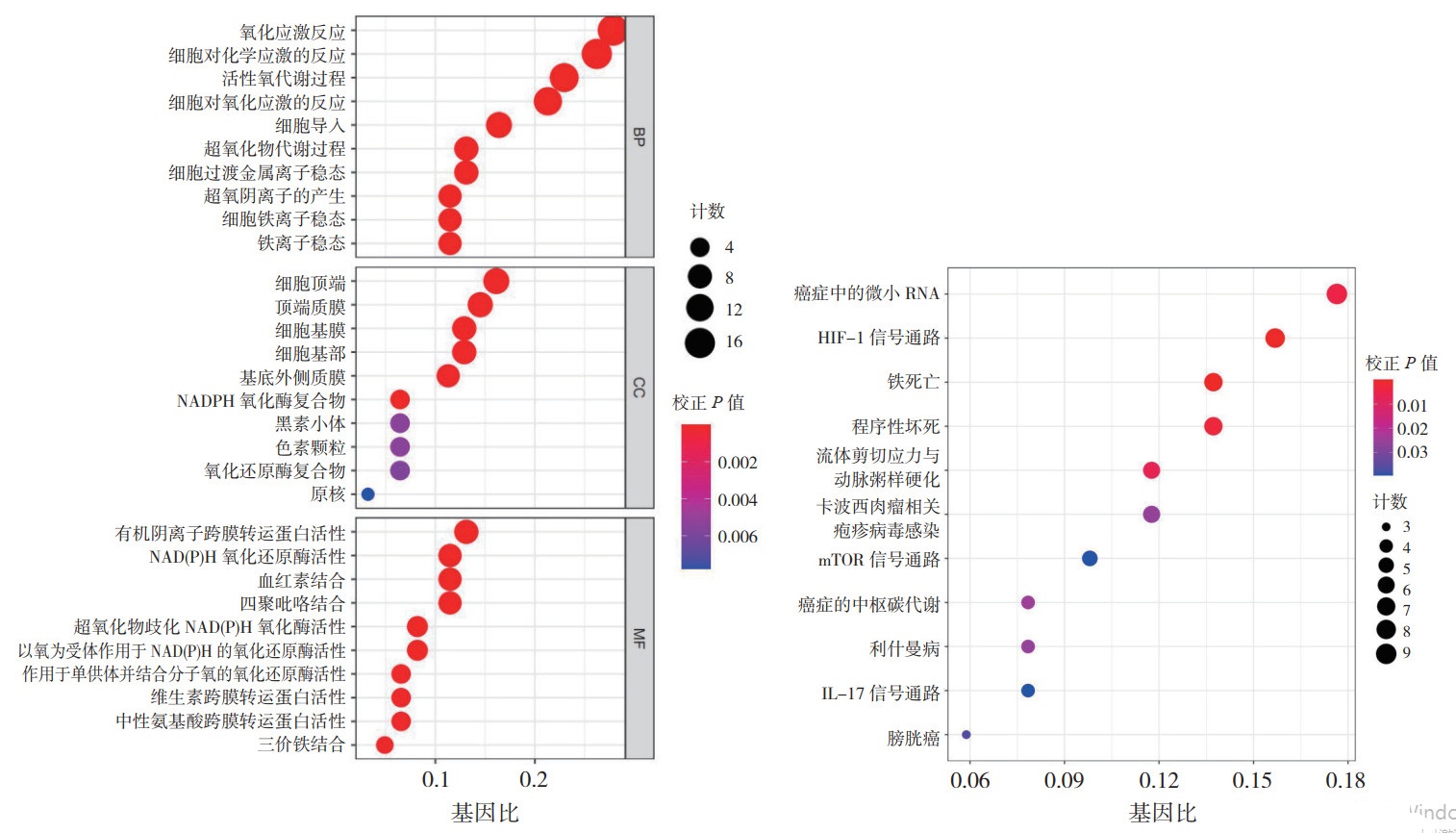
|
| 注 A:GO富集分析;B:KEGG通路富集分析;NADPH:烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate);HIF-1:低氧诱导因子-1(hypoxia-inducible factor-1);m-TOR:雷帕霉素靶蛋白(mammalian target of rapamycin);IL-17:白细胞介素17(interleukin-17) 图 2 HNSCC中铁死亡差异基因的GO富集分析和KEGG通路富集分析 Fig.2 GO enrichment analysis and KEGG pathway enrichment analysis of genes with significant difference in ferroptosis in HNSCC |
通过单因素Cox分析共筛选出72个与HNSCC预后相关的铁死亡相关lncRNA。在此基础上,利用LASSO回归降维处理和多因素Cox回归分析最终筛选得到11个与HNSCC患者预后相关的铁死亡lncRNA。对全部预后相关的铁死亡lncRNA进行可视化分析,绘制森林图(图 3)。
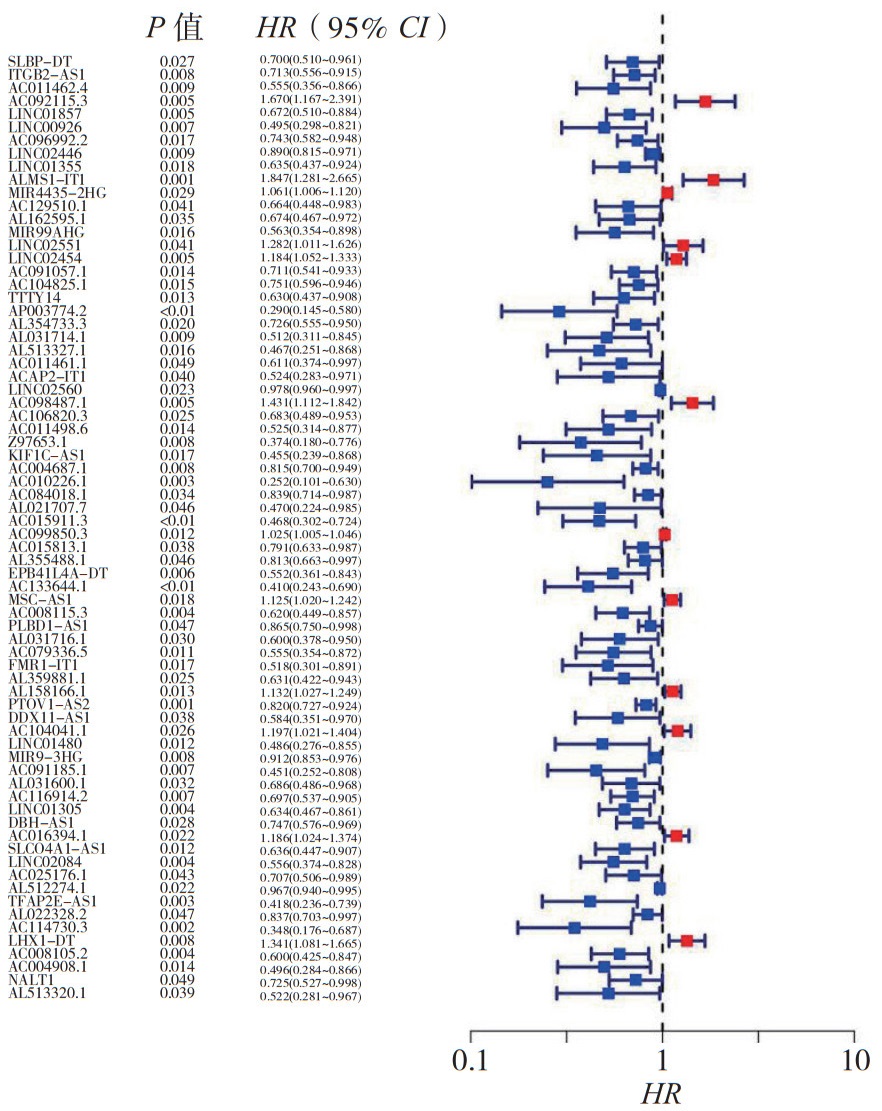
|
| 图 3 HNSCC预后相关的铁死亡lncRNA Fig.3 lncRNAs of ferroptosis associated with the prognosis of HNSCC |
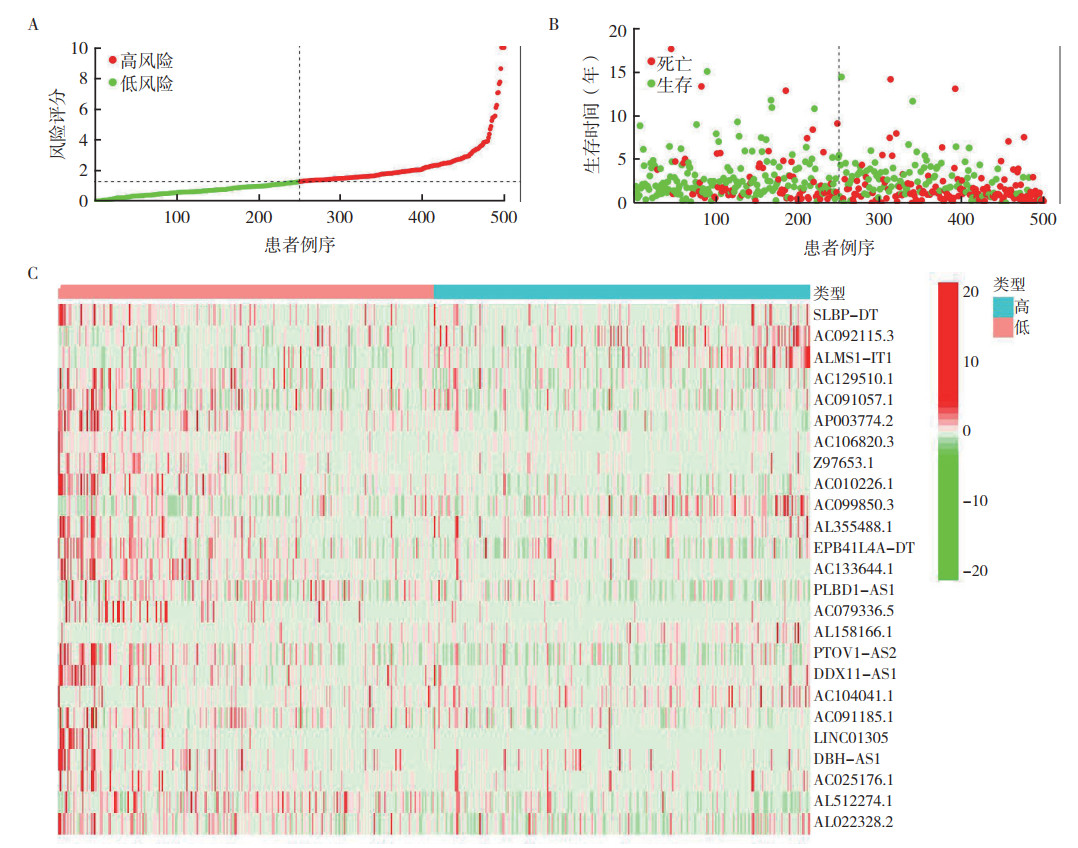
根据风险评分中位数,将TCGA队列中的食管癌患者分为高风险组和低风险组。Kaplan-Meier曲线分析显示,低风险组总生存时间长于高风险组(P < 0.01,图 4A),表明该模型具有良好的预测能力。同时,LncRNA预后模型对1、3和5年总生存率的AUC预测值分别为0.741、0.739和0.738(图 4B)。临床病理特征年龄、性别、肿瘤分级及分期预测总生存的ROC曲线的AUC分别为0.580、0.500、0.544和0.559(图 4C),优于其他一般临床性状。风险评分图、生存状态图和风险热图结果显示随着风险评分的增加,患者的死亡率逐步上升,低风险组的总生存期优于高风险组,高风险组和低风险组总生存比较,差异具有统计学意义(P < 0.05,图 5)。
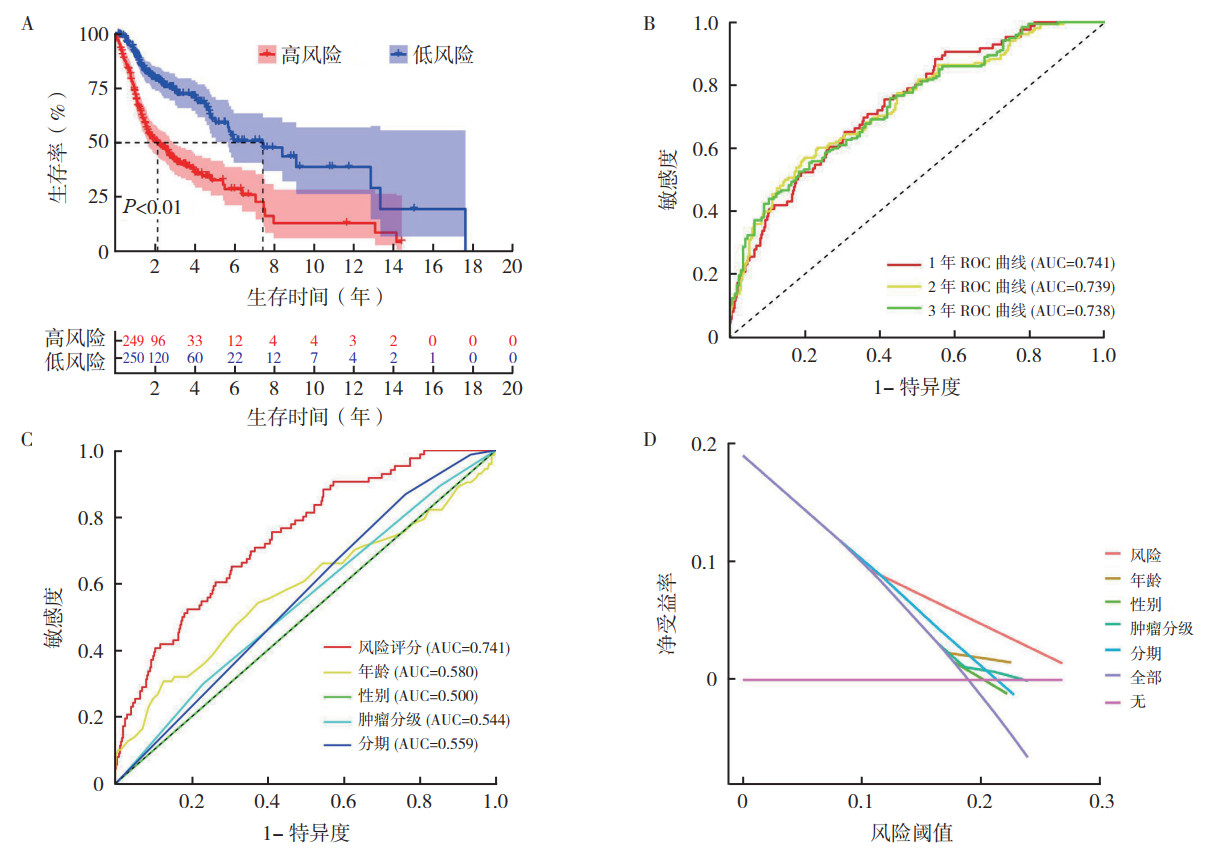
|
| 注 A:高风险组和低风险组HNSCC患者总生存曲线比较;B:预后模型的1、2和3年ROC曲线;C:各临床因素的ROC曲线;D:HNSCC患者预后风险因素的决策曲线分析 图 4 HNSCC中铁死亡相关基因预后模型的效能检验 Fig.4 Efficacy test of ferroptosis related gene prognostic model in HNSCC |
|
| 注 A:HNSCC患者中风险评分的分布情况;B:风险评分与生存时间的关系;黑色虚线是把患者分为高风险组和低风险组的最佳分界线;C:免疫核心基因与风险等级火山图 图 5 基于预后模型的HNSCC患者生存风险状态图 Fig.5 Survival risk status of HNSCC patients based on the prognostic model |
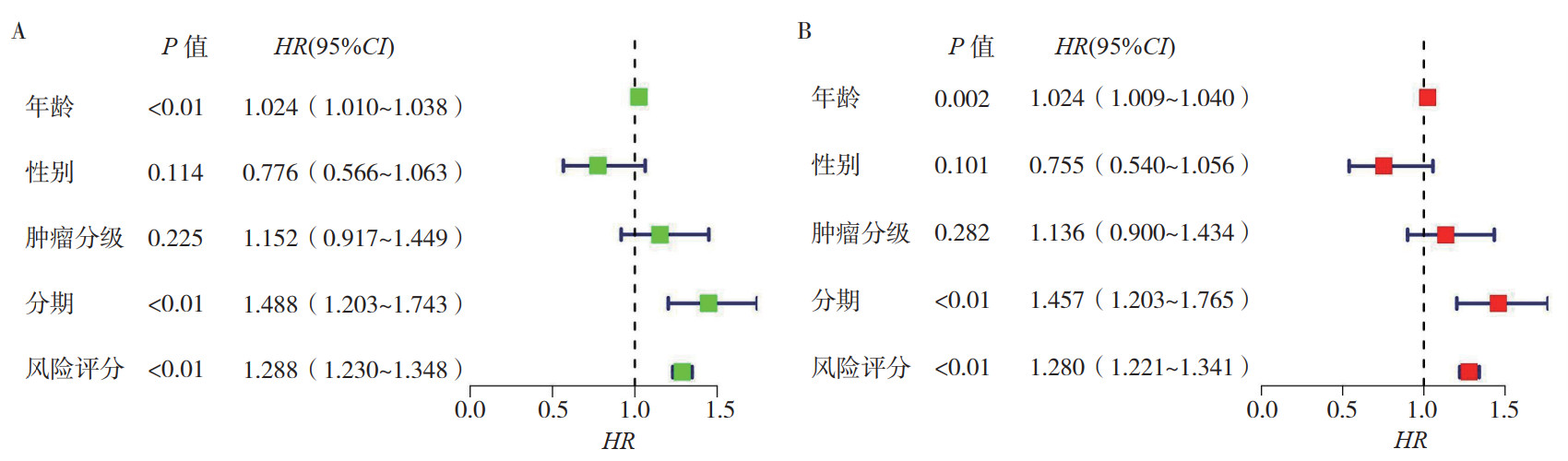
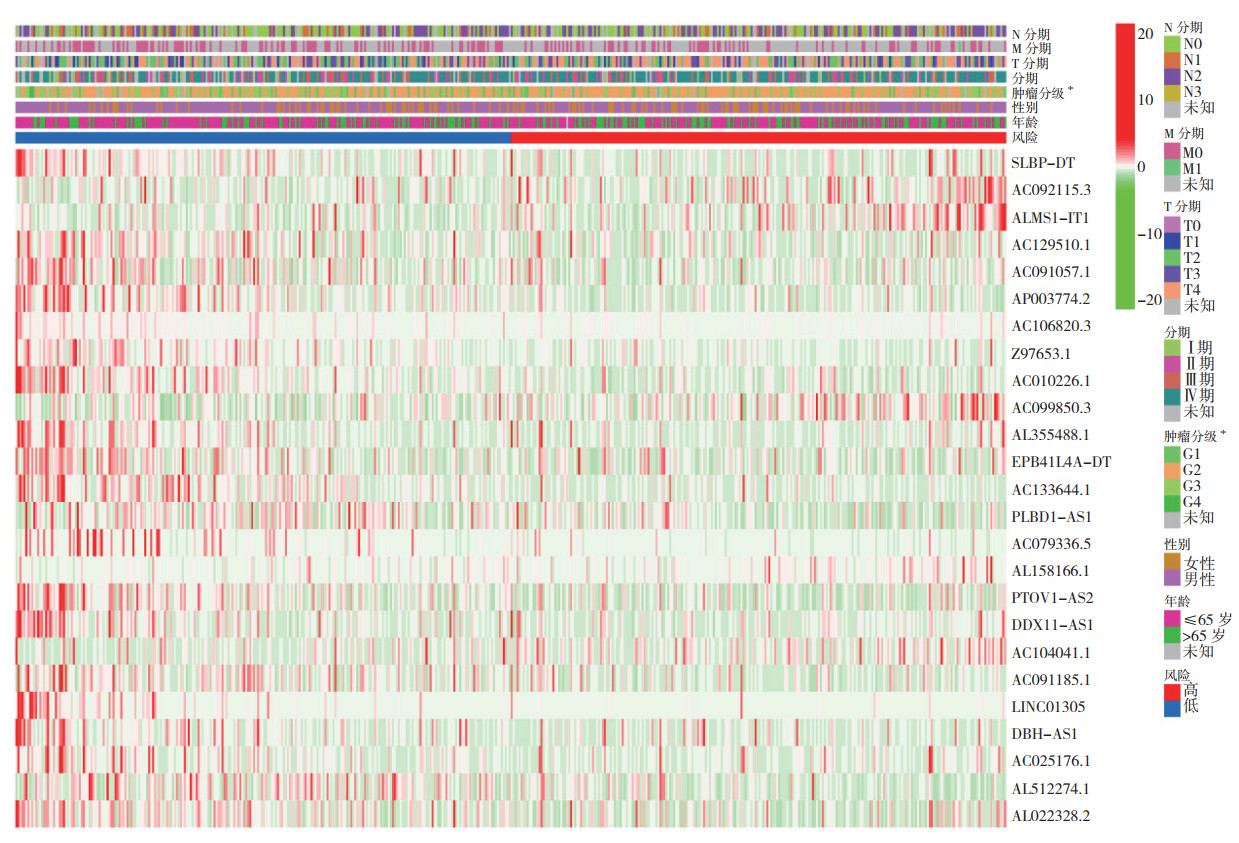
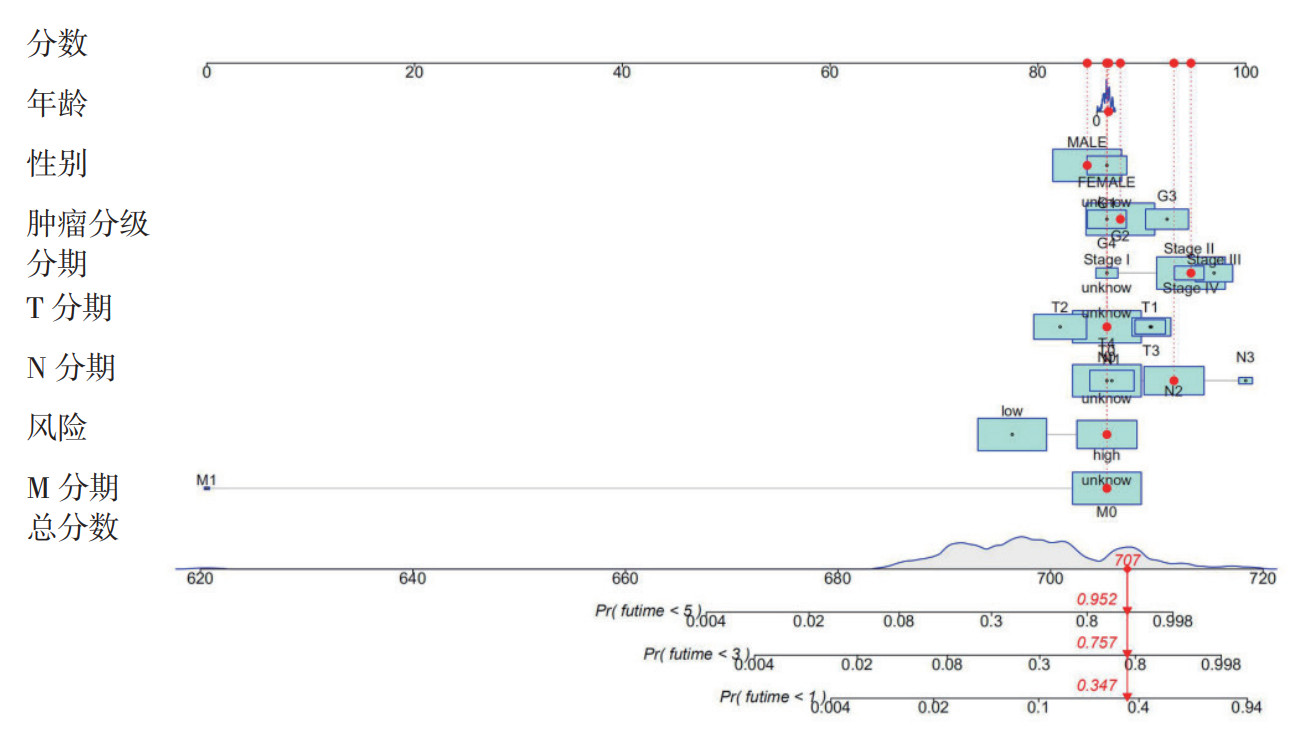
以生存时间为因变量,预后模型所计算的风险评分、年龄、性别和病理分期等临床指标作为协变量进行的单变量和多变量Cox分析显示,lncRNA特征(HR=1.280,95%CI:1.221~1.341,P < 0.01)、患者年龄(HR=1.024,95%CI:1.009~1.040,P=0.002)和肿瘤分期(HR=1.457,95%CI:1.203~1.765,P < 0.01)是HNSCC患者的独立预后因素(图 6)。铁死亡相关的lncRNA预后特征与临床病理表现之间关联的热图表明,多数新型lncRNA与风险模型呈负相关(P < 0.05,图 7)。结合临床病理特征和新型铁死亡相关lncRNA预后特征的混合列线图稳定准确(图 8),可用于HNSCC患者的临床观察。
|
| 注 A:单因素Cox回归风险评分与预后关系的森林图;B:多因素Cox回归风险评分与预后关系的森林图 图 6 单因素和多因素Cox回归分析风险评分与预后关系的森林图 Fig.6 Forest map of the relationship between risk score and prognosis by univariate and multivariate Cox regression analysis |
|
| 图 7 铁死亡相关lncRNA预后特征和临床病理表现的热图 Fig.7 Heat map of prognostic characteristics and clinicopathological manifestations of ferroptosis related lncRNAs |
|
| 注 futime:生存期 图 8 临床病理特征和铁死亡相关lncRNA预后特征的列线图 Fig.8 Nomogram of clinicopathological characteristics and prognostic features of ferroptosis related lncRNAs |
GSEA分析显示,多数新型铁死亡相关的lncRNA预后特征调节免疫和肿瘤相关通路,高风险组的通路主要与半乳糖代谢、黏着斑、蛋白酶体和类固醇生物合成等有关。低风险组原发性免疫缺陷、花生四烯酸代谢和B细胞受体信号通路等显著丰富(图 9)。
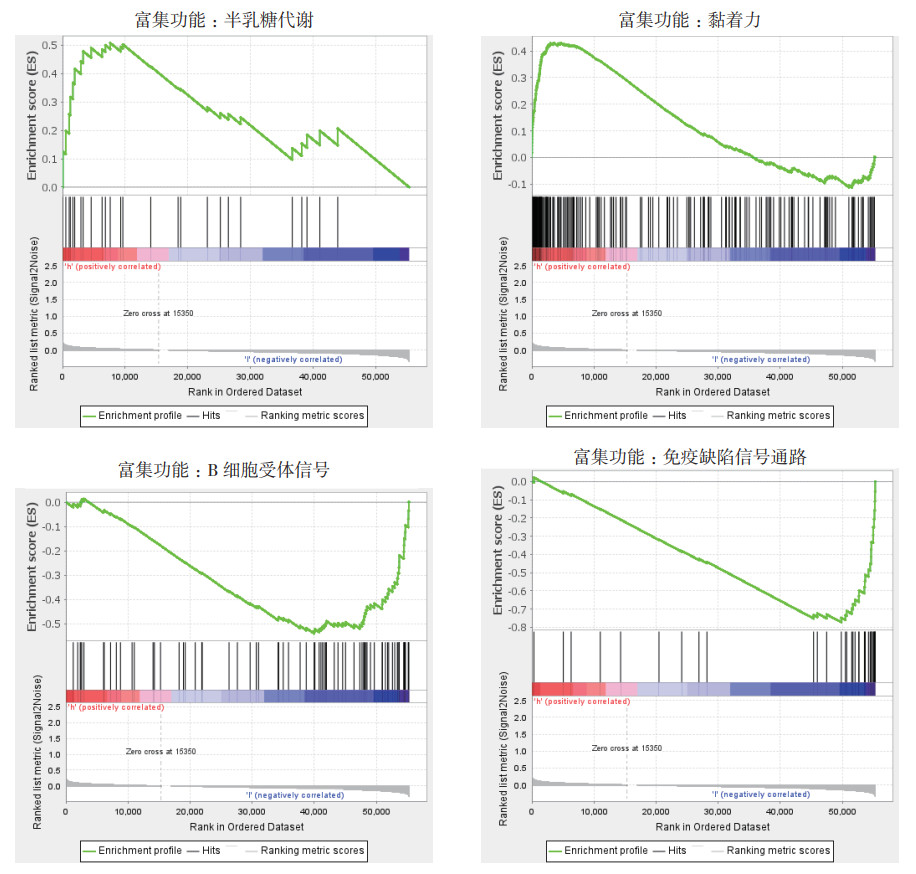
|
| 图 9 基于TCGA的铁死亡相关lncRNA的基因富集分析 Fig.9 Gene enrichment analysis of ferroptosis related lncRNAs based on TCGA |
基于TIMER、CIBERSORT、CIBERSORT-ABS、QUANTISEQ、MCPCOUNTER、XCELL和EPI平台的免疫反应热图显示,在所纳入的患者中发现B细胞、自然杀伤细胞(natural killer cell,NK细胞)、巨噬细胞M1、巨噬细胞M2和癌相关成纤维细胞等免疫细胞在高风险组和低风险组之间比较,差异均具有统计学意义(均P < 0.05,图 10A)。高风险组和低风险组在抗原呈递细胞的抑制与激活和Ⅰ型干扰素反应等方面比较,差异均无统计学意义(均P > 0.05);在T细胞共刺激(P < 0.01)、促炎(P < 0.01)和细胞溶解活性(P < 0.01)等免疫功能方面比较,差异均具有统计学意义(图 10B)。两组患者细胞毒性T淋巴细胞相关抗原4(cytotoxic T lymphocyte associated antigen-4,CTLA-4)、T细胞免疫球蛋白及其ITIM结构域(T-cell immunoreceptor with immunoglobulin and ITIM domains,TIGIT)、肿瘤坏死因子受体超家族(tumor necrosis factor receptor suberfamily 25,TNFRSF25)和细胞程序性死亡蛋白1(programmed cell death protein 1,PDCD1)等的表达情况比较,差异均具有统计学意义(均P < 0.01,图 10C)。
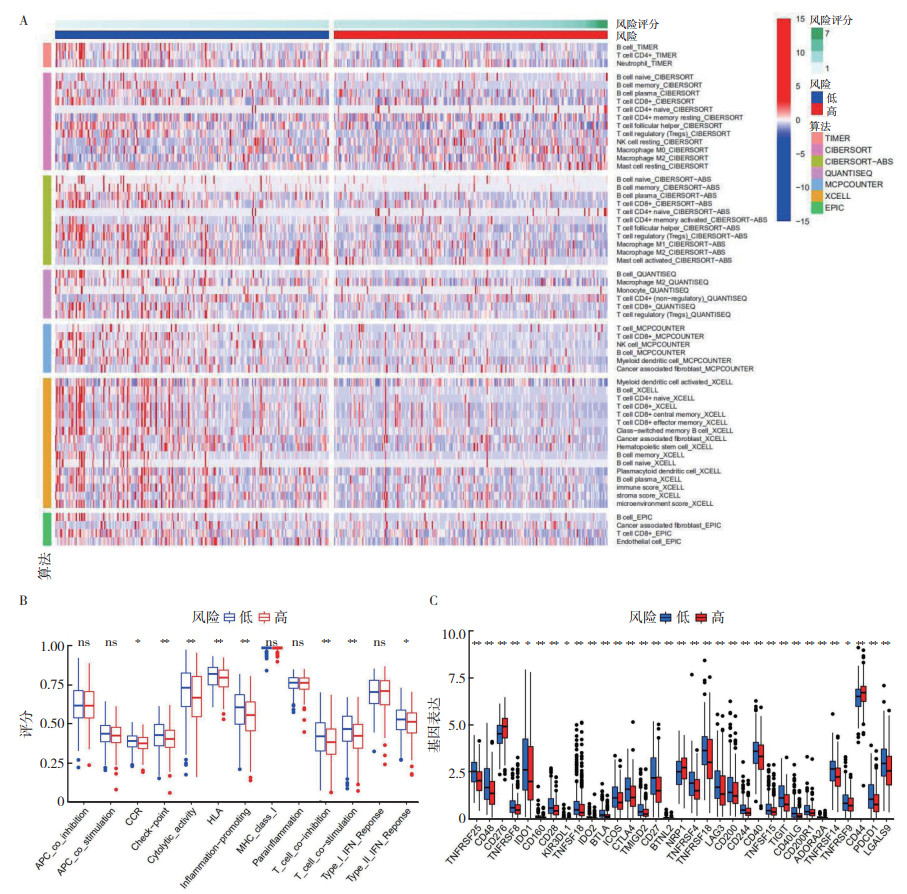
|
| 注 A: 基于TIMER、CIBERSORT、CIBERSORT-ABS、QUANTISEQ、MCPCOUNTER、XCELL和EPIC算法的高风险组和低风险组免疫反应热图;B: 高风险组和低风险组中免疫细胞亚群与相关功能之间的关系;C: 高风险组和低风险组免疫检查点的表达;CD4:分化群4(cluster of differentiation 4);APC:活化蛋白C (activated protein C);CCR:CC类趋化因子受体(C-C motif chemokine receptor);HLA:人类白细胞抗原(human leukocyte antigen);MHC:主要组织相容性复合体(major histocompatibility complex);IFN:干扰素(interferon);ns:差异无统计学意义(no significance,P > 0.05);*P < 0.05;**P < 0.01 图 10 高风险组和低风险组HNSCC患者的免疫反应及免疫细胞与免疫功能的关系 Fig.10 Immune responses of HNSCC patients in the high- and low-risk groups and the relationship between their immune cells and immune function |
高风险组和低风险组m6A相关mRNA表达量的比较显示,YTHDC2、YTHDC1、HNRNPC、RBM15和METTL14的表达在高风险组和低风险组间比较,差异均具有统计学意义(均P < 0.05,图 11)。
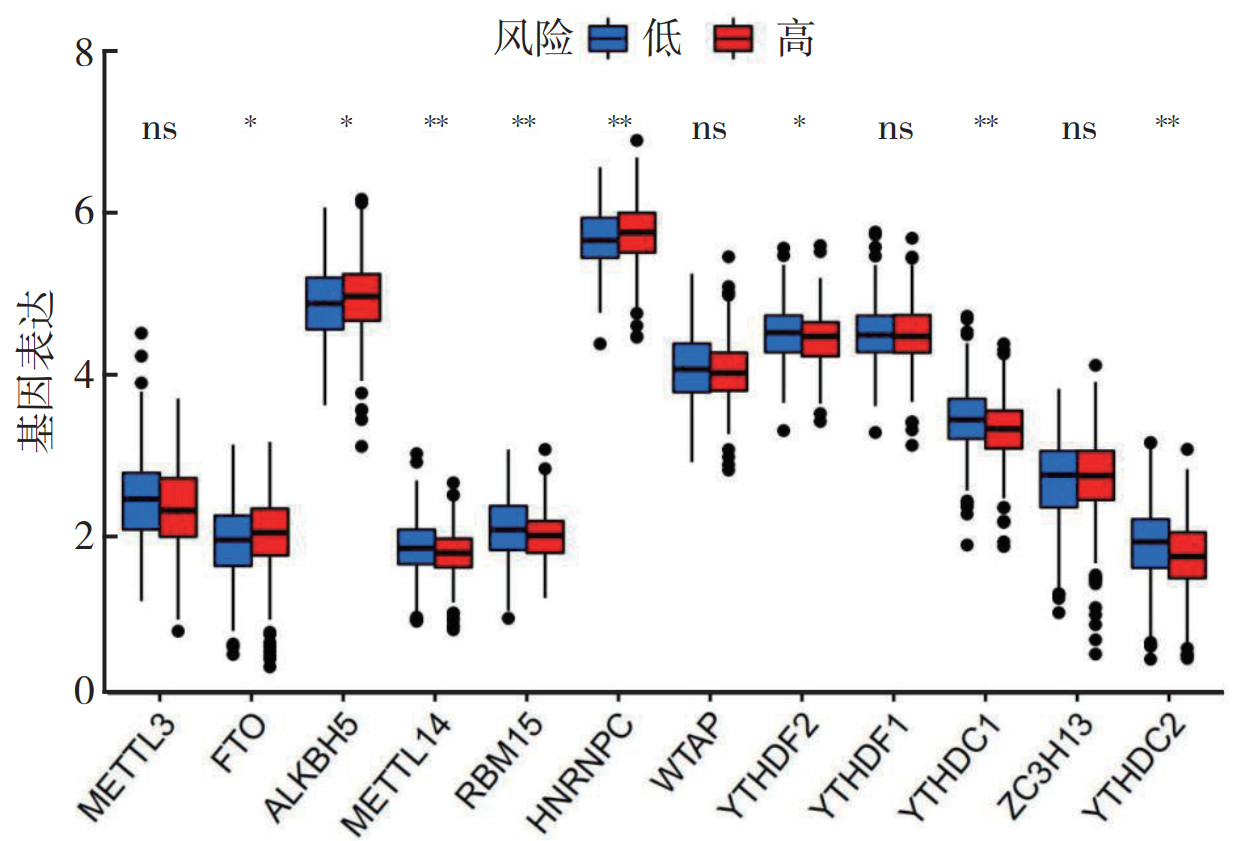
|
| 注 ns:差异无统计学意义(no significance,P > 0.05);*P < 0.05;**P < 0.01 图 11 HNSCC高风险组和低风险组m6A相关基因的表达 Fig.11 Expression of m6A related genes in the high-and low-risk groups of HNSCC |
HNSCC早期诊断非常困难,导致多数患者确诊时已为晚期,因此常伴随着高死亡率。而疾病分期在确定患者预后方面表现出强烈的预测效用。且在治疗方面,与晚期患者比较,早期HNSCC患者从化疗或手术中获益更多。因此,迫切需要寻找新的有效的诊断和预后生物标志物和潜在治疗靶点用于HNSCC的早期诊断、早期治疗和风险预测。目前,肿瘤分子生物学正在迅速发展,各种mRNA和基于microRNA(miRNA)的模型已被广泛报道为许多不同类型癌症的诊断、预后标志物和潜在治疗靶点[18]。其中,利用lncRNA作为诊断标志物、预后标志物和治疗靶点也已经出现在包括乳腺癌[19]、结肠癌[20]和膀胱癌[21]等不同类型的癌症中,然而,基于铁死亡相关的lncRNA对HNSCC患者的预后预测工具仍然缺乏。因此,本研究从铁死亡lncRNA着手,首先经筛选获得246个HNSCC的铁死亡相关基因和1 328个铁死亡相关的lncRNA,构建72个铁死亡相关的lncRNA模型来预测HNSCC患者的预后。经LASSO回归降维处理和多因素Cox分析发现,11个铁死亡相关lncRNA与HNSCC患者的预后有关,其中包括5个低风险lncRNA(AL512274.1、LINC01305、AC106820.3、AC129510.1和AC133644.1)和6个高风险lncRNA(ALMS1-IT1、AC025176.1、AL355488.1、DBH-AS1、AC104041.1和AC099850.3)。在探索影响HNSCC预后的研究中发现,lncRNA ALMS1-IT1具有最多的目标miRNA和蛋白质,提示ALMS1-IT1在HNSCC的预后中起重要作用[22]。ALMS1-IT1被认为是小细胞肺癌的关键基因[23],在ALMS1-IT1与肺腺癌(lung adenocarcinoma,LUAD)恶性进展关系的体外实验中发现,缺乏ALMS1-IT1的LUAD细胞的活力和集落数降低,而在与ALMS1-IT1和AVL9共转染的细胞中,增殖能力增强,集落数量增加,表明ALMS1-IT1的水平与LUAD细胞的恶性转化有关,并导致LUAD患者预后不良,可能是LUAD治疗的新生物标志物[24]。在探索调控DBH-AS1/BUD13/FN1轴对弥漫性大B细胞淋巴瘤(diffuse large B-cell lymphoma,DLBCL)进程影响的研究中,DBH-AS1的过表达促进细胞增殖,增强细胞迁移和侵袭能力,而沉默BUD13和FN1抵消DBH-AS1过表达对细胞增殖、迁移和侵袭的刺激影响。这表明DBH-AS1可通过调节BUD13和FN1介导DLBCL增殖、迁移和侵袭,加速DLBCL进展[25]。此外,lncRNA DBH-AS1促进肿瘤细胞增殖和肿瘤发展的作用在肝癌[26]和骨肉瘤[27]等的研究中均得到证明。自噬是一种维持细胞内环境稳定性和细胞内蛋白质代谢平衡的机制,可通过清除正常细胞中的异常细胞器和蛋白质,防止应激反应的产生,保护基因不受损伤,从而抑制各类肿瘤的发生。真核翻译启动因子2α激酶4(eukaryotic translation initiation factor 2 alpha kinase 4,EIF2AK4)属于蛋白激酶家族,可响应各种应激刺激而磷酸化真核起始因子2(eukaryotic initiation factor 2,EIF2)的亚基。为了响应细胞应激激活自噬相关基因表达,激活EIF2AK4-EIF2A激活转录因子4信号通路是必不可少的[28]。有研究显示,lncRNA AC099850.3与EIF2AK4表现出最大的正向共表达相关性[29]。AC099850.3可促进细胞周期分子苯并咪唑出芽抑制解除同源物蛋白1(budding uninhibited by benzimidazoles 1,BUB1)、周期蛋白依赖性激酶1(cyclin-dependent kinase 1,CDK1)、Polo样激酶1(Polo-like kinase 1, PLK1)和T细胞酪氨酸激酶(T cell tyrosine kinase,TTK)的表达,在体外可促进肝癌细胞的迁移和增殖[30]。AC099850.3高表达也与舌癌患者的生存率降低密切相关[31]。
本研究着重讨论与HNSCC预后相关的部分lncRNA的功能,除AC099850.3和ALMS1-IT1外,其他lncRNA在先前关于HNSCC的研究中尚未被报道,这意味着这些lncRNA可能是治疗HNSCC的新的潜在靶点,在HNSCC中的生物学作用将是未来研究的重点。
此外,本研究构建的风险预后模型具有良好的预测能力和稳定性,可以作为HNSCC的独立预测因子,并且优于其他临床因素。所有患者的风险评分与生存时间的关系提示高风险组的患者可能更早死亡。单因素和多因素Cox分析验证了该模型的预测能力,充分表明72个与铁死亡相关的lncRNA可能是HNSCC的良好预后因素。
近年来,诸多研究表明,铁死亡在清除癌细胞和克服治疗耐药性方面发挥了至关重要的作用[32]。另一方面,lncRNA的生物学功能也逐渐被阐明。一些lncRNA通过不同的生物学途径影响癌症进展和治疗效果[33]。然而,在铁死亡和lncRNA之间仍有许多未被探索的领域。本研究基于高通量测序数据确定72个与铁死亡相关的lncRNA。本研究仍存在一定的局限性,缺乏大型临床样本的验证,因此,为了进一步证实该模型的预测效果,需要进一步进行包含临床、放射学和组织病理学因素的多中心、大样本、纵向研究,以证明本研究的可靠性。此外,本研究所涉及的多个lncRNA的功能和机制值得进一步的体外和体内研究,以期提供其作为治疗HNSCC的新候选靶点的潜力。
| [1] |
Zhao JY, Liu DM, Yang H, et al. Long noncoding RNAs in head and neck squamous cell carcinoma: biological functions and mechanisms[J]. Mol Biol Rep, 2020, 47(10): 8075-8090. DOI:10.1007/s11033-020-05777-w |
| [2] |
叶显宗, 齐兴峰, 江碧兰, 等. 口腔梭形细胞鳞状细胞癌二例临床病理分析[J]. 实用肿瘤杂志, 2021, 36(4): 352-354. DOI:10.13267/j.cnki.syzlzz.2021.072 |
| [3] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [4] |
Simard EP, Torre LA, Jemal A. International trends in head and neck cancer incidence rates: differences by country, sex and anatomic site[J]. Oral Oncol, 2014, 50(5): 387-403. DOI:10.1016/j.oraloncology.2014.01.016 |
| [5] |
Global Burden of Disease Cancer Collaboration, Fitzmaurice C, Allen C, et al. Global, regional, and national cancer incidence, mortality, years of life lost, years lived with disability, and disability-adjusted life-years for 32 cancer groups, 1990 to 2015: a systematic analysis for the global burden of disease study[J]. JAMA Oncol, 2017, 3(4): 524-548. DOI:10.1001/jamaoncol.2016.5688 |
| [6] |
吴春晓, 鲍萍萍, 黄哲宙, 等. 2003-2012年上海市头颈部恶性肿瘤发病特征分析[J]. 中国癌症杂志, 2017, 27(6): 406-414. |
| [7] |
Lo Nigro C, Denaro N, Merlotti A, et al. Head and neck cancer: improving outcomes with a multidisciplinary approach[J]. Cancer Manag Res, 2017, 9: 363-371. DOI:10.2147/CMAR.S115761 |
| [8] |
Kumpitsch C, Moissl-Eichinger C, Pock J, et al. Preliminary insights into the impact of primary radiochemotherapy on the salivary microbiome in head and neck squamous cell carcinoma[J]. Sci Rep, 2020, 10(1): 16582. DOI:10.1038/s41598-020-73515-0 |
| [9] |
Yanamoto S, Yamada S, Takahashi H, et al. Clinicopathological risk factors for local recurrence in oral squamous cell carcinoma[J]. Int J Oral Maxillofac Surg, 2012, 41(10): 1195-1200. DOI:10.1016/j.ijom.2012.07.011 |
| [10] |
Lo Muzio L, Sartini D, Santarelli A, et al. Expression and prognostic significance of apoptotic genes in oral squamous cell carcinoma[J]. Mol Carcinog, 2014, 53(4): 264-271. DOI:10.1002/mc.21960 |
| [11] |
Ye J, Jiang XH, Dong ZH, et al. Low-concentration PTX and RSL3 inhibits tumor cell growth synergistically by inducing ferroptosis in mutant p53 hypopharyngeal squamous carcinoma[J]. Cancer Manag Res, 2019, 11: 9783-9792. DOI:10.2147/CMAR.S217944 |
| [12] |
Roh JL, Kim EH, Jang HJ, et al. Induction of ferroptotic cell death for overcoming cisplatin resistance of head and neck cancer[J]. Cancer Lett, 2016, 381(1): 96-103. DOI:10.1016/j.canlet.2016.07.035 |
| [13] |
Cao PB, Jin Q, Feng L, et al. Emerging roles and potential clinical applications of noncoding RNAs in hepatocellular carcinoma[J]. Semin Cancer Biol, 2021, 75: 136-152. DOI:10.1016/j.semcancer.2020.09.003 |
| [14] |
Mao C, Wang X, Liu YT, et al. A G3BP1-interacting lncRNA promotes ferroptosis and apoptosis in cancer via nuclear sequestration of p53[J]. Cancer Res, 2018, 78(13): 3484-3496. DOI:10.1158/0008-5472.CAN-17-3454 |
| [15] |
Wang ZL, Chen XW, Liu N, et al. A nuclear long non-coding RNA LINC00618 accelerates ferroptosis in a manner dependent upon apoptosis[J]. Mol Ther, 2021, 29(1): 263-274. DOI:10.1016/j.ymthe.2020.09.024 |
| [16] |
Friedmann Angeli JP, Krysko DV, Conrad M. Ferroptosis at the crossroads of cancer-acquired drug resistance and immune evasion[J]. Nat Rev Cancer, 2019, 19(7): 405-414. DOI:10.1038/s41568-019-0149-1 |
| [17] |
王佳峰, 葛明华. 头颈部黏膜鳞状细胞癌全球诊治新进展[J]. 实用肿瘤杂志, 2020, 35(1): 16-18. |
| [18] |
Chan JJ, Tay Y. Noncoding RNA: RNA regulatory networks in cancer[J]. Int J Mol Sci, 2018, 19(5): E1310. DOI:10.3390/ijms19051310 |
| [19] |
张莉, 张开炯, 吴立春, 等. 乳腺癌相关长链非编码RNA的生物信息学分析[J]. 肿瘤预防与治疗, 2018, 31(5): 305-312. |
| [20] |
Cai HJ, Zhuang ZC, Wu Y, et al. Development and validation of a ferroptosis-related lncRNAs prognosis signature in colon cancer[J]. Bosn J Basic Med Sci, 2021, 21(5): 569-576. |
| [21] |
Liu JC, Ma H, Meng LF, et al. Construction and external validation of a ferroptosis-related gene signature of predictive value for the overall survival in bladder cancer[J]. Front Mol Biosci, 2021, 8: 675651. DOI:10.3389/fmolb.2021.675651 |
| [22] |
Xing L, Zhang XQ, Chen AW. Prognostic 4-lncRNA-based risk model predicts survival time of patients with head and neck squamous cell carcinoma[J]. Oncol Lett, 2019, 18(3): 3304-3316. |
| [23] |
Lei YM, Shi YF, Duan J, et al. Identification of alternative splicing and lncRNA genes in pathogenesis of small cell lung cancer based on their RNA sequencing[J]. Adv Clin Exp Med, 2019, 28(8): 1043-1050. DOI:10.17219/acem/94392 |
| [24] |
Luan T, Zhang TY, Lv ZH, et al. The lncRNA ALMS1-IT1 may promote malignant progression of lung adenocarcinoma via AVL9-mediated activation of the cyclin-dependent kinase pathway[J]. FEBS Open Bio, 2021, 11(5): 1504-1515. DOI:10.1002/2211-5463.13140 |
| [25] |
Song YP, Gao F, Peng Y, et al. Long non-coding RNA DBH-AS1 promotes cancer progression in diffuse large B-cell lymphoma by targeting FN1 via RNA-binding protein BUD13[J]. Cell Biol Int, 2020, 44(6): 1331-1340. DOI:10.1002/cbin.11327 |
| [26] |
Bao J, Chen XQ, Hou YG, et al. LncRNA DBH-AS1 facilitates the tumorigenesis of hepatocellular carcinoma by targeting miR-138 via FAK/Src/ERK pathway[J]. Biomed Pharmacother, 2018, 107: 824-833. DOI:10.1016/j.biopha.2018.08.079 |
| [27] |
Huang JL, Ren TY, Cao SW, et al. HBx-related long non-coding RNA DBH-AS1 promotes cell proliferation and survival by activating MAPK signaling in hepatocellular carcinoma[J]. Oncotarget, 2015, 6(32): 33791-33804. DOI:10.18632/oncotarget.5667 |
| [28] |
B'chir W, Maurin AC, Carraro V, et al. The eIF2α/ATF4 pathway is essential for stress-induced autophagy gene expression[J]. Nucleic Acids Res, 2013, 41(16): 7683-7699. DOI:10.1093/nar/gkt563 |
| [29] |
Jiang QK, Xue DF, Shi FZ, et al. Prognostic significance of an autophagy-related long non-coding RNA signature in patients with oral and oropharyngeal squamous cell carcinoma[J]. Oncol Lett, 2021, 21(1): 29. |
| [30] |
Wu FH, Wei HZ, Liu GY, et al. Bioinformatics profiling of five immune-related lncRNAs for a prognostic model of hepatocellular carcinoma[J]. Front Oncol, 2021, 11: 667904. DOI:10.3389/fonc.2021.667904 |
| [31] |
Zhou RS, Zhang EX, Sun QF, et al. Integrated analysis of lncRNA-miRNA-mRNA ceRNA network in squamous cell carcinoma of tongue[J]. BMC Cancer, 2019, 19(1): 779. DOI:10.1186/s12885-019-5983-8 |
| [32] |
Woo Y, Lee HJ, Jung YM, et al. Regulated necrotic cell death in alternative tumor therapeutic strategies[J]. Cells, 2020, 9(12): E2709. DOI:10.3390/cells9122709 |
| [33] |
Zhang XY, Xie K, Zhou HH, et al. Role of non-coding RNAs and RNA modifiers in cancer therapy resistance[J]. Mol Cancer, 2020, 19(1): 47. |
 2022, Vol. 37
2022, Vol. 37


