文章信息
- 常梦悦, 曹喜兵, 赵振利, 范国强
- CHANG Mengyue, CAO Xibing, ZHAO Zhenli, FAN Guoqiang
- 泡桐WPR基因家族鉴定及其对丛枝植原体的响应
- Identification of the WPR gene family in Paulownia and its response to witches' broom phytoplasma
- 森林与环境学报,2022, 42(3): 312-319.
- Journal of Forest and Environment,2022, 42(3): 312-319.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2022.03.011
-
文章历史
- 收稿日期: 2021-12-21
- 修回日期: 2022-03-21
叶绿体运动相关蛋白(WEB1/PMI2-related,WPR) 具有调控叶绿体光重定位运动及叶片颜色的作用[1-2]。2005年,DEBLASIO et al[3]初次在拟南芥(Arabidopsis thaliana L.) 中发现WPR家族成员PMI1。WPR家族包含WEB1、PMI2和PMI15等成员[4],且多数WPR家族成员都包含WEMBL保守结构域[5]。拟南芥有14个WPR家族成员,根据过表达植株中叶绿体的定位情况可分为WEB1、PMI2、WPRa和WPRb四个亚家族[6]。拟南芥中PMI2和WEB1蛋白通过互作,调节叶绿体运动,可能导致叶片黄化[7-9]。目前,未见其他植物WPR基因家族的研究报道。泡桐[Paulownia fortunei (Seem.) Hemsl.] 为玄参科(Scrophulariaceae) 泡桐属(Paulownia) 植物,具有重要的经济和生态价值[10],其生长过程易受植原体感染,造成腋芽丛生、叶片黄化,严重影响泡桐产业发展[11-13]。植原体侵染后WPR表达量显著差异[14-15],但WPR家族与泡桐丛枝病引起的叶片黄化之间的关系尚未见报道。因此,本研究基于泡桐基因组测序结果[16],对WPR家族成员进行鉴定,并从进化树、基因结构和理化性质等方面进行分析,筛选出与泡桐丛枝病叶片黄化相关的WPR基因,该结果可为探究泡桐丛枝病叶片黄化原因提供参考。
1 材料与方法 1.1 试验材料以河南农业大学林木生物技术实验室同一无性系培养的白花泡桐健康组培苗(PF) 和植原体感染后的泡桐组培苗(PFI) 为试验材料,培养条件为:温度(25±2) ℃,光周期16 h/8 h (光/暗),光照强度130 μmol·m-2·s-1,培养30 d[16],取1.5 cm顶芽保存于-80 ℃冰箱中备用。
1.2 WPR成员鉴定及染色体定位根据WEMBL保守结构域的pfam号PF05701,从pfam数据库(http://pfam.xfam.org/) 中下载WPR家族的蛋白序列作为种子文件,利用HMMER软件对泡桐基因组进行分析(E-value < 0.1) [17],得到泡桐WPR家族的蛋白序列。从PLAZA (https://bioinformatics.psb.ugent.be/plaza/) 数据库中下载葡萄(Vitis vinifera L.)、毛果杨(Populus trichocarpa Torr.)、金丝小枣(Thread jujube Mill.)、橡胶树[Hevea brasiliensis (Willd. ex A. Juss) Muell. Arg]、拟南芥(Arabidopsis thaliana L.)、玉米(Zea mays L.) 及水稻(Oryza sativ L.) WPR家族成员的蛋白序列,利用ClustalW 2.0进行多序列比对,MEGA7构建系统发育树[18]。通过基因组Chr. change. GFF文件,获得基因所在的染色体信息,并利用Chr. fast文件找到基因长度,利用Mapchart (https://www.wur.nl/en/show/Mapchart.htm) [19]软件绘制WPR基因在白花泡桐染色体上的位置分布图,并依据染色体位置对亚家族成员命名。
1.3 WPR基因家族的共线性及选择压分析对拟南芥和白花泡桐WPR家族进行序列比对,整理基因对的染色体位置、长度、起止位点等信息,借助在线软件MCScanX (http://chibba.pgml.uga.edu/mcscan2/#tm) 得到共线性文件,借助circle_ plotter工具,将结果可视化。利用软件KaKs_ Calculator计算基因对间的同义替换(synonymous substitution, Ks)、非同义替换(non-synonymous substitution, Ka) 及选择压(Ka/Ks) [20]。
1.4 WPR家族理化性质及基序分析对基因组CDS文件搜索,获得WPR家族成员的CDS序列文件,利用全基因组GFF文件,借助activestate (https://www.activestate.com/) 软件得到基因全长文件,将全长文件和CDS文件放入软件GSDS (http://gsds.gao-lab.org/Gsds) 中得到基因结构图[19]。借助TBtools软件在GFF文件中选取上游2 000 bp作为启动子序列,利用软件Plant CARE (http://bioinformatics.psb.ugent.be/webtools/plantcARE/html/) 分析顺时调控元件[20]。利用软件ProtParam (https://web.expasy.org/protparam/)、ProtCompB (http://www.softberry.com/cgi-bin/programs/proloc/protcomppl.pl)、SOPMA (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsasopma.html) 和MEME (http://meme-suite.org/tools/meme),对泡桐WPR家族进行理化性质、亚细胞定位、二级结构和基序分析[19-20]。
1.5 植原体侵染后WPR基因家族转录组分析及验证基于美国国家生物信息中心(National Center for Biotechnology Information, NCBI, https://www.blast.ncbi.nlm.nih.gov/) 通过登录号SRR11787905、SRR11787916、SRR11787927、SRR11787938、SRR11787883和SRR11787894,查找WPR家族成员在PF和PFI中的表达量,利用pheatmap的R包绘制热图,预测WPR家族成员在泡桐丛枝病中的作用[21]。
为验证转录组数据是否可用,随机挑选5个差异基因进行实时荧光定量聚合酶链式反应(real time quantitative polymerase chain reaction, RT-qPCR) 验证,采用植物总RNA抽提试剂盒提取PF、PFI样品的总RNA。利用ReverTra Ace qPCR RT Master Mix with gDNA Remover试剂进行mRNA反转录合成cDNA。利用iQTMSYBRⒸGreen Supermix试剂进行RT-qPCR检测[22],每个样品重复3次。用Actin基因作为内参,数据统计采用2-△△Ct法,用SPSS 25软件进行数据分析,引物序列见表 1。
| 基因 Gene |
正向引物 Forward primer(5′-3′) |
反向引物 Reverse primer(5′-3′) |
| Actin | AATGGAATCTGCTGGAAT | ACTGAGGACAATGTTACC |
| PfWEB2 | CCTGATATTCCTATGATTCTC | TGTTCGTTCTATCTCTGT |
| PfWEB3 | AGATGAGGATGAATCAACTA | TACAACTGGCGAAGATAA |
| PfPMI3 | AAGAGTTGAATAAGTTGAAG | CCTAATTGCTGAATCCTT |
| PfPMI4 | GGAGAACTTAGAAGATGG | ATTACGAGGAGATGATTC |
| PfWPRb2 | ACTGCCTAACTTCTCTAA | CTCAATCTCTGGTTCATC |
基于模式植物拟南芥数据库,以泡桐与拟南芥WPR蛋白同源性比对结果为参照,将得分≥70的蛋白借助在线软件STRING (https://cn.string-db.org/) 构建互作蛋白网络,预测基因功能。
2 结果与分析 2.1 WPR基因家族的鉴定及进化分析在泡桐中共鉴定到16个WPR基因家族成员,对其构建系统发育树[图 1(a)],将其划分为WEB1、PMI2、WPRa和WPRb四个亚家族,泡桐WPR家族成员3对基因间同源性高。对不同物种间WPR家族成员构建系统发育树[图 1(b)],泡桐与木本植物橡胶树同源性较高,与草本植物之间同源性较低。
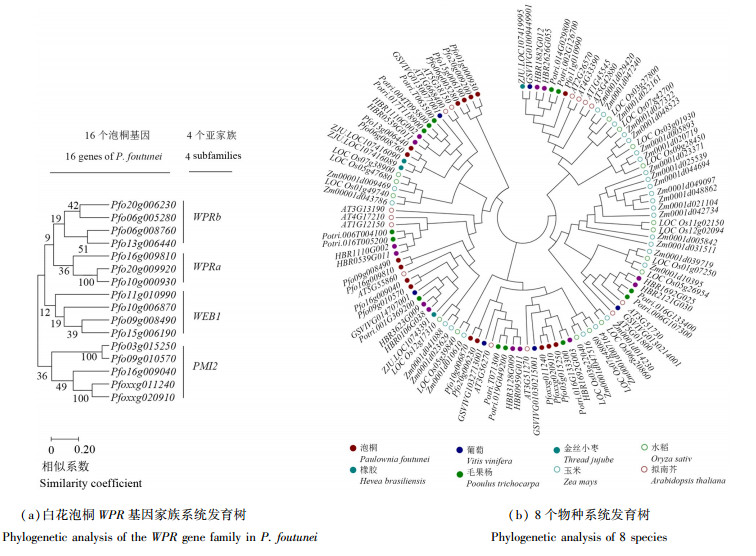
|
图 1 WPR基因家族系统发育树 Fig. 1 Phylogenetic analysis of the WPR gene family |
染色体定位分析发现16个WPR基因在染色体上的分布不均匀(图 2),2个基因没有定位到染色体上,其余14个分布在9条染色体上,其中6号、9号、10号、16号和20号染色体上有2个WPR基因,而3号、11号、13号和15号染色体上有1个WPR基因。将4个亚家族成员按照染色体从小到大的顺序进行命名,未定位到染色体上的家族成员放到最后命名,这为WPR基因家族的研究奠定了基础。
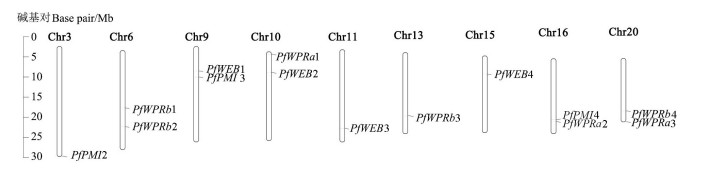
|
图 2 白花泡桐WPR基因家族在染色体上的分布 Fig. 2 Chromosome distribution of the WPR gene family in P. fortunei |
通过对泡桐内16个WPR基因进行共线性分析[图 3(a)],发现其中12个基因间具有共线性,属于旁系同源,说明在进化中发生了扩张事件。对拟南芥和泡桐的WPR基因构建共线性图谱[图 3(b)],发现有9个泡桐基因与拟南芥具有共线性,属于直系同源,说明在进化中发生了复制事件。
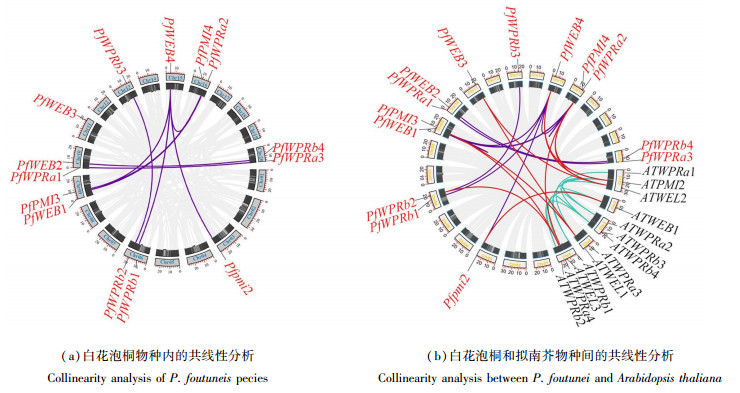
|
图 3 WPR基因家族共线性分析 Fig. 3 Collinearity analysis of the WPR gene family |
计算基因对之间的选择压(表 2),发现Ka介于0~0.31之间,Ks介于0.01~2.61之间,说明在进化过程中泡桐间进行非同义替换,泡桐与拟南芥间进行同义替换。Ka/Ks介于0.12~0.46之间,说明WPR基因家族在进化过程中受到纯化选择。
| 基因1 Gene 1 |
基因2 Gene 2 |
非同义替换率 Ka |
同义替换率 Ks |
选择压 Ka/Ks |
基因1 Gene 1 |
基因2 Gene 2 |
非同义替换率 Ka |
同义替换率 Ks |
选择压 Ka/Ks |
|
| ATWEB1 | ATWEL1 | 0.23 | 0.85 | 0.27 | PfPMI3 | PfPMI4 | 0.07 | 0.29 | 0.26 | |
| ATWPRa4 | PfPMI4 | 0.30 | 2.53 | 0.12 | PfPMI5 | PfPMI6 | 0.00 | 0.01 | 0.39 | |
| ATWPRa4 | PfPMI3 | 0.31 | 2.61 | 0.12 | PfWEB1 | PfWPRa1 | 0.15 | 0.33 | 0.46 | |
| PfPMI2 | PfPMI5 | 0.11 | 0.33 | 0.34 | PfWEB2 | PfWPRb1 | 0.10 | 0.35 | 0.28 | |
| PfPMI2 | PfPMI6 | 0.12 | 0.34 | 0.34 |
对16个WPR家族成员进行基因结构分析(图 4),发现WPR家族成员的外显子和内含子的数量均存在一定的规律。其中PfWEB1、PfWEB2、PfWPRb4含1个外显子且不含内含子,而PfPMI6和PfWPRa3含有2个内含子和3个外显子,其它基因均含有1个内含子和2个外显子,说明基因可能存在某种固定的结构。进一步分析WPR家族成员蛋白二级结构(表 3),发现不同结构占比由高到低依次是α-螺旋、无规则卷曲、延伸链、β-折叠,其中α-螺旋的占比为48.34%~93.27%,无规则卷曲的占比为5.94%~39.65%,而延伸链均小于12.80%,β-折叠不超过4.33%,说明α-螺旋和无规则卷曲是白花泡桐WPR蛋白的主要二级结构,可能跟保守结构域的形成相关。
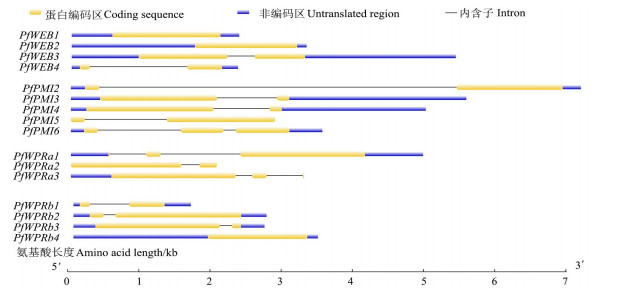
|
图 4 白花泡桐WPR家族成员的基因结构分析 Fig. 4 Gene structure analysis of WPR family members in P. fortunei |
| 基因名称 Gene Name |
二级结构比例Secondary structure proportion/% | 基因名称 Gene Name |
二级结构比例Secondary structure proportion/% | |||||||
| α-螺旋 α-helix |
β-折叠 β-angele |
无规则卷 Random coil |
延伸链 Extended strand |
α-螺旋 α-helix |
β-折叠 β-angele |
无规则卷 Random coil |
延伸链 Extended strand |
|||
| PfWEB1 | 93.27 | 0.00 | 5.94 | 0.79 | PfPMI6 | 54.69 | 0.59 | 39.65 | 5.08 | |
| PfWEB2 | 58.91 | 0.63 | 36.90 | 3.56 | PfWPRa1 | 82.64 | 0.00 | 14.86 | 2.50 | |
| PfWEB3 | 68.07 | 0.93 | 27.86 | 3.15 | PfWPRa2 | 76.69 | 0.59 | 22.72 | 0.00 | |
| PfWEB4 | 54.81 | 4.33 | 30.77 | 10.10 | PfWPRa3 | 76.73 | 0.61 | 22.67 | 0.00 | |
| PfPMI2 | 58.19 | 1.07 | 35.05 | 5.69 | PfWPRb1 | 48.34 | 3.70 | 35.07 | 12.80 | |
| PfPMI3 | 85.74 | 0.00 | 12.60 | 1.66 | PfWPRb2 | 75.76 | 1.37 | 20.73 | 2.13 | |
| PfPMI4 | 80.65 | 0.00 | 17.67 | 1.69 | PfWPRb3 | 58.19 | 0.00 | 37.50 | 3.66 | |
| PfPMI5 | 55.59 | 0.87 | 38.81 | 4.72 | PfWPRb4 | 81.22 | 0.00 | 16.85 | 1.93 | |
分析WPR家族蛋白质的理化性质(表 4),发现其理化性质存在明显差异。氨基酸数目介于208~858之间;等电点介于4.91~9.19之间;亲水性介于-0.657~-0.969之间;均含硫(S) 原子;有1个成员属于不稳定蛋白。对其家族成员进行亚细胞定位预测,发现家族成员在细胞内多个位置均有分布,主要定位在叶绿体、内质网、线粒体,少部分定位在细胞核、细胞膜和高尔基体。
| 基因 Gene |
氨基酸数 Number of amino acid |
分子质量 Molecular weight/u |
原子组成 Atomic composition |
等电点 PI |
不稳定指数 Instbility |
脂肪族 氨基酸指数Aliohatic index |
亲水性 Hydrophilicity |
| PfWEB1 | 505 | 56 959.13 | C2 450H4 069N703O827S11 | 5.16 | 47.28 | 85.01 | -0.803 |
| PfWEB2 | 477 | 54 529.49 | C2 372H3 877N689O732S24 | 8.65 | 41.60 | 78.07 | -0.687 |
| PfWEB3 | 858 | 94 930.27 | C4 081H6 704N1 178O1 380S18 | 5.21 | 53.86 | 77.56 | -0.738 |
| PfWEB4 | 208 | 23 450.76 | C1 023H1 664H290O321S9 | 7.74 | 63.65 | 70.77 | -0.657 |
| PfPMI2 | 562 | 63 232.75 | C2 738H4 519N803O879S15 | 9.19 | 46.50 | 76.58 | -0.764 |
| PfPMI3 | 603 | 66 945.50 | C2 865H4 802N836O971S14 | 5.64 | 39.86 | 77.86 | -0.848 |
| PfPMI4 | 651 | 72 248.37 | C4 629H7 733N1 357O1 576S26 | 5.68 | 41.46 | 76.18 | -0.824 |
| PfPMI5 | 512 | 57 211.72 | C2 752H4 576N810O899S14 | 8.35 | 43.56 | 81.54 | -0.771 |
| PfPMI6 | 572 | 63 844.33 | C2 466H4 093N725O808S12 | 8.50 | 46.79 | 81.71 | -0.731 |
| PfWPRa1 | 825 | 92 635.98 | C2 902H4 756N838O980S13 | 4.96 | 43.12 | 83.28 | -0.817 |
| PfWPRa2 | 599 | 67 483.49 | C4 090H6 747N1 167O1 424S21 | 5.00 | 50.52 | 81.05 | -0.806 |
| PfWPRa3 | 845 | 95 727.79 | C4 000H6 576N1 110O1 363S19 | 4.91 | 52.96 | 78.99 | -0.969 |
| PfWPRb1 | 211 | 23 511.97 | C1 032H1 715N291O324S4 | 8.98 | 47.54 | 84.03 | -0.688 |
| PfWPRb2 | 656 | 74 562.08 | C3 223H5 387N937O1 041S20 | 9.08 | 48.99 | 79.34 | -0.811 |
| PfWPRb3 | 464 | 52 890.26 | C2 295H3 738N664O722S22 | 6.79 | 48.18 | 76.51 | -0.730 |
| PfWPRb4 | 623 | 70 834.42 | C3 042H5 082N888O1 004S2 | 8.07 | 58.07 | 76.53 | -0.855 |
对WPR家族成员进行蛋白质基序预测,发现共有10个保守基序[图 5(a)],并对基序进行保守性分析[图 5(b)]。Motif 8存在于所有家族成员中,属于高度保守基序;在PfWEB2、PfPMI2、PfPMI5、PfPMI6及PfWPRb3中motif 4、motif 6及motif 9成对出现且高度保守,说明这些家族成员间可能具有某种固定的功能。

|
图 5 白花泡桐WPR家族成员蛋白质motif分析 Fig. 5 Motif of WPR family members in P. fortunei |
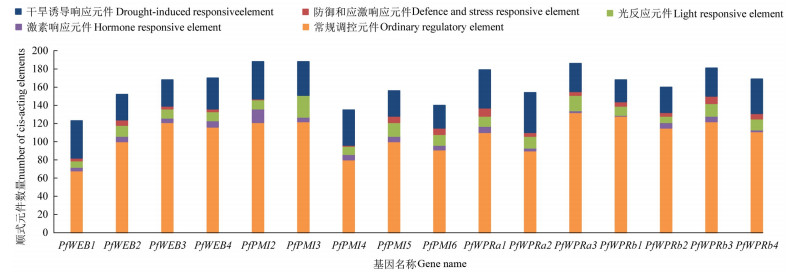
对WPR基因家族进行启动子顺式元件统计(图 6),发现有5大类55种不同的顺式调控元件,主要是常规调控、激素响应、光反应、防御和应激响应及干旱诱导响应元件。统计发现,PfWEB1基因所含顺式作用元件数量最少,共27种123个;PfPMI2和PfPMI3最多,分别含26种188个和23种188个;PfPMI2、PfPMI3顺式调控元件虽然数量多,但种类少,表明基因功能主要与光合作用及干旱胁迫相关。
|
图 6 白花泡桐WPR基因家族顺式作用元件统计 Fig. 6 Statistical analysis of the WPR gene family cis-acting elements in P. fortunei |
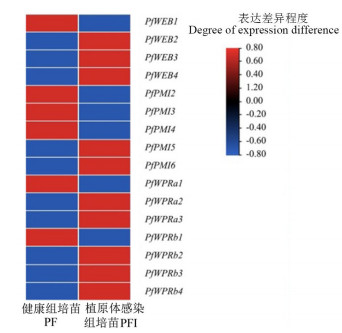
分析WPR家族基因在丛枝病病苗与健康苗中的转录表达水平(图 7),发现10个成员在病苗中表达量升高,6个成员在病苗中表达量降低,不同的亚家族成员在病健苗表达量中表现出不同的情况,说明发病过程中不同亚家族可能存在功能上的差异。
|
图 7 WPR家族基因在白花泡桐病健苗的表达量热图 Fig. 7 Expression heat map of WPR family gene in PF and PFI |
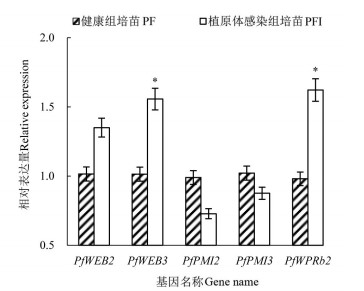
为验证转录组数据的准确性,从16个家族成员中随机挑5个基因进行RT-qPCR验证。结果(图 8) 表明,5个WPR基因中,3个基因在病苗中表达量上升,2个基因在病苗中表达量下降,与转录组结果一致,说明转录组结果的可靠性较高。
|
图 8 差异基因的RT-qPCR验证 Fig. 8 Validation of the differential gene using RT-qPCR |
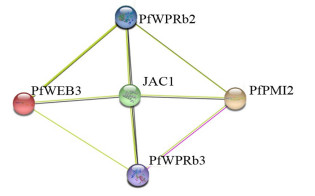
为了解WPR基因家族成员在泡桐丛枝病中发挥的作用,对其构建蛋白互作网络(图 9),发现在白花泡桐丛枝病病苗中PfWEB3、PfWPRb2、PfWPRb3和PfPMI2均与一种能导致叶片黄化的叶绿体运动相关的蛋白JAC1互作,推测其与JAC1具有相似功能。
|
图 9 WPR基因家族蛋白互作网络 Fig. 9 Protein interaction network of the WPR family genes |
WPR家族成员在拟南芥中会导致叶片黄化[7-10],前期研究发现,在泡桐丛枝植原体侵染后WPR基因家族的表达量发生变化[15-16],目前,关于WPR基因家族在泡桐丛枝病中的功能尚不清楚。白花泡桐基因组中共存在16个WPR家族成员,比葡萄、毛果杨、拟南芥WPR家族成员数量都多[5],推测泡桐WPR家族成员数量可能与全基因组复制事件有关[20]。对WPR家族进行进化分析,发现其与木本植物同源性较高;基因复制事件和选择压表明泡桐家族成员共线性产生的原因是片段复制。大部分家族成员具有相似的基因结构且所有家族成员都含有高度保守的基序(motif 8),暗示着家族成员间可能具有某种相似的功能,顺式作用元件表明WPR家族成员主要参与干旱胁迫和光合作用。综上所述,WPR家族成员在进化过程中可能高度保守并具有相似的功能。对WPR家族成员进行蛋白互作网络分析,发现白花泡桐丛枝病病苗中PfWEB3、PfWPRb2、PfWPRb3和PfPMI2与泡桐丛枝病引起的叶片黄化密切相关。据报道,拟南芥中AtWEB1、AtPMI2和AtPMI15表达量升高会导致植株叶片黄化[6]。PfWEB3、PfWPRb2和PfWPRb3表达量在泡桐丛枝病病苗中显著上调,且同源性分析发现,泡桐PfWEB3与拟南芥AtWEB1同源性为70%、泡桐PfWPRb2与拟南芥AtPMI2同源性为74%、泡桐PfWPRb3与拟南芥AtPMI15同源性为72%,同时,PfWEB3、PfWPRb2和PfWPRb3蛋白与一种能导致叶片黄化的叶绿体运动相关的蛋白JAC1互作,推测它们协同调节泡桐丛枝病发病后的叶片黄化过程。另外,研究发现,在调节拟南芥叶片黄化过程中,AtPMI1表达被抑制[3]。本研究的进化分析表明,泡桐PfPMI2与拟南芥AtPMI1同源性为77%,且泡桐PfPMI2表达量在丛枝病苗中显著下调,暗示低表达的PfPMI2可能也参与了叶片黄化过程。因此,推测PfWEB3、PfWPRb2、PfWPRb3和PfPMI2可能通过不同的调节方式调控泡桐丛枝病引起的叶片黄化,该结果为深入了解WPR家族成员在丛枝病叶片黄化中的作用及其参与的代谢途径奠定基础。
| [1] |
KODAMA Y, SUETSUGU N, KONG S G, et al. Two interacting coiled-coil proteins, WEB1 and PMI2, maintain the chloroplast photorelocation movement velocity in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(45): 19591-19596. DOI:10.1073/pnas.1007836107 |
| [2] |
SUETSUGU N, WADA M. Evolution of the Cp-actin-based motility system of chloroplasts in green plants[J]. Frontiers in Plant Science, 2016, 7: 561. |
| [3] |
DEBLASIO S L, LUESSE D L, HANGARTER R P. A plant-specific protein essential for blue-light-induced chloroplast movements[J]. Plant Physiology, 2005, 139(1): 101-114. DOI:10.1104/pp.105.061887 |
| [4] |
LUESSE D R, DEBLASIO S L, HANGARTER R P. Plastid movement impaired 2, a new gene involved in normal blue-light-induced chloroplast movements in Arabidopsis[J]. Plant Physiology, 2006, 141(4): 1328-1337. DOI:10.1104/pp.106.080333 |
| [5] |
KODAMA Y, SUETSUGU N, WADA M. Novel protein-protein interaction family proteins involved in chloroplast movement response[J]. Plant Signaling & Behavior, 2011, 6(4): 483-490. |
| [6] |
SUETSUGU N, WADA M. Two coiled-coil proteins, WEB1 and PMI2, suppress the signaling pathway of chloroplast accumulation response that is mediated by two phototropin-interacting proteins, RPT2 and NCH1, in seed plants[J]. International Journal of Molecular Sciences, 2017, 18(7): 1469. DOI:10.3390/ijms18071469 |
| [7] |
KASAHARA M, KAGAWA T, OIKAWA K, et al. Chloroplast avoidance movement reduces photodamage in plants[J]. Nature, 2002, 420(6917): 829-832. DOI:10.1038/nature01213 |
| [8] |
KÖNIGER M. Chloroplast movement in higher plants, ferns and bryophytes: a comparative point of view[M]//HANSON D T, RICE S K. Photosynthesis in bryophytes and early land plants. Dordrecht: Springer, 2014: 131-150.
|
| [9] |
DAVIS P A, CAYLOR S, WHIPPO C W, et al. Changes in leaf optical properties associated with light-dependent chloroplast movements[J]. Plant, Cell & Environment, 2011, 34(12): 2047-2059. |
| [10] |
蒋建平, 苌哲新, 舒寿兰, 等. 泡桐栽培学[M]. 北京: 中国林业出版社, 1990: 15-48.
|
| [11] |
田国忠, 林彩丽, 任争光, 等. 我国难培养原核植物病原生物-植原体研究现状[C]//第四届全国微生物资源学术暨国家微生物资源平台运行服务研讨会论文集. 成都: 中国微生物学会, 2012: 56-58.
|
| [12] |
田国忠, 温秀军, 李永, 等. 枣疯病和泡桐丛枝病原植原体分离物的组织培养保藏和嫁接传染研究[J]. 林业科学研究, 2005, 18(1): 1-9. DOI:10.3321/j.issn:1001-1498.2005.01.001 |
| [13] |
FAN G Q, CAO X B, ZHAO Z L, et al. Transcriptome analysis of the genes related to the morphological changes of Paulownia tomentosa plantlets infected with phytoplasma[J]. Acta Physiologiae Plantarum, 2015, 37(10): 202. DOI:10.1007/s11738-015-1948-y |
| [14] |
FAN G Q, CAO X B, NIU S Y, et al. Transcriptome, microRNA, and degradome analyses of the gene expression of Paulownia with phytoplamsa[J]. BMC Genomics, 2015, 16: 896. DOI:10.1186/s12864-015-2074-3 |
| [15] |
常梦悦, 翟晓巧. PMI2的研究进展[J]. 河南林业科技, 2019, 39(3): 13-16. DOI:10.3969/j.issn.1003-2630.2019.03.004 |
| [16] |
CAO Y B, SUN G L, ZHAI X Q, et al. Genomic insights into the fast growth of paulownias and the formation of Paulownia witches' broom[J]. Molecular Plant, 2021, 14(10): 1668-1682. DOI:10.1016/j.molp.2021.06.021 |
| [17] |
周丽霞, 曹红星. 油棕WRKY转录因子的全基因组鉴定与分析[J]. 广西植物, 2020, 40(7): 977-987. |
| [18] |
邹智. 蓖麻WRKY转录因子的全基因组鉴定及其进化分析[J]. 中国油料作物学报, 2013, 35(1): 36-42. |
| [19] |
罗玛妮娅, 王毅, 郑元, 等. 牛樟芝Zn(Ⅱ)2Cys6转录因子的全基因组鉴定与分析[J]. 森林与环境学报, 2021, 41(2): 157-163. |
| [20] |
VOORRIPS R E. MapChart: software for the graphical presentation of linkage maps and QTLs[J]. Journal of Heredity, 2002, 93(1): 77-78. DOI:10.1093/jhered/93.1.77 |
| [21] |
YAN L J, FAN G Q, LI X Y. Genome-wide analysis of three histone marks and gene expression in Paulownia fortunei with phytoplasma infection[J]. BMC Genomics, 2019, 20(1): 234. DOI:10.1186/s12864-019-5609-1 |
| [22] |
韩艳丽, 李静, 操庆国, 等. 梨树CDPK基因家族进化和表达分析[J]. 西北农业学报, 2017, 26(7): 1026-1032. |

 2022, Vol. 42
2022, Vol. 42


