文章信息
- 肖琳, 乐易迅, 孙维红, 张培兰, 邹双全
- XIAO Lin, LE Yixun, SUN Weihong, ZHANG Peilan, ZOU Shuangquan
- 秋茄和桐花树HDACs基因家族鉴定及特征分析
- Identification and characterization of HDACs gene family in Kandelia candel and Aegiceras corniculatum
- 森林与环境学报,2022, 42(2): 184-192.
- Journal of Forest and Environment,2022, 42(2): 184-192.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2022.02.009
-
文章历史
- 收稿日期: 2021-10-04
- 修回日期: 2022-01-16
2. 自然生物资源保育利用福建省高校工程技术研究中心, 福建 福州 350002
2. Fujian Colleges and Universities Engineering Research Institute of Conservation and Utilization of Natural Bioresources, Fuzhou, Fujian 350002, China
红树林是指生长在热带及亚热带潮间带,由不同灌木和乔木组成的木本植物群落复合体,具有重要的生态和经济价值。红树林为多种动物提供生存繁衍场所,而红树林的消失也将增加沿海地区自然灾害(如侵蚀、风暴潮、海啸和台风等) 对人类安全和沿海发展的威胁[1-2]。红树林的生存环境相对特殊,具有水淹、盐渍、高渗透压及强紫外照射等非生物胁迫,其中盐分对其生长和分布具有极其重要的作用。红树植物秋茄[Kandelia candel (Linn.) Druce] 和桐花树[Aegiceras corniculatum(Linn.) Blanco] 分别隶属红树科(Rhizophoraceae) 秋茄属(Kandelia) 和紫金牛科(Myrsinaceae) 蜡烛果属(Aegiceras),它们的木材坚实、耐腐,可用作薪炭材;树皮含单宁,可提制栲胶[3-4]。此外,它们拥有共同的耐盐机制——根系拒盐,即通过根系内皮层凯氏带对海水中的盐分进行过滤,以拒绝大部分盐离子由根部向地上部分运输[5-6]。
组蛋白去乙酰化酶(histone deacetylase, HDAC) 广泛参与了植物生长和非生物胁迫的响应过程,植物HDACs基因家族可分为RPD3/HDA1、SIR2和HD2等3个亚家族[7-8]。其中,HD2亚家族是植物HDACs基因家族所特有的,其结构不同于RPD3/HDA1和SIR2亚家族[9]。水稻(Oryza sativa L.) OsHDT701在种子萌发过程中的过度表达增强了水稻苗期的抗盐和抗渗透胁迫能力[10]。毛果杨(Populus trichocarpa Torr. & Gray) PtHDT903基因在盐胁迫下表达下调,而在冷胁迫下表达上调[11]。目前,对植物中HDACs基因家族的功能研究主要集中在拟南芥(Arabidopsis thaliana L.)、玉米(Zea mays L.)、水稻等草本植物中,对木本植物HDACs基因家族的研究较少,而HDACs是否参与红树植物的独特耐盐机制目前尚不清楚。因此,本研究对秋茄和桐花树HDACs基因家族蛋白进行鉴定,并对各个成员的蛋白质理化性质、系统进化树、染色体定位、非同义替换率/同义替换率(Ka/Ks) 进化分析、保守基序、保守蛋白结构域、基因结构、启动子顺式作用元件、蛋白质二级结构、多序列比对及表达量进行分析,以期为解析两种红树植物特有的耐盐机制提供参考,结果可为今后研究HDACs基因家族的结构和功能,识别和表征其他木本植物的HDACs提供一定的参考,同时为红树植物耐盐分子机制研究奠定理论基础。
1 材料与方法 1.1 试验材料供试材料由福建省泉州市泉州湾河口湿地省级自然保护区提供,分别采集秋茄和桐花树果实及根系,样本于-80 ℃冰箱低温保存备用。研究数据采用实验室测序获得的秋茄和桐花树基因组及转录组数据库。基因组序列已提交给国家基因组数据中心(National Genomics Data Center,NGDC),全基因组组装注释数据已保存在BioProject/GWH中(https://bigd.big.ac.cn/gwh),加入代码为PRJCA002330/GWHACBH00000000。
1.2 红树植物HDACs家族鉴定及基本分析通过在线软件Pfam (http://pfam.xfam.org/) 下载HDACs基因隐马尔可夫模型(RPD3/HDA1: PF00850、SIR2: PF02146、HD2: PF17800)。以拟南芥HDACs家族基因的氨基酸序列为对照,进行本地BLAST比对检索,并按照阈值条件为E-value<1×10-5将可信度较高的基因筛选出来,初步筛选秋茄和桐花树HDACs家族成员。然后通过美国国家生物信息中心(National Center for Biotechnology Information, NCBI, http://www.blast.ncbi.nlm.nih.gov/)验证候选基因的保守结构域,以及验证所查序列所属基因家族及其置信度。
用ExPASy (https://web.expasy.org/protparam/) 获取秋茄和桐花树HDACs相对分子质量,用NCBI (blast.ncbi.nlm.nih.gov/) 的BLASTn模块进行蛋白质长度预测,用Euk-mPLoc 2. 0 (http://www.csbio.sjtu.edu.cn/bioinf/euk-multi-2/) 对蛋白进行亚细胞定位,用Prabi (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html) 进行蛋白质二级结构的预测,用多重最大期望值的序列诱导(multiple em for motif elicitation,MEME) 算法(https://meme-suite.org/meme/doc/meme.html?man_type=web) 鉴定秋茄和桐花树HDACs基因家族蛋白序列的保守基序,motif数量选择20。利用NCBI获得的Hitdata文件进行结构域预测,用gff文件进行基因结构分析,用生物学家工具箱(toolbox for biologists,TBtoolsv 1. 09854) 软件进行可视化分析[12]。
利用TBtoolsv 1. 09854软件从秋茄和桐花树全基因组文件中提取含有2 000 bp DNA的HDACs基因家族启动子序列,用植物顺式作用调节元件(plant cis-acting regulatory element,PlantCARE, http://bioinformatics.psb.ugent.be/webtools/plantcare/html/) 网站预测秋茄和桐花树HDACs启动子顺式作用元件,用TBtoolsv 1. 09854软件进行可视化分析[12],然后用Excel 2019软件制图。
1.3 红树植物HDACs家族系统进化及表达模式分析分别从拟南芥信息资源网站(the arabidopsis information resource,TAIR,https://www.arabidopsis.org/)、Phytozome 13 (https://phytozome-next.jgi.doe.gov/pz/) 以及NCBI获得拟南芥、毛果杨和水稻3个物种的HDACs基因蛋白序列,用分子进化遗传学分析(Molecular Evolutionary Genetics Analysis,MEGA-XBF) 软件的ClustalW进行比对,采用邻近相邻法(Neighbor-Joining) 对秋茄、桐花树、拟南芥、水稻、毛果杨5个物种构建系统进化树,参数设置为1 000次(bootstrap) 检验、Poisson correction、pair-wise deletion,其他参数为默认值[13]。用在线网站EvolView (https://evolgenius.info//evolview-v2/#login) 和Adobe Illustrator 2021对进化树进行美化,用在线软件TBtoolsv 1. 09854进行染色体定位和Ka/Ks计算。用TBtoolsv 1. 09854软件对秋茄和桐花树的根部及果实转录组数据绘制基因表达量热图[12]。
1.4 红树植物HD2亚家族多序列比对分析将秋茄和桐花树中得到的HD2亚家族基因和已知的拟南芥HD2亚家族基因使用在线软件DNAMAN V6进行多序列比对,其中黑色表示序列同源性为100%,蓝色表示同源性≥75%,粉色表示同源性≥50%。
2 结果与分析 2.1 红树植物HDACs家族鉴定分析基于BLAST同源搜索方法,用已知的18个拟南芥HDACs家族成员为参考序列,在秋茄和桐花树基因组中共鉴定出28个HDACs基因,通过验证最终确定了24个具有HDACs结构域且序列完整的家族成员,并参照拟南芥HDACs家族命名方式进行命名。秋茄和桐花树HDACs家族的编码蛋白基本信息和二级结构信息见表 1。秋茄KcHAD1的蛋白质最长,为686个氨基酸残基(amino acid residues, aa),分子质量高达77 386. 83 u;KcHDT2的蛋白质长度为209 aa,分子质量为22 738. 32 u。秋茄HDACs家族成员(KcHDACs) 广泛存在于细胞核或细胞质。
| 基因名 Gene name |
分子质量 Molecular mass/u |
亚家族分类 Subfamily classification |
亚细胞定位 Subcellular localization |
蛋白质长度 Protein length/aa |
二级结构比例 Secondary structure proportion/% |
|||
| α-螺旋 α-helix |
延伸链 Extended chain |
β-转角 β-turn |
无规则卷曲 Random coil |
|||||
| KcSRT1(Maker00007783) | 42 277.47 | SIR2 | 细胞质 Cytoplasm |
388 | 34.54 | 15.21 | 6.96 | 43.30 |
| KcSRT2(Maker00010657) | 51 723.33 | SIR2 | 细胞质、细胞核 Cytoplasm, Nucleus |
466 | 29.83 | 17.38 | 6.22 | 46.57 |
| KcSRT3(Maker00012827) | 48 381.39 | SIR2 | 细胞质、细胞核 Cytoplasm, Nucleus |
441 | 41.95 | 14.06 | 6.58 | 37.41 |
| KcHDA1(Maker00000870) | 77 386.83 | RPD3/HDA1 | 细胞质Cytoplasm | 686 | 36.01 | 14.72 | 7.14 | 42.13 |
| KcHDA2(Maker00001161) | 39 151.90 | RPD3/HDA1 | 细胞质Cytoplasm | 353 | 35.13 | 18.98 | 8.22 | 37.68 |
| KcHDA3(Maker00008887) | 53 065.59 | RPD3/HDA1 | 细胞质、细胞核 Cytoplasm, Nucleus |
471 | 34.82 | 14.44 | 7.01 | 43.74 |
| KcHDA4(Maker00010156) | 61 385.54 | RPD3/HDA1 | 细胞质Cytoplasm | 561 | 32.62 | 14.44 | 6.95 | 45.99 |
| KcHDA5(Maker00012893) | 44 428.52 | RPD3/HDA1 | 细胞质Cytoplasm | 409 | 39.36 | 14.18 | 6.60 | 39.85 |
| KcHDA6(Maker00015452) | 49 220.50 | RPD3/HDA1 | 细胞质、细胞核 Cytoplasm, Nucleus |
430 | 36.28 | 16.98 | 7.91 | 38.84 |
| KcHDA7(Maker00015474) | 41 214.94 | RPD3/HDA1 | 细胞质Cytoplasm | 379 | 33.51 | 16.89 | 5.80 | 43.80 |
| KcHDA8(Maker00017258) | 52 159.50 | RPD3/HDA1 | 细胞核Nucleus | 463 | 35.85 | 12.96 | 5.83 | 45.36 |
| KcHDT1(Maker00016977) | 35 503.64 | HD2 | 细胞核Nucleus | 324 | 14.15 | 17.59 | 6.79 | 61.11 |
| KcHDT2(Maker00017857) | 22 738.32 | HD2 | 细胞核Nucleus | 209 | 6.70 | 16.75 | 3.83 | 72.73 |
| AcoSRT1(Aco015608) | 21 175.33 | SIR2 | 细胞质、细胞核、 线粒体、细胞外 Cytoplasm, Nucleus, Mitochondria, Extracellular |
186 | 37.63 | 10.22 | 6.45 | 45.70 |
| AcoSRT2(Aco022210) | 52 115.30 | SIR2 | 细胞质、细胞核 Cytoplasm, Nucleus |
466 | 28.11 | 18.03 | 6.44 | 47.42 |
| AcoSRT3(Aco025061) | 27 357.79 | SIR2 | 细胞质、细胞核 Cytoplasm, Nucleus |
240 | 25.42 | 22.50 | 8.33 | 43.75 |
| AcoHDA1(Aco000775) | 55 819.91 | RPD3/HDA1 | 细胞质、细胞核 Cytoplasm, Nucleus |
493 | 34.69 | 13.18 | 6.90 | 45.23 |
| AcoHDA2(Aco008936) | 39 909.78 | RPD3/HDA1 | 细胞质、细胞核 Cytoplasm, Nucleus |
355 | 36.06 | 22.25 | 7.89 | 33.80 |
| AcoHDA3(Aco010789) | 45 996.94 | RPD3/HDA1 | 细胞质Cytoplasm | 423 | 35.70 | 15.60 | 7.33 | 41.37 |
| AcoHDA4(Aco012134) | 49 022.92 | RPD3/HDA1 | 细胞质Cytoplasm | 452 | 37.61 | 16.15 | 7.74 | 38.50 |
| AcoHDA5(Aco019178) | 76 699.16 | RPD3/HDA1 | 细胞质Cytoplasm | 688 | 34.01 | 18.46 | 6.83 | 40.70 |
| AcoHDA6(Aco021153) | 52 971.59 | RPD3/HDA1 | 细胞质Cytoplasm | 472 | 36.44 | 16.10 | 7.20 | 40.25 |
| AcoHDA7(Aco022050) | 50 816.29 | RPD3/HDA1 | 细胞质、细胞核 Cytoplasm, Nucleus |
454 | 34.14 | 15.86 | 6.83 | 43.17 |
| AcoHDT1(Aco010402) | 51 641.82 | HD2 | 细胞核Nucleus | 470 | 15.32 | 14.68 | 3.40 | 66.60 |
从表 1还可以看出,桐花树AcoHDACs的蛋白质长度为186~688 aa,分子质量为21 175. 33~76 699. 16 u。桐花树HDACs家族成员(AcoHDACs) AcoSRT1 (Aco015608) 同时位于细胞核、细胞质、线粒体和细胞外,其他多位于细胞核或细胞质。
预测蛋白二级结构可为研究其功能及二级结构之间的相互作用模式提供理论基础。在线分析结果(表 1) 表明,秋茄和桐花树HDACs家族蛋白二级结构主要为无规则卷曲结构,其中HD2亚家族成员二级结构差异较为显著,与其他2个亚家族二级结构的相似度低。在PRD3/HDA1亚家族,无规则卷曲结构在KcHDACs中占比最大,其次以α-螺旋为主,最后是延伸链和β-转角;桐花树7个蛋白除AcoHDA2无规则卷曲小于α-螺旋外,其他蛋白均为无规则卷曲>α-螺旋,其次是延伸链>β-转角。SIR2亚家族中,除KcSRT3无规则卷曲α-螺旋,其余蛋白二级结构均以无规则卷曲>α-螺旋>延伸链>β-转角排列。秋茄和桐花树HD2亚家族无规则卷曲显著大于α-螺旋,均超过60%,其中秋茄的2个HD2蛋白二级结构大小排序与其他2个亚家族不同,无规则卷曲>延伸链>α-螺旋>β-转角。
2.2 红树植物HDACs家族系统进化树分析为了研究秋茄和桐花树与模式植物之间HDACs基因家族的进化关系,用MEGA X构建秋茄、桐花树、拟南芥、水稻和毛果杨共计74个HDACs家族成员序列的系统发育树。根据拟南芥HDACs基因家族的分类方法,将秋茄和桐花树的24个序列划分为3个亚家族,其中RPD3/HDA1亚家族有15个成员,HD2亚家族有3个成员,SIR2有6个成员(图 1)。

|
▲表示秋茄, ★表示桐花树。Note: ▲ indicates K. candel, ★ indicates Aegiceras corniculatum. 图 1 红树植物与模式植物HDACs基因家族的系统进化树 Fig. 1 Phylogenetic tree of HDACs gene family in mangrove and model plants |
秋茄和桐花树HDACs基因在染色体上的分布如图 2所示。秋茄HDACs基因家族成员多数以成对的方式出现在8条染色体上,5条染色体(Chr 1、Chr 3、Chr 4、Chr 9和Chr 16) 各分布2个基因,其余3条染色体各分布1个基因,其中,Chr 1 (KcHDT1) 染色体和Chr 17 (KcHDT2) 染色体、Chr 2 (KcSRT1) 染色体和Chr 3 (KcSRT3) 染色体均发生过片段复制事件。桐花树HDACs主要是单条基因出现在不同的染色体上,除染色体Chr 6和Chr 17上有2个基因外,其他染色体都是1个基因对应1条染色体,其中,Chr 1 (AcoSRT3) 染色体和Chr 5 (AcoSRT2) 染色体发生了片段复制事件。同时,通过计算3对片段复制基因的Ka/Ks值,发现秋茄和桐花树HDACs家族同源基因对Ka/Ks都小于1,说明纯化选择是秋茄和桐花树HDACs基因进化过程中的动力来源。

|
图 2 秋茄和桐花树HDACs基因在染色体上的分布 Fig. 2 Distribution of HDACs genes on chromosomes of K. candel and Aegiceras corniculatum |
秋茄和桐花树HDACs保守基序、保守蛋白结构域及基因结构如图 3所示。PRD3/HDA1亚家族中外显子-内含子分布数量差异较大,其中KcHDA4外显子(17) -内含子(13) 和AcoHDA6外显子(16) -内含子(13) 数量最多,KcHDA7和AcoHDA3最少,均为4个;HD2亚家族外显子-内含子数量差异较小,分别为8~11个和6~10个;同时秋茄SIR2亚家族的外显子为11~16个,内含子为10~13个,而桐花树SIR2亚家族外显子-内含子数量差异大,AcoSRT2外显子(14) -内含子(13) 数量最多,AcoSRT1外显子(6) -内含子(5) 数量最少。对蛋白质保守基序进行分析后发现,motif 1和motif 11存在于PRD3/HDA1亚家族所有成员,同时,除KcHDA2、AcoHDA2和AcoHDA5外,PRD3/HDA1亚家族所有成员都包含motif 2和motif 9;HD2亚家族中所有成员都包含motif 19;SIR2亚家族成员除AcoSRT1,其余成员均包含motif 8、motif 10和motif 15。结构域分析发现,秋茄和桐花树HDACs家族在结构上都相对保守,15个PRD3/HDA1亚家族成员均具有1个典型的HDACs结构域。HD2亚家族蛋白序列结构与肽基脯氨酸顺反异构酶(peptidyl-prolyl cis-trans isomerase, PPIase) 具有相似性,3个HD2亚家族成员具有Nucleoplasmin结构域。SIR2亚家族成员的蛋白结构与其他2个亚家族不具有同源性且保守性高,6个成员均具有SIR2结构域。

|
图 3 秋茄和桐花树HDACs的保守基序、保守蛋白结构域及基因结构 Fig. 3 Conserved motifs, conserved protein domains and gene structures of HDACs in K. candel and Aegiceras corniculatum |
在线对秋茄和桐花树24个基因进行2 000 bp启动子顺式作用元件分析,结果(图 4) 表明,秋茄和桐花树HDACs家族基因序列包含了5种激素响应元件、3种胁迫响应元件及一些特异性元件。秋茄和桐花树HDACs家族基因序列都具有光响应元件,且光响应元件总数量占比最大,分别有139个和128个。5种激素响应元件分布不均,其中茉莉酸甲酯(meth-yljasmonate, MeJA) 响应元件和脱落酸(abscisic acid, ABA) 响应元件最多。

|
图 4 秋茄和桐花树HDACs启动子的顺式作用元件 Fig. 4 Cis-acting elements of K. candel and Aegiceras corniculatum HDACs promoters |
此外,秋茄和桐花树HDACs家族中还存在少量与昼夜节律调控、茎尖生长发育、胚乳发育和伤口有关的特异性元件。对秋茄和桐花树HDACs的启动子元件数量及分布研究发现,其基因表达在生长过程中会受到至少2种元件共同调控,而MeJA响应元件和ABA响应元件在诱导秋茄和桐花树HDACs基因激素表达过程中起主要作用,同时,干旱和低温等逆境胁迫响应元件总数仅次于光响应和激素响应元件总数。
2.6 红树植物HD2亚家族多序列比对分析HD2是植物所特有的基因家族,它与HDACs其他两个家族的蛋白序列不具有同源性,因此,分别对秋茄和拟南芥、桐花树和拟南芥HD2亚家族进行多序列比对,结果(图 5) 表明,秋茄和桐花树HD2序列N端缺少特有的保守五肽基序-MEFWG保守区域,但仍具有2个保守氨基酸残基(组氨酸和天冬氨酸) 和富含天冬氨酸和谷氨酸氨基酸残基的中心区域,以及C端的一个C2H2锌指结构域。

|
图 5 秋茄和桐花树HD2亚家族多序列比对 Fig. 5 Multiple sequence alignment of HD2 subfamily in K. candel and Aegiceras corniculatum |
通过分析秋茄和桐花树根与果实的转录组数据,分别得到它们HDACs家族基因的表达量模式(图 6)。结果显示,KcSRT3、KcHDT2和KcHDA8在根中高表达,而KcHDT2和KcHDA8在果实的5个时期也都有高表达。AcoHDA1和AcoHDT1在根中高表达,同时AcoHDT1在果实中表达量也较高。
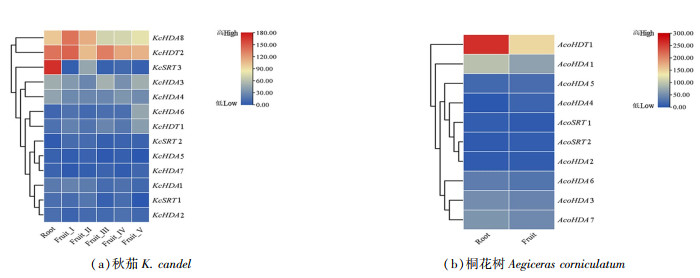
|
图 6 秋茄和桐花树HDACs基因根和果表达分析 Fig. 6 Expression analysis of HDACs gene in roots and fruits of K. candel and Aegiceras corniculatum |
红树植物秋茄和桐花树HDACs基因组中分别鉴定出13个和11个HDACs基因家族成员,进化树分析发现,这些基因主要分为RPD3/HDA1、SIR2和HD2共3个亚家族。基因复制是基因家族进化过程中最主要的扩张方式。片段复制事件是秋茄和桐花树HDACs家族扩张的主要方式,而从基因表达还可以看出,3对片段复制基因中的2个成员KcHDT2和KcSRT3主要在根和果实中表达。3个亚家族之间的蛋白结构和基序的数量及排列不同,但它们在同一分枝上高度保守,表明亚家族不同分枝间可能具有功能的差异性。
启动子元件与转录因子结合能促进基因进行特异性表达。秋茄和桐花树HDACs基因包含多种激素响应及胁迫响应的作用元件,其中光响应元件数目最多,MeJA和ABA元件数目次之。研究发现,外源ABA与沉默表达ABA分解基因均能提高水稻对盐碱胁迫的抗性[14];MeJA可抑制小麦(Triticum aestivum L.) 受盐胁迫引起的过量活性氧,从而增强其抗盐性[15];有关茶树[Camellia sinensis (L.) O. Ktze.] 类胡萝卜素裂解双加氧酶(carotenoid cleavage dioxygenase, CCD) 基因家族研究表明,在光照处理后CCD基因表达量显著上调,其中包含了大量光响应元件[16]。而在本研究中,由于受元件数目及其作用影响,推测秋茄和桐花树HDACs基因中的表达调控模式也会受多重因子影响,较为复杂,且多与植物抗逆性相关。大麦的HD2基因对茉莉酸(jasmonic acid, JA)、ABA和水杨酸(salicylic acid, SA) 与植物胁迫相关激素有响应[17],拟南芥AtHD2D (AtHDT4) 的转基因植物对干旱、盐和冷胁迫等非生物胁迫表现出高耐受性[18],这说明HD2基因在应对不同非生物胁迫方面发挥着关键作用。
HD2亚家族在植物的生长过程中具有特定功能[19],其具有的去乙酰化酶结构域与RPD3/HDA1、SIR2亚家族明显的差异性也证实了这一点。无规则卷曲作为连接其他二级结构的重要区域,有利于构成酶的活性部位和蛋白质的功能部位[20]。二级结构分析结果显示,HD2亚家族蛋白中4种结构占比与其他2个亚家族不同,秋茄和桐花树HD2亚家族蛋白中存在大量无规则卷曲结构,占比60%以上。秋茄和桐花树HD2序列N端都缺少固定的MEFWG motif,并被连续存在的酸性和碱性氨基酸残基包围,与拟南芥和玉米不同[21],这可能是导致秋茄和桐花树HD2亚家族α-螺旋程度极低的原因之一。以上这些结果进一步表明了HD2亚家族的特异性。
拟南芥AtHD2C (AtHDT3) 转基因植物通过降低自身蒸腾作用,以增强对盐和干旱胁迫的耐受性[22];毛果杨PtHDT902过度表达抑制了不定根的形成,并调控其耐盐性[23];转录因子SCARECROW与AtHDA19相互作用,可调控根系发育[24];SRT1基因在水稻中过度表达提高了对氧胁迫的耐受性[25]。秋茄和桐花树中共有7个基因(KcHDT1、KcHDT2、KcSRT3、KcHDA8、AcoSRT1、AcoHDA1和AcoHDT1) 与上述基因聚类紧密,其中KcHDT2、KcSRT3、KcHDA8和AcoHDT1在根和果实中存在特异性表达,KcHDT2和AcoHDT1在根和果实的表达显著升高。因此,推测秋茄和桐花树的HD2基因具有抗盐碱,适应水淹、缺氧及过量光照等胁迫环境的能力,且通过调控根部进而完成根系拒盐这一过程。
| [1] |
王友绍. 红树林分子生态学[M]. 北京: 科学出版社, 2019.
|
| [2] |
LEE S Y, PRIMAVERA J H, DAHDOUH-GUEBAS F, et al. Ecological role and services of tropical mangrove ecosystems: a reassessment[J]. Global Ecology and Biogeography, 2014, 23(7): 726-743. DOI:10.1111/geb.12155 |
| [3] |
王文卿, 陈琼. 南方滨海耐盐植物资源[M]. 厦门: 厦门大学出版社, 2013.
|
| [4] |
中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 1979: 33.
|
| [5] |
王文卿, 林鹏. 红树植物体内元素分布特点与抗盐机理[J]. 林业科学, 2003, 39(4): 30-36. DOI:10.3321/j.issn:1001-7488.2003.04.005 |
| [6] |
张宜辉, 王文卿, 林鹏. 红树植物的盐分平衡机制[J]. 海洋科学, 2007, 31(11): 86-90. DOI:10.3969/j.issn.1000-3096.2007.11.017 |
| [7] |
SENDRA R, RODRIGO I, SALVADOR M L, et al. Characterization of pea histone deacetylases[J]. Plant Molecular Biology, 1988, 11(6): 857-866. DOI:10.1007/BF00019525 |
| [8] |
CHEN X S, DING A B, ZHONG X H. Functions and mechanisms of plant histone deacetylases[J]. Science China Life Sciences, 2020, 63(2): 206-216. DOI:10.1007/s11427-019-1587-x |
| [9] |
MA X J, LÜ S B, ZHANG C, et al. Histone deacetylases and their functions in plants[J]. Plant Cell Reports, 2013, 32(4): 465-478. DOI:10.1007/s00299-013-1393-6 |
| [10] |
ZHAO J H, ZHANG J X, ZHANG W, et al. Expression and functional analysis of the plant-specific histone deacetylase HDT701 in rice[J]. Frontiers in Plant Science, 2015, 5: 764. |
| [11] |
TONG B T, XIA D A, LÜ S B, et al. Cloning and expression analysis of PtHDT903, a HD2-type histone deacetylase gene in Populus trichocarpa[J]. Biotechnology & Biotechnological Equipment, 2018, 32(5): 1 098-1 104. |
| [12] |
CHEN C J, CHEN H, ZHANG Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1 194-1 202. DOI:10.1016/j.molp.2020.06.009 |
| [13] |
ERFIANTI T, WIJANARKA W, KUSUMANINGRUM H P. Molecular identification of inulinolytic yeast isolated from cherry fruit (Muntingia calabura L.)based on internal transcribed spacer sequence[J]. Journal of Physics: Conference Series, 2021, 1943: 012 059. DOI:10.1088/1742-6596/1943/1/012059 |
| [14] |
刘晓龙. 脱落酸(ABA)对水稻耐碱胁迫的诱抗效应及机理研究[D]. 长春: 中国科学院大学, 2019.
|
| [15] |
QIU Z B, GUO J L, ZHU A J, et al. Exogenous jasmonic acid can enhance tolerance of wheat seedlings to salt stress[J]. Ecotoxicology and Environmental Safety, 2014, 104: 202-208. DOI:10.1016/j.ecoenv.2014.03.014 |
| [16] |
倪子鑫, 武清扬, 杨云, 等. 茶树CsCCD基因家族全基因组鉴定及乌龙茶LED补光晾青下表达分析[J]. 生物工程学报, 2022, 38(1): 359-373. |
| [17] |
DEMETRIOU K, KAPAZOGLOU A, TONDELLI A, et al. Epigenetic chromatin modifiers in barley: I Cloning, mapping and expression analysis of the plant specific HD2 family of histone deacetylases from barley, during seed development and after hormonal treatment[J]. Physiologia Plantarum, 2009, 136(3): 358-368. DOI:10.1111/j.1399-3054.2009.01236.x |
| [18] |
HAN Z F, YU H M, ZHAO Z, et al. AtHD2D Gene plays a role in plant growth, development, and response to abiotic stresses in Arabidopsis thaliana[J]. Frontiers in Plant Science, 2016, 7: 310. |
| [19] |
TAHIR M S, TIAN L N. HD2-type histone deacetylases: unique regulators of plant development and stress responses[J]. Plant Cell Reports, 2021, 40(9): 1 603-1 615. DOI:10.1007/s00299-021-02688-3 |
| [20] |
朱圣庚, 徐长法. 生物化学[M]. 4版. 北京: 高等教育出版社, 2017.
|
| [21] |
DANGL M, BROSCH G, HAAS H, et al. Comparative analysis of HD2 type histone deacetylases in higher plants[J]. Planta, 2001, 213(2): 280-285. DOI:10.1007/s004250000506 |
| [22] |
SRIDHA S, WU K Q. Identification of AtHD2C as a novel regulator of abscisic acid responses in Arabidopsis[J]. The Plant Journal, 2006, 46(1): 124-133. DOI:10.1111/j.1365-313X.2006.02678.x |
| [23] |
MA X J, LIANG X Y, LÜ S B, et al. Histone deacetylase gene PtHDT902 modifies adventitious root formation and negatively regulates salt stress tolerance in poplar[J]. Plant Science, 2020, 290: 110 301. DOI:10.1016/j.plantsci.2019.110301 |
| [24] |
GAO M J, PARKIN I, LYDIATE D, et al. An auxin-responsive SCARECROW-like transcriptional activator interacts with histone deacetylase[J]. Plant Molecular Biology, 2004, 55(3): 417-431. DOI:10.1007/s11103-004-0892-9 |
| [25] |
HUANG L M, SUN Q W, QIN F J, et al. Down-regulation of a SILENT INFORMATION REGULATOR2-related histone deacetylase gene, OsSRT1, induces DNA fragmentation and cell death in rice[J]. Plant Physiology, 2007, 144(3): 1 508-1 519. DOI:10.1104/pp.107.099473 |

 2022, Vol. 42
2022, Vol. 42


