文章信息
- 江小羊, 曹喜兵, 赵振利, 邓敏捷, 范国强
- JIANG Xiaoyang, CAO Xibing, ZHAO Zhenli, DENG Minjie, FAN Guoqiang
- 泡桐E2基因家族分析及对丛枝植原体的响应
- Analysis of E2 gene family in Paulownia and their responses to witches' broom phytoplasmas
- 森林与环境学报,2022, 42(2): 174-183.
- Journal of Forest and Environment,2022, 42(2): 174-183.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2022.02.008
-
文章历史
- 收稿日期: 2021-10-07
- 修回日期: 2021-12-10
植物泛素/蛋白酶体系统是细胞内蛋白质降解的重要途径之一,在植物生长发育和免疫调节过程中起着重要作用[1-2]。泛素结合酶(ubiquitin-conjugating enzyme, UBC)E2是参与泛素化过程中重要的酶类之一,含有1个140~220氨基酸的UBC保守催化结构域,能够通过UBC结构域与E3的RING结构域相互作用并特异性识别底物蛋白,进而影响植物的形态建成[3]。目前,关于植物E2基因家族的功能研究已有较多报道: 如在拟南芥[Arabidopsis thaliana (L.) Heynh.]中,高表达的AtUBC1与AtUBC2可以抑制转基因拟南芥开花[4];AtUBC1-1与AtUBC2-1双突变体植株呈现莲座叶数量减少,叶片面积增大[5]; 在小麦(Triticum aestivum L.)中,MILLYARD et al[6]发现E2基因Tau4沉默后会导致小麦黑穗病(Tilletia foetida)发病延迟; 在烟草(Nicotiana tabacum L.)中, UNVER et al[7]阐明了UBC2基因沉默可导致转基因植株抗烟草花叶病毒水平提高; ZHOU et al[8]发现过表达大豆[Glycine max (Linn.)Merr.]GmUBC2能增强转基因植物的抗旱性;陈曙等[9]报道玉米(Zea maysLinn.)UBC亚家族UBC2基因受低氮胁迫的影响其表达量发生下调。然而关于E2基因家族在泡桐丛枝病(Paulownia witches′ broom, PaWB)发生过程中的作用尚未见报道。
泡桐为玄参科泡桐属落叶乔木,原产于我国,因其木材具有耐腐蚀、不易变形、燃点高等优良特性[10],且其对温度的适应范围也较大,近年来,在世界多个国家和地区均有引种和种植的报道[11-12]。然而,泡桐在生长过程中很容易遭受植原体感染产生丛枝病,进而导致植株生长缓慢,严重时会造成植株死亡[13-14]。本课题组前期研究发现,泡桐丛枝病发生与泛素介导的蛋白水解有关,且其表达量在植原体感染前后发生了显著变化[15-16]。为进一步研究E2基因家族在泡桐丛枝病发病中的作用,利用生物信息学方法对白花泡桐[Paulownia fortunei(Seem.) Hemsl.]E2基因家族进行鉴定,并详细分析了其家族成员的结构和功能以及对泡桐丛枝病的响应情况,以期为泡桐丛枝病的发病机理提供理论基础。
1 材料与方法 1.1 试验材料以河南农业大学林木生物技术实验室培养的二倍体白花泡桐健康组培苗(PF)和植原体感染的组培幼苗(PFI)为试验材料,将生长30 d的两种幼苗接种到350 mL含有1/2 MS的培养基的培养瓶中进行培养,组培室温度为(25±2) ℃,光照强度为130 μmol·m-2·s-1,光周期为16 h/8 h(光/暗),详细的条件参见FAN et al[17]方法。培养30 d后,剪取两种幼苗1.5 cm的顶芽,保存于-80 ℃冰箱中备用。
1.2 E2基因家族成员的鉴定与理化性质的分析首先采用两种方法进行E2基因家族成员的初期鉴定:一种是从NCBI数据库(https://www.ncbi.nlm.nih.gov/)中下载拟南芥和水稻(Oryza sativaL.)E2基因的蛋白序列,利用白花泡桐基因组数据建库,进行本地blast分析(E-value < 0.05),获得与已有拟南芥和水稻的同源蛋白(覆盖度>40%, 同源性>75%);另一种是从Pfam数据库(http://pfam.xfam.org/)中下载具有UBC结构域(PF00179)的隐马尔可夫模型(hidden markov model, HMM),使用hmmsearch程序在白花泡桐基因组的蛋白质数据库中进行检索(E-value < 0.05),获得含有UBC结构域的蛋白。然后将上述两种方法获取的蛋白成员取交集,进而确定为泡桐E2基因家族成员,最后,利用NCBI中保守域(conserved domain database, CDD)数据库进行确定E2基因家族的UBC结构域检测,剔出冗余序列,最终获得E2基因家族成员。进一步借助在线软件ProtParam(https://web.expasy.org/protparam/)进行蛋白质理化性质的预测[18]。
1.3 E2基因家族成员的染色体位置与命名在TBtools 1.049软件中同时输入泡桐基因组Chr.change.GFF(V1.0)[19]文件和上述鉴定到的E2基因家族成员对应的基因ID号,绘制出E2基因家族成员在染色体上的位置分布图,使用参数为默认值,最后,根据其在染色体Chr01~Chr20上的先后顺序进行命名。
1.4 E2基因家族系统发育树的构建为了解白花泡桐E2基因与其他植物E2基因之间的进化关系,使用邻接算法(neighbor-joining, NJ)构建E2基因家族系统发育进化树。步骤如下:首先将1.1已经鉴定到的白花泡桐与拟南芥、水稻的E2基因家族成员的蛋白序列合并整理在一个Fasta文件中;然后利用ClustalW 2.0软件对该文件进行多序列比对,得到多序列比对结果,设置参数Gap Opening为10、Extention为0.2、Delay Divergent Sequences为30、DNA Transition Weight为0.5;最后将多序列比对结果输入MEGA7.0软件进行系统发育进化树构建,采用1000次Bootstrap抽样进行误差检验,其他参数为默认设置。
1.5 E2基因家族成员的motif分析与基因结构分析利用MEME在线软件(http://meme-suite.org/tools/meme)对E2基因家族成员的蛋白序列进行motif分析,motif个数显示设置为10,然后采用TBtools软件进行motif的可视化分析。基因结构分析首先通过对基因组CDS文件进行搜索,得到E2基因的CDS序列;然后利用借助activestate软件(https://www.activestate.com/)得到基因全长文件;最后将全长文件和CDS文件放入在线软件GSDS(http://gsds.gao-lab.org/Gsds)中,绘制E2基因的基因结构图。
1.6 E2基因家族成员的启动子作用元件分析为研究白花泡桐E2基因启动子区域中与基因表达相关的特异性顺式作用元件,从泡桐基因组中提取E2基因家族成员启动子区域的DNA序列(即编码区上游2 000 bp),然后利用PlantCARE数据库(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测泡桐E2基因家族成员启动子区的顺式作用元件。
1.7 E2基因家族成员的基因重复事件与共线性分析为了解泡桐E2基因家族的重复事件及其与拟南芥的共线性,通过本地blast对白花泡桐自身蛋白序列、白花泡桐和拟南芥蛋白序列进行比对,分别得到白花泡桐物种内、白花泡桐和拟南芥物种间的基因对,使用KaKs_Calculator 2.0软件计算异义替换(non-synonymous substitution, Ka),同义替换(synonymous substitution, Ks)及Ka/Ks值,然后用借助TBtools软件来分析E2基因家族成员的基因重复事件以及拟南芥与泡桐的共线性关系。
1.8 白花泡桐E2基因表达量变化分析在前期建立的泡桐丛枝病发生模拟系统的基础上[16],从NCBI数据库中下载白花泡桐丛枝病幼苗在不同浓度甲基磺酸甲酯(methyl methanesulfonate, MMS)和利福平(rifampin, Rif),不同时间点处理下的转录组数据[19],登录号为SRR11787883、SRR11787892~SRR117-87905、SRR11787916、SRR11787919~SRR117-87945、SRR11787947、SRR11787949、SRR11-787960、SRR11787970、SRR11787971。通过分析E2基因家族成员在不同条件处理下与病苗之间表达量的变化,采用图 1的方案进行与泡桐丛枝病发病相关E2基因的筛选,同时利用pheatmap的R包进行热图的绘制。
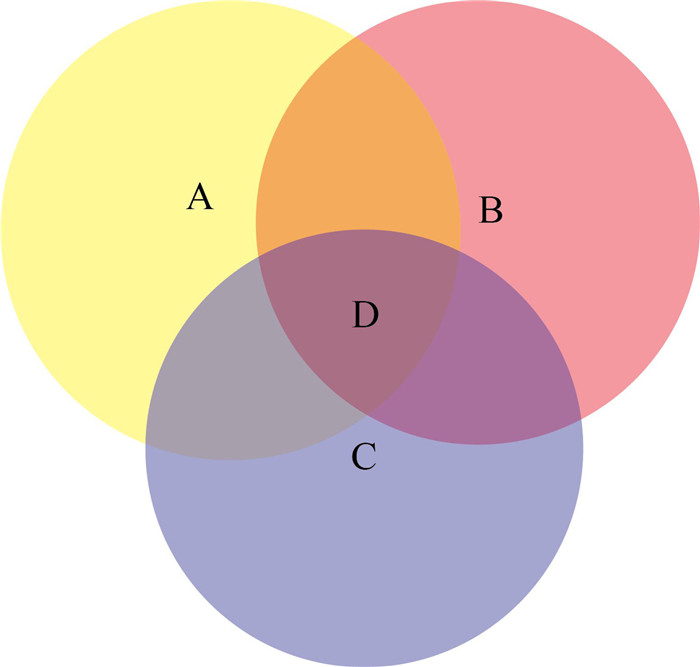
|
注:A.PF/PFI的差异基因; B.不同MMS处理的病苗/PFI的差异基因;C.不同Rif处理的病苗/PFI的差异基因;D.与泡桐丛枝病发病相关的E2基因。 Note: A.PF/PFI differential gene; B.diseased vaccines under different MMS/PFI differential gene; C.diseased vaccines under different Rif/PFI differential gene; D.E2 gene related to Paulownia witches′ broom. 图 1 与泡桐丛枝病发病相关的E2基因的筛选方案 Fig. 1 Screening scheme of E2 gene related to Paulownia witches′ broom |
采用植物RNA抽提试剂盒(北京爱普拜生物技术有限公司)提取PF和PFI幼苗1.5 cm顶芽的总RNA。随机挑选5个E2基因进行实时荧光定量聚合酶链式反应(real-time quantitative polymerase chain reaction, RT-qPCR)分析,聚合酶链式反应(polymerase chain reaction, PCR)反应采用Taq SYBR Green qPCR Premix试剂进行,PCR程序在美国Bio-Rad公司的CFX96TM Real-Time Syste荧光定量PCR仪上进行。PCR反应体系和扩增程序参照CAO et al[20]方法进行,每个样品3个生物学重复。使用PfActin作为内参基因,2-△△Ct法进行表达量的分析。利用GraphPad Prism 8进行数据统计分析并作图,引物序列见表 1。
| 基因Gene | 正向引物(5′-3′) Forward primer (5′-3′) | 反向引物(5′-3′) Reverse primer (5′-3′) |
| PfActin | AATGGAATCTGCTGGAAT | ACTGAGGACAATGTTACC |
| PfUBC13 | TTCCAAGTTACCATCCATT | TCCAAGCATATATTCCCATT |
| PfUBC44 | GTCGGTTAATCTGATGGT | ATTGTAAGAGGATAGTAGCC |
| PfUBC45 | GTAGAATCATCAAGGAGAC | TCATAAGGAGACTGTGTT |
| PfUBC46 | CAAATATCAACAGCAATG | ACTCTATCAGTCTTGTAC |
| PfUBC51 | AGTTCTAAGCATCATATCAATG | TCTTACGCAACGACTAAC |
在白花泡桐基因组中共鉴定到56个具有UBC保守结构域的E2基因家族成员,蛋白理化性质分析发现其成员间存在明显差异,氨基酸个数介于109~1 148个之间;蛋白的分子量介于12~127 kDa之间;等电点在4.35~9.29之间;其中E2基因家族中不稳定蛋白有10个(蛋白不稳定指数低于40),且56个成员均含有硫(S)原子,表明硫原子在白花泡桐E2基因家族中是不可缺少的组成原子(表 2)。这说明E2基因家族的不同成员间理化性质差异较大,推测其在生物体中具有不同的功能。
| 基因ID Gene ID | 基因名称Gene name | 染色体Chromosome | 氨基酸数Number of amino acids | 分子量Molecular weight/Da | 原子组成Atomic composition | 等电点PI | 不稳定指数Instbility index |
| Pfo01g009490.1 | PfUBC1 | Chr01 | 221 | 23 936.43 | C1071H1719N291O321S4 | 9.29 | 33.01 |
| Pfo01g009500.1 | PfUBC2 | Chr01 | 263 | 28 524.68 | C1270H2040N346O384S7 | 8.94 | 39.77 |
| Pfo02g001120.1 | PfUBC3 | Chr02 | 148 | 16 576.14 | C751H1163N193O214S8 | 7.72 | 48.09 |
| Pfo02g001780.1 | PfUBC4 | Chr02 | 183 | 20 846.46 | C924H1401N233O291S13 | 4.46 | 53.08 |
| Pfo02g004670.1 | PfUBC5 | Chr02 | 184 | 20 572.60 | C919H1447N243O274S9 | 7.74 | 56.11 |
| Pfo03g010980.1 | PfUBC6 | Chr03 | 146 | 16 602.87 | C734H1144N204O220S8 | 6.20 | 28.03 |
| Pfo03g015410.1 | PfUBC7 | Chr03 | 148 | 16 387.98 | C747H1159N189O211S7 | 8.42 | 39.37 |
| Pfo04g000520.1 | PfUBC8 | Chr04 | 1148 | 12 741.23 | C5530H8619N1525O1855S31 | 4.60 | 38.37 |
| Pfo04g013600.1 | PfUBC9 | Chr04 | 183 | 20 859.53 | C921H1400N232O291S15 | 4.43 | 57.77 |
| Pfo05g007040.1 | PfUBC10 | Chr05 | 185 | 20 267.98 | C903H1403N237O277S8 | 5.61 | 63.04 |
| Pfo05g009200.1 | PfUBC11 | Chr05 | 148 | 16 508.06 | C748H1159N191O214S8 | 7.72 | 44.40 |
| Pfo05g012640.1 | PfUBC12 | Chr05 | 191 | 21 506.17 | C943H1466N250O303S11 | 4.81 | 61.64 |
| Pfo06g004820.1 | PfUBC13 | Chr06 | 148 | 16 775.33 | C754H1168N202O214S9 | 8.36 | 50.31 |
| Pfo06g008250.1 | PfUBC14 | Chr06 | 180 | 20 594.72 | C935H1449N249O262S7 | 8.35 | 36.93 |
| Pfo06g010840.1 | PfUBC15 | Chr06 | 160 | 18 000.53 | C816H1240N222O228S6 | 8.41 | 51.24 |
| Pfo06g015030.1 | PfUBC16 | Chr06 | 187 | 21 257.60 | C934H1422N242O306S10 | 4.35 | 44.08 |
| Pfo07g006240.1 | PfUBC17 | Chr07 | 164 | 18 732.39 | C808H1285N231O251S15 | 5.12 | 38.56 |
| Pfo07g011950.1 | PfUBC18 | Chr07 | 183 | 20 862.41 | C920H1401N233O295S13 | 4.40 | 52.21 |
| Pfo07g015440.1 | PfUBC19 | Chr07 | 157 | 17 796.56 | C799H1248N218O225S9 | 8.70 | 52.85 |
| Pfo08g002840.1 | PfUBC20 | Chr08 | 192 | 21 756.34 | C962H1460N244O308S12 | 4.41 | 48.80 |
| Pfo08g003950.1 | PfUBC21 | Chr08 | 152 | 17 330.53 | C769H1185N215O231S6 | 5.37 | 73.04 |
| Pfo08g003960.1 | PfUBC22 | Chr08 | 152 | 17 334.53 | C768H1181N213O232S7 | 5.10 | 72.55 |
| Pfo08g008730.1 | PfUBC23 | Chr08 | 215 | 23 746.18 | C1076H1673N273O318S7 | 4.91 | 35.11 |
| Pfo08g014760.1 | PfUBC24 | Chr08 | 237 | 26 787.90 | C1205H1891N317O349S12 | 8.78 | 53.58 |
| Pfo09g005110.1 | PfUBC25 | Chr09 | 919 | 10 278.58 | C4568H7085N1195O1413S32 | 4.74 | 42.97 |
| Pfo09g010030.1 | PfUBC26 | Chr09 | 148 | 16 404.96 | C747H1158N188O213S7 | 7.72 | 42.11 |
| Pfo09g014020.1 | PfUBC27 | Chr09 | 151 | 16 947.57 | C767H1212N200O222S5 | 6.13 | 52.38 |
| Pfo09g016430.1 | PfUBC28 | Chr09 | 153 | 17 219.87 | C777H1232N206O225S5 | 6.74 | 48.04 |
| Pfo10g002850.1 | PfUBC29 | Chr10 | 133 | 14 727.16 | C683H1042N168O183S6 | 7.80 | 41.36 |
| Pfo11g000500.1 | PfUBC30 | Chr11 | 327 | 37 001.43 | C1668H2613N443O486S11 | 9.19 | 42.61 |
| Pfo11g000650.1 | PfUBC31 | Chr11 | 299 | 33 414.46 | C1477H2299N411O459S8 | 5.28 | 50.31 |
| Pfo11g011650.1 | PfUBC32 | Chr11 | 192 | 22 081.29 | C1003H1556N256O293S6 | 5.31 | 41.60 |
| Pfo11g012180.1 | PfUBC33 | Chr11 | 161 | 18 242.79 | C815H1258N224O237S8 | 8.34 | 65.92 |
| Pfo12g004390.1 | PfUBC34 | Chr12 | 190 | 20 883.57 | C925H1444N242O292S8 | 5.59 | 45.90 |
| Pfo12g007460.1 | PfUBC35 | Chr12 | 188 | 21 326.02 | C938H1450N254O293S11 | 5.54 | 68.24 |
| Pfo12g010490.1 | PfUBC36 | Chr12 | 148 | 16 508.06 | C748H1159N191O214S8 | 7.72 | 48.81 |
| Pfo12g012010.1 | PfUBC37 | Chr12 | 190 | 20 654.72 | C944H1445N247O263S6 | 8.87 | 39.22 |
| Pfo13g010340.1 | PfUBC38 | Chr13 | 152 | 17 337.56 | C773H1186N212O231S6 | 5.37 | 67.74 |
| Pfo13g010730.1 | PfUBC39 | Chr13 | 183 | 20 789.36 | C921H1410N240O289S10 | 4.69 | 44.67 |
| Pfo14g000360.1 | PfUBC40 | Chr14 | 236 | 27 122.19 | C1227H1897N317O353S12 | 7.61 | 58.50 |
| Pfo14g008270.1 | PfUBC41 | Chr14 | 189 | 21 409.71 | C955H1495N257O276S13 | 5.15 | 61.98 |
| Pfo15g006780.1 | PfUBC42 | Chr15 | 161 | 18 238.80 | C816H1258N224O236S8 | 8.34 | 66.65 |
| Pfo15g007700.1 | PfUBC43 | Chr15 | 207 | 23 776.56 | C1083H1687N283O301S9 | 8.72 | 44.25 |
| Pfo15g011040.1 | PfUBC44 | Chr15 | 160 | 18 104.44 | C813H1224N224O234S7 | 7.69 | 51.77 |
| Pfo16g005410.1 | PfUBC45 | Chr16 | 153 | 17 178.82 | C775H1229N205O225S5 | 6.74 | 50.28 |
| Pfo16g008650.1 | PfUBC46 | Chr16 | 148 | 16 495.02 | C747H1156N190O215S8 | 7.71 | 43.21 |
| Pfo16g011700.1 | PfUBC47 | Chr16 | 911 | 101 878.79 | C4508H7016N1186O1423S40 | 4.70 | 45.30 |
| Pfo18g002610.1 | PfUBC48 | Chr18 | 157 | 17 753.54 | C798H1247N217O224S9 | 8.70 | 49.99 |
| Pfo18g007500.1 | PfUBC49 | Chr18 | 183 | 20 824.36 | C917H1399N233O295S13 | 4.35 | 54.93 |
| Pfo19g000990.1 | PfUBC50 | Chr19 | 296 | 33 004.16 | C1466H2281N405O448S8 | 5.50 | 52.53 |
| Pfo20g003820.1 | PfUBC51 | Chr20 | 168 | 18 792.26 | C839H1293N223O254S7 | 5.05 | 53.48 |
| Pfoxxg012850.1 | PfUBC52 | tig00000997 | 148 | 16 461.00 | C744H1158N190O215S8 | 7.72 | 42.35 |
| Pfoxxg021220.1 | PfUBC53 | tig00016940 | 148 | 16 510.08 | C749H1161N189O215S8 | 7.71 | 43.95 |
| Pfoxxg030860.1 | PfUBC54 | wtdbg01169 | 151 | 16 956.49 | C764H1171N205O215S9 | 8.76 | 51.12 |
| Pfoxxg016110.1 | PfUBC55 | tig00015986 | 152 | 17 129.70 | C769H1206N206O223S7 | 5.79 | 43.16 |
| Pfoxxg004680.1 | PfUBC56 | Contig01576 | 109 | 12 752.55 | C568H874N156O165S7 | 6.50 | 32.29 |
为了解E2基因家族成员在泡桐染色体上的分布情况,利用TBtools将56个E2基因在染色体上的定位进行可视化分析。发现其分布在泡桐的19条染色体上(图 2),但在8号染色体上分布数量最多,为5个家族成员,6、9、11、12号染色体上各有4个家族成员,10、19、20号染色体上分布最少,为1个家族成员。然后根据E2基因家族成员在染色体从小到大顺序和染色体上的位置分布对其进行命名,把未定位到染色体上的家族成员放到最后进行命名(表 2),该结果为后期E2基因家族成员功能的研究奠定了基础。
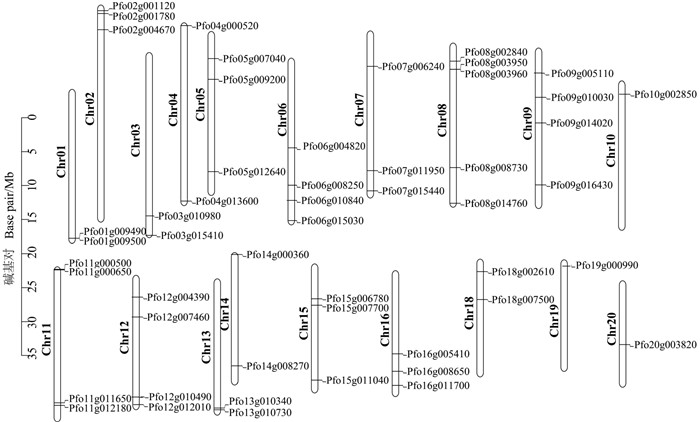
|
图 2 白花泡桐家族E2基因家族成员在染色体上的分布 Fig. 2 Distribution of P. fortunei E2 gene family on different chromosomes |
将泡桐56个家族成员与拟南芥E2基因家族成员(AtUBCs)、水稻E2基因家族成员(OsUBCs)的蛋白序列进行了系统发育树的构建,发现泡桐与拟南芥的同源性较高(图 3)。随后根据拟南芥E2基因家族成员的分类进行白花泡桐家族成员(PfUBCs)的分类,发现泡桐E2基因在Ⅵ亚家族中含有成员数最多, 为11个,这与拟南芥中家族成员个数最多的亚家族一样[21];但在ⅩⅥ亚家族没有一个泡桐E2基因家族成员,可能是E2基因家族在进化过程中发生了一些适应性的变异。
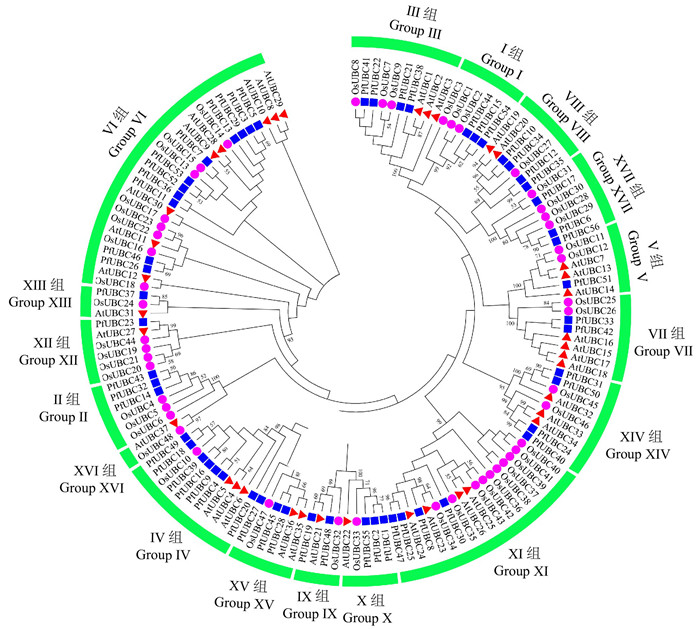
|
图 3 E2基因家族进化树 Fig. 3 Phylogenetic analysis of the E2 gene family |
为研究白花泡桐E2基因家族成员之间进化关系结构特点,借助MEME软件对PfUBCs蛋白序列进行分析。结果显示, PfUBCs中含有motif个数为2~7个,所有家族成员都有motif1,说明motif1是泡桐E2基因家族的保守基序,且较多的基因成员按照motif6、motif5、motif1、motif4、motif2、motif3的顺序排列(图 4),该结果可能与E2基因的特定功能有关。
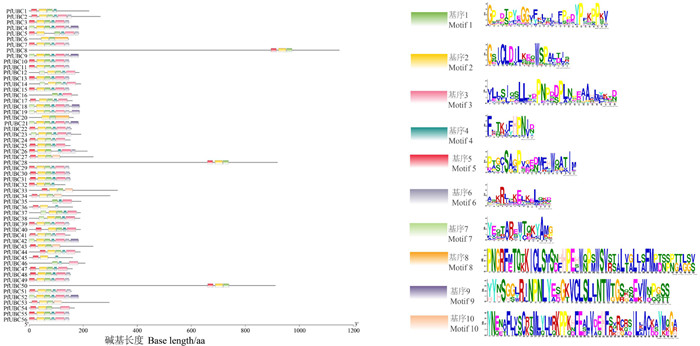
|
图 4 白花泡桐E2基因家族成员motif分析 Fig. 4 Motif analysis of P. fortunei E2 gene family members |
利用GSDS软件对56个PfUBCs进行基因结构分析,结果表明, E2家族各成员的内含子和外显子数量均存在较大差异[图 5(a)]。基因家族成员的内含子数量介于0~8个之间,含有3~5个内含子的基因数占83.9%,但PfUBC30和PfUBC47不含内含子。所有的基因家族成员均含有外显子,其数量介于4~9个之间,但有11个基因家族成员无非编译区(untranslated region, UTR)。该结果说明E2基因家族成员的功能在家族进化过程中发生了较大的变化。
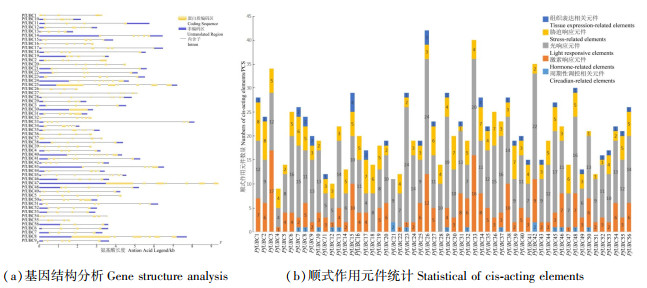
|
图 5 白花泡桐E2基因家族基因结构和顺式作用元件分析 Fig. 5 Analysis of P. fortunei E2 gene family gene structure and cis-acting elements |
利用Plant-CARE预测到了97种不同的顺式作用元件,根据其功能可将其分为5类:组织表达相关元件、胁迫响应元件、光响应元件、激素响应元件和周期性调控相关元件[图 5(b)]。56个E2基因家族成员均含有光响应原件,除PfUBC21无胁迫响应元件,PfUBC18和PfUBC28无激素响应元件外,其他成员均含有胁迫响应元件和激素响应元件。该结果说明光响应元件、胁迫和激素响应元件在E2基因转录过程中具有重要的功能。
2.6 E2基因家族成员的基因重复事件及共线性分析对PfUBCs进行基因对的计算以及共线性分析,发现E2基因家族成员中有45个基因参与了28个片段重复事件[图 6 (a)],说明片段重复在泡桐E2基因家族的进化中发挥了重要的作用。此外,泡桐基因对之间Ka/Ks值远小于1,说明该基因家族在进化过程中受基因纯化选择的作用。为了进一步研究E2基因家族的进化,对泡桐和拟南芥E2基因家族成员进行了共线性分析。结果显示,白花泡桐和拟南芥E2基因家族的相似性较高,两个物种间存在41个共线性事件[图 6 (b)],说明该基因家族在进化过程中是高度保守的,而有些PfUBCs与AtUBCs不存在共线性关系,推测产生该结果的原因是E2基因在后来的家族进化过程中产生了新的基因。
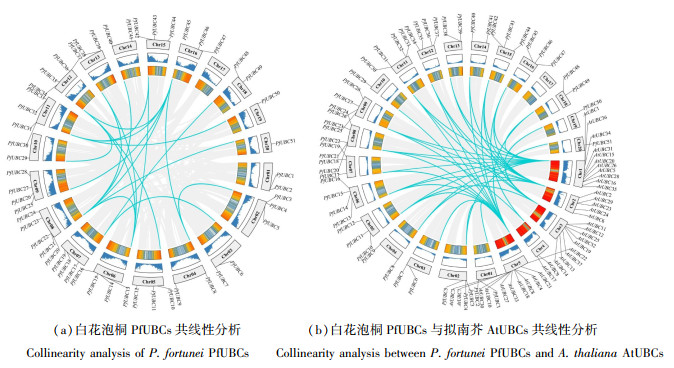
|
图 6 不同物种间共线性分析 Fig. 6 Collinearity analysis between different species |
通过转录组数据对病健苗和不同浓度试剂处理下病苗的泡桐E2基因进行了表达量的分析(图 7)。根据泡桐丛枝病发生模拟系统的变化趋势,在PF/PFI中发现28个E2基因上调,11个E2基因下调,其余的17个检测不到表达量。然后,在MMS试剂处理的比较组中(MMS20-10/PFI、MMS20-15/PFI、MMS20-30/PFI、MMS60-10/PFI、MMS60-20/PFI),发现56个E2基因中,同时上调的有4个(PfUBC5、PfUBC44、PfUBC45、PfUBC51),无同时下调基因;在Rif试剂处理的比较组中(Rif30-10/PFI、Rif30-15/PFI、Rif30-30/PFI、Rif100-10/PFI、Rif100-20/PFI),同时上调的有4个(PfUBC14、PfUBC44、PfUBC45、PfUBC51),无同时下调基因。为了排除不同试剂对病苗处理结果的影响,依据图 1的方案最终发现有3个共同基因(PfUBC44、PfUBC45和PfUBC51)在植原体感染后的病苗中表达量同时上升,鉴于适宜浓度的MMS和Rif试剂处理能使白花泡桐丛枝病病苗转变成健康形态[22-23],且其基因表达趋势在不同比较组中相同,因此推测PfUBC44、PfUBC45和PfUBC51可能与泡桐丛枝病发生有关。
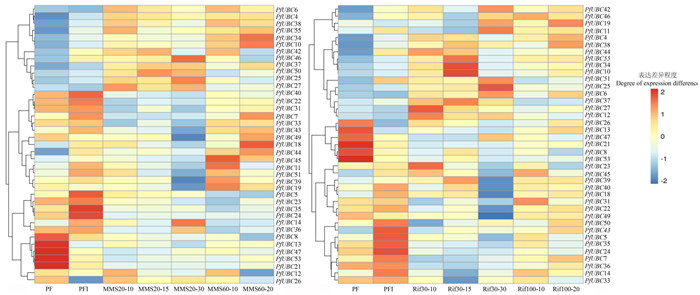
|
注:高表达水平为红色,低表达水平为蓝色。 Note: high expression levels are shown in red, and low expression levels are shown in blue. 图 7 不同试剂处理下E2基因家族成员的表达量热图 Fig. 7 Heat map of expression of E2 gene family members in different reagents treatment |
为了验证转录组数据中E2基因数据的准确性,从56个基因家族成员中随机挑选5个差异表达的E2基因进行RT-qPCR分析,结果表明(图 8),5个E2基因中,4个(PfUBC44、PfUBC45、PfUBC46和PfUBC51)基因的表达量在病苗中上升,1个(PfUBC13)基因的表达量在病苗中下降,且上述基因的表达趋势与转录组数据结果一致,该结果说明转录组测序中E2基因的数据可靠性较高。
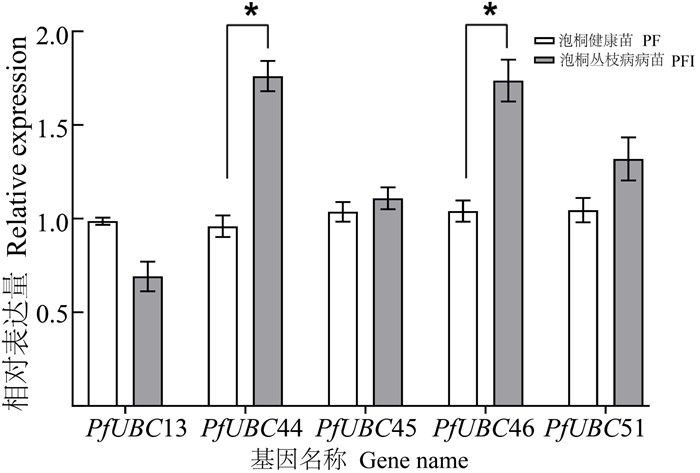
|
注:*表示PF与PFI之间差异显著(P < 0.05)。 Note: * indicates significant difference between PF and PFI (P < 0.05). 图 8 白花泡桐E2基因RT-qPCR验证 Fig. 8 Bar plot displaying the RT-qPCR validation of P. fortunei E2 genes |
E2泛素结合酶是泛素-蛋白酶体系统中关键酶,参与植物生长发育和免疫调节等多个生物学过程[1-2],但是其在泡桐丛枝病中的功能未见报道,鉴于本课题组前期发现泛素介导的蛋白表达量在植原体感染泡桐后上升[16],且泛素化结合酶E2基因在丛枝病研究中作用尚不清楚。因此,本研究在白花泡桐全基因组范围内进行了泛素结合酶E2基因家族的鉴定,共鉴定到56个E2基因家族成员,高于拟南芥的37个[21]、水稻的48个[24],低于玉米的75个[25],推测基因家族成员的个数可能与物种基因组大小有关。同时分析了泡桐与水稻、拟南芥的E2基因家族成员进化关系,发现泡桐与模式植物拟南芥的同源性较高,但基因亚家族分类中存在较大差异,说明E2基因家族在物种进化中发生了较大的分化。基因重复事件是基因在适应环境变化而自主发生的事件,研究发现白花泡桐56个E2基因家族成员中共发生了28个基因重复事件,并且通过计算Ka/Ks值发现该基因家族在进化过程中受基因纯化选择作用的影响。另外白花泡桐和拟南芥E2基因家族相似性较高,但有些PfUBCs与AtUBCs不存在共线性关系,推测产生该结果的原因是E2基因在后来的家族进化过程中产生了新的基因。因此,推测基因重复事件在E2基因家族扩张和功能多样化中有着重要的作用。
植物先天免疫反应与泛素化介导的蛋白降解密切相关[26]。在拟南芥中,WAND et al[27]报道AtUBC13基因的失活损害了含有核苷酸结合结构域和富含亮氨酸重复序列的蛋白介导的免疫反应,导致病原体的快速繁殖,证明该基因在效应蛋白激发的免疫反应(effector-triggered immunity, ETI)过程中起着重要作用;TUREK et al[28]发现拟南芥接种番茄紫丁香假单胞菌后,过表达AtUBC35植株增强了泛素化介导的免疫反应。在稻瘟菌侵染水稻过程中,林艺娟[29]发现OsUBC2基因是泛素化过程中的一个关键性酶,过表达OsUBC2基因可提高转基因水稻泛素化降解而抵抗病原菌的入侵。本研究中PfUBC44、PfUBC45和PfUBC51是响应泡桐丛枝病的关键基因,在植原体侵染后表达量升高,并且其分别与OsUBC2、AtUBC35和AtUBC13有较高的同源性。因此,推测泡桐丛枝病发生后,PfUBC44、PfUBC45和PfUBC51基因通过提高泛素化介导的蛋白降解以激活植物的免疫防御机制,从而响应了丛枝植原体的侵染。
综上所述,文中利用生物信息学方法从全基因组水平上对白花泡桐E2基因家族成员进行鉴定及分析,共鉴定到56个泛素化结合酶E2基因,并结合丛枝病发生过程中E2基因表达量的变化分析,最终发现PfUBC44、PfUBC45和PfUBC51基因与泡桐丛枝病发生密切相关,为研究白花泡桐E2基因家族在泡桐丛枝病发生过程中的作用提供了候选基因,其具体的功能可进一步通过转基因等实验进行验证。
| [1] |
陈默, 于丽杰, 金晓霞, 等. 植物泛素/26S蛋白酶体途径的研究进展[J]. 中国生物工程杂志, 2014, 34(4): 118-126. |
| [2] |
宋素胜, 谢道昕. 泛素蛋白酶体途径及其对植物生长发育的调控[J]. 植物学通报, 2006, 23(5): 564-577. |
| [3] |
PRUNEDA J N, STOLL K E, BOLTON L J, et al. Ubiquitin in motion: structural studies of the ubiquitin-conjugating enzyme-ubiquitin conjugate[J]. Biochemistry, 2011, 50(10): 1 624-1 633. DOI:10.1021/bi101913m |
| [4] |
GU X F, JIANG D H, WANG Y Q, et al. Repression of the floral transition via histone H2B monoubiquitination[J]. The Plant Journal, 2009, 57(3): 522-533. DOI:10.1111/j.1365-313X.2008.03709.x |
| [5] |
XU L, MÉNARD R, BERR A, et al. The E2 ubiquitin-conjugating enzymes, AtUBC1 and AtUBC2, play redundant roles and are involved in activation of FLC expression and repression of flowering in Arabidopsis thaliana[J]. The Plant Journal, 2009, 57(2): 279-288. DOI:10.1111/j.1365-313X.2008.03684.x |
| [6] |
MILLYARD L, LEE J, ZHANG C, et al. The ubiquitin conjugating enzyme, TaU4 regulates wheat defence against the phytopathogen zymoseptoria tritici[J]. Scientific Reports, 2016, 6: 35 683. DOI:10.1038/srep35683 |
| [7] |
UNVER T, TURKTAS M, BUDAK H. In planta evidence for the involvement of a ubiquitin conjugating enzyme (UBC E2 clade) in negative regulation of disease resistance[J]. Plant Molecular Biology Reporter, 2013, 31(2): 323-334. DOI:10.1007/s11105-012-0502-y |
| [8] |
ZHOU G A, CHANG R Z, QIU L J. Overexpression of soybean ubiquitin-conjugating enzyme gene GmUBC2 confers enhanced drought and salt tolerance through modulating abiotic stress-responsive gene expression in Arabidopsis[J]. Plant Molecular Biology, 2010, 72(4/5): 357-367. |
| [9] |
陈曙, 赵秋芳, 陈宏良, 等. 玉米泛素结合酶基因家族分析及低氮胁迫下亚家族UBC2的表达分析[J]. 热带作物学报, 2020, 41(2): 305-314. |
| [10] |
YADAV N K, VAIDYA B N, HENDERSON K, et al. A review of Paulownia biotechnology: a short rotation, fast growing multipurpose bioenergy tree[J]. American Journal of Plant Sciences, 2013, 4(11): 2 070-2 082. DOI:10.4236/ajps.2013.411259 |
| [11] |
蒋建平, 苌哲新, 舒寿兰, 等. 泡桐栽培学[M]. 北京: 中国林业出版社, 1990.
|
| [12] |
WANG Z, LIU W S, FAN G Q, et al. Quantitative proteome-level analysis of Paulownia witches' broom disease with methyl methane sulfonate assistance reveals diverse metabolic changes during the infection and recovery processes[J]. Peer J, 2017, 5: e3495. DOI:10.7717/peerj.3495 |
| [13] |
王洁, 田国忠, 徐启聪, 等. 泡桐丛枝病病树周围几种植物上植原体的分子检测[J]. 中国农业科学, 2010, 43(2): 304-312. |
| [14] |
GRIFFITHS H M, SINCLAIR W A, SMART C D, et al. The phytoplasma associated with ash yellows and lilac witches'broom: 'Candidatus phytoplasma fraxini'[J]. International Journal of Systematic and Evolutionary Microbiology, 1999, 49(4): 1 605-1 614. DOI:10.1099/00207713-49-4-1605 |
| [15] |
李文杨, 李永生, 曹亚兵, 等. 泡桐丛枝病相关lncRNAs的表达和调控[J]. 森林与环境学报, 2019, 39(2): 181-187. |
| [16] |
王哲. 白花泡桐丛枝病发生相关ceRNA研究[D]. 郑州: 河南农业大学, 2020.
|
| [17] |
FAN G Q, CAO X B, NIU S Y, et al. Transcriptome, microRNA, and degradome analyses of the gene expression of Paulownia with phytoplamsa[J]. BMC Genomics, 2015, 16: 896. DOI:10.1186/s12864-015-2074-3 |
| [18] |
罗玛妮娅, 王毅, 郑元, 等. 牛樟芝Zn(Ⅱ)2Cys6转录因子的全基因组鉴定与分析[J]. 森林与环境学报, 2021, 41(2): 157-163. |
| [19] |
CAO Y B, SUN G L, ZHAI X Q, et al. Genomic insights into the fast growth of paulownias and the formation of Paulownia witches' broom[J]. Molecular Plant, 2021, 14(10): 1 668-1 682. DOI:10.1016/j.molp.2021.06.021 |
| [20] |
CAO X B, ZHAI X Q, ZHANG Y F, et al. Comparative analysis of microRNA expression in three Paulownia species with phytoplasma infection[J]. Forests, 2018, 9(6): 302. DOI:10.3390/f9060302 |
| [21] |
KRAFT E, STONE S L, MA L G, et al. Genome analysis and functional characterization of the E2 and RING-type E3 ligase ubiquitination enzymes of Arabidopsis[J]. Plant Physiology, 2005, 139(4): 1 597-1 611. DOI:10.1104/pp.105.067983 |
| [22] |
翟晓巧, 曹喜兵, 范国强. 甲基磺酸甲酯处理的豫杂一号泡桐丛枝病幼苗的生长及SSR分析[J]. 林业科学, 2010, 46(12): 176-181. |
| [23] |
范国强, 张变莉, 翟晓巧, 等. 利福平对泡桐丛枝病幼苗形态和内源植物激素变化的影响[J]. 河南农业大学学报, 2007, 41(4): 387-390, 395. |
| [24] |
BAE H, KIM W T. Classification and interaction modes of 40 rice E2 ubiquitin-conjugating enzymes with 17 rice ARM-U-box E3 ubiquitin ligases[J]. Biochemical and Biophysical Research Communications, 2014, 444(4): 575-580. |
| [25] |
JUE D W, SANG X L, LU S Q, et al. Genome-wide identification, phylogenetic and expression analyses of the ubiquitin-conjugating enzyme gene family in maize[J]. PLoS One, 2015, 10(11): e0143488. |
| [26] |
XIE D X, FEYS B F, JAMES S, et al. COI1:an Arabidopsis gene required for jasmonate-regulated defense and fertility[J]. Science, 1998, 280(5 366): 1 091-1 094. |
| [27] |
WANG L P, WEN R, WANG J H, et al. Arabidopsis UBC13 differentially regulates two programmed cell death pathways in responses to pathogen and low-temperature stress[J]. New Phytologist, 2019, 221(2): 919-934. |
| [28] |
TUREK I, TISCHER N, LASSIG R, et al. Multi-tiered pairing selectivity between E2 ubiquitin-conjugating enzymes and E3 ligases[J]. Journal of Biological Chemistry, 2018, 293(42): 16 324-16 336. |
| [29] |
林艺娟. OsRBCS和OsUBC2在水稻抗病防御中的作用机制[D]. 福州: 福建农林大学, 2014.
|

 2022, Vol. 42
2022, Vol. 42


