文章信息
- 谢德金, 任可, 陈凌艳, 何天友, 荣俊冬, 郑郁善
- XIE Dejin, REN Ke, CHEN Lingyan, HE Tianyou, RONG Jundong, ZHENG Yushan
- 草珊瑚不同光质条件下RT-qPCR内参基因筛选与验证
- Selection and validation of internal reference genes for RT-qPCR in Sarcandra glabra exposed to different LED lights
- 森林与环境学报,2021, 41(5): 536-544.
- Journal of Forest and Environment,2021, 41(5): 536-544.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2021.05.012
-
文章历史
- 收稿日期: 2021-06-05
- 修回日期: 2021-08-10
2. 福建农林大学园林学院, 福建 福州 350002
2. College of Landscape, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
内参基因是生物组织中能够稳定表达,且不受外界生物和非生物胁迫影响的一类管家基因,如:甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、18S/25S核糖体(18S/25S ribosomal RNA,18S/25S rRNA)、泛素蛋白(ubiquitin,UBQ)、β-微管蛋白(β-tubulin,TUB)和激动蛋白(actin,ACT)等。目前,许多物种在不同发育阶段、不同组织部位和各种胁迫条件下,已被筛选出各自合适的内参基因,例如:在拟南芥缺铁条件下,SAND家族蛋白(SAND family protein,SAND)、有丝分裂蛋白YLS8(mitosis protein YLS8,YLS8)和TIP41-like家族蛋白(TIP41-like family protein,TIP41-like)被选为最适的内参基因,且3个基因在真核生物中都是保守的[1]。在对红薯的各类非生物胁迫中,内参基因L25核糖体蛋白(L25 ribosomal protein,L25)、延伸因子1α(elongation factor 1 α,ef1α)和泛素结合酶E2(ubiquitin-conjugating enzyme E2,Ntubc2)的稳定性最好[2]。在对番茄的内参基因筛选中,GAPDH、UBQ、ACT和尿苷酸激酶(uridylate kinase,UK)基因在所有组织中的稳定性最高[3]。除此之外,药用植物内参基因筛选的研究也相继报道。杨阳等[4]以不同发育时期和茉莉酸甲酯处理的多花黄精的块茎为材料,对8个候选内参基因进行最优筛选,结果表明β微管蛋白-泛素结合酶-E2-10(β-tubulin-ubiquitin-conjugating enzyme-E2-10,UBQ-E2-10)和ef1α2的稳定性最好。李娟等[5]选用不同生长年限和不同组织部位的远志为试验材料,对8个候选内参基因进行筛选,经过比对分析,内参基因ef1α的表达最稳定。叶友杰等[6]的研究结果表明,GAPDH+UBQ的双内参组合能更加准确地分析目的基因在巴戟天不同组织中的相对表达量,而GAPDH+ACT的内参组合对目的基因在不同处理叶中的相对表达量的分析可靠性更高。乔永刚等[7]以不同温度处理的药用蒲公英为研究对象,通过对10个不同的候选内参基因的分析评价,其中18 S rRNA和GAPDH是最适合的内参基因。王浩等[8]选取5个常用的候选内参基因,并验证其在何首乌不同组织中的可靠性,最终选择丝氨酸苏氨酸磷酸化酶(serinethreonine-protein phosphatase,PP2A)作为最佳内参基因。杨林林等[9]选取5个常用的候选内参基因,验证其在狭叶柴胡的不同组织的稳定性,其中TUB的稳定性最好,被选为最优内参基因。筛选不同物种的不同组织或不同环境条件下的最优内参基因,并且结合实时荧光定量PCR(real-time quantitative PCR,RT-qPCR)方法能够更加准确和可靠地对组织中目的基因表达量的变化得出较为准确的结果。
草珊瑚(Sarcandra glabra)是金栗兰科草珊瑚属植物,为常绿半灌木,以干燥全草入药,有清热凉血、活血消斑和祛风通络等功效[10]。目前,有关草珊瑚内参基因筛选的研究较少,而前人的研究多集中在采用DNA分子标记技术,开展草珊瑚种质资源遗传多样性方面的研究[11-13]。本试验以不同光质培养条件下的草珊瑚幼苗为材料,研究不同光质处理和不同组织部位中内参基因的表达稳定性,旨在筛选出最优内参基因。同时,采用筛选的最优内参基因结合RT-qPCR方法,测定不同光质培养条件下草珊瑚叶片中碳代谢关键酶基因的表达量变化。结果可以为分析校正目的基因在草珊瑚不同组织的表达模式提供准确性和可靠性的参考。
1 材料与方法 1.1 供试材料2019年5月15日,将草珊瑚幼苗置于人工气候培养室进行3种不同光质培养:白光(white LED light,WL)、红光(red LED light,RL)和蓝光(blue LED light,BL),光合有效辐射80 μmol·m-2·s-1,光周期比为16∶8,室温为25 ℃。待培养60 d后,分别选取每种光质下生长一致的草珊瑚幼苗3株,共9个样本(表 1)。将选取的幼苗用超纯水洗净擦干后,按不同光质处理和根茎叶3个不同组织部位分别采集混匀,液氮速冻并放置于-80 ℃超低温冰箱备用。
| 处理 Treatment |
组织部位 Tissue |
样本名称 Sample |
处理 Treatment |
组织部位 Tissue |
样本名称 Sample |
处理 Treatment |
组织部位 Tissue |
样本名称 Sample |
||
| 白光WL | 叶Leaf | WY | 红光RL | 叶Leaf | RY | 蓝光BL | 叶Leaf | BY | ||
| 茎Stem | WJ | 茎Stem | RJ | 茎Stem | BJ | |||||
| 根Root | WG | 根Root | RG | 根Root | BG |
采用传统CTAB法分别提取草珊瑚不同光质处理下根、茎和叶3个不同组织的总RNA,并测定其总RNA的浓度和完整性。将总RNA用反转录试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser(Takara,RR047A)合成cDNA第一链,测定其浓度后,分别等体积吸取9个样本的cDNA第一链原液并混合,将混合液以5倍梯度稀释的方式从高浓度向低浓度依次稀释,共形成5个不同浓度的cDNA混合液模板:600.00、120.00、24.00、4.80和0.96 ng·μL-1,最后将不同浓度的cDNA混合液模板放于-20 ℃备用。
1.2.2 内参基因的选择及qPCR特异性引物的设计参考相关文献和ICG网站(Internal Control Genes,http://icg.big.ac.cn)提供的其他物种内参基因信息,选择10个常用的内参基因作为候选基因:Actin、TUB、SAND、细胞色素P450(Cytochrome P450,CYP450)、UBQ、ef1α、CAC、泛素结合酶2(ubiquitin-conjugating enzyme2,UBC2)、GAPDH和泛素结合酶38(ubiquitin-conjugating enzyme38,UBC38)。根据草珊瑚的转录组数据和荧光定量引物设计原则,用Primer premier 5软件对选择的10个候选内参基因进行引物设计(表 2)。
| 基因Gene | 引物序列Primer sequence | 长度Length/bp | 扩增效率Efficiency/% |
| Actin | F:5′-TCATCGAAGTCCAAGGCAAC-3′;R:5′-ATTTGGCCGGTCGTGATTTG-3′ | 139 | 107.9 |
| GAPDH | F:5′-AATTGCTGGCAACTCCCTTG-3′;R:5′-AAGTCGCCAATTTCGGAACC-3′ | 144 | 100.6 |
| TUB | F:5′-ATGTTTCCGCACTCTCAAGC-3′;R:5′-AAGTTCACCGCAAGCTTTCG-3′ | 141 | 99.4 |
| SAND | F:5′-AGTGGATGCTACGGAGAAAGTG-3′;R:5′-AAAGCAAACCCTGACGCAAC-3′ | 147 | 105.8 |
| CYP450 | F:5′-AAAGAAGCGCAGTTGGGAAG-3′;R:5′-TTGCGTTTCACACCAATGGG-3′ | 82 | 102.5 |
| UBQ | F:5′-TTCTTTGAACGCGTCCTCAC-3′;R:5′-AACAGTGACGAAGGCACAAC-3′ | 111 | 108.9 |
| ef1α | F:5′-TTGCCTGCAAATTCGCTGAG-3′;R:5′-ACACACATTGGCTTGGATGG-3′ | 133 | 102.1 |
| CAC | F:5′-TCCGACAAATTGGAGGTTGC-3′;R:5′-TGCTGCTGACAACAATCACG-3′ | 75 | 100.9 |
| UBC2 | F:5′-TTTTTGGCCCTAGTCAGCTG-3′;R:5′-TGCAAATTCTGAGGCAGCAC-3′ | 106 | 101.8 |
| UBC38 | F:5′-AAATGAAGCTCCAGGATGCG-3′;R:5′-AGTGGTTCTGGCACAAAAGC-3′ | 112 | 100.7 |
在前期的研究中,3种光质培养条件下草珊瑚幼苗的生长状态和相关生理生化指标已经进行测定,包括可溶性糖、蔗糖、还原糖含量[14]。在本研究中,采用筛选的最优内参基因结合RT-qPCR方法,测定影响碳代谢关键酶基因表达量的变化,检验基因表达量的变化与相应的生理生化指标含量的变化是否一致。相关酶基因如下:1, 5-二磷酸核酮糖羧化酶/加氧酶(ribulose-1, 5-bisphosphate carboxylase/oxygenase,Rubisco),蔗糖合成酶(sucrose synthase,SS),细胞壁酸性转化酶(cell wall acid invertase,AI-cell wall)和液泡酸性转化酶(vacuolar acid invertase,AI-vacuolar),碱性转化酶(neutral invertase,NI-1和NI-2),蔗糖磷酸合成酶(sucrose phosphate synthase,SPS),这7个关键酶基因的引物序列经草珊瑚转录组数据筛选相关基因片段后,用Primer premier 5软件设计并送福州铂尚生物技术有限公司进行合成(表 3)。
| 基因Gene | 引物序列Primer sequence |
| Rubisco | F:5′-AGCTCGTTGTTCACATCACC-3′;R:5′-GCAAACACAAGTTCGCACTG-3′ |
| SS | F:5′-AATGGATCGGGTTCGCAATG-3′;R:5′-AGTCGCAAATGTGGGCAAAC-3′ |
| SPS | F:5′-AAGCTTGAGAAGGTGTTGCG-3′;R:5′-TTCCTGAGCAACAACGTTGC-3′ |
| NI-1 | F:5′-TCTTGGCCGACTTTGCTTTG-3′;R:5′-TGCAACCTCAACAGCCTTTG-3′ |
| NI-2 | F:5′-TCGCCTCGTTTTTCAAACCG-3′;R:5′-ATGCGGCTCGAATGAATTGC-3′ |
| AI-vacuolar | F:5′-TCGTGCCACTTAATGTTGGC-3′;R:5′-TTGTGCTGCAGTTGTAACCC-3′ |
| AI-cell wall | F:5′-TGGATGAATGAGTCGGATAGCG-3′;R:5′-TGGCCATTGCACAAGTTGTC-3′ |
扩增体系和反应程序按照Takara公司的TB Green Premix Ex Taq试剂盒的说明进行操作,每个样本设置3个平行试验,每个平行试验3个重复,另外每个候选内参基因再设置3个阴性对照,验证设计的内参基因引物有无引物二聚体或其他因素干扰。试验所用仪器为ABI公司的7500 Real Time PCR System。
1.2.5 数据处理与分析使用RefFinder(https://www.heartcure.com.au/reffinder/)和4种程序软件geNorm[15]、Bestkeeper[16]、NormFinder[17]和The comparative delta-Ct method[18]对草珊瑚所有候选的内参基因进行分析比较,从而筛选最佳内参基因。分析每个候选内参基因和在关键酶基因不同样本中扩增的循环阈值(cycle threshold,Ct),采用Ct(2-ΔΔCt)法计算关键酶基因在不同光质处理下草珊瑚叶片中的相对表达量。
2 结果与分析 2.1 样本的总RNA质量检测由图 1的琼脂糖凝胶电泳检测结果可知,不同光质下草珊瑚根、茎和叶3个组织部位提取的总RNA条带清晰,提取的总RNA质量较好,可以用于接下来的cDNA的合成,及候选内参基因RT-qPCR筛选与验证。
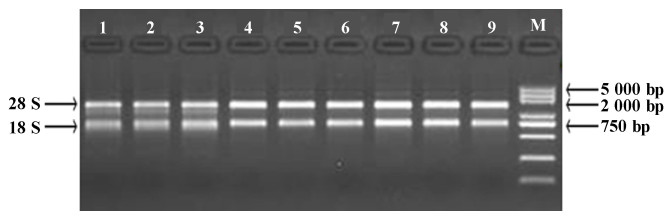
|
注:1.白光处理的叶片;2. 红光处理的叶片;3. 蓝光处理的叶片;4.白光处理的茎组织;5. 红光处理的茎组织;6. 蓝光处理的茎组织;7. 白光处理的根组织;8. 红光处理的根组织;9. 蓝光处理的根组织;M.Trans 2k plus marker。 Note: 1.WY; 2.RY; 3.BY; 4.WJ; 5.RJ; 6.BJ; 7.WG; 8.RG; 9.BG; M.Trans 2k plus marker. 图 1 不同光质处理下草珊瑚幼苗不同组织总RNA琼脂糖凝胶电泳检测 Fig. 1 Agarose gel analysis of total RNA extracted from different tissues of S. glabra seedlings exposed to different LED lights |
10个候选内参基因在不同浓度梯度的cDNA混合液中的Ct值平均分布在20~35个循环之间,其中UBC2基因Ct值较小,表达丰度最高(图 2)。由图 3的候选内参基因的溶解曲线可知所有的引物特异性较高。同时,表 2中列出的所有内参基因的的扩增效率均在90%~110%,相关系数均在0.99以上,这表明筛选的10条候选内参基因引物均符合RT-qPCR的试验要求,可以进行后续内参基因筛选试验。
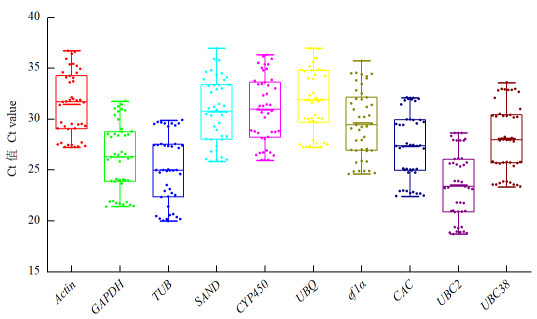
|
图 2 不同候选内参基因在草珊瑚幼苗样本中的Ct值 Fig. 2 Ct values of candidate reference genes in all S. glabra samples |
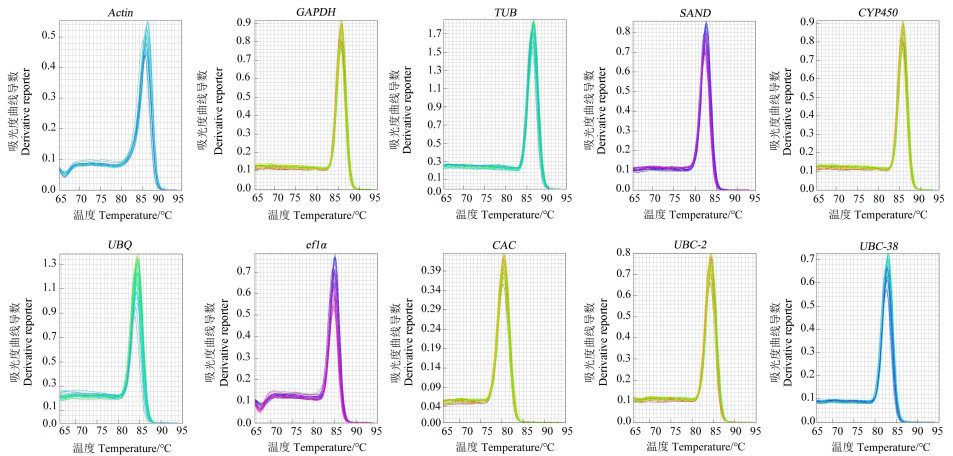
|
图 3 候选内参基因溶解曲线 Fig. 3 Melting curves of the candidate reference genes |
geNorm软件可以筛选任何样本的任意数量的内参基因,从而确定最佳的内参基因以及最合适内参基因数目,最佳的内参基因的确定主要依赖于软件算出的基因平均表达稳定值(M值),M值越小,内参基因稳定性越好。除了M值之外,该软件还有另外一个配对变异因子(V值),用Vn和Vn+1的比值(默认为0.15)来确定所需最适内参基因的数目,如果该值小于0.15,则最适合内参基因的数量为n(n≥2)个。如果该值大于0.15,最适内参基因的数量为n+1个[15]。由图 4(a)可知CAC的稳定性最好,UBC38和UBC2次之,SAND的稳定性最差。图 4(b)的配对变异因子V值中V2/V3的值小于0.15,软件推荐排在前两位的CAC和UBC38作为一对内参基因对目的基因的相对表达量进行校正。
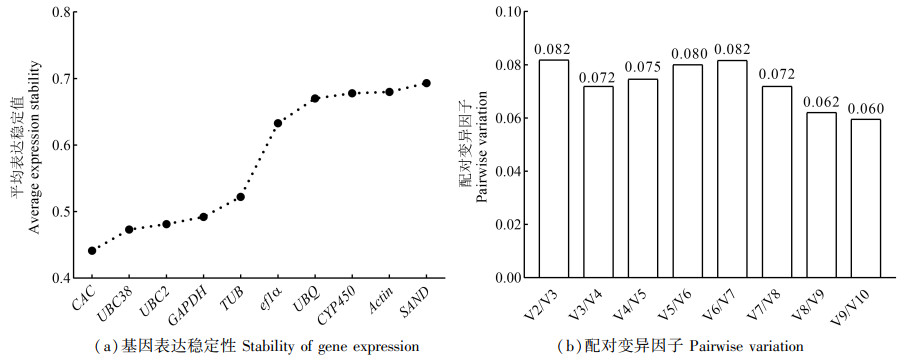
|
图 4 候选内参基因GeNorm软件分析结果 Fig. 4 Analysis results of the candidate reference genes calculated using GeNorm |
NormFinder软件计算原理与geNorm相似,都是通过获得内参基因表达稳定性M值,再根据M值的大小进行排序。该软件也是以M值越小,内参基因稳定性越好为筛选标准,但是不能给出最合适内参基因数目,只能筛选一个最佳内参基因[17]。图 5中显示内参基因CAC的表达稳定性最好,所以选择CAC作为最佳内参。
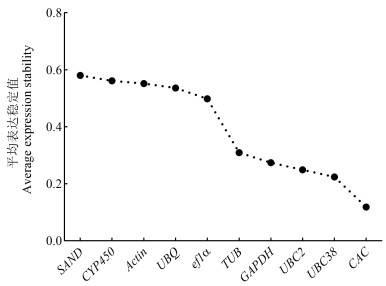
|
图 5 候选内参基因NormFinder软件分析结果 Fig. 5 Analysis results of the candidate reference genes calculated using NormFinder |
BestKeeper软件最多可以进行100个样本中的10个内参基因稳定性和10个目的基因表达量的筛选和比较,不需要对原始Ct值进行变换。通过软件可以得出每个内参基因的显著性水平(P值)、标准偏差(standard deviation,SD)、变异系数(variable coefficient,CV)和相关系数变量(correlation coefficient variable,r)等参数[16]。在进行比较之前,候选内参基因的显著性水平在P < 0.05时,内参基因的筛选才有统计学意义。内参基因的r值越大,CV和SD的值越小,内参基因稳定性越好。表 4中10个内参基因的P值都小于0.05,其中CAC内参基因的r值最高,表明其稳定性最高,除此之外,GAPDH、TUB、UBC2和UBC38的r值也较高,稳定性也较好。
| 基因 Gene |
标准偏差 SD |
变异系数 CV |
相关系数变量 r |
P值 P value |
基因 Gene |
标准偏差 SD |
变异系数 CV |
相关系数变量 r |
P值 P value |
|
| Actin | 2.664 | 8.463 | 0.987 | 0.001 | UBQ | 2.618 | 8.224 | 0.988 | 0.001 | |
| GAPDH | 2.819 | 10.725 | 0.996 | 0.001 | ef1α | 2.831 | 9.551 | 0.988 | 0.001 | |
| TUB | 2.844 | 11.372 | 0.996 | 0.001 | CAC | 2.803 | 10.254 | 0.998 | 0.001 | |
| SAND | 2.749 | 8.949 | 0.984 | 0.001 | UBC2 | 2.800 | 11.897 | 0.996 | 0.001 | |
| CYP450 | 2.824 | 9.130 | 0.987 | 0.001 | UBC38 | 2.809 | 8.463 | 0.996 | 0.001 |
method软件分析Delta-Ct(ΔCt)法筛选内参基因的依据是通过计算任意2个内参基因在所有样本之间的Ct值之差(ΔCt),然后求得这对内参基因的平均ΔCt值和标准偏差,最后将每一个内参基因的平均ΔCt值和标准偏差进行归类,并计算各内参基因的平均标准偏差,平均标准偏差越小,则内参基因稳定性越高[18]。图 6中显示了10个候选内参基因的平均标准偏差,其中CAC的值最小,稳定性最好,其次是UBC2,稳定性较差的是Actin基因。
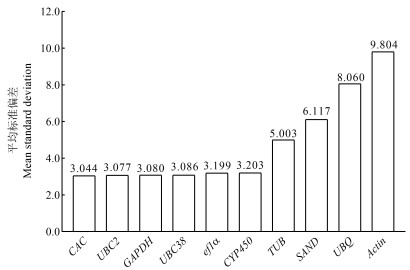
|
图 6 候选内参基因在Delta Ct中的排名和平均标准偏差 Fig. 6 Ranking of reference genes and their CV calculated using Delta Ct |
RefFinder是一款基于互联网技术评价和筛选内参基因的综合性应用程序,它将目前流行的4种筛选内参基因的软件重新整合并综合分析,从而对候选内参基因进行比较和排名。RefFinder程序会根据4种内参基因软件的筛选指标分别算出候选内参基因在每个软件的排名,之后给予每个内参基因相应的权重值并计算每个基因权重值的几何平均数,最终依据权重值的几何平均数的大小进行排名,值越小,内参基因稳定性越好[19]。表 5列出了候选内参基因在4种软件中的排名,并通过RefFinder程序整合计算出了最终每个候选内参基因的权重值的几何平均数大小,其中CAC的数值最小,稳定性最高,软件推荐CAC作为草珊瑚幼苗目标基因相对定量的内参基因。
| 排名Ranking | Delta Ct | BestKeeper | NormFinder | geNorm | 综合排名(值) Comprehensive ranking (value) |
| 1 | CAC | UBC2 | CAC | GAPDH/UBC38 | CAC (1.565) |
| 2 | UBC2 | CAC | UBC2 | UBC2 (2.000) | |
| 3 | GAPDH | UBC38 | GAPDH | CAC | GAPDH (2.449) |
| 4 | UBC38 | GAPDH | CYP450 | UBC2 | UBC38 (2.783) |
| 5 | ef1α | CYP450 | UBC38 | ef1α | CYP450 (5.180) |
| 6 | CYP450 | ef1α | ef1α | CYP450 | ef1α (5.477) |
| 7 | TUB | TUB | TUB | TUB | TUB (7.000) |
| 8 | SAND | SAND | SAND | SAND | SAND (8.000) |
| 9 | UBQ | UBQ | UBQ | UBQ | UBQ (9.000) |
| 10 | Actin | Actin | Actin | Actin | Actin (10.000) |
图 7(a)表示的是草珊瑚叶片中筛选的碳代谢部分关键酶基因的RT-qPCR结果,图 7(b)表示的是草珊瑚转录组数据库中碳代谢部分关键酶基因在叶片中的每千个碱基的转录每百万映射读取的碎片(fragments per kilobase of transcript per million mapped transcript,FPKM)值经过对数函数归一化后的结果。从图 7(a)和图 7(b)中可以看出,草珊瑚叶中各个关键酶基因在不同光质处理下的RT-qPCR的变化趋势与转录组的大致相似,以CAC为内参基因,可以对RT-qPCR的结果的准确性进行校正。
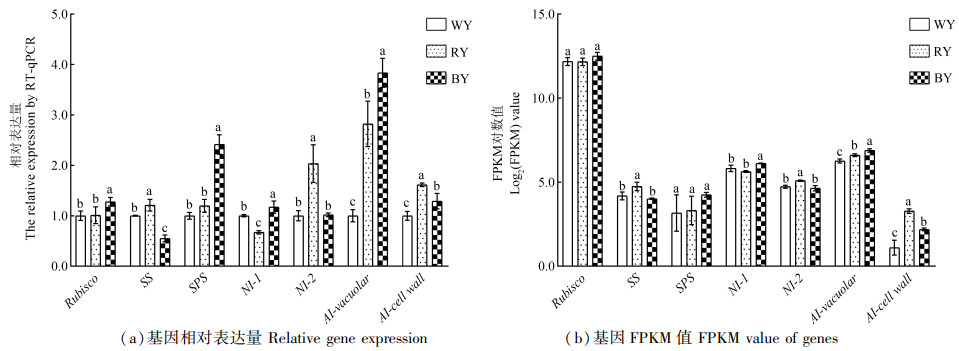
|
注:不同小写字母在不同处理间表示差异显著(P < 0.05)。 Note: different small letters indicate significant differences among the treatments(P < 0.05). 图 7 草珊瑚幼苗叶片碳代谢关键酶基因的相对表达量 Fig. 7 Relative expression of key enzymes of the carbon metabolism in leaves of S. glabra seedlings |
草珊瑚幼苗叶片碳代谢过程中关键酶基因的相对表达量结果表明:Rubisco是碳同化羧化阶段的关键酶,能够使CO2进行羧化反应。图 7(a)中蓝光处理下Rubisco基因的转录水平显著高于白光和红光处理,而红光和白光之间几乎没有差异;红光处理下SS基因的表达水平上最高,而在蓝光诱导下,SS基因的转录水平明显受到了抑制,导致其表达量明显低于白光处理;蓝光处理下SPS基因的表达量显著高于白光和红光处理,而白光和红光处理之间几乎没有差异;红光和蓝光处理下的NI-1和NI-2基因在叶中的相对表达量的结果呈现相反的趋势,蓝光处理下NI-1基因表达量显著增加,而该基因在红光诱导下的表达量显著降低;红光处理下的NI-2基因的表达量显著高于蓝光和白光处理,而蓝光和白光处理之间无显著差异。相较于白光处理,AI-vacuolar和AI-cell wall基因分别经过红光和蓝光处理后,两者的表达量都显著增加,但在蓝光处理下AI-vacuolar基因的表达量比红光处理的显著增加,而AI-cell wall基因在蓝光处理下的表达量相比红光处理显著降低。
3 讨论与结论RT-qPCR技术能够方便准确地对目的基因的表达谱进行绝对和相对定量。通过RT-qPCR技术获得准确的基因表达结果的前提条件是能够对目的基因的表达进行标准化处理。内参基因的选择是进行标准化处理的关键,在生物体中,一般选择保守的管家基因作为内参基因。筛选到的最优内参基因能准确分析不同组织或处理条件下目的基因的表达量变化。在某种处理条件下筛选的内参基因,不一定适用于其他生物样本或处理条件,具体的试验对象或条件下,都需要重新筛选最优的内参基因[20]。例如,内参基因ef1α、TUB、亲环蛋白(cyclophilin,CYP)和真核细胞翻译起始因子(eukaryotic initiation factor 4A,EIF4A)在玉米非生物胁迫(温度、盐和PEG)条件下和不同组织(根、茎和叶)中的稳定性最好,ACT2的稳定性较低[21]。相反,在玉米籽粒的发育过程中,内参基因TUB和ACT的稳定性最好[22]。在番茄的生长发育阶段,内参基因CAC、TIP41和SAND的稳定性最好[23]。不同非生物胁迫(氮饥饿、低温和低光照)条件下,番茄幼苗的最优内参基因有区别,如:在低光照条件下,GAPDH和磷酸甘油酸激酶(phosphoglycerate kinase,PGK)稳定性最好,而在氮饥饿、低温条件下,ef1α的稳定性最好[24]。
在对内参基因进行评价的4个统计算法中:geNorm推荐CAC和UBC38作为一对内参基因对目的基因的相对表达量进行校正,其中CAC的M值最小,UBC38次之,两者内参基因稳定性最好,另外配对变异因子V值中V2/V3的比值< 0.15, 所以最终软件选择这两个内参基因同时对目的基因的表达量进行校正。同理,基于M值的大小筛选标准,NormFinder软件也得出CAC为最佳内参基因。BestKeeper软件综合分析每个内参基因的P值、标准偏差和相关系数变量的参数,最终得出UBC38和CAC的SD和CV值都较小,且稳定性较高,适宜作为内参基因。同样,ΔCt法筛选到的最佳内参基因也是CAC。最后经过RefFinder的综合性分析,推荐CAC作为草珊瑚幼苗目标基因相对定量的内参基因。CAC基因是一种网格蛋白适配器复合物基因,也是一种比较保守的管家基因。在其他物种中,如:番茄[23]、盐角草[25]和西葫芦[26]的内参基因筛选中,CAC的稳定性都比较高,适合作为内参基因;相反,在侧柏中,CAC的稳定性比较低,并不适于侧柏中目的基因的表达量分析[27]。
碳氮代谢是植物最基本的代谢过程,两者一直处于动态的变化中,并且相辅相成。氮代谢需要碳代谢提供碳源和能量,而碳代谢需要氮代谢提供各种酶和光和色素[28]。在前期的研究中,相较于白光处理,蓝光培养条件下草珊瑚幼苗叶片的蔗糖和淀粉含量显著增加,红光处理则加速了蔗糖的分解和淀粉的积累[14]。SS酶能够可逆性的催化蔗糖和核苷二磷酸的反应从而生成核苷二磷酸葡萄糖和果糖,由此可知SS能够加速蔗糖的分解,进而参与植物的淀粉和纤维素的生物合成[29]。SPS是控制蔗糖生物合成代谢的关键酶,在植物叶中能够对碳的再分配起到重要的作用,其主要是调控淀粉和蔗糖在光合组织叶中的再分配,SPS酶活性的增加能够在一定程度上促进蔗糖的合成代谢加强,分解代谢降低[30-31]。图 7(a)中,蓝光能够使得草珊瑚幼苗叶片的SS基因的表达量下降和SPS基因表达量的增加,从而促使叶片中蔗糖的合成,降低蔗糖的分解,红光处理下SS基因的表达量升高,更有利于淀粉的积累,但会导致蔗糖的含量有所下降。转化酶能够不可逆的将蔗糖水解为葡萄糖和果糖,但图 7(a)中草珊瑚幼苗的4个转化酶基因NI-1、NI-2、AI-vacuolar和AI-cell wall在不同光质下的相对表达量的趋势不同,这也从侧面反应了转化酶基因表达网络的复杂性。结合RT-qPCR中碳代谢关键酶基因的相对表达量与蔗糖、淀粉等含量的测定结果可知两者的变化趋势基本一致,也能反映结果的可靠性。
综上所述,草珊瑚幼苗经过不同光质的诱导后,其根、茎和叶3种组织中,筛选的候选内参基因CAC稳定性最好,为后续目的基因在不同光质处理条件下的相对表达量分析提供相对准确的结果。
| [1] |
HAN B, YANG Z, SAMMA M K, et al. Systematic validation of candidate reference genes for qRT-PCR normalization under iron deficiency in Arabidopsis[J]. BioMetals, 2013, 26(3): 403-413. DOI:10.1007/s10534-013-9623-5 |
| [2] |
PARK S, KIM Y, JI C Y, et al. Stable internal reference genes for the normalization of real-time PCR in different sweetpotato cultivars subjected to abiotic stress conditions[J]. PloS One, 2012, 7(12): e51502. DOI:10.1371/journal.pone.0051502 |
| [3] |
MASCIA T, SANTOVITO E, GALLITELLI D, et al. Evaluation of reference genes for quantitative reverse-transcription polymerase chain reaction normalization in infected tomato plants[J]. Molecular Plant Pathology, 2010, 11(6): 805-816. |
| [4] |
杨阳, 叶碧欢, 宋其岩, 等. 多花黄精块茎发育和胁迫条件下qPCR内参基因的筛选与验证[J]. 中国中药杂志, 2020, 45(24): 5967-5975. |
| [5] |
李娟, 孔冉冉, 梅月菊, 等. 栽培远志qRT-PCR内参基因筛选与P450s基因的时空表达分析[J]. 中草药, 2019, 50(24): 6084-6090. DOI:10.7501/j.issn.0253-2670.2019.24.024 |
| [6] |
叶友杰, 谢德金, 杨德明, 等. 巴戟天实时荧光定量PCR内参基因的选择[J]. 中草药, 2020, 51(04): 1060-1068. |
| [7] |
乔永刚, 王勇飞, 曹亚萍, 等. 药用蒲公英低温和高温胁迫下内参基因筛选与相关基因表达分析[J]. 园艺学报, 2020, 47(6): 1153-1164. |
| [8] |
王浩, 蔡启忠, 刘露, 等. 何首乌实时荧光定量PCR内参基因筛选[J]. 中国中药杂志, 2021, 46(1): 80-85. |
| [9] |
杨林林, 孙卓, 杨利民, 等. 狭叶柴胡内参基因筛选及皂苷合成关键酶基因组织表达分析[J]. 中草药, 2018, 49(15): 3651-3658. DOI:10.7501/j.issn.0253-2670.2018.15.026 |
| [10] |
国家药典委员会编. 中华人民共和国药典[M]. 北京: 中国医药科技出版社, 2015.
|
| [11] |
魏艺聪, 陈莹, 牛广俊, 等. 不同居群草珊瑚的ITS序列分析及模式识别研究[J]. 中草药, 2014, 45(20): 2982-2986. DOI:10.7501/j.issn.0253-2670.2014.20.021 |
| [12] |
倪开诚, 闵芳, 郭卫东, 等. 采用ISSR分子标记进行草珊瑚8个种源的遗传多样性分析[J]. 中草药, 2008, 39(9): 1392-1396. DOI:10.3321/j.issn:0253-2670.2008.09.037 |
| [13] |
罗育, 黄春喜, 吴耀生, 等. 3种DNA分子标记法联合鉴别草珊瑚及其混伪品[J]. 中草药, 2020, 51(3): 733-740. |
| [14] |
谢德金, 李静文, 叶友杰, 等. 光质对草珊瑚幼苗生长及其生理生化基础的影响[J]. 草业学报, 2020, 29(8): 104-115. |
| [15] |
VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): 34. |
| [16] |
PFAFFL M W, TICHOPAD A, PRGOMET C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [17] |
ANDERSEN C, JENSEN J, ORNTOFT T. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64: 5245-5250. DOI:10.1158/0008-5472.CAN-04-0496 |
| [18] |
SILVER N, BEST S, JIANG J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Molecular Biology, 2006, 7: 33. DOI:10.1186/1471-2199-7-33 |
| [19] |
XIE F, XIAO P, CHEN D, et al. miRDeepFinder: a miRNA analysis tool for deep sequencing of plant small RNAs[J]. Plant Molecular Biology, 2012, 80(1): 75-84. DOI:10.1007/s11103-012-9885-2 |
| [20] |
RACHANA K E, RAJESH M K. Selection and validation of reference genes for gene expression normalization in coconut (Cocos nucifera L.) under biotic stress and hormone stimuli[J]. Plant Gene, 2019, 19: 100184. DOI:10.1016/j.plgene.2019.100184 |
| [21] |
LIN Y, ZHANG C, LAN H, et al. Validation of potential reference genes for qPCR in maize across abiotic stresses, hormone treatments, and tissue types[J]. PloS One, 2014, 9(5): e95445. DOI:10.1371/journal.pone.0095445 |
| [22] |
GALLI V, DA SILVA MESSIAS R, DOS ANJOS E SILVA S D, et al. Selection of reliable reference genes for quantitative real-time polymerase chain reaction studies in maize grains[J]. Plant Cell Reports, 2013, 32(12): 1869-1877. DOI:10.1007/s00299-013-1499-x |
| [23] |
EXPÓSITO-RODRÍGUEZ M, BORGES A A, BORGES-PÉREZ A, et al. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process[J]. BMC plant biology, 2008, 8: 131. DOI:10.1186/1471-2229-8-131 |
| [24] |
LOVDAL T, LILLO C. Reference gene selection for quantitative real-time PCR normalization in tomato subjected to nitrogen, cold, and light stress[J]. Analytical Biochemistry, 2009, 387(2): 238-242. DOI:10.1016/j.ab.2009.01.024 |
| [25] |
XIAO X, MA J, WANG J, et al. Validation of suitable reference genes for gene expression analysis in the halophyte Salicornia europaea by real-time quantitative PCR[J]. Frontiers in Plant Science, 2015, 5: 788. |
| [26] |
OBRERO Á, DIE J V, ROMÁN B, et al. Selection of reference genes for gene expression studies in zucchini (Cucurbita pepo) using qPCR[J]. Journal of Agricultural and Food Chemistry, 2011, 59(10): 5402-5411. DOI:10.1021/jf200689r |
| [27] |
CHANG E, SHI S, LIU J, et al. Selection of reference genes for quantitative gene expression studies in Platycladus orientalis(Cupressaceae) using real-time PCR[J]. PloS One, 2012, 7(3): e33278. DOI:10.1371/journal.pone.0033278 |
| [28] |
陆飞伍, 罗兴录, 李红雨, 等. 不同木薯品种叶片碳氮代谢与块根淀粉积累特性研究[J]. 中国农学通报, 2009, 25(10): 120-124. |
| [29] |
SCHMÖLZER K, GUTMANN A, DIRICKS M, et al. Sucrose synthase: a unique glycosyltransferase for biocatalytic glycosylation process development[J]. Biotechnology Advances, 2016, 34(2): 88-111. |
| [30] |
宁宇, 邓惠惠, 李清明, 等. 红蓝光质对芹菜碳氮代谢及其关键酶活性的影响[J]. 植物生理学报, 2015, 51(1): 112-118. |
| [31] |
HASHIDA Y, HIROSE T, OKAMURA M, et al. A reduction of sucrose phosphate synthase (SPS) activity affects sucrose/starch ratio in leaves but does not inhibit normal plant growth in rice[J]. Plant Science, 2016, 253: 40-49. |

 2021, Vol. 41
2021, Vol. 41


