文章信息
- 于瀛龙, 贺兴江, 周文才, 黎华君, 李应, 韦小平
- YU Yinglong, HE Xingjiang, ZHOU Wencai, LI Huajun, LI Ying, WEI Xiaoping
- 梵净山地区中华蜜蜂种群遗传分析
- Population genetic analysis of honeybee Apis cerana cerana in Fanjingshan
- 森林与环境学报,2021, 41(4): 425-430.
- Journal of Forest and Environment,2021, 41(4): 425-430.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2021.04.013
-
文章历史
- 收稿日期: 2021-04-26
- 修回日期: 2021-06-10
蜜蜂(Apis)是重要的传粉昆虫,对维持和促进植物的遗传多样性至关重要,蜜蜂还有保护濒危植物物种和帮助生态系统恢复等重要功能,在许多自然生态系统中不可或缺[1]。中华蜜蜂(Apis cerana cerana Fabricius)自然分布于中国大部分地区,具有抗螨、耐寒、善于利用零星蜜粉源等优良的生产性状[2]。但近年来,中华蜜蜂的自然分布面积缩小,种群规模减小,遗传多样性下降。其面临农药、病害、西方蜜蜂竞争、盲目引种、人为不当管理等多种因素的威胁[3],有效开展本土蜜蜂遗传资源的保护愈发重要。
自然保护区主要保护特定的濒危物种、亚种和自然栖息地[4]。现有保护区内物种资源研究主要集中在受保护的濒危动物和脊椎动物上,而对包括昆虫在内非濒危物种关注较少。贵州梵净山国家级自然保护区(保护区)是联合国教科文组织世界遗产地,是联合国“人与生物圈计划”项目的一部分,是濒危物种黔金丝猴(Rhinopithecus brelichi Thomas)的唯一栖息地,保护区内分布有梵净山冷杉(Abies fanjingshanensis W.L.Huang.)、珙桐(Davidia involucrate Baill)等濒危植物,植被覆盖率约96.5%,包括针叶林、阔叶林、竹林和灌木林[5]。而以梵净山为代表的自然保护区对本土的蜜蜂种群的保护效果,保护区内中华蜜蜂的遗传多样性程度和种群规模,保护区内的蜜蜂能否形成独特的种群仍然未知。因此,本研究利用15个微卫星分子遗传标记, 分析保护区内外的中华蜜蜂的遗传结构,阐明梵净山地区(涵盖保护区内外)中华蜜蜂的遗传多样性水平、有效种群大小和遗传分化情况,明确自然保护区在蜜蜂遗传资源保护上的作用,为本土蜜蜂种质资源的保护开展提供重要依据。
1 研究区概况与研究方法 1.1 研究区概况在保护区内共采集了107群中华蜜蜂样本(表 1),主要用于分析保护区内蜜蜂遗传多样性和种群大小。保护区内蜜蜂生存状况良好,较少有人为干扰。所有样本均取自土法圆桶饲养蜂群,蜂群为保护区原住民野外放置空蜂箱收蜂而来,没有蜂王、蜂群的外来引入和人工育王行为。样本采集于2020年3月至5月,涵盖了在保护区管理局登记的原住民的所有蜂群。对保护区周边江口、印江、松桃县3个县域的蜜蜂进行了采集与分析,此样本代表保护区外的中华蜜蜂,主要用于分析保护区内外中华蜜蜂遗传分化水平。保护区外与保护区内的采集地点之间的距离为8.93~97.96 km,样品为当地活框饲养、土法饲养的蜂群,蜂群来源多为野外自然收蜂。部分蜂场(15号蜂场2017年从贵州遵义购买蜂群20群,2019年已全部淘汰;20号蜂场2018、2019年从四川、江西各购买两只蜂王,采样时其情况已不详,取样蜂群为本场自然收蜂蜂群;21号蜂场2017年从福建购买蜂群30群,2019年已全部换王淘汰)存在购买外地蜂群的情况,但引入的蜂群多数不适合当地环境,早已自然淘汰。部分蜂场(13号、20号、21号蜂场)存在人工育王现象,为排除人工育王操作对遗传分化结果的干扰,人工育王的蜂群仅保留1群用作分析。实际使用的蜂群样本数为90群。
| 序号 Order |
保护区内外 Inside or outside of the nature reserve |
样本数 Number of samples/colony |
纬度 Latitude |
经度 Longitude |
海拔 Altitude /m |
| 1 | 保护区内Inside | 10 | 27°54′54″N | 108°35′56″E | 1 023 |
| 2 | 保护区内Inside | 9 | 27°55′07″N | 108°36′03″E | 1 167 |
| 3 | 保护区内Inside | 4 | 27°55′56″N | 108°35′58″E | 864 |
| 4 | 保护区内Inside | 3 | 27°55′23″N | 108°36′60″E | 1 143 |
| 5 | 保护区内Inside | 2 | 27°55′03″N | 108°38′23″E | 1 207 |
| 6 | 保护区内Inside | 24 | 27°49′31″N | 108°36′31″E | 1 166 |
| 7 | 保护区内Inside | 12 | 27°50′46″N | 108°36′22″E | 890 |
| 8 | 保护区内Inside | 10 | 27°56′27″N | 108°38′33″E | 1 244 |
| 9 | 保护区内Inside | 13 | 27°56′27″N | 108°38′36″E | 1 175 |
| 10 | 保护区内Inside | 15 | 27°57′59″N | 108°37′52″E | 982 |
| 11 | 保护区内Inside | 5 | 27°49′58″N | 108°45′38″E | 790 |
| 12 | 保护区外Outside | 10 | 28°05′11″N | 108°46′29″E | 838 |
| 13 | 保护区外Outside | 2 | 28°02′22″N | 109°12′22″E | 654 |
| 14 | 保护区外Outside | 3 | 28°03′18″N | 109°12′20″E | 737 |
| 15 | 保护区外Outside | 15 | 27°59′31″N | 108°56′20″E | 516 |
| 16 | 保护区外Outside | 10 | 27°40′46″N | 108°46′06″E | 561 |
| 17 | 保护区外Outside | 10 | 27°44′49″N | 108°35′28″E | 563 |
| 18 | 保护区外Outside | 10 | 27°43′57″N | 108°36′46″E | 1 107 |
| 19 | 保护区外Outside | 16 | 27°56′41″N | 108°26′17″E | 668 |
| 20 | 保护区外Outside | 5 | 28°05′27″N | 108°29′00″E | 602 |
| 21 | 保护区外Outside | 9 | 27°05′29″N | 108°29′42″E | 548 |
在每个蜂群中取1只工蜂,用于分子遗传标记检测。利用UNIQ-10柱式试剂盒[生工生物工程(上海)股份有限公司]从工蜂胸部提取基因组。使用2×SanTaq PCR Mix试剂盒[生工生物工程(上海)股份有限公司]扩增UN244T、Ac-1、Ac-2、Ac-5、AC011、Ac-26、Ac-35、Ap085、AP189、AP313、AT004、AT185、BI314、K0715、UN117共15个微卫星位点[6-7]。扩增产物委托生工生物工程(上海)股份有限公司用ABI 3730xl自动测序仪进行毛细管电泳检测,GeneScanTM LIZR[500]作为内标,GeneMapper 4.0软件生成电泳数据。所获得原数据全部经人工校验,统一峰图读数准则,排除位点扩增中加A情况的影响,以保证数据的准确性。
1.3 数据分析在遗传多样性和有效种群大小分析中,使用Mstools计算期望杂合度(expected heterozygosity,He)、观察杂合度(observed heterozygosity,Ho)、多态信息含量(polymorphic information content,PIC)和等位基因数(allele number,Na),有效等位基因数(effective allele number,Ne)和香农指数(Shannon index,I)用GenAlEx 6.5计算[8]。Ne estimator 2.01用于研究有效种群大小,采用随机交配模型中的连锁不平衡方法,评判标准设定为0.05[9]。
在遗传分化分析中,利用R 4.0.5软件进行主成分判别分析(discriminant analysis of principal components,DAPC)[10],样本预先分组,保留主成分解释的80%变异用于判别分析,通过样本的空间分布来评价样本的遗传分化程度,引入外群四川阿坝蜜蜂样本以呈现二纬空间分布[6]。利用GenAlEx 6.5软件计算保护区内外中华蜜蜂的遗传分化系数(genetic differentiation coefficient,FST)、基因流(number of migrants,Nm)和Nei氏遗传距离。用Structure 2.3.4软件[11]分析样本的遗传结构分组情况,K值设为2~5,重复10次,采用混合模型,马尔可夫链重复次数设置为100 000,burn-in设置为10 000,原始结果利用CLUMPP 1.1.2软件汇总,最后利用Distruct 1.1软件呈现[12]。利用GenAlEx 6.5软件计算样本间的遗传分化程度与地理距离、海拔差异的相关性。
2 结果与分析 2.1 梵净山地区中华蜜蜂的遗传分化遗传结构分析显示,在分2~5组的假设下,保护区外中华蜜蜂样本(Fanjingshan exterior,FE)和保护区内中华蜜蜂样本(Fanjingshan interior,FI)均未出现遗传结构上明显的差异(图 1),说明没有遗传分化发生。
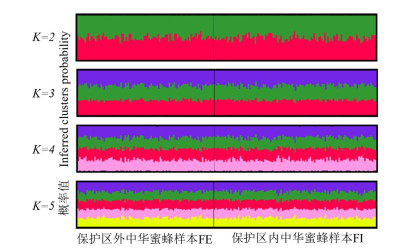
|
图 1 梵净山地区中华蜜蜂的遗传结构分析 Fig. 1 Structure analysis of Apis cerana cerana inside and outside Fanjingshan |
DAPC结果显示,保护区内外的中华蜜蜂在特征值最高的判别函数1上没有明显分离,仅在判别函数2上呈现轻微分离(图 2)。其中,判别函数1的特征值为3 137;判别函数2的特征值为95.95。
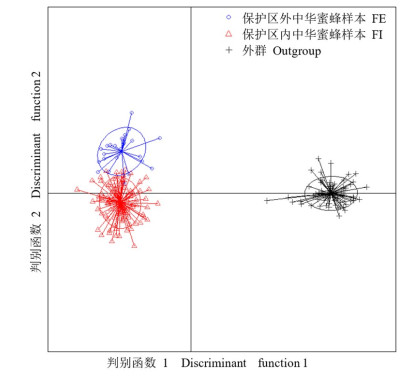
|
图 2 梵净山地区中华蜜蜂的主成分判别分析 Fig. 2 Discriminant analysis of principal component for Apis cerana cerana inside and outside Fanjingshan |
保护区内外的中华蜜蜂在15个微卫星位点上的FST在0.001~0.009之间,平均为0.004±0.003,属于弱分化或无分化状态。Nm在26.071~344.389之间,平均为120.491±104.995,基因流动情况明显。Nei氏遗传距离为0.017。相关性分析显示中华蜜蜂个体之间的遗传分化与地理距离之间没有显著的相关关系(R2=0.049 7),未因地理距离造成遗传分化(图 3)。同样,中华蜜蜂个体间的遗传分化程度与海拔高度之间没有显著的相关关系(R2=0.000 3),未因海拔差异造成遗传分化(图 4)。通过综合遗传结构分析、主成分判别分析、相关性分析和FST、Nm、Nei氏遗传距离的结果,保护区内的中华蜜蜂与保护区外中华蜜蜂之间没有发生遗传分化。
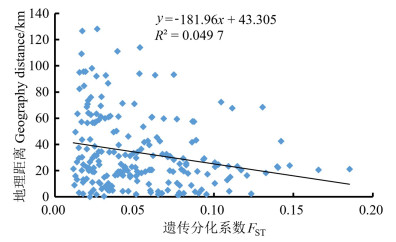
|
图 3 梵净山地区中华蜜蜂遗传分化与地理距离的相关性分析 Fig. 3 Mantel test for genetic distance FST and logarithm of geographic distance among Apis cerana cerana inside and outside Fanjingshan |
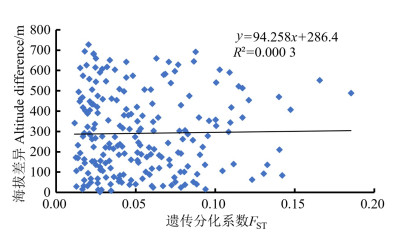
|
图 4 梵净山地区中华蜜蜂遗传分化与海拔差异的相关性分析 Fig. 4 Mantel test for genetic difference FST and logarithm of altitude difference among Apis cerana cerana inside and outside Fanjingshan |
保护区内中华蜜蜂的平均He为0.689 1±0.040 5、Ho为0.656 2±0.011 9、Na为9.80±4.81、Ne为3.99±0.50、I为1.508 6±0.133 3、PIC为0.647 3±0.170 9(表 2)。保护区内中华蜜蜂有效种群大小为561.2(248.0~无限)。保护区外中华蜜蜂的平均He为0.691 4±0.037 1、Ho为0.646 7±0.013 0、Na为9.67±4.51、Ne为3.92±0.47、I为1.512 4±0.127 7、PIC为0.647 4± 0.163 4(表 2)。保护区外中华蜜蜂有效种群大小为4 749.6(383.3~无限)。整体上,梵净山地区的中华蜜蜂平均He为0.691 0±0.038 7、Ho为0.651 9±0.008 8、Na为11.27±5.40、Ne为4.00±0.49、I为1.529 6±0.132 8、PIC值为0.650 2±0.167 1,有效种群大小为8 171.2(807.8~无限)。
| 位点 Locus |
期望杂合度 He |
多态信息含量 PIC |
观察杂合度 Ho |
等位基因数 Na |
有效等位基因数 Ne |
香农指数 I |
传分化系数 FST | 基因流 Nm |
|||||||||||
| FI | FE | FI | FE | FI | FE | FI | FE | FI | FE | FI | FE | ||||||||
| UN244T | 0.857 9 | 0.853 5 | 0.837 8 | 0.832 5 | 0.609 5 | 0.505 6 | 17 | 15 | 6.84 | 6.61 | 2.180 3 | 2.150 3 | 0.003 | 73.437 | |||||
| Ac-1 | 0.662 2 | 0.589 1 | 0.596 0 | 0.527 5 | 0.644 9 | 0.584 3 | 6 | 8 | 2.93 | 2.41 | 1.213 9 | 1.124 7 | 0.009 | 27.316 | |||||
| Ac-2 | 0.655 7 | 0.667 5 | 0.605 4 | 0.624 4 | 0.654 2 | 0.633 3 | 9 | 12 | 2.88 | 2.97 | 1.316 9 | 1.440 6 | 0.001 | 344.389 | |||||
| Ac-5 | 0.667 4 | 0.608 6 | 0.623 9 | 0.559 6 | 0.682 2 | 0.606 7 | 8 | 6 | 2.98 | 2.53 | 1.373 3 | 1.192 9 | 0.003 | 84.096 | |||||
| AC011 | 0.851 9 | 0.830 3 | 0.836 4 | 0.812 7 | 0.820 8 | 0.811 1 | 20 | 20 | 6.57 | 5.74 | 2.334 8 | 2.257 9 | 0.004 | 60.254 | |||||
| Ac-26 | 0.753 7 | 0.761 4 | 0.712 2 | 0.718 2 | 0.723 8 | 0.655 6 | 9 | 9 | 4.00 | 4.12 | 1.582 6 | 1.597 1 | 0.004 | 62.095 | |||||
| Ac-35 | 0.766 2 | 0.812 8 | 0.726 2 | 0.782 5 | 0.735 8 | 0.755 6 | 12 | 11 | 4.21 | 5.22 | 1.710 4 | 1.875 2 | 0.007 | 34.001 | |||||
| Ap085 | 0.874 7 | 0.869 1 | 0.857 7 | 0.849 3 | 0.867 9 | 0.800 0 | 15 | 12 | 7.73 | 7.37 | 2.247 5 | 2.120 5 | 0.001 | 174.567 | |||||
| AP189 | 0.829 0 | 0.826 1 | 0.801 2 | 0.797 3 | 0.691 6 | 0.744 4 | 7 | 9 | 5.72 | 5.60 | 1.801 9 | 1.830 8 | 0.006 | 40.336 | |||||
| AP313 | 0.565 3 | 0.635 5 | 0.537 5 | 0.608 7 | 0.523 8 | 0.611 1 | 13 | 14 | 2.29 | 2.72 | 1.328 3 | 1.556 1 | 0.003 | 98.325 | |||||
| AT004 | 0.605 4 | 0.580 5 | 0.536 7 | 0.511 3 | 0.607 5 | 0.606 7 | 9 | 7 | 2.52 | 2.37 | 1.176 0 | 1.097 8 | 0.001 | 306.916 | |||||
| AT185 | 0.792 8 | 0.783 4 | 0.757 7 | 0.745 9 | 0.800 0 | 0.806 8 | 8 | 8 | 4.74 | 4.52 | 1.714 6 | 1.676 5 | 0.002 | 129.268 | |||||
| BI314 | 0.681 8 | 0.673 2 | 0.641 7 | 0.633 1 | 0.700 9 | 0.622 2 | 8 | 8 | 3.11 | 3.03 | 1.409 3 | 1.402 0 | 0.001 | 268.890 | |||||
| K0715 | 0.444 2 | 0.494 9 | 0.348 5 | 0.376 5 | 0.481 1 | 0.556 8 | 3 | 3 | 1.79 | 1.97 | 0.655 6 | 0.710 5 | 0.009 | 26.071 | |||||
| UN117 | 0.327 7 | 0.385 6 | 0.290 2 | 0.331 2 | 0.299 1 | 0.400 0 | 3 | 3 | 1.48 | 1.62 | 0.583 6 | 0.653 5 | 0.003 | 77.406 | |||||
| 平均 Mean±SD |
0.689 1±0.040 5 | 0.691 4±0.037 1 | 0.647 3±0.170 9 | 0.647 4±0.163 4 | 0.656 2±0.011 9 | 0.646 7±0.013 0 | 9.80±4.81 | 9.67±4.51 | 3.99±0.50 | 3.92±0.47 | 1.508 6±0.133 3 | 1.512 4±0.127 7 | 0.004±0.003 | 120.491±104.995 | |||||
| 注:FI为保护区内中华蜜蜂样本;FE为保护区外中华蜜蜂样本。Note:FI represents samples inside the nature reserve, while FE represents samples outside the reserve. | |||||||||||||||||||
贵州梵净山国家级自然保护区内外的中华蜜蜂没有发生明显的种群遗传分化,保护区内暂未形成中华蜜蜂的独特种群。保护区内蜜蜂没有遗传分化、形成独特种群的主要原因是缺乏强烈的隔离作用,没有能够有效阻碍蜜蜂基因流的地理隔离、距离隔离等。
保护区的森林覆盖率为96.5%,植物种类约4 394种,而保护区外自然环境同样优越,保护区外的江口、印江、松桃3个县域森林覆盖率分别达到69.62%、60.23%、58.80%,且有严格限制重工业企业等的管控措施,这使得保护区内外均维持了良好的蜜粉源和营巢环境。蜜蜂具备广泛分布在保护区内外的环境基础。另一方面,蜜蜂的婚飞、分蜂和弃巢的最远距离分别为15、10和19.56 km[13-15],在自然环境相似、没有明显的地理屏障且蜜蜂蜂群能够连续分布的情况下,中华蜜蜂能维持较强的基因流,即使在650 km范围内也没有发现蜜蜂的遗传分化[16]。在本研究中,遗传分化系数与地理距离、海拔差异之间的相关性分析也证实距离、海拔没有造成有效的隔离,基因流维持在26.071~344.389之间,此种强度足以抑制遗传分化的产生[17]。因此,缺乏有效的隔离作用使得保护区内的中华蜜蜂没有与外界产生遗传分化。
根据本研究结果,与同类研究中的中华蜜蜂遗传多样性比较,梵净山地区中华蜜蜂处于较高的遗传多样性水平。在本研究与前人研究[18-22]中相同的8个微卫星位点上(表 3),梵净山保护区内中华蜜蜂在Ac-1、Ac-2、Ac-35上的Na超过以往研究;Ac-5的Na与前人研究中的最高值持平;Ac-26的Na为9,He为0.753 7,PIC为0.712 2,超过前人研究中的最高值;其余3个位点上,保护区内中华蜜蜂也维持了较高的遗传多样性。保护区外的中华蜜蜂在Ac-1、Ac-2、Ac-26位点上的多样性水平更为突出,Ac-1、Ac-2位点上的Na,Ac-26位点上的He、PIC均超过保护区内中华蜜蜂,为现有研究的最高值。
| 位点 Locus |
等位基因数Na | 期望杂合度He | 多态信息含量PIC | ||||||||
| 前人研究 Previous study |
梵净山内 Fanjignshan inside |
梵净山外 Fanjingshan outside |
前人研究 Previous study |
梵净山内 Fanjignshan inside |
梵净山外 Fanjingshan outside |
前人研究 Previous study |
梵净山内 Fanjignshan inside |
梵净山外 Fanjingshan outside |
|||
| AC011 | 8~45 | 20 | 20 | 0.641 8~0.962 6 | 0.851 9 | 0.830 3 | 0.602 2~0.961 6 | 0.836 4 | 0.812 7 | ||
| Ap085 | 2~37 | 15 | 12 | 0.142 7~0.935 6 | 0.874 7 | 0.869 1 | 0.131 9~0.932 2 | 0.857 7 | 0.849 3 | ||
| AP313 | 1~15 | 13 | 14 | 0.00~0.78 | 0.565 3 | 0.635 5 | 0.00~0.75 | 0.537 5 | 0.608 7 | ||
| Ac-1 | 3~5 | 6 | 8 | 0.55~0.67 | 0.662 2 | 0.589 1 | 0.45~0.61 | 0.596 0 | 0.527 5 | ||
| Ac-2 | 3~7 | 9 | 12 | 0.51~0.70 | 0.655 7 | 0.667 5 | 0.44~0.66 | 0.605 4 | 0.624 4 | ||
| Ac-5 | 4~8 | 8 | 6 | 0.44~0.79 | 0.667 4 | 0.608 6 | 0.41~0.74 | 0.623 9 | 0.559 6 | ||
| Ac-26 | 5~8 | 9 | 9 | 0.60~0.75 | 0.753 7 | 0.761 4 | 0.56~0.70 | 0.712 2 | 0.718 2 | ||
| Ac-35 | 2~10 | 12 | 11 | 0.30~0.84 | 0.766 2 | 0.812 8 | 0.26~0.82 | 0.726 2 | 0.782 5 | ||
梵净山地区中华蜜蜂的遗传多样性高,重要原因是蜜蜂以大种群形式存在。估算保护区内中华蜜蜂的有效种群大小为561.2,保护区外为4 749.6,整体计算梵净山地区中华蜜蜂的有效种群大小为8 171.2,和同类研究比较,种群规模偏大[6]。同等速率的遗传突变下,蜂群数量多产生的遗传变异就多,遗传多样性水平便高[23]。对以梵净山为代表的野生生物类保护区保护效果评估的重要标准包括有效种群大小、遗传多样性的高低等[23-24]。梵净山地区中华蜜蜂能够以约8 000群的种群规模存在,并表现出的同类研究中高水平的遗传多样性,这两个角度体现出保护区和周边对中华蜜蜂种群的保护作用。推测保护区内高森林覆盖率、丰富的植物种类、人为活动的严格管控等措施维持了良好的栖息地环境、充足的食物来源和较少的人为干扰,使中华蜜蜂维持大种群,形成高遗传多样性。在全国本土中华蜜蜂遗传多样性下降、遗传资源广受威胁的背景下,自然保护区及周边区域蕴藏了遗传多样性丰富的蜜蜂种质资源。
致谢: 感谢贵州梵净山国家级自然保护区管理局在本研究的样本采集和保护区基本信息提供上给予的帮助;感谢样本采集中给予支持协助的蜂农朋友们。| [1] |
褚亚芳, 胡福良. 蜜蜂与生态平衡[J]. 蜜蜂杂志, 2009, 29(3): 8-10. DOI:10.3969/j.issn.1003-9139.2009.03.004 |
| [2] |
杨冠煌. 中华蜜蜂[M]. 北京: 中国农业科技出版社, 2001.
|
| [3] |
杨冠煌. 引入西方蜜蜂对中蜂的危害及生态影响[J]. 昆虫学报, 2005, 48(3): 401-406. DOI:10.3321/j.issn:0454-6296.2005.03.016 |
| [4] |
CHI X L, ZHANG Z J, XU X T, et al. Threatened medicinal plants in China: distributions and conservation priorities[J]. Biological Conservation, 2017, 210: 89-95. |
| [5] |
郭艳清, 周俊, 宋先华, 等. 贵州省梵净山自然保护区黔金丝猴的种群数量[J]. 兽类学报, 2017, 37(1): 104-108. |
| [6] |
YU Y L, ZHOU S J, ZHU X J, et al. Genetic differentiation of eastern honey Bee (Apis cerana) populations across Qinghai-Tibet Plateau-Valley landforms[J]. Frontiers in Genetics, 2019, 10: 483. DOI:10.3389/fgene.2019.00483 |
| [7] |
于瀛龙, 周姝婧, 徐新建, 等. 贵州省东方蜜蜂微卫星DNA遗传分化与遗传多样性分析[J]. 福建农林大学学报(自然科学版), 2017, 46(3): 323-328. |
| [8] |
PEAKALL R, SMOUSE P E. GenAlEx 6.5:genetic analysis in Excel.Population genetic software for teaching and research-an update[J]. Bioinformatics, 2012, 28(19): 2537-2539. DOI:10.1093/bioinformatics/bts460 |
| [9] |
DO C, WAPLES R S, PEEL D, et al. Ne estimator v2:re-implementation of software for the estimation of contemporary effective population size (Ne) from genetic data[J]. Molecular Ecology Resources, 2014, 14(1): 209-214. DOI:10.1111/1755-0998.12157 |
| [10] |
R CORE TEAM. R: a language and environment for statistical computing[R]. Vienna: R Foundation for Statistical Computing, 2016.
|
| [11] |
PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155(2): 945-959. DOI:10.1093/genetics/155.2.945 |
| [12] |
ROSENBERG N A. Distruct: a program for the graphical display of population structure[J]. Molecular Ecology Notes, 2004, 4(1): 137-138. |
| [13] |
JENSEN A B, PALMER K A, CHALINE N, et al. Quantifying honey bee mating range and isolation in semi-isolated valleys by DNA microsatellite paternity analysis[J]. Conservation Genetics, 2005, 6(4): 527-537. |
| [14] |
SCHNEIDER S S, MCNALLY L C. Waggle dance behavior associated with seasonal absconding in colonies of the African honey bee, Apis mellifera scutellata[J]. Insectes Sociaux, 1994, 41(2): 115-127. DOI:10.1007/BF01240472 |
| [15] |
VILLA J D. Swarming behavior of honey bees (Hymenoptera: Apidae) in southeastern Louisiana[J]. Annals of the Entomological Society of America, 2004, 97(1): 111-116. DOI:10.1603/0013-8746(2004)097[0111:SBOHBH]2.0.CO;2 |
| [16] |
郭慧萍, 周姝婧, 朱翔杰, 等. 秦巴山区中华蜜蜂种群微卫星DNA遗传分析[J]. 昆虫学报, 2016, 59(3): 337-345. |
| [17] |
WANG P, ZHANG M, LIU J J, et al. Genetic diversity and structure of Libanotis buchtormensis (Fisch.) DC. in disjunct populations along the bilateral sides of deserts in northwestern China[J]. Plant Systematics and Evolution, 2015, 301(9): 2219-2230. DOI:10.1007/s00606-015-1225-y |
| [18] |
JI T, YIN L, CHEN G H. Genetic diversity and population structure of Chinese honeybees (Apis cerana) under microsatellite markers[J]. African Journal of Biotechnology, 2011, 10(9): 1712-1720. |
| [19] |
陈晶, 陈国宏, 吉挺, 等. 沂蒙山中华蜜蜂微卫星DNA遗传多样性分析[J]. 中国蜂业, 2008, 59(4): 11-13. |
| [20] |
徐新建, 周姝婧, 朱翔杰, 等. 海南岛中华蜜蜂遗传多样性的微卫星DNA分析[J]. 昆虫学报, 2013, 56(5): 554-560. |
| [21] |
徐新建, 周姝婧, 朱翔杰, 等. 黄土高原中华蜜蜂遗传多样性的微卫星DNA分析[J]. 福建农林大学学报(自然科学版), 2013, 42(6): 638-642. |
| [22] |
于瀛龙, 周姝婧, 徐新建, 等. 长白山中华蜜蜂(Apis cerana cerana)遗传多样性分析[J]. 福建农林大学学报(自然科学版), 2013, 42(6): 643-647. |
| [23] |
FRANKHAM R, BALLOU J D, BRISCOE D A, et al. Introduction to conservation genetics[M]. Cambridge: Cambridge University Press, 2002.
|
| [24] |
何刚, 郭松涛, 金学林, 等. 佛坪自然保护区川金丝猴种群动态[J]. 兽类学报, 2018, 38(3): 247-259. |

 2021, Vol. 41
2021, Vol. 41


