文章信息
- 李云琴, 王毅, 张子蕴, 原晓龙, 杨焱, 杨文忠
- LI Yunqin, WANG Yi, ZHANG Ziyun, YUAN Xiaolong, YANG Yan, YANG Wenzhong
- 桑黄9个萜类合成酶基因的鉴定及表达分析
- Identification and expression analysis of nine terpene synthase genes in Sanghuangporus sanghuang
- 森林与环境学报,2021, 41(4): 402-409.
- Journal of Forest and Environment,2021, 41(4): 402-409.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2021.04.010
-
文章历史
- 收稿日期: 2021-03-19
- 修回日期: 2021-05-31
2. 云南省森林植物培育与开发利用重点实验室, 国家林草局云南珍稀濒特森林植物保护和繁育重点实验室, 云南 昆明 650201;
3. 上海市农业科学院食用菌研究所, 上海 201106
2. Key Laboratory of Forestry Plant Cultivation and Utilization, Key Laboratory of Rare and Endangered Forest Plants of National Forestry and Grassland Administration, Kunming, Yunnan 650201, China;
3. Institute of Edible Fungi, Shanghai Academy of Agricultural Sciences, Shanghai 201106, China
萜类化合物又称类异戊二烯类化合物,是地球上最大、结构和功能最丰富的一类天然化合物,到目前为止,已发现了80 000多种不同结构的萜类化合物,按碳原子数目分为单萜(C10)、倍半萜(C15)、二萜(C20)、三萜(C30)和多萜等;按照碳环数分为链萜、单环萜、双环萜和三环萜等[1]。萜类化合物有抑菌、抗病毒、抗炎、抗氧化、抗肿瘤及增强机体免疫力等功能[2],具有巨大的医学及商业潜力。萜类化合物在高等真菌中的含量极为丰富,高等真菌中的萜类化合物主要有倍半萜、三萜等,倍半萜类化合物是高等真菌中结构最丰富的一类,三萜类也是其主要的次生代谢产物[3]。研究发现,高等真菌中不同类型的萜类在抗癌、抗病毒等方面具有显著的作用,如THANH et al[4]从火木层孔菌(Phellinus igniarius)子实体中分离得到一个新的三萜化合物对MCF-7等肿瘤细胞株具有较好的抑制作用;SANDARGO et al[5]从掌状玫耳(Rhodotus palmatus)中分离的12个倍半萜类化合物可显著抑制丙型肝炎病毒。
萜类化合物均是以萜类合成酶(terpene synthase, TPS)为关键酶来合成的[6],萜类合成酶根据催化产物的不同,分为单萜、倍半萜和三萜合成酶,分别以牻牛儿基焦磷酸(geranyl diphosphate,GPP) 或橙花基焦磷酸(neryl diphosphate,NPP)、法呢基焦磷酸(farnesyl diphosphate,FPP)和牻牛儿基牻牛儿基焦磷酸(geranylgeranyl diphosphate,GGPP)为前体底物合成相应的单萜、倍半萜[7],FPP和GGPP又可以进一步耦合产生三萜[8]。萜类合成酶是萜类化合物生物合成的关键酶,也是萜类合成过程中研究最深入的酶类[3]。高等植物中的TPS基因组已被大量研究,例如拟南芥基因组中11个AtTPS基因的研究[9],水稻TPS基因家族11个OsTPS基因的研究[10]等等。随着分子生物学的发展,学者们开展了一些关于真菌萜类合成酶基因组的研究,如WAWRZYN et al[11]对发光类脐菇(Omphalotus olearius)中9个TPS基因进行克隆和功能鉴定,这些基因主要诱导生成倍半萜类化合物(α-muurolene、δ-cadinene、γ-cadinene等);MISCHKO et al[12]研究粉孢革菌(Coniophora puteana)TPS基因时,通过大肠杆菌异源表达,从中鉴定出2种倍半萜合成酶,这2种酶能够高选择性地催化β-copaene和cubebol合成。
桑黄[Sanghuangporus sanghuang (Sheng H. Wu, T. Hatt. & Y.C. Dai) Sheng H.Wu, L.W. Zhou & Y.C.Dai]是担子菌门(Basidiomycotas)层菌纲(Hymenomycetes)非褶菌目(Aphyllophorales)锈革孔菌科(Hymenochaetaceae)桑黄孔菌属(Sanghuangporus)的多年生高等药用真菌[13],具有活血、止血和排毒等功效,药用历史已达2 000多年。近年来对桑黄的深入研究发现,其包含多糖类、黄酮类、酚类、萜类、甾体、香豆素类和生物碱等活性成分,桑黄中的萜类主要是倍半萜和三萜[14],这些萜类活性成分具有较大的药用及商业利用价值。但是,目前鲜见关于桑黄萜类合成过程中起关键作用的TPS基因的报道。本研究从桑黄全基因组中分离得到9个SsTPS基因,对其进行生物信息学分析、系统进化树构建,并分析在不同培养基培养条件下的基因表达情况,为进一步研究桑黄SsTPS基因功能和相关活性萜类化合物的高效开发提供理论基础。
1 材料与方法 1.1 试验材料野生型桑黄菌株由上海市农业科学院食用菌研究所提供,菌株基因组原始数据及原始编号见参考文献[15]。采用组织分离法,将子实体用体积分数为75%的酒精消毒,在无菌条件下将子实体切碎后接种在酵母麦芽浸粉肉汤(malt medium, MY)斜面培养基上,28 ℃避光培养15 d,当菌落直径达到1 cm左右时,选择无杂菌污染的菌丝转接到新的MY培养基上。将经转录间隔区序列(internal transcribed spacer,ITS)鉴定的菌丝体接种在含不同碳氮源添加物的培养基上,28 ℃培养15 d后,采集菌丝体用于检测桑黄萜类合成酶基因(terpene synthase of Sanghuangporus sanghuang,SsTPS)的具体表达情况。
1.2 培养基配制液体碳源培养基配制:以MY(青岛日水生物技术有限公司生产)21 g·L-1、琼脂粉15 g·L-1为基础,分别添加20 g·L-1的甘露醇、麦芽糖、山梨醇、蔗糖、乳糖、肌醇、果糖配置成不同碳源培养基。
液体氮源培养基配制:以麦芽糖1.8 g·L-1、葡萄糖6 g·L-1为基础,分别添加4 g·L-1的牛肉浸粉、大豆蛋白粉、酵母粉、香蕉粉、淀粉配置成不同氮源培养基;高温高压蒸汽灭菌(121 ℃,20 min)。
1.3 总RNA提取和cDNA合成将菌丝接种于上述培养基,28 ℃,150 r·min-1摇床培养15 d,过滤菌液后将获得的菌丝体压干并分装为小份样品(每份含菌丝50 mg),在液氮中粉碎,按北京康为世纪生物科技有限公司的RNA提取试剂盒的方法提取样品总RNA,使用微量分光光度计(型号Thermo NanoDrop 2000)和质量分数为1.5%的琼脂糖凝胶电泳分别检测RNA的浓度和质量。参照北京全式金生物技术有限公司的TransScript© Reverse Transcriptase[M-MLV,RNaseH-]试剂盒说明书进行RNA反转录。
1.4 蛋白质理化性质分析用在线程序Expasy protparam tool(https://web.expasy.org/protparam)分析TPS蛋白质理化性质,利用在线程序NetPhos-3.1 service(https://services.healthtech.dtu.dk/service.php?NetPhos-3.1)和DictyOGlyc-1.1 service(https://services.healthtech.dtu.dk/service.php?DictyOGlyc-1.1)分别预测氨基酸序列磷酸化和O-糖基化修饰情况,利用在线程序SignalP-5.0(http://www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽,TargetP-2.0 Server(http://www.cbs.dtu.dk/services/TargetP/)预测蛋白质亚细胞定位,利用在线程序Prabi(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)预测蛋白质的二级结构。
1.5 蛋白质序列分析和系统进化树构建将SsTPS蛋白质序列导入到DNAMAN软件中进行多重序列比对,利用在线程序MEME(http://meme.nbcr.net/meme/tools/meme)对9个SsTPS蛋白质保守结构域进行预测;通过在美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI, https://www.ncbi.nlm.nih.gov/)上比对,并查阅相关文献,获得其他真菌的TPS蛋白质序列,利用MEGA 6.0软件对桑黄SsTPS蛋白质进行系统进化树构建。
1.6 基因表达分析使用DNAMAN在线程序primer进行桑黄SsTPS基因检测引物设计,并由上海生工生物工程有限公司合成引物。以cDNA作为模板与桑黄9个SsTPS基因检测引物进行聚合酶链式反应(polymerase chain reaction,PCR)扩增,同时以内参Tubulin引物作为对照。反应总体系为20 μL(mRNA模板1 μL,引物2 μL,PCR Master Mix with Dye 10 μL,ddH2O 7 μL),反应程序:95 ℃预变性5 min,95 ℃预变性30 s,58 ℃退火30 s,72 ℃延伸1 min,35个循环,最后72 ℃延伸10 min,12 ℃保存。用质量分数为1.5%的琼脂糖凝胶电泳检测,利用Azure Biosystems成像系统软件将PCR凝胶图像信息转化为基因表达量数值导入到Excel表格中并计算相对表达量,利用在线程序Genescloud(https://www.genescloud.cn/chart/)将相对表达量数据做成热图。
| $ F=X / T $ |
式中:F表示相对表达量;X表示不同培养基中桑黄SsTPS的表达量;T表示与X对应的不同培养基中桑黄Tubulin的表达量。
1.7 基因簇分析使用在线程序antiSMASH (https://fungismash.secondarymetabolites.org)对9个桑黄SsTPS基因所在contig进行基因簇预测分析。
2 结果与分析 2.1 桑黄萜类合成酶蛋白质理化性质分析桑黄全基因组中共分离得到9个SsTPS基因,将其命名为SsTPS1~SsTPS9,分别对应相应的SsTPS蛋白质。桑黄SsTPS蛋白质理化性质分析结果如表 1所示。蛋白质SsTPS1的氨基酸长度最长,为1 688个氨基酸残基(amino acid residue, aa);SsTPS7的氨基酸长度最短,为272个aa。SsTPS分子量为31 433.65~185 193.11 u之间,其中,SsTPS1分子量最大,SsTPS7分子量最小;桑黄TPS蛋白质的理论等电点为5.23~7.00,其中,SsTPS8的理论等电点最小,SsTPS6的理论等电点值最大,平均值为5.95,除了SsTPS6的理论等电点为7.00外,所有蛋白质的理论等电点均小于7.00,说明其蛋白质分子中富含酸性氨基酸且酸性氨基酸数量多于碱性氨基酸;蛋白质的磷酸化位点个数的平均值为87.11,O-糖基化位点个数的平均值为0.89。桑黄SsTPS蛋白质的不稳定系数介于31.56~48.95之间,其中,SsTPS2、SsTPS4、SsTPS5、SsTPS6和SsTPS7为不稳定蛋白质(不稳定系数大于40),SsTPS1、SsTPS3、SsTPS8、SsTPS9为稳定性蛋白质(不稳定系数小于40);脂肪系数介于74.61~91.71之间,SsTPS1蛋白的脂肪系数最大,SsTPS9蛋白质的脂肪系数最小;蛋白质亲水性平均系数介于-0.444~-0.067之间,均表现为亲水性。
| 蛋白质名称 Protein name |
氨基酸数 Number of amino acids |
分子量 Molecular weight /u |
理论等电点 Theoretical isoelectric point |
磷酸化位点 Phosphory-lation site |
O-糖基化位点 O-glycosy-lation site |
不稳定系数 Instability index |
| SsTPS1 | 1 688 | 185 193.11 | 6.50 | 273 | 2 | 34.78 |
| SsTPS2 | 900 | 102 513.73 | 5.54 | 113 | 0 | 48.95 |
| SsTPS3 | 308 | 35 176.07 | 6.11 | 44 | 1 | 31.56 |
| SsTPS4 | 314 | 35 406.10 | 5.37 | 71 | 3 | 42.41 |
| SsTPS5 | 359 | 41 346.71 | 5.62 | 53 | 0 | 40.87 |
| SsTPS6 | 366 | 41 106.94 | 7.00 | 72 | 0 | 41.09 |
| SsTPS7 | 272 | 31 433.65 | 6.67 | 42 | 1 | 40.99 |
| SsTPS8 | 466 | 51 645.60 | 5.23 | 63 | 0 | 33.71 |
| SsTPS9 | 284 | 32 454.61 | 5.55 | 53 | 1 | 39.61 |
| 基因名称 Gene name |
脂肪系数 Aliphatic index |
亲水性平均系数 Grand average of hydrop athicity |
二级结构所占比例Secondary structure/% | |||
| α-螺旋 α-helix |
β-转角 β-turn |
无规则卷曲 Random coil |
延伸链 Extended strand |
|||
| SsTPS1 | 91.71 | -0.067 | 37.03 | 9.00 | 33.00 | 20.97 |
| SsTPS2 | 84.34 | -0.294 | 39.44 | 6.56 | 40.33 | 13.67 |
| SsTPS3 | 80.36 | -0.353 | 52.60 | 2.60 | 36.69 | 8.12 |
| SsTPS4 | 79.62 | -0.303 | 57.01 | 2.87 | 32.48 | 7.64 |
| SsTPS5 | 84.51 | -0.437 | 53.48 | 2.23 | 35.38 | 8.91 |
| SsTPS6 | 77.08 | -0.219 | 54.37 | 2.73 | 34.97 | 7.92 |
| SsTPS7 | 83.46 | -0.444 | 48.90 | 4.41 | 31.62 | 15.07 |
| SsTPS8 | 88.22 | -0.118 | 53.65 | 3.65 | 31.76 | 10.94 |
| SsTPS9 | 74.61 | -0.328 | 55.28 | 2.11 | 33.10 | 9.51 |
所有SsTPS蛋白质的二级结构均有α-螺旋、β-转角、无规卷曲及延伸链4种,除了SsTPS2蛋白质的二级结构以无规则卷曲为主外,其余蛋白质均以α-螺旋为主,无规则卷曲为次要结构,延伸链及β-转角所占比例较少。
2.2 桑黄萜类合成酶蛋白质保守基序分析桑黄SsTPS蛋白质保守基序分析结果如图 1所示。Motif1、Motif3、Motif6为桑黄SsTPS蛋白质的保守结构域,Motif2、Motif4、Motif5为未知保守结构域;除了SsTPS2没有基序外,其余桑黄SsTPS蛋白质均含TPS家族的保守结构域DXXD[16]和NSE[16]基序。SsTPS2经Blast同源性比对和保守结构域分析,证明其属于TPS家族,可能在进化过程中,结构域发生了变异。SsTPS5、SsTPS6和SsTPS8含有Motif数目最多,均包含了6个Motif,其次是SsTPS1和SsTPS4,各包含5个Motif,SsTPS7的Motif数目最少(3个)。

|
图 1 桑黄萜类合成酶蛋白质保守基序分析 Fig. 1 Analysis of the conserved motifs of terpene synthase proteins in Sanghuangporus sanghuang |
桑黄SsTPS蛋白质系统进化分析结果如图 2所示。桑黄的9个SsTPS蛋白质大致聚为2个大支系,其中,SsTPS1、SsTPS4、SsTPS7、SsTPS8和SsTPS9为一个支系,SsTPS2、SsTPS3、SsTPS5、SsTPS6为另一个支系。
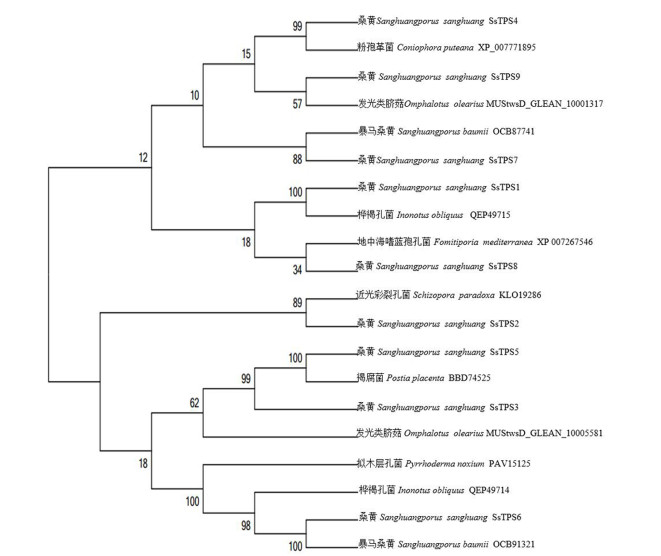
|
图 2 桑黄萜类合成酶蛋白质的系统发育树 Fig. 2 A phylogenetic tree of terpene synthase proteins |
SsTPS1与桦褐孔菌(Inonotus obliquus,登录号:QEP49715)的倍半萜合成酶聚在一起,由此初步推断,SsTPS1为倍半萜合成酶,且可以催化合成倍半萜化合物(α-muurolene)[17];SsTPS2与近光彩裂孔菌(Schizopora paradoxa,登录号:KLO19286)三萜羊毛甾醇合成酶聚在一起,由此推断,SsTPS2为三萜羊毛甾醇合成酶;SsTPS3和SsTPS5与褐腐菌(Postia placenta,登录号:BBD74525)和发光类脐菇(Omphalotus olearius,登录号:MUStwsD_GLEAN_10005581)的倍半萜合成酶聚在一起,且发光类脐菇主要产生δ-杜松烯(δ-cadinene)[11],由此推断,SsTPS3和SsTPS5属于倍半萜合成酶,而且可能与合成δ-cadinene的类似物或衍生物有关;SsTPS4与粉孢革菌(Coniophora puteana,登录号:XP_007771895)倍半萜合成酶聚集在一起,且粉孢革菌的倍半萜合成酶主要产生β-拮靶烯(β-copaene)[12],由此推断,SsTPS4也是倍半萜合成酶,其主要功能与合成β-copaene的类似物或衍生物有关;SsTPS9与发光类脐菇(Omphalotus olearius,登录号:MUStwsD_GLEAN_10001317)的倍半萜合成酶聚集在一起,且发光类脐菇的这个酶主要生成α-衣兰油烯(α-muurolene)[11],由此推断,SsTPS9属于倍半萜合成酶,而且可能与合成α-muurolene的类似物或衍生物有关。
2.4 桑黄萜类合成酶基因的表达验证及分析通过在线程序Genescloud(https://www.genescloud.cn/chart/)绘制不同培养条件下桑黄SsTPS基因表达热图,结果如图 3所示。桑黄9个SsTPS基因在不同碳源和氮源培养条件下的表达情况各不相同,SsTPS1、SsTPS4、SsTPS6基因在所有培养基中均不表达;SsTPS7仅在添加肌醇为碳源、大豆蛋白粉为氮源的培养基中低量表达,在其它培养基中均不表达;其余基因呈现差异化表达,SsTPS2、SsTPS3、SsTPS5、SsTPS8、SsTPS9分别在添加果糖为碳源、大豆蛋白粉为氮源、牛肉浸粉为氮源、酵母粉为氮源、香蕉粉为氮源的培养基中表达量最高。
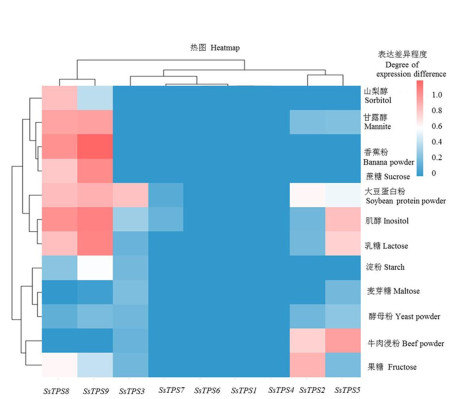
|
图 3 不同培养条件下桑黄萜类合成酶基因的表达热图 Fig. 3 A heat map depicting SsTPS gene expression in different carbon and nitrogen source media |
对桑黄9个SsTPS基因所在contig进行基因簇分析(图 4)后发现,SsTPS1的编码基因所在contig有锌指转录因子和血红蛋白超级家族细胞色素P450氧化还原酶基因存在,该基因簇的核心基因可能是锌指蛋白质、SsTPS与P450组成的一个基因簇。SsTPS4基因所在contig发现与其呈簇出现的有还原型烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate, NADPH)和黄素腺嘌呤二核苷酸(flavin adenine dinucleotide, FAD)氧化还原酶基因以及1个萜类合成酶基因。
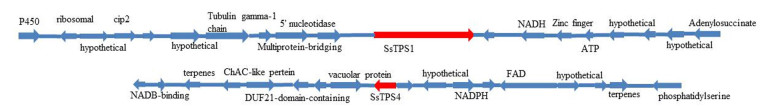
|
图 4 桑黄萜类合成酶基因的基因簇预测分析 Fig. 4 Prediction and analysis of gene clusters |
真菌产生的萜类化合物种类繁多,具有多种生物活性功能,其中许多萜类化合物可供人类使用[8]。真菌是天然次生代谢产物的重要来源,通过发酵工程可大量且快速地获得萜类等活性成分[3]。这些萜类活性成分合成过程中需要许多酶的参与,萜类合成酶就是其中的关键酶,因此,深入了解萜类合成酶是进行萜类化合物生物合成的第一步[8]。随着越来越多基因组数据库的建立,从基因组层面分析和研究基因家族成为可能[18]。目前许多真菌的萜类合成酶基因组已被鉴定,并且已取得一些进展。本研究分离出9个桑黄SsTPS基因,除SsTPS2蛋白质的保守结构域位点比较特殊外,大部分桑黄TPS蛋白质均具有TPS的典型特征结构域。此外,除了含有高度保守的TPS结构域,还有其他未知结构域的motif存在,说明虽然多数SsTPS具有相似的蛋白质保守结构域和功能冗余特性,但仍存在motif的多样性,它们可能作为潜在的蛋白质-蛋白质间的相互作用结构域辅助TPS蛋白质发挥作用[6]。SsTPS1、SsTPS4、SsTPS6基因在培养基中均不表达,可能是因为这3个基因属于沉默基因,或者是假基因(任何情况都不表达)。其余SsTPS基因在不同培养基培养的菌丝体中呈现差异化表达。HEWAGE et al[19]的研究结果也表明,真菌基因在不同碳氮源培养基上呈现表达差异,有些基因在有些培养基中大量表达,而在另外的培养基中完全不表达。YU et al[20]以胡枝子(Lespedeza bicolor)的星形拟盘多毛孢(Pestalotiopsis lespedezae)为内生真菌,在固体水稻培养基上加入质量分数为3.5%的NaI进行发酵,得到10个新的氨苯甲酸衍生物;GAO et al[21]在固体水稻培养基上加入质量分数为3.5%的NaNO3或谷氨酸钠发酵,诱导内生真菌(Aplosporella javeedii)产生了11种新的内酰胺衍生物;构巢曲霉(Aspergillus nidulans)与吸水链霉菌(Streptomyces hygroscopicus)联合培养之后,产生新的化合物可用于抑制骨质疏松[22];盘针孢属真菌(Libertella sp.)与a-变形杆菌(a-proteobacterium)联合培养得到了分别培养没有得到的化合物,对人结肠癌具有较强的细胞毒活性[23]。由此可知,培养基成分、培养条件和添加剂能够影响基因表达,进而促使其产生不同的次级代谢产物,得到大量活性不同甚至新型的代谢产物,这些案例正好印证了真菌培养的单菌多产物策略(one strain-many compounds,OSMAC)[24]。
桑黄TPS基因包含5个倍半萜合酶基因,1个三萜合酶基因;SsTPS1和SsTPS4基因所在contig有基因簇存在,但这2个基因在不同碳源和氮源培养条件下均不表达。下一步的研究可根据OSMAC以及一些激活沉默基因和基因簇的方法(如基因的过表达、异源表达、重构生物合成基因簇[25]等等)来刺激或激活不同的基因或基因簇,提高其表达以得到目标化合物或获取一些新的活性物质。
真菌萜类合成酶基因的克隆和分析对萜类化合物生物合成的分子调控具有重要意义,有利于阐明真菌萜类生物合成机理,也有利于利用代谢工程手段定向改造生产菌种,从而提高萜类化合物的产量或产生新的萜类化合物[1]。
| [1] |
刘莉, 胡昌华. 真菌二萜环化酶的研究进展[J]. 微生物学报, 2010, 50(11): 1438-1445. |
| [2] |
DAI Y C, ZHOU L W, CUI B K, et al. Current advances in Phellinus sensu lato: medicinal species, functions, metabolites and mechanisms[J]. Applied Microbiology and Biotechnology, 2010, 87(5): 1587-1593. DOI:10.1007/s00253-010-2711-3 |
| [3] |
周国峰, 杨郁, 徐锐, 等. 蘑菇来源萜类化合物研究进展[J]. 国际药学研究杂志, 2020, 47(11): 928-945. |
| [4] |
THANH N T, TUAN N N, KUO P C, et al. Chemical constituents from the fruiting bodies of Phellinus igniarius[J]. Natural Product Research, 2018, 32(20): 2392-2397. DOI:10.1080/14786419.2017.1413572 |
| [5] |
SANDARGO B, MICHEHL M, STADLER M, et al. Antifungal sesquiterpenoids, rhodocoranes, from submerged cultures of the wrinkled peach mushroom, Rhodotus palmatus[J]. Journal of Natural Products, 2020, 83(3): 720-724. DOI:10.1021/acs.jnatprod.9b00871 |
| [6] |
AGGER S, LOPEZ-GALLEGO F, SCHMIDT-DANNERT C. Diversity of sesquiterpene synthases in the basidiomycete Coprinus cinereus[J]. Molecular Microbiology, 2009, 72(5): 1181-1195. DOI:10.1111/j.1365-2958.2009.06717.x |
| [7] |
SCHILMILLER A L, SCHAUVINHOLD I, LARSON M, et al. Monoterpenes in the glandular trichomes of tomato are synthesized from a neryl diphosphate precursor rather than geranyl diphosphate[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(26): 10865-10870. DOI:10.1073/pnas.0904113106 |
| [8] |
WAWRZYN G T, BLOCH S E, SCHMIDT-DANNERT C. Discovery and characterization of terpenoid biosynthetic pathways of fungi[J]. Methods in Enzymology, 2012, 515: 83-105. |
| [9] |
VANDESTEENE L, RAMON M, LE ROY K, et al. A single active trehalose-6-psynthase(TPS) and a family of putative regulatory TPS-like proteins in Arabidopsis[J]. Molecular Plant, 2010, 3(2): 406-419. DOI:10.1093/mp/ssp114 |
| [10] |
ZANG B S, LI H W, LI W J, et al. Analysis of trehalose-6-phosphate synthase(TPS) gene family suggests the formation of TPS complexes in rice[J]. Plant Molecular Biology, 2011, 76(6): 507-522. DOI:10.1007/s11103-011-9781-1 |
| [11] |
WAWRZYN G T, QUIN M B, CHOUDHARY S, et al. Draft genome of Omphalotus olearius provides a predictive framework for sesquiterpenoid natural product biosynthesis in Basidiomycota[J]. Chemistry & Biology, 2012, 19(6): 772-783. |
| [12] |
MISCHKO W, HIRTE M, FUCHS M, et al. Identification of sesquiterpene synthases from the Basidiomycota Coniophora puteana for the efficient and highly selective β-copaene and cubebol production in E. coli[J]. Microbial Cell Factories, 2018, 17(1): 164. DOI:10.1186/s12934-018-1010-z |
| [13] |
吴声华, 戴玉成. 药用真菌桑黄的种类解析[J]. 菌物学报, 2020, 39(5): 781-794. |
| [14] |
陈万超, 杨焱, 张劲松, 等. 桑黄类真菌活性代谢产物的研究进展[J]. 食用菌学报, 2020, 27(4): 188-201. |
| [15] |
曲德辉, 杨焱, 张赫男, 等. 桑黄菌株活力评价及优良菌株筛选[J]. 菌物学报, 2016, 35(10): 1187-1198. |
| [16] |
MAFU S, KARUNANITHI P S, PALAZZO T A, et al. Biosynthesis of the microtubule-destabilizing diterpene pseudolaric acid B from golden larch involves an unusual diterpene synthase[J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(5): 974-979. DOI:10.1073/pnas.1612901114 |
| [17] |
褚丽华, 王丽芝, 陈士林, 等. 一个紫芝多产物倍半萜合酶的功能鉴定[J]. 中国实验方剂学杂志, 2019, 25(12): 151-157. |
| [18] |
赵荣荣, 刘斌, 郭超, 等. 梨萜类合成酶基因家族生物信息学与表达模式分析[J]. 生物技术通报, 2018, 34(5): 131-141. |
| [19] |
HEWAGE R T, AREE T, MAHIDOL C, et al. One strain-many compounds(OSMAC) method for production of polyketides, azaphilones, and an isochromanone using the endophytic fungus Dothideomycete sp.[J]. Phytochemistry, 2014, 108: 87-94. DOI:10.1016/j.phytochem.2014.09.013 |
| [20] |
YU X Q, GAO Y, FRANK M, et al. Induction of ambuic acid derivatives by the endophytic fungus Pestalotiopsis lespedezae through an OSMAC approach[J]. Tetrahedron, 2021, 79: 131876. DOI:10.1016/j.tet.2020.131876 |
| [21] |
GAO Y, STUHLDREIER F, SCHMITT L, et al. Induction of new lactam derivatives from the endophytic fungus Aplosporella javeedii through an OSMAC approach[J]. Frontiers in Microbiology, 2020, 11: 600983. DOI:10.3389/fmicb.2020.600983 |
| [22] |
SCHROECKH V, SCHERLACH K, NVTZMANN H W, et al. Intimate bacterial-fungal interaction triggers biosynthesis of archetypal polyketides in Aspergillus nidulans[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(34): 14558-14563. DOI:10.1073/pnas.0901870106 |
| [23] |
OH D C, JENSEN P R, KAUFFMAN C A, et al. Libertellenones A-D: induction of cytotoxic diterpenoid biosynthesis by marine microbial competition[J]. Bioorganic & Medicinal Chemistry, 2005, 13(17): 5267-5273. |
| [24] |
BODE H B, BETHE B, HÖFS R, et al. Big effects from small changes: possible ways to explore nature's chemical diversity[J]. Chembiochem, 2002, 3(7): 619-627. DOI:10.1002/1439-7633(20020703)3:7<619::AID-CBIC619>3.0.CO;2-9 |
| [25] |
关丹丹, 赫卫清. 通过重构来激活沉默的次级代谢产物生物合成基因簇[J]. 中国医药生物技术, 2020, 15(5): 504-507. DOI:10.3969/j.issn.1673-713X.2020.05.011 |

 2021, Vol. 41
2021, Vol. 41


