文章信息
- 赵森, 邓力华, 陈芬
- ZHAO Sen, DENG Lihua, CHEN Fen
- 秋茄叶绿体基因组密码子使用偏好性分析
- Codon usage bias of chloroplast genome in Kandelia obovata
- 森林与环境学报,2020, 40(5): 534-541.
- Journal of Forest and Environment,2020, 40(5): 534-541.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2020.05.011
-
文章历史
- 收稿日期: 2020-06-11
- 修回日期: 2020-07-02
2. 中国科学院亚热带农业生态研究所亚热带农业生态过程重点实验室, 湖南 长沙 410125
2. Key Laboratory of Agro-ecological Processes in Subtropical Region, Institute of Subtropical Agriculture, Chinese Academy of Sciences, Changsha, Hunan 410125, China
密码子是联结生物体内遗传物质和蛋白质翻译的纽带。生物体内的20种氨基酸,除了蛋氨酸(Met)和色氨酸(Trp)具有单一密码子外,其余氨基酸均由2~6个同义密码子编码。同义密码子的使用具有非均一性,某一物种或某一基因倾向使用一种或几种特定的同义密码子的现象称为同义密码子的使用偏好性[1]。密码子使用偏好性是自然界广泛存在的现象,是一种分子进化形式,同一物种或亲缘关系近的物种中基因一般表现为相似的密码子使用模式。由于物种在进化过程中受到的突变压力和选择作用影响不同,不同物种的密码子使用偏好性不同。通过对密码子偏好性的研究,能够更好地了解物种的进化发展[2]。越来越多的研究表明,密码子使用偏好性与基因表达水平、基因长度、基因翻译的起始信号、蛋白质氨基酸的组成、蛋白质结构、tRNA的丰度、突变模式和频率、GC含量等因素有关[3-5]。叶绿体基因组是核基因组之外相对独立的遗传物质,能够半自主地进行复制、转录和翻译等生命过程。以叶绿体为受体的基因工程其遗传方式为母性遗传,与一般的核转化体系相比具有外源基因高效表达、安全性高、多基因共表达、无位置效应和基因沉默等多方面的优势[6-7]。已在植物抗性[8]、医用蛋白高效表达[9-10]和农艺性状改良[11]等方面有重要应用。目前,在油茶(Camellia oleifera Abel.)[12]、巨桉(Eucalyptus grandis Hill ex Maiden)[13]、沙枣(Elaeagnus angustifolia Linn.)[14]、酸枣[Ziziphus jujuba var. spinosa (Bunge) Hu ex H. F. Chow.][15]、灰毛浆果楝[Cipadessa cinerascens (Pell.) Hand.-Mazz.][16]等林木树种中开展了叶绿体基因组的相关研究。
秋茄(Kandelia obovata Sheue, Liu & Yong)是一种生活在热带、亚热带潮间带的红树植物,是红树林生态系统的重要组成部分,在海岸生态系统中发挥重要功能[17]。红树林除了具有重要的生态功能外,还是人类所需的食物、药物以及化工原料的提供者[18-19]。近年来,我国红树林陆续出现多种害虫种群爆发的现象,对红树林湿地的可持续利用带来严峻挑战,如广西北部湾地区的秋茄就遭受过小袋蛾(Acanthopsyche subferalbata Hampson)、白囊袋蛾(Chalioides kondonis Matsumura)、蜡彩袋蛾(Chalia larminati Heylaerts)等多种害虫的危害[20]。通过叶绿体基因工程来提高植物抗虫性是一种有效手段,但叶绿体基因密码子使用偏好性一般与核基因不一致,直接转化可能会造成转化效率低和基因沉默现象。通过对叶绿体密码子使用特性分析,有助于提高外源基因的转化效率和高效表达,同时,可为下一步开展叶绿体基因工程应用和分子进化研究奠定基础。
1 材料与方法 1.1 基因序列的获取从GenBank数据库中搜索并下载秋茄叶绿体基因组序列(登录号:MN117072),选择长度大于300 bp的蛋白质编码序列为分析样本[12]。
1.2 密码子偏好性相关参数计算以筛选后的非重复的蛋白质编码序列为研究对象,利用CodonW 1.4.2计算样本的同义密码子相对使用度(relative synonymous codon usage, URSC)和有效密码子数(effective number of codons, Nec);计算样本中密码子第3位核苷酸T、C、A和G的含量,分别记为T3、C3、A3和G3;利用CUSP在线程序(http://www.bioinformatics.nl/emboss-explorer/)计算样本总体GC含量,记为GCall;计算密码子第1、第2和第3位上GC含量,分别记为GC1、GC2和GC3。应用SPSS 22.0软件,基于Pearson相关系数法进行各参数间的相关性分析。
1.3 密码子偏好性成因分析 1.3.1 中性绘图分析计算各基因密码子第1、第2位核苷酸上GC含量的平均值GC12,即GC1和GC2的平均值,以GC12为纵坐标,以GC3为横坐标作散点图,分析GC3和GC12之间的相关性[21]。
1.3.2 有效密码子数绘图分析若同义密码子使用频率仅由碱基突变引起,则Nec=2+GC3+29/[GC32+(1-GC3)2],并绘制标准曲线,以各基因的GC3为横坐标,实际Nec值为纵坐标,绘制各基因的散点图进行有效密码子数绘图(Nec-plot)分析,通过散点图可以判断造成密码子使用偏好性的原因[22]。
1.3.3 奇偶偏好性分析在分析计算各基因密码子第3位上核苷酸含量(A3、T3、C3、G3)的基础上,以G3/(G3+C3)为横坐标,以A3/(A3+T3)为纵坐标作散点图进行奇偶偏好性(PR2-plot)分析,对密码子第3位上的4种碱基组成进行分析[12]。
1.4 最优密码子分析根据样本Nec值大小进行排序,分别选取排在前、后10%的基因定义为高偏性库和低偏性库,计算两个库的ΔURSC值。如果ΔURSC>0.3,同时在低偏性库中URSC<1,在高偏性库中URSC>1,则认定为最优密码子[23-24]。
1.5 系统发育分析 1.5.1 基于同义密码子相对使用度的聚类分析从GenBank中下载桐花树[Aegiceras corniculatum (L.) Blanco]、白骨壤[Avicennia marina (Forsk.) Vierh.]、银叶树(Heritiera littoralis Dryand.)、红榄李[Lumnitzera littorea (Jack) Voigt]、榄李(Lumnitzera racemosa Willd.)、红海榄(Rhizophora stylosa Griff.)和木果楝(Xylocarpus granatum J. Koenig)等红树植物叶绿体基因组CDS序列,按1.1的方法对各物种的CDS序列进行筛选,连同秋茄的CDS序列,在CodonW软件中计算各物种不同密码子的URSC值,除去蛋氨酸(AUG)、色氨酸(UGG)和3个终止密码子(UAA、UAG和UGA)后的59个密码子用于分析。以每个物种作为一个分析对象,以各密码子对应的URSC值作为变量,在SPSS 22.0中采用系统聚类法进行聚类分析。
1.5.2 基于rbcL基因序列的系统发育分析以桐花树、白骨壤、银叶树、红榄李、榄李、红海榄、秋茄和木果楝等红树植物的rbcL基因序列为分析对象,在MEGA 5.0软件中对各物种的rbcL基因序列进行Clustal W比对,分析中产生的空位处理为缺失,运用最大似然法(maximum likelihood, ML)和邻接法(neighbor joining, NJ)分别构建系统发育树,ML采用K2P双参数模型估算核苷酸的替代数。两种方法均采用1 000次的Bootstrap检验各分支的置信度,其它参数为软件默认设置。
2 结果与分析 2.1 秋茄叶绿体基因组的密码子组成对筛选到的50条秋茄叶绿体蛋白质编码序列进行分析,结果如表 1所示。密码子第1、2和3位上的GC平均含量分别为46.23%、38.68%和27.62%,GCall平均含量为37.51%,说明密码子不同位置上的GC含量不同,其中又以GC3含量最低,即偏好使用以A/U结尾的密码子。Nec范围在35.40~55.50之间,平均值为47.02。以Nec < 45说明密码子使用偏好性强,Nec>45说明密码子使用偏好性弱的标准来判断[16],其中Nec大于45的基因有35个(表 1)。可以看出,秋茄叶绿体基因密码子使用偏好非随机选择,偏好性较弱。
| 基因 Gene |
GC含量Content of GC/% | Nec | |||
| GC1 | GC2 | GC3 | GCall | ||
| rps12 | 52.80 | 47.20 | 30.40 | 43.47 | 43.76 |
| psbA | 50.28 | 43.22 | 32.20 | 41.90 | 41.40 |
| matK | 38.51 | 30.06 | 26.33 | 31.63 | 49.71 |
| ycf3 | 48.82 | 38.24 | 30.59 | 39.22 | 55.50 |
| psaA | 51.53 | 43.01 | 30.49 | 41.68 | 49.05 |
| psaB | 48.98 | 42.86 | 30.75 | 40.86 | 48.28 |
| rps14 | 44.55 | 46.53 | 29.70 | 40.26 | 35.85 |
| psbC | 54.43 | 45.57 | 31.22 | 43.74 | 47.35 |
| psbD | 51.98 | 43.22 | 33.90 | 43.03 | 44.95 |
| rpoB | 49.58 | 36.88 | 28.94 | 38.47 | 49.39 |
| rpoC1 | 50.36 | 36.87 | 25.82 | 37.69 | 48.18 |
| rpoC2 | 43.66 | 36.72 | 28.27 | 36.22 | 49.05 |
| rps2 | 42.31 | 42.74 | 29.49 | 38.18 | 49.55 |
| atpI | 48.79 | 37.50 | 25.81 | 37.37 | 45.14 |
| atpF | 45.26 | 35.79 | 25.79 | 35.61 | 43.94 |
| atpA | 55.31 | 39.57 | 27.17 | 40.68 | 48.26 |
| rps4 | 49.01 | 36.63 | 26.24 | 37.29 | 49.01 |
| ndhJ | 47.80 | 37.74 | 32.70 | 39.41 | 45.80 |
| ndhK | 43.81 | 43.81 | 23.89 | 37.17 | 50.00 |
| ndhC | 45.45 | 33.06 | 29.75 | 36.09 | 52.50 |
| atpE | 52.99 | 39.55 | 30.60 | 41.04 | 55.01 |
| atpB | 55.71 | 41.08 | 27.66 | 41.48 | 44.90 |
| rbcL | 58.19 | 44.12 | 28.15 | 43.49 | 47.35 |
| accD | 39.59 | 35.51 | 27.96 | 34.35 | 45.64 |
| ycf4 | 42.16 | 41.08 | 31.35 | 38.20 | 47.35 |
| petA | 52.96 | 36.76 | 29.28 | 39.67 | 49.90 |
| rps18 | 34.31 | 37.25 | 24.51 | 32.03 | 35.40 |
| rpl20 | 32.59 | 38.52 | 21.48 | 30.86 | 52.55 |
| clpP | 56.63 | 35.71 | 30.10 | 40.82 | 53.57 |
| psbB | 55.01 | 45.78 | 28.49 | 43.09 | 46.89 |
| petB | 47.27 | 40.45 | 29.09 | 38.94 | 44.24 |
| petD | 50.57 | 36.78 | 22.99 | 36.78 | 38.11 |
| rpoA | 46.76 | 31.47 | 27.06 | 35.10 | 50.45 |
| rps11 | 53.24 | 51.08 | 28.06 | 44.12 | 50.07 |
| rps8 | 43.70 | 40.74 | 25.93 | 36.79 | 46.22 |
| rpl14 | 47.15 | 37.40 | 29.27 | 37.94 | 52.74 |
| rps3 | 42.53 | 30.77 | 22.17 | 31.83 | 46.81 |
| rpl22 | 35.03 | 35.03 | 22.93 | 31.00 | 42.77 |
| rpl2 | 49.48 | 48.45 | 29.55 | 42.50 | 51.61 |
| ycf2 | 42.08 | 33.64 | 36.07 | 37.26 | 52.46 |
| ndhB | 42.07 | 38.55 | 31.90 | 37.51 | 48.72 |
| rps7 | 51.92 | 42.95 | 23.72 | 39.53 | 42.89 |
| ndhF | 37.37 | 35.35 | 23.25 | 31.99 | 44.55 |
| ccsA | 32.92 | 36.05 | 24.76 | 31.24 | 45.91 |
| ndhE | 37.25 | 31.37 | 25.49 | 31.37 | 43.40 |
| ndhG | 39.66 | 34.08 | 24.02 | 32.59 | 41.83 |
| ndhI | 44.24 | 36.97 | 23.03 | 34.75 | 45.57 |
| ndhA | 41.71 | 36.36 | 21.93 | 33.33 | 42.16 |
| ndhH | 49.24 | 36.55 | 26.40 | 37.39 | 47.94 |
| ycf1 | 34.03 | 27.24 | 24.43 | 28.57 | 47.31 |
| 平均值 Average |
46.23 | 38.68 | 27.62 | 37.51 | 47.02 |
秋茄叶绿体基因组密码子各参数的相关性分析结果显示(表 2),GC1、GC2、GC3和GCall两两之间的相关性均达到了极显著水平,说明密码子不同位置上的碱基组成十分相似。Nec与GC3之间相关系数为0.329,呈显著相关,说明GC3对密码子使用偏好性有影响。Nec与GCall和密码子个数(N)的相关系数分别为0.182和0.205,相关性较低,均未达到显著水平,说明编码区长度和GCall含量对密码子偏好性的影响较小。
| 因素Factor | GC1 | GC2 | GC3 | GCall | Nec |
| GC2 | 0.540** | ||||
| GC3 | 0.447** | 0.357* | |||
| GCall | 0.891** | 0.803** | 0.667** | ||
| Nec | 0.213 | -0.062 | 0.329* | 0.182 | |
| N | -0.119 | -0.280* | 0.237 | -0.114 | 0.205 |
| 注:**表示在0.01水平上极显著相关;*表示在0.05水平上显著相关。Note: ** extremely significant correlation at P < 0.01; * significant correlation at P < 0.05. | |||||
秋茄叶绿体各氨基酸相对同义密码子使用度分析结果表明(表 3),URSC>1的密码子有30个。其中,UUA密码子的URSC值最大,为2.05,以U结尾的密码子16个,以A结尾的密码子13个,以G结尾的密码子1个。这一结果进一步证明,秋茄叶绿体基因组密码子偏好使用以A/U结尾的密码子而非G/C结尾的密码子。
| 氨基酸 Amino acid |
密码子 Codon |
数量 Number |
URSC |
| 苯丙氨酸 Phe |
UUU | 785 | 1.35 |
| UUC | 381 | 0.65 | |
| 半胱氨酸 Cys |
UGU | 188 | 1.55 |
| UGC | 55 | 0.45 | |
| 谷氨酰胺 Gln |
CAA | 589 | 1.56 |
| CAG | 168 | 0.44 | |
| 缬氨酸 Val |
GUU | 424 | 1.49 |
| GUC | 138 | 0.48 | |
| GUA | 432 | 1.51 | |
| GUG | 148 | 0.52 | |
| 甘氨酸 Gly |
GGU | 455 | 1.3 |
| GGC | 158 | 0.45 | |
| GGA | 537 | 1.54 | |
| GGG | 246 | 0.7 | |
| 亮氨酸 Leu |
UUA | 740 | 2.05 |
| UUG | 439 | 1.22 | |
| CUU | 436 | 1.21 | |
| CUC | 129 | 0.36 | |
| CUA | 289 | 0.8 | |
| CUG | 129 | 0.36 | |
| 天冬氨酸 Asp |
GAU | 674 | 1.57 |
| GAC | 184 | 0.43 | |
| 组氨酸 His |
CAU | 382 | 1.54 |
| CAC | 113 | 0.46 | |
| 天冬酰胺 Asn |
AAU | 842 | 1.59 |
| AAC | 214 | 0.41 | |
| 脯氨酸 Pro |
CCU | 331 | 1.59 |
| CCC | 153 | 0.73 | |
| CCA | 250 | 1.2 | |
| CCG | 101 | 0.48 | |
| 丙氨酸 Ala |
GCU | 494 | 1.81 |
| GCC | 174 | 0.64 | |
| GCA | 304 | 1.11 | |
| GCG | 122 | 0.45 | |
| 丝氨酸 Ser |
UCU | 405 | 1.61 |
| UCC | 252 | 1 | |
| UCA | 306 | 1.22 | |
| UCG | 142 | 0.56 | |
| AGU | 317 | 1.26 | |
| AGC | 87 | 0.35 | |
| 谷氨酸 Glu |
GAA | 858 | 1.56 |
| GAG | 240 | 0.44 | |
| 酪氨酸 Tyr |
UAU | 636 | 1.66 |
| UAC | 131 | 0.34 | |
| 赖氨酸 Lys |
AAA | 871 | 1.56 |
| AAG | 245 | 0.44 | |
| 苏氨酸 Thr |
ACU | 421 | 1.63 |
| ACC | 184 | 0.71 | |
| ACA | 326 | 1.26 | |
| ACG | 100 | 0.39 | |
| 异亮氨酸 Ile |
AUU | 932 | 1.52 |
| AUC | 324 | 0.53 | |
| AUA | 579 | 0.95 | |
| 精氨酸 Arg |
CGU | 268 | 1.34 |
| CGC | 80 | 0.4 | |
| CGA | 280 | 1.4 | |
| CGG | 82 | 0.41 | |
| AGA | 372 | 1.86 | |
| AGG | 116 | 0.58 |
秋茄叶绿体基因组密码子使用的中性绘图分析结果如图 1所示,GC3的取值范围在0.215~0.361之间,GC12的取值范围在0.306~0.522之间。相关分析显示,GC3和GC12之间的相关系数为0.464 0,在0.01水平上相关性显著,回归系数为0.706 4,其中突变压力对密码子使用偏好性的效应占70.64%。由此可以看出,突变是秋茄叶绿体基因组密码子使用偏好性的重要因素。
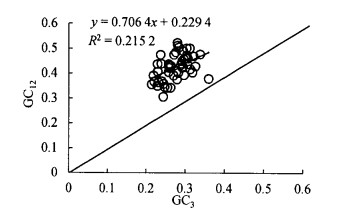
|
图 1 秋茄叶绿体基因组密码子使用的中性绘图分析 Fig. 1 Neutral-plot analysis of the codon usage of K. obovata |
在没有选择作用的情况下,基因会分布在标准曲线上或附近。Nec-plot绘图(图 2)显示,除了小部分基因分布在标准曲线上或附近外,大部分基因远离标准曲线,说明自然选择效应也是影响因素之一。
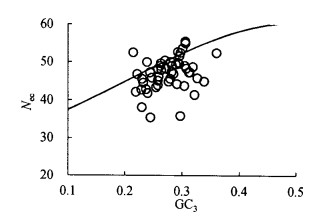
|
图 2 秋茄叶绿体基因组的有效密码子数绘图分析 Fig. 2 Nec-plot analysis of the codon usage of K. obovata |
秋茄叶绿体基因组密码子的奇偶偏好性分析结果(图 3)显示,平面上4个区域内基因分布并不均匀。G3/(G3+C3)小于0.5的基因有26个,A3/(A3+T3)小于0.5的基因有30个,说明密码子第3位上C的使用频率高于G,T的使用频率高于A,即秋茄叶绿体基因组密码子第3位上嘧啶使用频率高于嘌呤。如果只受到突变单一因素影响时,4种碱基会被均衡使用,而秋茄4种碱基使用不均衡,表明秋茄叶绿体基因组密码子使用的偏好性除了受到突变压力的影响外,还可能受到选择压力等其它因素的影响。
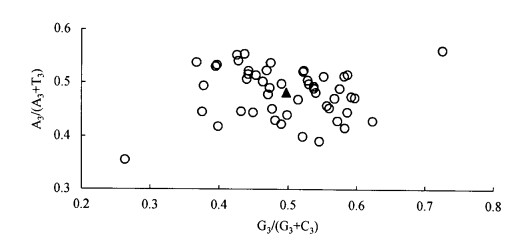
|
注:三角形表示每个基因平均分布位置,其值为x=0.497 224 43±0.081 186 424, y=0.481 708 133±0.046 540 165。 Note: the angle indicates the average position for each gene, calculated as follows: x=0.497 224 43±0.081 186 424, y=0.481 708 133±0.046 540 165. 图 3 秋茄叶绿体基因组密码子的奇偶偏好性分析 Fig. 3 PR2-plot analysis of codon usage of chloroplast genome of K. obovata |
根据Nec值大小,分别取最大的5条和最小的5条序列建立高低偏性库,并计算两库的ΔURSC值,其中ΔURSC值>0.3的密码子有UUU、CUC、CUG、GUC、GUG、UCA、ACC、ACA、GCA、UAU、UGC、CGA、AGA、GGC、GGA等共15个(表 4),其中AGA密码子ΔURSC值最大,为0.66。以A/U结尾的密码子有8个,以G/C结尾的密码子7个。结合URSC在高低偏性库中的值进行分析,最终得出3个最优密码子,分别是:ACC、ACA和GCA。其中Thr密码子2个(ACC和ACA),Ala密码子1个(GCA)。
| 氨基酸 Amino acid |
密码子 Codon |
高表达基因 High expression gene |
低表达基因 Low expression gene |
ΔURSC | |||
| URSC | 数目Number | URSC | 数目Number | ||||
| 苯丙氨酸 Phe |
UUU** | 1.52 | 19 | 1.18 | 36 | 0.34 | |
| UUC | 0.48 | 6 | 0.82 | 25 | -0.34 | ||
| 酪氨酸 Tyr |
UAU** | 1.70 | 23 | 1.38 | 18 | 0.32 | |
| UAC | 0.30 | 4 | 0.62 | 8 | -0.32 | ||
| 组氨酸 His |
CAU | 1.20 | 6 | 1.33 | 12 | -0.13 | |
| CAC* | 0.80 | 4 | 0.67 | 6 | 0.13 | ||
| 谷氨酰胺 Gln |
CAA | 1.45 | 24 | 1.69 | 22 | -0.24 | |
| CAG* | 0.55 | 9 | 0.31 | 4 | 0.24 | ||
| 天冬酰胺 Asn |
AAU* | 1.63 | 39 | 1.51 | 34 | 0.12 | |
| AAC | 0.38 | 9 | 0.49 | 11 | -0.11 | ||
| 赖氨酸 Lys |
AAA | 1.59 | 27 | 1.53 | 23 | 0.06 | |
| AAG | 0.41 | 7 | 0.47 | 7 | -0.06 | ||
| 天冬氨酸 Asp |
GAU | 1.42 | 22 | 1.48 | 17 | -0.06 | |
| GAC | 0.58 | 9 | 0.52 | 6 | 0.06 | ||
| 谷氨酸 Glu |
GAA | 1.66 | 39 | 1.65 | 33 | 0.01 | |
| GAG | 0.34 | 8 | 0.35 | 7 | -0.01 | ||
| 半胱氨酸 Cys |
UGU | 1.43 | 5 | 2.00 | 8 | -0.57 | |
| UGC*** | 0.57 | 2 | 0 | 0 | 0.57 | ||
| 缬氨酸 Val |
GUU | 1.30 | 14 | 1.77 | 27 | -0.47 | |
| GUC** | 0.47 | 5 | 0.13 | 2 | 0.34 | ||
| GUA | 1.49 | 16 | 1.84 | 28 | -0.35 | ||
| GUG** | 0.74 | 8 | 0.26 | 4 | 0.48 | ||
| 脯氨酸 Pro |
CCU* | 1.60 | 10 | 1.39 | 16 | 0.21 | |
| CCC | 0.64 | 4 | 0.78 | 9 | -0.14 | ||
| CCA | 1.28 | 8 | 1.22 | 14 | 0.06 | ||
| CCG | 0.48 | 3 | 0.61 | 7 | -0.13 | ||
| 苏氨酸 Thr |
ACU | 1.37 | 12 | 2.23 | 24 | -0.86 | |
| ACC*** | 1.26 | 11 | 0.74 | 8 | 0.52 | ||
| ACA** | 1.14 | 10 | 0.84 | 9 | 0.30 | ||
| ACG | 0.23 | 2 | 0.19 | 2 | 0.04 | ||
| 甘氨酸 Gly |
GGU | 1.07 | 11 | 1.80 | 31 | -0.73 | |
| GGC** | 0.98 | 10 | 0.52 | 9 | 0.46 | ||
| GGA** | 1.76 | 18 | 1.39 | 24 | 0.37 | ||
| GGG | 0.20 | 2 | 0.29 | 5 | -0.09 | ||
| 丙氨酸 Ala |
GCU | 1.55 | 26 | 2.39 | 37 | -0.84 | |
| GCC* | 0.66 | 11 | 0.39 | 6 | 0.27 | ||
| GCA** | 1.07 | 18 | 0.77 | 12 | 0.30 | ||
| GCG* | 0.72 | 12 | 0.45 | 7 | 0.27 | ||
| 异亮氨酸 Ile |
AUU | 1.55 | 43 | 1.88 | 54 | -0.33 | |
| AUC | 0.47 | 13 | 0.42 | 12 | 0.05 | ||
| AUA* | 0.98 | 27 | 0.70 | 20 | 0.28 | ||
| 亮氨酸 Leu |
UUA | 1.64 | 18 | 2.16 | 37 | -0.52 | |
| UUG | 1.18 | 13 | 1.69 | 29 | -0.51 | ||
| CUU* | 1.36 | 15 | 1.17 | 20 | 0.19 | ||
| CUC** | 0.45 | 5 | 0.06 | 1 | 0.39 | ||
| CUA | 0.73 | 8 | 0.82 | 14 | -0.09 | ||
| CUG*** | 0.64 | 7 | 0.12 | 2 | 0.52 | ||
| 丝氨酸 Ser |
UCU | 1.11 | 7 | 1.55 | 17 | -0.44 | |
| UCC | 0.79 | 5 | 1.18 | 13 | -0.39 | ||
| UCA** | 1.58 | 10 | 1.09 | 12 | 0.49 | ||
| UCG | 0.32 | 2 | 0.27 | 3 | 0.05 | ||
| AGU* | 1.58 | 10 | 1.36 | 15 | 0.22 | ||
| AGC* | 0.63 | 4 | 0.55 | 6 | 0.08 | ||
| 精氨酸 Arg |
CGU | 0.86 | 9 | 2.12 | 18 | -1.26 | |
| CGC | 0.29 | 3 | 0.47 | 4 | -0.18 | ||
| CGA*** | 1.81 | 19 | 1.18 | 10 | 0.63 | ||
| CGG | 0.38 | 4 | 0.35 | 3 | 0.03 | ||
| AGA*** | 2.19 | 23 | 1.53 | 13 | 0.66 | ||
| AGG* | 0.48 | 5 | 0.35 | 3 | 0.13 | ||
| 注:*表示0.08≤ΔURSC < 0.30,**表示0.30≤ΔURSC < 0.50,***表示ΔURSC≥0.50。Note: * means 0.08≤ΔURSC < 0.30, ** means 0.30≤ΔURSC < 0.50, *** means ΔURSC≥0.50. | |||||||
基于叶绿体基因组的rbcL基因序列,分别通过ML和NJ方法构建桐花树、白骨壤、银叶树、红榄李、榄李、红海榄、秋茄和木果楝等红树植物的系统发育树,经比较,两种方法构建的系统发育树的拓扑结构基本一致。基于NJ方法构建的系统发育树[图 4(a)]显示,同为使君子科(Combretaceae)的榄李和红榄李以及同为红树科(Rhizophoraceae)的秋茄和红海榄都以100%的支持率分别聚为一枝,梧桐科(Sterculiaceae)的银叶树和楝科(Meliaceae)的木果楝以67%的支持率聚在一起,紫金牛科(Myrsinaceae)的桐花树和马鞭草科(Verbenaceae)的白骨壤与其它红树植物的关系较远。基于密码子使用偏好性的聚类结果与上述系统发育树不完全一致,榄李与红榄李、秋茄与红海榄、银叶树与木果楝首先聚在一起。白骨壤先与银叶树和木果楝聚在一起后再与使君子科的2种植物聚为一枝,桐花树则先与红树科的2物种聚在一起后再与其它红树植物聚为一类[图 4(b)]。基于rbcL基因序列的系统进化树和基于URSC值的聚类分析有一定的相似性,表明密码子使用偏好性与物种之间的亲缘关系有关。

|
注:进化树分支上的数值表示置信度。 Note: the value on the branch of the phylogenetic tree represents the confidence. 图 4 8种红树植物的系统发育分析 Fig. 4 Phylogenetic analysis of 8 mangrove plants |
GC含量是生物体基因组碱基组成的重要指标,由于密码子第3位上受到的选择压力比前两位小,故GC3常被用做衡量密码子使用偏好性的指标[2]。对秋茄叶绿体基因组密码子不同位置上的GC含量分析发现,GC1(46.23%)>GC2(38.68%)>GC3(27.62%),说明秋茄叶绿体基因组偏好使用NNU和NNA类型的密码子,URSC分析结果也证明了这一点,在对普通油茶[12]、沙枣[14]、籽粒苋(Amaranthus hypochondriacus L.)[22]、菊科(Asteraceae)[23]等植物的研究中也发现,在这些植物的叶绿体基因组中偏好使用A/U结尾的密码子。各蛋白质编码基因的Nec取值介于35.40~55.50之间,平均值为47.02,显示秋茄叶绿体基因密码子偏性较弱。这与巨桉[13]、沙枣[14]、酸枣[15]等叶绿体密码子使用偏好性一致,这可能与叶绿体基因功能较为保守有关。密码子使用偏好性是由突变和自然选择相互作用的强度和方向决定的,相关性分析和中性绘图结果显示,GC3和GC1、GC2、GC12及GCall之间均极显著相关,说明秋茄叶绿体基因密码子前两位和第3位的碱基组成无显著差异,则暗示突变压力是影响密码子偏好性的重要因素[24]。PR2-plot分析显示,各基因在平面内的分布并不均匀,位于左下方的基因多于其他区域,说明秋茄叶绿体蛋白质编码基因密码子中第3位上嘧啶使用频率高于嘌呤,表明秋茄叶绿体基因密码子使用模式除了受到突变影响外,还可能受到自然选择等其它因素的影响,其密码子使用模式形成过程较为复杂,Nec-plot分析也证明了这一点。这与籽粒苋[22]、沙枣[14]、文心兰(Oncidium Gower Ramsey)[25]等植物叶绿体基因组的特征一致,而与普通油茶[12]、苦荞(Fagopyrum taraicum Gaerth)[26]、酸枣[15]等植物主要受自然选择作用的影响不同,也由此看出,造成不同植物密码子使用偏好性差异的因素也不尽相同。
比较基于rbcL基因序列的系统发育树和基于URSC值的聚类分析可以看出,亲缘关系较近的物种首先聚在一起,但对亲缘关系较远的物种,两者的分析结果并不一致,这可能与基于URSC的聚类分析只能在较小的分类单元中提供较为可靠的结果,而在较大的分类单元中只能作为传统分类的补充有关[27]。
秋茄叶绿体基因组密码子偏好使用以A/U结尾的密码子,密码子使用偏好性较低。突变是引起密码子使用偏好性的主要因素,除此之外,自然选择、碱基组成等其它方面的因素也发挥一定作用。密码子使用偏好性与物种之间的亲缘关系有关,经综合分析,确定了ACC、ACA和GCA等3个密码子为秋茄叶绿体基因组的最优密码子。
| [1] |
SHARP P M, COWE E. Synonymous codon usage in Saccharomyces cerevisiae[J]. Yeast, 1991, 7(7): 657-678. DOI:10.1002/yea.320070702 |
| [2] |
吴彦庆, 赵大秋, 陶俊. 芍药花色调控基因的密码子使用模式及其影响因素分析[J]. 中国农业科学, 2016, 49(12): 2368-2378. |
| [3] |
ANGELLOTTI M C, BHUIYAN S B, CHEN G R, et al. CodonO:codon usage bias analysis within and across genomes[J]. Nucleic Acids Research, 2007, 35(Web Server issue): 132-136. |
| [4] |
ROMERO H, ZAVALA A, MUSTO H. Codon usage in Chlamydia trachomatis is the result of strand-specific mutational biases and a complex pattern of selective forces[J]. Nucleic Acids Research, 2000, 28(10): 2084-2090. DOI:10.1093/nar/28.10.2084 |
| [5] |
刘庆坡, 薛庆中. 粳稻叶绿体基因组的密码子用法[J]. 作物学报, 2004, 30(12): 1220-1224. |
| [6] |
程琳, 瞿波, 李和平, 等. 植物叶绿体基因工程研究进展[J]. 华中农业大学学报, 2011, 30(2): 249-259. |
| [7] |
李宏韬, 赵淑青, 赵彦修, 等. 叶绿体基因工程简介[J]. 遗传, 2003, 25(4): 495-498. |
| [8] |
李轶女, 孙丙耀, 苏宁, 等. 水稻叶绿体表达体系的建立及抗PPT叶绿体转化植株的获得[J]. 中国农业科学, 2007, 40(9): 1849-1855. |
| [9] |
LEE S B, LI B C, JIN S X, et al. Expression and characterization of antimicrobial peptides Retrocyclin-101 and Protegrin-1 in chloroplasts to control viral and bacterial infections[J]. Plant Biotechnology Journal, 2011, 9(1): 100-115. |
| [10] |
赵雅坤, 史贤明, 张忠明. 人白细胞介素4在衣藻叶绿体中的高效转化及表达[J]. 华中农业大学学报, 2006, 25(2): 110-116. |
| [11] |
CHAKRABARTI S K, LUTZ K A, LERTWIRIYAWONG B, et al. Expression of the cry9Aa2 B.t. gene in tobacco chloroplasts confers resistance to potato tuber moth[J]. Transgenic Research, 2006, 15(4): 481-488. |
| [12] |
王鹏良, 杨利平, 吴红英, 等. 普通油茶叶绿体基因组密码子偏好性分析[J]. 广西植物, 2018, 38(2): 135-144. |
| [13] |
王鹏良, 吴双成, 杨利平, 等. 巨桉叶绿体基因组密码子偏好性分析[J]. 广西植物, 2019, 39(12): 1583-1592. |
| [14] |
王婧, 王天翼, 王罗云, 等. 沙枣叶绿体全基因组序列及其使用密码子偏性分析[J]. 西北植物学报, 2019, 39(9): 1559-1572. |
| [15] |
胡晓艳, 许艳秋, 韩有志, 等. 酸枣叶绿体基因组密码子使用偏性分析[J]. 森林与环境学报, 2019, 39(6): 621-628. |
| [16] |
原晓龙, 王毅, 张劲峰. 灰毛浆果楝叶绿体基因组密码子使用特征分析[J]. 森林与环境学报, 2020, 40(2): 195-202. |
| [17] |
杜照奎, 陈河, 李钧敏. 红树植物秋茄分子生物学研究进展[J]. 生物工程学报, 2017, 33(2): 196-204. |
| [18] |
林鹏. 中国红树林研究进展[J]. 厦门大学学报(自然科学版), 2001, 40(2): 592-603. |
| [19] |
BASYUNI M, OKU H, INAFUKU M, et al. Molecular cloning and functional expression of a multifunctional triterpene synthase cDNA from a mangrove species Kandelia candel (L.) Druce[J]. Phytochemistry, 2006, 67(23): 2517-2524. DOI:10.1016/j.phytochem.2006.09.016 |
| [20] |
李志刚, 戴建青, 叶静文, 等. 中国红树林生态系统主要害虫种类、防控现状及成灾原因[J]. 昆虫学报, 2012, 55(9): 1109-1118. |
| [21] |
YANG X, LUO X N, CAI X P. Analysis of codon usage pattern in Taenia saginata based on a transcriptome dataset[J]. Parasites & Vectors, 2014, 7(1): 527. |
| [22] |
冯瑞云, 梅超, 王慧杰, 等. 籽粒苋叶绿体基因组密码子偏好性分析[J]. 中国草地学报, 2019, 41(4): 8-15. |
| [23] |
NIE X J, DENG P C, FENG K W, et al. Comparative analysis of codon usage patterns in chloroplast genomes of the Asteraceae family[J]. Plant Molecular Biology Reporter, 2014, 32(4): 828-840. DOI:10.1007/s11105-013-0691-z |
| [24] |
罗火林, 张亚楠, 罗丽萍, 等. 兰科植物FT基因的密码子偏性分析[J]. 分子植物育种, 2016, 14(1): 51-58. |
| [25] |
李冬梅, 吕复兵, 朱根发, 等. 文心兰叶绿体基因组密码子使用的相关分析[J]. 广东农业科学, 2012, 39(10): 61-65. |
| [26] |
胡莎莎, 罗洪, 吴琦, 等. 苦荞叶绿体基因组密码子偏爱性分析[J]. 分子植物育种, 2016, 14(2): 309-317. |
| [27] |
赵春丽, 彭丽云, 王晓, 等. 苋菜AtGAI基因密码子偏好性与进化分析[J]. 中国农业大学学报, 2019, 24(12): 10-22. |

 2020, Vol. 40
2020, Vol. 40

