文章信息
- 杜超群, 许业洲, 孙晓梅
- DU Chaoqun, XU Yezhou, SUN Xiaomei
- 日本落叶松种子园群体遗传多样性评价
- Genetic diversity evaluation of Larix kaempferi population in seed orchard
- 森林与环境学报,2020, 40(4): 406-411.
- Journal of Forest and Environment,2020, 40(4): 406-411.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2020.04.010
-
文章历史
- 收稿日期: 2020-04-21
- 修回日期: 2020-05-06
2. 中国林业科学研究院林木遗传育种国家重点实验室, 北京 100091
2. State Key Laboratory of Tree Genetics and Breeding, Chinese Academy of Forestry, Beijing 100091, China
种子园的建立是针叶树育种最有效的方法之一。种子园通过有性过程生产改良种子,既要获得较高的遗传增益,又必需保持较宽的遗传基础[1],保证所生产的种子具有一定的遗传多样性,这对确保种子育苗所营造的林分具有较强的适应性,尤其对避免极端气候等环境因子造成林分损失至关重要[2]。
日本落叶松[Larix kaempferi (Lamb.) Carr.]在我国已有一百多年的引种栽培历史,是我国北方重要的用材树种之一,目前已成为鄂西亚高山地区的主要造林树种。湖北省建始县长岭岗林场是我国南方唯一的日本落叶松国家良种基地,该基地营建的日本落叶松种子园是当地及周边地区主要的种子来源地,近20 a累计供应良种超过3 000 kg,大大提高了该区域日本落叶松人工林的良种化水平[3]。针对日本落叶松种子园的早期研究大多是基于表型数据的评估和选择[3-5]。由于建园的材料来源于多个省份,可推测其遗传多样性十分丰富,但其遗传多样性的现状及特点目前尚未有研究报道。目前日本落叶松种子园仍然同时具有生产群体和育种群体的功能,只有清楚地掌握种子园群体的遗传基础和遗传结构,才能确保大规模人工造林的生态安全,才能有效地开展具有发展潜力的新品种创制[6]。
随着生物技术的不断发展,分子标记已成为生物信息学最重要的研究手段之一。利用分子标记技术可以实现物种各群体遗传学参数及亲缘关系的快速分析[6],已被普遍应用于群体遗传多样性和遗传结构等研究中[7],其中简单重复序列(simple sequence repeat, SSR)标记由于具有操作简单、多态性高、信息量大和共显性等优越性,且在种间的通用性高,能够很好地满足遗传多样性等研究要求[8-9]。目前,虽然SSR标记已被用于日本落叶松遗传多样性的研究中[10-12],但报道不多,而且以往的研究所涉及的SSR标记数量较少。本研究以湖北省建始县长岭岗林场日本落叶松改良种子园无性系为对象,利用本课题组开发出来的、多态性较好的16个表达序列标签SSR (expressed sequence tag-SSR, EST-SSR)标记进行遗传多样性分析和评价,旨在明确种子园的遗传基础,为后期育种策略的制定和遗传改良的可持续发展提供参考依据。
1 材料与方法 1.1 试验材料日本落叶松改良种子园位于湖北省建始县长岭岗林场日本落叶松国家良种基地,北纬30°48′,东经110°03′,海拔1 600~1 720 m,属内陆性冷凉气候带高山多雨潮湿区,年平均气温7~8 ℃,平均最低气温-9.4 ℃,年降水量1 600~1 800 mm,土壤为山地黄棕壤。该种子园的建园时间为1989—1997年,面积49.5 hm2,是在初级种子园的基础上通过去劣留优改建而成的,建园的材料为来自辽宁、山东、内蒙古、甘肃及本场母树林的优树穗条,以2年生实生苗为砧木嫁接成无性系,初植密度为416~625株· hm-2。2008年,根据生长和结实情况对种子园进行去劣疏伐,保留密度为250~318株· hm-2,保存无性系145个,不同来源的群体分别标记为湖北(Hubei, HB)、辽宁(Liaoning, LN)、内蒙古(Inner Mongolia, IM)、山东(Shandong, SD)、甘肃(Gansu, GS),具体情况如表 1所示。
| 群体Population | 来源Source | 无性系数量Number of clones |
| HB | 长岭岗林场Changlinggang Forest Farm | 35 |
| LN | 草河口林场、朝阳林场、土壤研究所 Caohekou Forest Farm, Chaoyang Forest Farm, Institute of Soil |
52 |
| IM | 旺业甸林场Wangyedian Forest Farm | 18 |
| SD | 天麻林场Tianma Forest Farm | 27 |
| GS | 小陇山林业实验局Xiaolongshan Forestry Experimental Bureau | 13 |
采集种子园145个无性系(每系号1株)的嫩叶,硅胶干燥后置于超低温冰箱(-80 ℃)中保存备用。采用改良的十六烷基三甲基溴化铵(cetyltrimethylammonium ammonium bromide, CTAB)法提取基因组DNA[10]。试验所用的16个SSR标记见表 2,引物信息与CHEN et al[12]相同,由北京睿博兴科生物技术有限公司合成。标记扩增和数据读取整理等参照CHEN et al[12]的方法进行。
| 位点 Locus |
观测等位基因数 Na |
有效等位基因数 Ne |
观测杂合度 Ho |
期望杂合度 He |
Shannon多样性指数I | 多态性信息含量指数 PIC |
| Y27 | 6 | 2.319 | 0.587 | 0.566 | 0.971 | 0.505 |
| QL397 | 12 | 5.831 | 1.000 | 0.828 | 1.966 | 0.818 |
| QL386 | 12 | 4.949 | 0.821 | 0.790 | 1.857 | 0.792 |
| QL375 | 3 | 1.759 | 0.444 | 0.425 | 0.727 | 0.383 |
| QL322 | 2 | 1.815 | 0.528 | 0.448 | 0.641 | 0.354 |
| OH299 | 6 | 2.713 | 0.606 | 0.610 | 1.204 | 0.628 |
| HL391 | 4 | 1.192 | 0.173 | 0.158 | 0.315 | 0.156 |
| HL215 | 3 | 1.952 | 0.590 | 0.487 | 0.727 | 0.381 |
| H339 | 3 | 1.276 | 0.205 | 0.209 | 0.370 | 0.175 |
| H233 | 5 | 2.332 | 0.597 | 0.563 | 0.996 | 0.528 |
| H217 | 4 | 2.832 | 0.652 | 0.646 | 1.081 | 0.574 |
| H197 | 2 | 1.606 | 0.288 | 0.364 | 0.545 | 0.318 |
| H177 | 3 | 1.901 | 0.469 | 0.472 | 0.708 | 0.387 |
| H46 | 4 | 2.614 | 0.617 | 0.614 | 1.034 | 0.562 |
| H52 | 5 | 2.925 | 1.000 | 0.657 | 1.185 | 0.608 |
| H140 | 8 | 2.658 | 0.666 | 0.619 | 1.220 | 0.596 |
| 均值Mean | 5.125 | 2.542 | 0.578 | 0.528 | 0.972 | 0.485 |
| 标准差SE | 0.274 | 0.141 | 0.028 | 0.021 | 0.052 |
利用GenAlex 6.41软件进行数据分析,包括遗传参数的估算、各位点哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)卡方检验、分子方差分析(analysis of molecular variance, AMOVA)、群体间遗传距离和遗传相似性以及主坐标分析(principal coordinates analysis, PCoA)。估算的遗传参数有:观测等位基因数(number of observed alleles, Na)、有效等位基因数(number of effective alleles, Ne)、观测杂合度(observed heterozygosity, Ho)、期望杂合度(expected heterozygosity, He)、Shannon多样性指数、群体内近交系数(inbreeding coefficient in population, Fis)、群体总近交系数(total inbreeding coefficient of population, Fit)、基因分化系数(genetic differentiation coefficient, Fst)、基因流(gene flow, Nm)。利用Power Marker version 3.25软件计算群体多态性信息含量指数(polymorphism information content index, PIC)。
2 结果与分析 2.1 简单重复序列位点多态性分析16对引物的多态性表现见表 2。145份材料共检测到82个等位基因,每个位点的Na为2~12个不等,平均Ne为2.542,平均Ho为0.578,平均He为0.528,平均Shannon多样性指数为0.972。其中QL397位点检测到的等位基因数最多,Ne和He最大(分别为5.831和0.828),Shannon多样性指数最高(1.966);HL391位点的Ne和He最小(分别为1.192和0.158),Shannon多样性指数最低(0.315), 说明各个位点对研究群体遗传多样性的检测能力和效果不同。16个位点的PIC在0.156~0.818之间,平均值为0.485,其中PIC大于0.500的位点有9个,为高度多态性位点,表明所选SSR引物能有效揭示群体遗传多样性。
2.2 位点哈迪-温伯格平衡卡方检验16个SSR位点的F统计量检测结果显示(表 3),日本落叶松改良种子园的Fst平均值为0.023,说明群体间遗传分化程度较小;Fis、Fit均为负值,群体表现为轻微杂合子过量,不存在近交衰退;Nm平均值为13.932,远高于自然群体,也说明遗传分化较小[2]。
| 位点Locus | 群体内近交系数Fis | 群体总近交系数Fit | 基因分化系数Fst | 基因流Nm |
| Y27 | -0.037 | -0.020 | 0.016 | 15.494 |
| QL397 | -0.208 | -0.192 | 0.014 | 18.191 |
| QL386 | -0.039 | -0.016 | 0.023 | 10.753 |
| QL375 | -0.045 | -0.025 | 0.019 | 12.737 |
| QL322 | -0.177 | -0.111 | 0.056 | 4.199 |
| OH299 | 0.006 | 0.053 | 0.047 | 5.039 |
| HL391 | -0.096 | -0.083 | 0.012 | 20.360 |
| HL215 | -0.212 | -0.189 | 0.019 | 12.679 |
| H339 | 0.021 | 0.042 | 0.021 | 11.662 |
| H233 | -0.059 | -0.042 | 0.015 | 15.899 |
| H217 | -0.009 | 0.000 | 0.009 | 28.175 |
| H197 | 0.208 | 0.243 | 0.045 | 5.335 |
| H177 | 0.007 | 0.037 | 0.030 | 8.059 |
| H46 | -0.004 | 0.011 | 0.015 | 16.432 |
| H52 | -0.523 | -0.486 | 0.024 | 10.039 |
| H140 | -0.076 | -0.066 | 0.009 | 27.867 |
| 均值Mean | -0.078 | -0.053 | 0.023 | 13.932 |
| 标准差SE | 0.039 | 0.038 | 0.004 | 1.808 |
分别对不同来源的5个群体进行HWE卡方检验(表 4),整体来看,5个群体中大部分位点均符合HWE (P≥0.05),其中GS群体所有位点均符合,HB和IM群体均有1个位点偏离平衡,LN群体中偏离平衡的位点最多(4个)。另外分析结果还发现H52位点在4个群体中均显著偏离HWE。
| 位点 Locus |
群体 Population |
χ2检验值 Chi-square test value |
P值 P value |
| Y27 | HB | 5.754 | 0.124 |
| LN | 5.780 | 0.833 | |
| IM | 3.000 | 0.809 | |
| SD | 3.426 | 0.754 | |
| GS | 0.738 | 0.864 | |
| QL397 | HB | 63.606 | 0.198 |
| LN | 75.889 | 0.003 | |
| IM | 24.000 | 0.996 | |
| SD | 56.784 | 0.408 | |
| GS | 21.419 | 0.434 | |
| QL386 | HB | 31.601 | 0.935 |
| LN | 56.346 | 0.120 | |
| IM | 37.957 | 0.762 | |
| SD | 37.776 | 0.769 | |
| GS | 27.141 | 0.511 | |
| QL375 | HB | 2.951 | 0.399 |
| LN | 2.835 | 0.418 | |
| IM | 1.045 | 0.790 | |
| SD | 1.343 | 0.719 | |
| GS | 4.116 | 0.249 | |
| QL322 | HB | 0.015 | 0.901 |
| LN | 0.023 | 0.880 | |
| IM | 3.485 | 0.062 | |
| SD | 6.750 | 0.009 | |
| GS | 0.008 | 0.928 | |
| OH299 | HB | 11.685 | 0.307 |
| LN | 8.960 | 0.536 | |
| IM | 16.925 | 0.323 | |
| SD | 8.793 | 0.552 | |
| GS | 2.866 | 0.825 | |
| HL391 | HB | 0.207 | 0.649 |
| LN | 1.061 | 0.983 | |
| IM | 0.149 | 0.985 | |
| SD | 0.040 | 0.842 | |
| GS | 0.221 | 0.638 | |
| HL215 | HB | 2.793 | 0.425 |
| LN | 3.918 | 0.048 | |
| IM | 0.281 | 0.596 | |
| SD | 2.390 | 0.495 | |
| GS | 1.364 | 0.714 | |
| H339 | HB | 0.308 | 0.579 |
| LN | 1.828 | 0.176 | |
| IM | 0.149 | 0.700 | |
| SD | 4.683 | 0.197 | |
| GS | 0.737 | 0.391 | |
| H233 | HB | 7.778 | 0.255 |
| LN | 3.852 | 0.697 | |
| IM | 1.858 | 0.602 | |
| SD | 2.113 | 0.909 | |
| GS | 1.003 | 0.985 | |
| H217 | HB | 1.261 | 0.738 |
| LN | 4.747 | 0.191 | |
| IM | 2.612 | 0.856 | |
| SD | 0.836 | 0.841 | |
| GS | 2.649 | 0.449 | |
| H197 | HB | 2.472 | 0.116 |
| LN | 0.063 | 0.801 | |
| IM | 0.126 | 0.723 | |
| SD | 10.547 | 0.001 | |
| GS | 0.860 | 0.354 | |
| H177 | HB | 6.355 | 0.096 |
| LN | 3.756 | 0.053 | |
| IM | 0.000 | 0.990 | |
| SD | 1.452 | 0.693 | |
| GS | 0.090 | 0.764 | |
| H46 | HB | 4.861 | 0.562 |
| LN | 3.754 | 0.710 | |
| IM | 1.419 | 0.701 | |
| SD | 9.207 | 0.162 | |
| GS | 0.897 | 0.826 | |
| H52 | HB | 24.861 | 0.006 |
| LN | 52.466 | 0.000 | |
| IM | 15.149 | 0.019 | |
| SD | 27.000 | 0.003 | |
| GS | 8.233 | 0.222 | |
| H140 | HB | 6.778 | 0.964 |
| LN | 17.811 | 0.661 | |
| IM | 6.396 | 0.781 | |
| SD | 6.687 | 0.755 | |
| GS | 3.115 | 0.979 |
5个群体的遗传多样性参数见表 5。群体之间各参数值相差不大,Ne在2.363~2.675之间,Ho为0.559~0.600,He为0.507~0.545,平均Shannon多样性指数为0.933~1.007。在遗传多样性评价中,多用Shannon多样性指数、He和Ne这3个指标来衡量群体遗传多样性的大小[2, 6, 9]。相对来说,LN群体的Ne和Shannon多样性指数最高,IM群体最低;HB和LN群体的He相等,在5个群体中最高,IM群体仍为最低。按照Shannon多样性指数值进行排序,5个群体遗传多样性从高到低的顺序为:LN>HB>SD>GS>IM。
| 群体Population | 观测等位基因数Na | 有效等位基因数Ne | 观测杂合度Ho | 期望杂合度He | Shannon多样性指数I |
| HB | 4.250 | 2.588 | 0.600 | 0.545 | 0.998 |
| LN | 4.375 | 2.675 | 0.592 | 0.545 | 1.007 |
| IM | 4.063 | 2.363 | 0.559 | 0.507 | 0.933 |
| SD | 4.313 | 2.581 | 0.565 | 0.518 | 0.977 |
| GS | 3.563 | 2.503 | 0.572 | 0.527 | 0.943 |
通过计算群体间遗传距离和遗传相似性可知(表 6):5个群体遗传距离较小,在0.002~0.015之间,遗传相似性的平均值为0.992。其中遗传距离最小的群体为SD和GS、HB和LN,遗传距离最大的为HB和IM之间。AMOVA分析结果表明,群体间的变异仅占1.71%,98.29%的变异存在于群体内,说明群体间的遗传分化非常小。基于SSR遗传距离的PCoA分析结果如图 1所示。5个群体的分布区域相互交叉重叠,群体之间没有明显的聚集,甚至可以观察到来自不同群体的个体有重合现象,说明遗传基础比较相似。
| 群体 Population |
HB | LN | IM | SD | GS |
| HB | 0.998 | 0.985 | 0.990 | 0.989 | |
| LN | 0.002 | 0.992 | 0.989 | 0.991 | |
| IM | 0.015 | 0.008 | 0.994 | 0.996 | |
| SD | 0.010 | 0.011 | 0.006 | 0.998 | |
| GS | 0.011 | 0.009 | 0.004 | 0.002 |
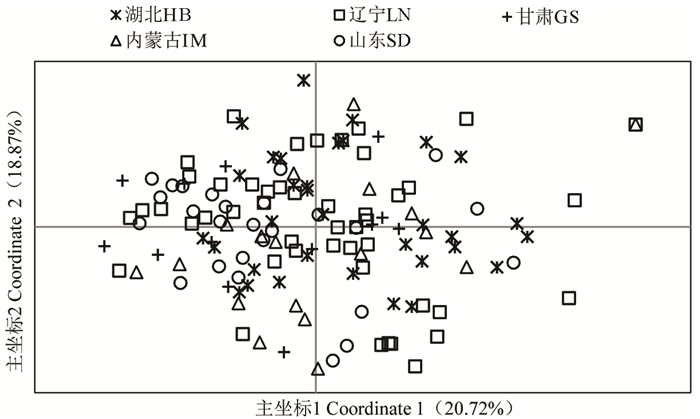
|
图 1 基于遗传距离的主坐标分析 Fig. 1 PCoA analysis based on genetic distance |
日本落叶松原产地位于日本本州岛中部山区,欧洲和北美均有引种,我国是最早引种的国家之一,山东省是我国有历史记载引种最早的省份,辽宁、吉林、甘肃、内蒙古、黑龙江、河南、四川、湖北、湖南等地均有大规模引种栽培[13]。目前国内所有的资源均为20世纪引种资源,虽然日本落叶松育种已进入第二代,但是二代育种群体主要来源于种子园子代测定林[14],二代生产群体(二代种子园)还未进入生产阶段,因此,目前改良代种子园仍然是我国日本落叶松人工林良种的主要来源,也是国内种质资源遗传基础的重要保障。
松属树种的遗传多样性研究已获得较大的进展,马尾松(Pinus massoniana Lamb.)种子园Shannon多样性指数为0.468~0.477[15],湿加松(Pinus elliottii×P. oaribaea)亲本群体平均杂合度为0.144~0.884[16],白皮松(Pinus bungeana Zucc.)天然群体He和Shannon多样性指数分别为0.118~0.333和0.195~0.495[17],红松(Pinus koraiensis Sieb. et Zucc.)天然群体及种子园Shannon多样性指数分别为0.262和0.267[18],华北落叶松(Larix principis-rupprechtii Mayr.) 1~3代种子园的He和Shannon多样性指数分别为0.418~0.479和0.705~0.791[2]。本研究对日本落叶松种子园145个无性系进行了遗传多样性分析,发现每个标记检测到的等位基因数目为2~12个,平均Ne为2.542,平均Ho为0.578,平均He为0.528,平均Shannon多样性指数为0.972,平均PIC为0.485。与其他松属树种相比,日本落叶松种子园遗传多样性相对较高。
一般来说,地理环境比如海拔、经纬度等的差异容易使同一物种不同居群之间产生明显的遗传分化[9],如万爱华等[15]对来自8个省份的马尾松种子园无性系进行研究,发现亲缘关系的远近与纬度和地理距离的相关性均比较明显;王小平等[19]、赵罕等[17]分析了白皮松群体的地理变异规律,无论是表型性状(球果、种子)还是遗传结构均存在明显的差异。日本落叶松种子园不同来源的群体按照Shannon多样性指数进行排序,从高到低的顺序为:LN>HB>SD>GS>IM,但是群体间遗传多样性各参数值差异不大;计算群体间的遗传距离发现, 5个群体之间遗传距离较小(0.002~0.015之间)。分子方差分析结果表明,遗传变异主要存在于群体内,仅有1.71%存在于群体间,说明群体间的遗传分化非常小;PCoA分析结果更为直观地反映出5个群体遗传基础相似,没有明显的聚类。种子园中的材料虽然引自5个不同省份,但是日本落叶松作为外来树种,直接从原产地引种的机会较少,国内种植材料引种交流非常频繁,因此遗传背景逐渐趋于类似,检测不到明显的分化。
在多世代育种进程中,维持育种群体较宽的遗传基础对于提升育种效果至关重要[10]。虽然国内日本落叶松种子园群体遗传多样性较高,但是与原产地相比(He为0.717~0.795,PIC为0.720) [11],还有很大的提升空间,从育种区外或者说原产地引进优良资源补充进入育种群体,可以有效提高遗传多样性,拓宽遗传基础。另外,遗传多样性随着遗传改良进程的变化状况也是育种者和生产者十分关注的问题,下一步可开展相关研究和探讨。
| [1] |
李义良, 赵奋成, 张应中, 等. 分子标记在松树遗传与进化研究中的应用[J]. 分子植物育种, 2009, 7(5): 1004-1009. |
| [2] |
于大德, 袁定昌, 张登荣, 等. 华北落叶松种子园不同世代间遗传多样性变化[J]. 植物遗传资源学报, 2014, 15(5): 940-947. |
| [3] |
杜超群, 赵虎, 袁慧, 等. 日本落叶松种子园母树生长及种实性状评价[J]. 森林与环境学报, 2019, 39(1): 32-36. |
| [4] |
周旭昌. 日本落叶松无性系种子园母树生长性状聚类分析[J]. 北华大学学报(自然科学版), 2014, 15(4): 531-534. |
| [5] |
隋娟娟, 张卓文, 李时元, 等. 长岭岗日本落叶松种子园不同家系种子品质的研究[J]. 种子, 2008, 27(10): 108-112. |
| [6] |
卢万鸿.尾叶桉多世代育种群体的遗传多样性及其改良性状的关联分析[D].北京: 中国林业科学研究院, 2017. http://cdmd.cnki.com.cn/Article/CDMD-82201-1017270617.htm
|
| [7] |
陈新民, 何中虎, 史建荣, 等. 利用SSR标记进行优质冬小麦品种(系)的遗传多样性研究[J]. 作物学报, 2003, 29(1): 13-19. |
| [8] |
DU Q Z, WANG B W, WEI Z Z, et al. Genetic diversity and population structure of Chinese white poplar(Populus tomentosa) revealed by SSR markers[J]. The Journal of Heredity, 2012, 103(6): 853-862. |
| [9] |
包文泉, 乌云塔娜, 杜红岩, 等. 基于SSR标记的西藏光核桃群体遗传多样性和遗传结构分析[J]. 林业科学, 2018, 54(2): 30-41. |
| [10] |
杨秀艳, 孙晓梅, 张守攻, 等. 日本落叶松EST-SSR标记开发及二代优树遗传多样性分析[J]. 林业科学, 2011, 47(11): 52-58. |
| [11] |
NISHIMURA M, SETOGUCHI H. Homogeneous genetic structure and variation in tree architecture of Larix kaempferi along altitudinal gradients on Mt. Fuji[J]. Journal of Plant Research, 2011, 124(2): 253-263. |
| [12] |
CHEN X B, SUN X M, DONG L M, et al. Mating patterns and pollen dispersal in a Japanese larch (Larix kaempferi) clonal seed orchard:a case study[J]. Science China Life Sciences, 2018, 61(9): 1011-1023. |
| [13] |
田志和. 日本落叶松育种学[M]. 北京: 北京农业大学出版社, 1995.
|
| [14] |
杨秀艳, 张守攻, 孙晓梅, 等. 北亚热带高山区日本落叶松自由授粉家系遗传测定与二代优树选择[J]. 林业科学, 2010, 46(8): 45-50. |
| [15] |
万爱华, 徐有明, 管兰华, 等. 马尾松种子园无性系遗传结构的地理变异[J]. 东北林业大学学报, 2006, 34(4): 12-14. |
| [16] |
李义良, 赵奋成, 吴惠姗, 等. 湿加松亲本间遗传距离与杂种优势的相关性分析[J]. 林业科学研究, 2012, 25(2): 138-143. |
| [17] |
赵罕, 郑勇奇, 李斌, 等. 白皮松天然群体遗传结构的地理变异分析[J]. 植物遗传资源学报, 2013, 14(3): 395-401. |
| [18] |
冯富娟, 张冬东, 韩士杰. 红松种子园优良无性系的遗传多样性[J]. 东北林业大学学报, 2007, 35(9): 9-11. |
| [19] |
王小平, 刘晶岚, 王九龄. 白皮松种子及球果形态特征的地理变异[J]. 北京林业大学学报, 1998, 20(3): 25-31. |

 2020, Vol. 40
2020, Vol. 40


