文章信息
- 胡晓艳, 许艳秋, 韩有志, 杜淑辉
- HU Xiaoyan, XU Yanqiu, HAN Youzhi, DU Shuhui
- 酸枣叶绿体基因组密码子使用偏性分析
- Codon usage bias analysis of the chloroplast genome of Ziziphus jujuba var. spinosa
- 森林与环境学报,2019, 39(6): 621-628.
- Journal of Forest and Environment,2019, 39(6): 621-628.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.06.009
-
文章历史
- 收稿日期: 2019-08-15
- 修回日期: 2019-09-12
自然界存在的20种氨基酸(蛋氨酸和色氨酸由1个密码子编码)均由2-6个密码子编码[1]。这些编码同一种氨基酸的密码子称为同义密码子。在没有基因突变和进化压力等因素的影响下,信使核糖核酸(message ribonucleic acid,mRNA)在翻译时同义密码子的使用频率应相等[2],但是在实际情况中,同义密码子的使用频率是不相同的,表现出很大的差异性,这就形成了同义密码子使用偏性(codon usage bias,CUB),即某物种或某个基因偏向使用一种或几种特定的同义密码子[3]。密码子使用偏性因物种、组织、基因的差异而表现出多样性[4],容易在高表达基因中检测到[5],也会对mRNA翻译的速率、效率以及多肽链的折叠产生影响[6-7]。分析密码子的使用偏性及其影响因素不但可以确定最优密码子,从而进一步设计基因表达载体,提高目的基因的表达量,而且有助于研究生物适应外界环境的分子机制。
叶绿体基因组主要编码与植物光合作用相关的蛋白质[8]。对植物叶绿体基因组密码子使用模式进行分析,可为阐明叶绿体分子的进化特征提供理论参考。目前关于叶绿体基因组密码子使用模式的研究,已在陆地棉(Gossypium hirsutum L.)[9]、樟树[Cinnamomum camphora (L.) Presl.][10]和竹亚科(Bambusoideae)[1]等多种植物上开展。酸枣[Ziziphus jujuba Mill. var. spinosa (Bunge) Hu ex H. F. Chow]为鼠李科(Rhamnaceae)枣属(Ziziphus)植物,是枣的变种。酸枣作为中药应用已有2 000多年的历史,所含生物活性物质如酸枣多糖、黄酮类等,对人体有多种功效[11]。目前,酸枣叶绿体基因组序列已经公布,酸枣叶绿体基因组全长161 211 bp,包含85个蛋白质编码基因,8个核糖体RNA(ribosomal RNA,rRNA)及37个转运RNA(transfer RNA,tRNA)[12]。以美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)中登录的酸枣叶绿体基因组为研究对象,讨论酸枣叶绿体基因组密码子使用模式及其影响因素,将为明确酸枣的系统发育位置,叶绿体基因组的遗传变异及基因功能提供重要的理论依据。
1 材料与方法 1.1 数据来源从NCBI数据库中获取酸枣叶绿体基因组序列(KX266830)。为减少分析误差,删除长度小于300 bp的编码序列(coding sequence,CDS)及重复序列,最终获得58条序列用于后续分析。
1.2 研究方法 1.2.1 碱基组成和同义密码子偏好性分析利用CodonW 1.4.2软件对酸枣叶绿体基因组58条序列进行密码子组成和偏好性参数分析。密码子组成的度量指标包括:同义密码子在第3位上胸腺嘧啶(thymine,T)、胞嘧啶(cytosine,C)、腺嘌呤(adenine,A)、鸟嘌呤(guanine,G)的出现频率(T3S、C3S、A3S、G3S),同义密码子第3位的G+C含量(GC3),密码子中总的鸟嘌呤和胞嘧啶含量(GC),芳香族氨基酸频率(aromatic amino acid frequency,Aro)、密码子适应指数(codon adaptation index,CAI)、密码子偏好指数(codon bias index,CBI)、最优密码子频率(frequency of optimal codons,Fop)、氨基酸长度(length of amino acid,Laa)等。密码子使用偏好性的度量指标包括同义密码子相对使用度(relative synonymous codon usage,URSC)和有效密码子数(effective number of codons,Nec)。URSC是指对于某个特定的密码子,在编码相对应的氨基酸时,该密码子在所有同义密码子中的相对概率,计算时去除氨基酸组成对密码子使用产生的影响。URSC为某一特定密码子的使用频率与其在无偏好性使用时预期频率间的比值[13-14]。若URSC=1,则该密码子使用无偏好性;若URSC>1,则该密码子使用频繁;若URSC < 1,则该密码子使用频率较低[1]。Nec取值范围为20~60,是检测单个基因密码子使用偏好程度的指标。当Nec=20时,表示在某一特定基因中,每个氨基酸都只使用1个特定的密码子;当Nec=61时,每个氨基酸的各个同义密码子均衡使用,即Nec值越大,密码子使用的随机性越高,使用偏性越弱[15]。
1.2.2 密码子使用偏性影响因素分析对GC1和GC2的平均值(GC12)与GC3的相关性进行分析,解析密码子3个位置碱基组成的差异性[16]。二者显著相关说明,密码子3个碱基的变异模式相同,突变对密码子的使用影响较大;反之则表示基因保守性较高,选择压力对密码子使用影响大。
以各基因GC3和Nec的值分别作X和Y轴,衡量各基因GC3对同义密码子使用的影响。当没有选择压力时,Nec=2.5-GC3+29.5/[GC32+(1-GC3)2],当密码子使用仅受GC3影响时,其Nec值将分布于期望值曲线上或附近;当受到较强选择作用时,其Nec值将分布在曲线下方较远的位置[17]。
根据△URSC法预测最优密码子,将各基因按Nec值大小排序,取两端各10%的基因组成高样本组与低样本组,比较2组数据中各基因密码子的URSC值。若△URSC>0.08,且该密码子的URSC值在高样本组中>1,在低样本组中 < 1,则该密码子为最优密码子[8]。
为衡量AT或GC组成偏差是否对密码子使用造成影响,选择4种密码子编码的氨基酸进行密码子第3位上4种碱基组成情况分析。分别以A3/(A3+T3)/4和G3/(G3+C3)/4为横、纵坐标作图,从图的中心点(0.5, 0.5,表示A=T且C=G)发出的矢量表示密码子使用偏性的方向和程度[16]。
为阐明密码子使用模式的变异趋势,基于URSC值,用CodonW 1.4.2软件进行对应性分析。经软件处理后所有基因分布于一个57维的向量空间中,根据基因在向量空间中的相对位置可判断影响密码子使用偏性的主要因素。基因在主向量轴(第1、2轴)之间的分散程度,代表了密码子的主要变化趋势,可作为推断其密码子使用变异的依据[18]。
2 结果与分析 2.1 酸枣叶绿体基因组密码子组成和偏好性分析在NCBI下载的酸枣叶绿体基因组(KX266830)去除长度小于300 bp的CDS及重复序列后,剩余CDS全长70 699 bp(占基因组全长的43.85%)。利用CodonW 1.4.2软件对酸枣叶绿体基因组58条CDS密码子组成和偏好性进行统计分析(表 1)。酸枣叶绿体基因组编码的58个氨基酸序列长度范围为100~2 287,平均长度为304;不同CDS间G+C含量的变化范围为0.30~0.45,平均值为0.39;同义密码子第3位上胸腺嘧啶T3S、胞嘧啶C3S、腺嘌呤A3S、鸟嘌呤G3S出现的频率变化范围分别为:0.38~0.55、0.09~0.25、0.29~0.56、0.07~0.27;同义密码子第3位G+C的含量变化范围为0.19~0.34,平均值为0.26;Nec的平均值为47.72,表明酸枣叶绿体基因组密码子使用偏性较弱。
| 基因 Gene |
T3S | C3S | A3S | G3S | Nec | GC3 | GC | Aro | Laa |
| rps12 | 0.42 | 0.20 | 0.44 | 0.13 | 41.81 | 0.27 | 0.42 | 0.04 | 123 |
| psbA | 0.53 | 0.25 | 0.29 | 0.09 | 39.65 | 0.29 | 0.42 | 0.14 | 353 |
| matK | 0.49 | 0.14 | 0.46 | 0.20 | 49.57 | 0.25 | 0.34 | 0.16 | 506 |
| atpA | 0.48 | 0.15 | 0.41 | 0.18 | 47.40 | 0.26 | 0.41 | 0.06 | 507 |
| atpF | 0.41 | 0.18 | 0.47 | 0.24 | 42.82 | 0.31 | 0.37 | 0.09 | 184 |
| atpI | 0.45 | 0.20 | 0.41 | 0.13 | 48.82 | 0.27 | 0.38 | 0.13 | 247 |
| rps2 | 0.49 | 0.11 | 0.42 | 0.22 | 49.23 | 0.24 | 0.39 | 0.07 | 236 |
| rpoC2 | 0.45 | 0.16 | 0.46 | 0.20 | 50.32 | 0.27 | 0.37 | 0.09 | 1 389 |
| rpoC1 | 0.46 | 0.16 | 0.45 | 0.19 | 50.24 | 0.27 | 0.40 | 0.09 | 682 |
| rpoB | 0.45 | 0.14 | 0.45 | 0.21 | 48.62 | 0.26 | 0.39 | 0.08 | 1 070 |
| psbD | 0.49 | 0.22 | 0.36 | 0.13 | 47.73 | 0.29 | 0.43 | 0.17 | 353 |
| psbC | 0.46 | 0.20 | 0.35 | 0.17 | 47.58 | 0.30 | 0.45 | 0.14 | 473 |
| rps14 | 0.38 | 0.14 | 0.46 | 0.23 | 39.35 | 0.30 | 0.42 | 0.07 | 100 |
| psaB | 0.49 | 0.16 | 0.37 | 0.19 | 49.34 | 0.28 | 0.41 | 0.15 | 734 |
| psaA | 0.48 | 0.18 | 0.38 | 0.16 | 48.85 | 0.27 | 0.43 | 0.13 | 750 |
| ycf3 | 0.43 | 0.20 | 0.42 | 0.27 | 59.39 | 0.34 | 0.41 | 0.14 | 168 |
| rps4 | 0.47 | 0.17 | 0.46 | 0.15 | 48.42 | 0.24 | 0.39 | 0.07 | 201 |
| ndhJ | 0.50 | 0.14 | 0.43 | 0.20 | 51.71 | 0.26 | 0.40 | 0.15 | 158 |
| ndhK | 0.44 | 0.18 | 0.44 | 0.15 | 54.79 | 0.27 | 0.40 | 0.10 | 226 |
| ndhC | 0.51 | 0.11 | 0.44 | 0.15 | 36.77 | 0.20 | 0.36 | 0.20 | 120 |
| atpE | 0.42 | 0.18 | 0.41 | 0.22 | 53.28 | 0.31 | 0.42 | 0.02 | 133 |
| atpB | 0.42 | 0.18 | 0.44 | 0.16 | 49.17 | 0.28 | 0.43 | 0.06 | 498 |
| rbcL | 0.52 | 0.19 | 0.37 | 0.15 | 47.40 | 0.27 | 0.44 | 0.10 | 475 |
| accD | 0.54 | 0.14 | 0.43 | 0.18 | 45.76 | 0.23 | 0.34 | 0.11 | 569 |
| ycf4 | 0.47 | 0.20 | 0.37 | 0.19 | 50.99 | 0.30 | 0.39 | 0.16 | 184 |
| cemA | 0.50 | 0.21 | 0.45 | 0.12 | 51.92 | 0.25 | 0.32 | 0.15 | 229 |
| petA | 0.48 | 0.18 | 0.37 | 0.22 | 51.31 | 0.31 | 0.41 | 0.08 | 320 |
| rps18 | 0.49 | 0.13 | 0.43 | 0.20 | 39.01 | 0.25 | 0.36 | 0.07 | 101 |
| rpl20 | 0.47 | 0.09 | 0.44 | 0.25 | 48.33 | 0.25 | 0.36 | 0.09 | 117 |
| rps12 | 0.42 | 0.20 | 0.44 | 0.13 | 41.81 | 0.27 | 0.42 | 0.04 | 123 |
| clpP | 0.43 | 0.18 | 0.46 | 0.17 | 56.04 | 0.27 | 0.42 | 0.10 | 200 |
| psbB | 0.49 | 0.17 | 0.37 | 0.16 | 46.39 | 0.27 | 0.44 | 0.15 | 508 |
| petB | 0.49 | 0.16 | 0.36 | 0.16 | 46.53 | 0.26 | 0.41 | 0.13 | 215 |
| petD | 0.41 | 0.16 | 0.41 | 0.20 | 42.52 | 0.29 | 0.41 | 0.11 | 160 |
| rpoA | 0.48 | 0.16 | 0.49 | 0.18 | 49.73 | 0.24 | 0.35 | 0.07 | 331 |
| rps11 | 0.43 | 0.12 | 0.45 | 0.15 | 49.27 | 0.22 | 0.44 | 0.04 | 138 |
| rps8 | 0.46 | 0.16 | 0.50 | 0.11 | 33.72 | 0.20 | 0.34 | 0.05 | 134 |
| rpl14 | 0.42 | 0.18 | 0.51 | 0.11 | 42.58 | 0.23 | 0.40 | 0.03 | 122 |
| rpl16 | 0.38 | 0.18 | 0.52 | 0.07 | 40.06 | 0.21 | 0.44 | 0.08 | 135 |
| rps3 | 0.44 | 0.17 | 0.56 | 0.11 | 49.37 | 0.21 | 0.35 | 0.07 | 216 |
| rpl22 | 0.47 | 0.15 | 0.50 | 0.16 | 45.46 | 0.23 | 0.36 | 0.09 | 138 |
| rpl2 | 0.41 | 0.19 | 0.43 | 0.18 | 52.94 | 0.30 | 0.44 | 0.05 | 274 |
| ycf2 | 0.44 | 0.21 | 0.40 | 0.26 | 52.92 | 0.34 | 0.37 | 0.12 | 2 287 |
| ndhB | 0.44 | 0.21 | 0.43 | 0.13 | 47.55 | 0.27 | 0.37 | 0.13 | 510 |
| rps7 | 0.40 | 0.16 | 0.53 | 0.12 | 46.50 | 0.22 | 0.41 | 0.05 | 155 |
| ycf1 | 0.43 | 0.20 | 0.47 | 0.19 | 52.69 | 0.30 | 0.35 | 0.13 | 379 |
| ndhF | 0.54 | 0.11 | 0.44 | 0.13 | 43.95 | 0.19 | 0.32 | 0.15 | 752 |
| ccsA | 0.46 | 0.16 | 0.45 | 0.14 | 51.44 | 0.24 | 0.34 | 0.15 | 321 |
| ndhD | 0.46 | 0.14 | 0.44 | 0.16 | 47.08 | 0.23 | 0.35 | 0.15 | 512 |
| ndhG | 0.48 | 0.12 | 0.41 | 0.19 | 47.63 | 0.23 | 0.34 | 0.13 | 176 |
| ndhI | 0.55 | 0.16 | 0.42 | 0.15 | 48.35 | 0.23 | 0.35 | 0.11 | 167 |
| ndhA | 0.46 | 0.13 | 0.45 | 0.15 | 44.13 | 0.23 | 0.36 | 0.13 | 363 |
| ndhH | 0.47 | 0.16 | 0.48 | 0.15 | 51.07 | 0.23 | 0.39 | 0.11 | 393 |
| ycf1 | 0.49 | 0.16 | 0.55 | 0.15 | 48.28 | 0.22 | 0.30 | 0.12 | 1 907 |
| rps7 | 0.40 | 0.16 | 0.53 | 0.12 | 46.50 | 0.22 | 0.41 | 0.05 | 155 |
| ndhB | 0.44 | 0.21 | 0.43 | 0.13 | 47.55 | 0.27 | 0.37 | 0.13 | 510 |
| ycf2 | 0.44 | 0.21 | 0.40 | 0.26 | 52.92 | 0.34 | 0.37 | 0.12 | 2 287 |
| rpl2 | 0.41 | 0.19 | 0.43 | 0.18 | 52.94 | 0.30 | 0.44 | 0.05 | 274 |
| 平均值 | 0.46 | 0.17 | 0.44 | 0.17 | 47.72 | 0.26 | 0.39 | 0.10 | 304 |
酸枣叶绿体基因组中性绘图结果如图 1所示。相关性分析表明GC12与GC3无显著相关关系(R2=-0.508 89),表明酸枣叶绿体基因组各编码基因保守性较高,选择压力对密码子使用的影响较大。
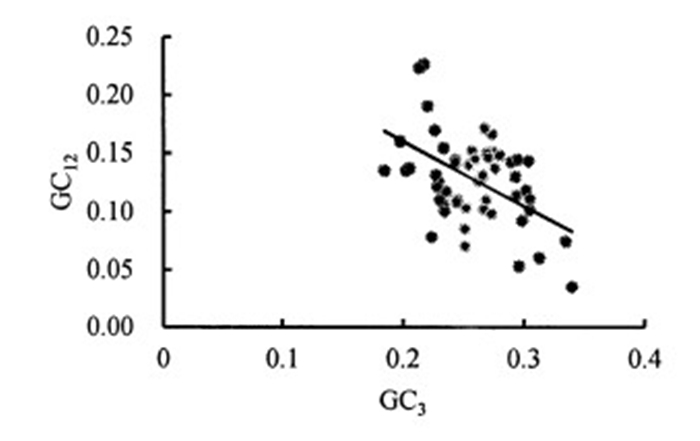
|
图 1 密码子使用的中性绘图分析 Fig. 1 Neutrality plot analysis of the codon use |
酸枣叶绿体基因组密码子使用的有效密码子数绘图分析(Nec-plot绘图分析)结果见图 2。相关性分析表明Nec与GC3无显著相关性(R2=0.177),这就表明除核苷酸组成影响密码子使用偏性外,自然选择等因素对密码子使用的影响更严重。同时从图 2还可以看出,绝大部分基因的Nec值都偏离了Nec预期值的标准曲线,同样表明来自外界的选择效应等因素对酸枣叶绿体基因组密码子使用偏性的影响更大。
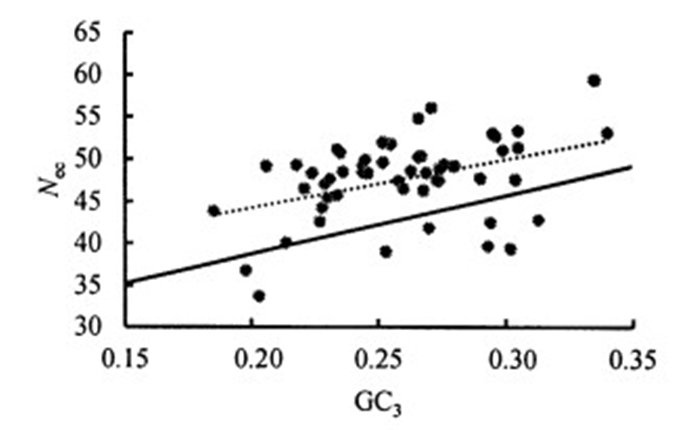
|
图 2 密码子使用的有效密码子数绘图分析 Fig. 2 Nec-plot analysis of the codon use |
酸枣叶绿体基因组最优密码子分析结果见表 2。URSC>1的密码子为高频率密码子,即UUU、UUA、UUG等密码子为酸枣叶绿体基因组高频率密码子(表 2中以下划线表示)。△URSC>0.08的密码子CUC、CUG、AUC等为酸枣叶绿体高表达优越密码子(表 2中以*表示)。结合高频密码子和高表达优越密码子,将同时满足2个条件的密码子确定为酸枣叶绿体基因组的最优密码子。最终确定UUU、GUA、UCA、ACA、AAU、AGA、GGG等7个密码子为最优密码子,除密码子GGG外,其他均以A或U结尾,其中2个以U结尾,4个以A结尾。
| 氨基酸 Amino acid |
密码子 Codon |
基因表达数 Number of expressed gene |
同义密码子相对使用度URSC | △URSC | |||||
| 高表达基因 High expressed gene |
低表达基因 Low expressed gene |
基因组 Genome |
高表达基因 High expressed gene |
低表达基因 Low expressed gene |
基因组 Genome |
||||
| 苯丙氨酸 Phe |
UUU** | 21 | 26 | 932 | 1.40 | 1.08 | 1.30 | 0.32 | |
| UUC | 9 | 22 | 501 | 0.60 | 0.92 | 0.70 | -0.32 | ||
| 亮氨酸 Leu |
UUA | 26 | 25 | 829 | 1.93 | 1.88 | 1.88 | 0.05 | |
| UUG | 12 | 20 | 526 | 0.89 | 1.50 | 1.19 | -0.61 | ||
| CUU | 17 | 16 | 562 | 1.26 | 1.20 | 1.27 | 0.06 | ||
| CUC*** | 8 | 1 | 179 | 0.59 | 0.08 | 0.41 | 0.51 | ||
| CUA | 10 | 15 | 375 | 0.74 | 1.13 | 0.85 | -0.39 | ||
| CUG** | 8 | 3 | 179 | 0.59 | 0.23 | 0.41 | 0.36 | ||
| 异亮氨酸 Ile |
AUU | 36 | 43 | 1 039 | 1.30 | 1.72 | 1.44 | -0.42 | |
| AUC* | 19 | 14 | 419 | 0.67 | 0.56 | 0.58 | 0.11 | ||
| AUA* | 28 | 18 | 702 | 1.01 | 0.72 | 0.98 | 0.29 | ||
| 蛋氨酸 Met |
AUG | 26 | 25 | 562 | 1.00 | 1.00 | 1.00 | 0.00 | |
| 缬氨酸 Val |
GUU | 19 | 24 | 426 | 1.33 | 2.23 | 1.32 | -0.90 | |
| GUC* | 6 | 2 | 165 | 0.42 | 0.19 | 0.51 | 0.23 | ||
| GUA* | 22 | 15 | 507 | 1.54 | 1.40 | 1.57 | 0.14 | ||
| GUG*** | 10 | 2 | 197 | 0.70 | 0.19 | 0.61 | 0.51 | ||
| 丝氨酸 Ser |
UCU | 15 | 22 | 537 | 1.40 | 2.00 | 1.52 | -0.60 | |
| UCC | 7 | 10 | 292 | 0.65 | 0.91 | 0.83 | -0.26 | ||
| UCA*** | 12 | 4 | 378 | 1.12 | 0.36 | 1.07 | 0.76 | ||
| UCG* | 9 | 8 | 202 | 0.84 | 0.73 | 0.57 | 0.11 | ||
| AGU** | 18 | 15 | 128 | 1.80 | 1.30 | 1.27 | 0.50 | ||
| AGC | 2 | 8 | 73 | 0.20 | 0.70 | 0.73 | -0.50 | ||
| 脯氨酸 Pro |
CCU | 22 | 14 | 389 | 1.69 | 1.70 | 1.50 | -0.01 | |
| CCC* | 11 | 5 | 202 | 0.85 | 0.61 | 0.78 | 0.24 | ||
| CCA* | 13 | 7 | 293 | 1.00 | 0.85 | 0.13 | 0.15 | ||
| CCG | 6 | 7 | 154 | 0.46 | 0.85 | 0.59 | -0.39 | ||
| 苏氨酸 Thr |
ACU | 13 | 18 | 511 | 0.91 | 2.06 | 1.61 | -1.15 | |
| ACC | 15 | 10 | 239 | 1.05 | 1.14 | 0.75 | -0.09 | ||
| ACA*** | 24 | 6 | 372 | 1.68 | 0.69 | 1.17 | 0.99 | ||
| ACG* | 5 | 1 | 145 | 0.35 | 0.11 | 0.46 | 0.24 | ||
| 丙氨酸 Ala |
GCU | 29 | 36 | 594 | 1.53 | 2.62 | 1.82 | -1.09 | |
| GCC** | 16 | 3 | 217 | 0.84 | 0.22 | 0.67 | 0.62 | ||
| GCA | 18 | 13 | 342 | 0.95 | 0.95 | 1.05 | 0.00 | ||
| GCG** | 13 | 3 | 152 | 0.68 | 0.22 | 0.47 | 0.46 | ||
| 酪氨酸 Tyr |
UAU | 32 | 21 | 756 | 1.60 | 1.56 | 1.60 | 0.04 | |
| UAC | 8 | 6 | 187 | 0.40 | 0.44 | 0.40 | -0.04 | ||
| 终止密码子 TER |
UAA | 3 | 4 | 36 | 1.20 | 1.60 | 1.53 | -0.40 | |
| UAG** | 2 | 1 | 11 | 0.80 | 0.40 | 0.47 | 0.40 | ||
| UGA | 0 | 0 | 11 | 0.00 | 0.00 | 1.00 | 0.00 | ||
| 组氨酸 His |
CAU | 13 | 10 | 467 | 1.24 | 1.25 | 1.51 | -0.01 | |
| CAC | 8 | 6 | 150 | 0.76 | 0.75 | 0.49 | 0.01 | ||
| 谷氨酰胺 Gln |
CAA | 25 | 17 | 686 | 1.47 | 1.79 | 1.53 | -0.32 | |
| CAG** | 9 | 2 | 212 | 0.53 | 0.21 | 0.47 | 0.32 | ||
| 天门冬酰胺 Asn |
AAU*** | 37 | 18 | 938 | 1.42 | 0.90 | 1.51 | 0.52 | |
| AAC | 15 | 22 | 307 | 0.58 | 1.10 | 0.49 | -0.52 | ||
| 赖氨酸 Lys |
AAA | 40 | 29 | 1 040 | 1.57 | 1.71 | 1.53 | -0.14 | |
| AAG*** | 11 | 5 | 320 | 0.43 | 0.29 | 0.47 | 0.14 | ||
| 天门冬氨酸 Asp |
GAU | 31 | 14 | 858 | 1.48 | 1.40 | 1.60 | 0.08 | |
| GAC | 11 | 6 | 217 | 0.52 | 0.60 | 0.40 | -0.08 | ||
| 谷氨酸 Glu |
GAA | 39 | 37 | 1 020 | 1.37 | 1.61 | 1.50 | -0.24 | |
| GAG* | 18 | 9 | 337 | 0.63 | 0.39 | 0.50 | 0.24 | ||
| 半胱氨酸 Cys |
UGU | 9 | 6 | 81 | 1.50 | 2.00 | 1.29 | -0.50 | |
| UGC** | 3 | 0 | 45 | 0.50 | 0.00 | 0.71 | 0.50 | ||
| 色氨酸 Trp |
UGG | 12 | 18 | 442 | 1.00 | 1.00 | 1.00 | 0.00 | |
| 精氨酸 Arg |
CGU | 13 | 15 | 207 | 1.18 | 1.36 | 1.62 | -0.18 | |
| CGC | 5 | 5 | 81 | 0.45 | 0.45 | 0.63 | 0.00 | ||
| CGA | 20 | 20 | 155 | 1.82 | 1.82 | 1.21 | 0.00 | ||
| CGG | 6 | 4 | 69 | 0.55 | 0.36 | 0.54 | 0.19 | ||
| AGA* | 25 | 12 | 139 | 1.52 | 1.33 | 1.20 | 0.19 | ||
| AGG | 8 | 6 | 93 | 0.48 | 0.67 | 0.80 | -0.19 | ||
| 甘氨酸 Gly |
GGU | 23 | 31 | 240 | 1.15 | 2.10 | 1.07 | -0.95 | |
| GGC* | 9 | 5 | 154 | 0.45 | 0.34 | 0.69 | 0.11 | ||
| GGA** | 32 | 19 | 217 | 1.60 | 1.29 | 0.97 | 0.31 | ||
| GGG*** | 16 | 4 | 288 | 0.80 | 0.27 | 1.28 | 0.53 | ||
| 注:加下划线的密码子代表基因组的△URSC>1,斜体的密码子为最优密码子(*代表△URSC>0.08,**代表△URSC>0.3,***代表△URSC>0.5)。Note:the codon with underline means △URSC>1 in the genome and the italic codon indicates the optimal codon (*means △URSC>0.08,** means △URSC>0.3,*** means △URSC>0.5). | |||||||||
酸枣叶绿体基因组奇偶偏差分析结果见图 3。由图 3可知,各编码基因在平面图的4个区域中分布不均匀,较多分布在右上方,说明密码子第3位碱基的使用存在偏好性,嘌呤的使用比嘧啶更频繁。从使用频率上看,A高于T,G高于C。当密码子的使用仅受突变影响时,这4种碱基从使用频率上看是一致的。由此推断本研究中酸枣叶绿体基因组密码子使用模式,受突变影响的同时,还受其他因素的影响,如选择压力等。
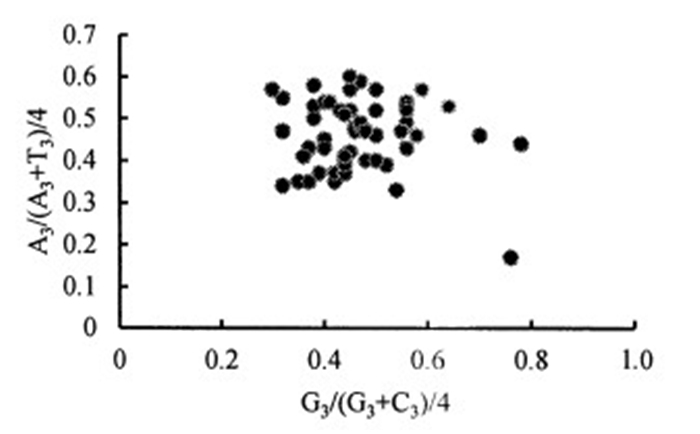
|
图 3 奇偶偏差分析 Fig. 3 PR bias-plot analysis |
以各基因在主向量轴(第1、2轴)的取值为横、纵坐标作图,对酸枣叶绿体基因组密码子使用的对应性分析结果如图 4所示。从图 4可知,酸枣叶绿体基因组58个编码基因的分布相对集中。相关性分析表明(表 3),第一轴与GC含量呈显著相关关系,与其他参数相关性均未达到显著水平,说明酸枣叶绿体基因组密码子使用偏性的影响因素中GC含量影响最大,其他因素也有一定影响。
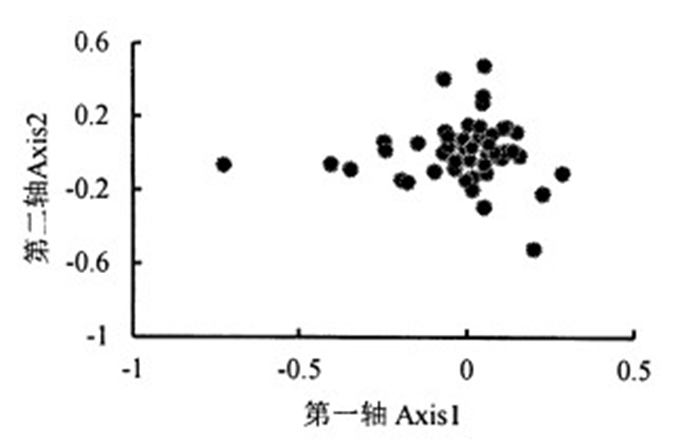
|
图 4 对应性分析 Fig. 4 Result of correspondence analysis |
| 指标 Index |
第一轴 Axis1 |
CAI | CBI | Fop | GC3 | Nec |
| CAI | 0.211 | |||||
| CBI | 0.043 | 0.765** | ||||
| Fop | 0.016 | 0.784** | 0.975** | |||
| GC3 | 0.087 | 0.173 | 0.155 | 0.197 | ||
| Nec | 0.101 | -0.007 | -0.293* | -0.210 | 0.413** | |
| GC | 0.272* | 0.290* | 0.383** | 0.389** | 0.433** | 0.070 |
| 注:**在0.01水平上显著相关,*在0.05水平上显著相关。Note:** means significant correlation at 0.01 level and * means significant correlation at 0.05 level. | ||||||
基因密码子使用参数之间的相关性分析如表 4所示,有效密码子数(Nec)与同义密码子第3位碱基胞嘧啶(C3S)、鸟嘌呤(G3S)、胞嘧啶和鸟嘌呤(GC3)的含量、基因总的GC含量、基因编码氨基酸的长度(Laa)、芳香族氨基酸频率(Aro)呈极显著正相关(P < 0.01),与同义密码子第3位胸腺嘧啶(T3S)、腺嘌呤(A3S)含量呈极显著负相关(P < 0.01)。由参数间的相关性关系可知,同义密码子第3位的碱基含量直接影响着基因密码子使用偏好性程度的大小。密码子使用偏好性强(Nec值越小)的基因多为A/T结尾的密码子。
| 使用参数 Index of codon use |
T3S | C3S | A3S | G3S | Nec | GC3 | GC | Laa |
| C3S | -0.20** | |||||||
| A3S | -0.43** | -0.30** | ||||||
| G3S | -0.04* | -0.20** | -0.24** | |||||
| Nec | -0.05** | 0.21** | -0.11** | 0.42** | ||||
| GC3 | -0.26** | 0.61** | -0.55** | 0.61** | 0.42** | |||
| GC | -0.39** | 0.32** | -0.39** | -0.15** | 0.08** | 0.43** | ||
| Laa | 0.11** | 0.18** | -0.06** | 0.32** | 0.26** | 0.29** | -0.21** | |
| Aro | 0.55** | 0.76** | -0.41** | 0.05** | 0.10** | 0.05** | -0.33** | 0.219 |
| 注:**在0.01水平上显著相关,*在0.05水平上显著相关。Note:** means significant correlation at 0.01 level and * means significant correlation at 0.05 level. | ||||||||
以酸枣叶绿体基因组58条CDS为研究对象,对其密码子使用偏性进行分析,中性绘图分析、Nec绘图分析和奇偶偏差分析的结果表明,酸枣叶绿体基因组密码子的使用受碱基突变等因素的影响较小,而受选择压力等其他因素的影响较大。在漫长的进化历史中,酸枣受到了长期的人工选择,从而选育出数量庞大的栽培枣品种,栽培枣和酸枣比较基因组学研究发现酸枣驯化过程中受到选择作用的基因多是涉及糖酸合成运输途径等的核基因[18]。本研究发现酸枣叶绿体基因组密码子使用受外界选择作用的影响较大,选择作用是否对酸枣叶绿体功能基因的进化产生影响仍有待进一步研究明确。
密码子使用偏性是物种对自身基因组组成和外界环境选择压力长期适应的结果[2],在影响密码子使用偏性的诸多因素中,密码子的碱基组成是最普遍的因素。由于密码子第3位上碱基受到的选择压力较第1、2位上碱基的小,因此GC3通常作为分析密码子使用偏性的重要参数。本研究中酸枣叶绿体基因组58条编码基因密码子的GC12与GC3无显著相关关系,而奇偶偏差分析结果表明密码子第3位碱基的使用存在偏性,嘌呤的使用比嘧啶更频繁。这与大花香水月季(Rosa odorata Andr. var. gigantea)[2]、陆地棉[8]、小麦(Triticum aestivum L.)[19]等叶绿体基因组密码子使用偏性的研究结果存在差异,上述研究发现大花香水月季等物种叶绿体基因组密码子第3位嘧啶的使用频率比嘌呤频繁。蒺藜苜蓿(Medicago truncatula Gaertn)[20]及禾本科植物(Poaceae)[21]叶绿体基因组密码子第3位碱基使用上T的频率高于A,这与本研究的结果一致,但是G和C的使用频率物种间存在较大的差异。上述分析结果表明,不同物种间不仅叶绿体基因组的碱基组成存在差异,密码子使用偏性的影响因素也存在一定的差异。
最优密码子分析确定了UUU、GUA、UCA、ACA、AAU、AGA、GGG等7个密码子为酸枣叶绿体基因组最优密码子,并且除GGG外,其他均以A或U结尾,其中2个以U结尾,4个以A结尾。这与大花香水月季[2]、蝴蝶兰(Phalaenopsis Aphrodite Rchb. F.)[22]及陆地棉[8]等物种叶绿体基因组最优密码子分析结果一致,上述物种都喜欢使用以A/T结尾的密码子,这可能与不同物种间叶绿体基因组进化的相对保守性有关系。本研究揭示了与酸枣叶绿体基因组有关的编码基因进化以及基因表达的重要信息,将为今后开展酸枣叶绿体基因工程、异源表达基因修饰奠定基础,同时也将为在叶绿体基因组水平上建立枣属及鼠李科植物分类的新方法提供重要依据。
| [1] |
吴妙丽, 陈世品, 陈辉. 竹亚科叶绿体基因组的密码子使用偏性分析[J]. 森林与环境学报, 2019, 39(1): 9-14. |
| [2] |
牛元, 徐琼, 王嵛德, 等. 大花香水月季叶绿体基因组密码子使用偏性分析[J]. 西北林学院学报, 2018, 33(3): 123-130. DOI:10.3969/j.issn.1001-7461.2018.03.19 |
| [3] |
DURET L. Evolution of synonymous codon usage in metazoans[J]. Current Opinion in Genetics and Development, 2002, 12(6): 640-649. DOI:10.1016/S0959-437X(02)00353-2 |
| [4] |
PAULET D, DAVID A, RIVALS E. Ribo-seq enlightens codon usage bias[J]. DNA Research, 2017, 24(3): 303-310. DOI:10.1093/dnares/dsw062 |
| [5] |
HANSON G, COLLER J. Codon optimality, bias and usage in translation and mRNA decay[J]. Nature Reviews Molecular Cell Biology, 2018, 19(1): 20-30. |
| [6] |
BRULE C E, GRAYHACK E J. Synonymous codons:choose wisely for expression[J]. Trends in Genetics, 2017, 33(4): 283-297. DOI:10.1016/j.tig.2017.02.001 |
| [7] |
MAN O, PILPEL Y. Differential translation efficiency of orthologous genes is involved in phenotypic divergence of yeast species[J]. Nature Genetics, 2007, 39(3): 415-421. DOI:10.1038/ng1967 |
| [8] |
尚明照, 刘方, 华金平, 等. 陆地棉叶绿体基因组密码子使用偏性的分析[J]. 中国农业科学, 2011, 44(2): 245-253. DOI:10.3864/j.issn.0578-1752.2011.02.003 |
| [9] |
秦政, 郑永杰, 桂丽静, 等. 樟树叶绿体基因组密码子偏好性分析[J]. 广西植物, 2018, 38(10): 1346-1355. DOI:10.11931/guihaia.gxzw201805023 |
| [10] |
曾路, 张如意, 王序. 酸枣仁化学成分研究[J]. 植物学报, 1986, 28(5): 517-522. |
| [11] |
HUANG J, CHEN R H, LI X G. Comparative analysis of the complete chloroplast genome of four known Ziziphus species[J]. Genes, 2017, 8(12): 340. DOI:10.3390/genes8120340 |
| [12] |
LIU Q P, FENG Y, XUE Q Z. Analysis of factors shaping codon usage in the mitochondrion genome of Oryza sativa[J]. Mitochondrion, 2004, 4(4): 313-320. DOI:10.1016/j.mito.2004.06.003 |
| [13] |
SHARP P M, TUOHY T M F, MOSURSKI K R. Codon usage in yeast:cluster analysis clearly differentiates highly and lowly expressed genes[J]. Nucleic Acids Research, 1986, 14(13): 5125-5143. DOI:10.1093/nar/14.13.5125 |
| [14] |
WRIGHT F. The 'effective number of codons' used in a gene[J]. Gene, 1990, 87(1): 23-29. DOI:10.1016/0378-1119(90)90491-9 |
| [15] |
SUEOKA N. Directional mutation pressure and neutral molecular evolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8): 2653-2657. DOI:10.1073/pnas.85.8.2653 |
| [16] |
LIU X E. A more accurate relationship between 'effective number of codons' and GC3s under assumptions of no selection[J]. Computational Biology and Chemistry, 2013, 42: 35-39. DOI:10.1016/j.compbiolchem.2012.11.003 |
| [17] |
Mcinerney J O. Replicational and transcriptional selection on codon usage in Borrelia burgdorferi[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(18): 10698-10703. DOI:10.1073/pnas.95.18.10698 |
| [18] |
HUANG J, ZHANG C, ZHAO X, et al. The Jujube genome provides insights into genome evolution and the domestication of sweetness/acidity taste in fruit trees[J]. PLoS Genetics, 2016, 12(12): e1006433. DOI:10.1371/journal.pgen.1006433 |
| [19] |
ZHANG W J, ZHOU J, LI Z F, et al. Comparative analysis of codon usage patterns among mitochondrion, chloroplast and nuclear genes in Triticum aestivum L.[J]. Journal of Integrative Plant Biology, 2007, 49(2): 246-254. DOI:10.1111/j.1744-7909.2007.00404.x |
| [20] |
杨国锋, 苏昆龙, 赵怡然, 等. 蒺藜苜蓿叶绿体密码子偏好性分析[J]. 草业学报, 2015, 24(12): 171-179. DOI:10.11686/cyxb2015016 |
| [21] |
张月荣.禾本科叶绿体基因组密码子使用模式及紫茎泽兰叶绿体RNA编辑分析[D].杨凌: 西北农林科技大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10712-1013346227.htm
|
| [22] |
续晨, 贲爱玲, 蔡晓宁. 蝴蝶兰叶绿体基因组密码子使用的相关分析[J]. 分子植物育种, 2010, 8(5): 945-950. DOI:10.3969/mpb.008.000945 |

 2019, Vol. 39
2019, Vol. 39


