文章信息
- 刘丹, 刘斌, 曾钦朦, 陈世品, 刘宝, 李煜
- LIU Dan, LIU Bin, ZENG Qinmeng, CHEN Shipin, LIU Bao, LI Yu
- 闽楠优良基因型遗传差异的SSR分析
- Genetic diversity of the superior genotypes of Phoebe bournei using SSR markers
- 森林与环境学报,2019, 39(5): 449-453.
- Journal of Forest and Environment,2019, 39(5): 449-453.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.05.001
-
文章历史
- 收稿日期: 2019-03-28
- 修回日期: 2019-06-14
闽楠[Phoebe bournei (Hemsl.) Yang]为樟科(Lauraceae)楠属(Phoebe)树种,染色体数目为2n=24,是我国特有的珍贵用材树种,具有重要的生态和经济价值[1-2]。其木材呈淡黄色,幽香,纹理美观,剖面更有闪光点或金色丝线,主要用于上等建筑、高档家具和名贵工艺品等。闽楠自然分布于我国福建、江西、浙江、广东、广西、湖南、湖北和贵州等省。由于巨大的经济价值,其野生资源遭到严重破坏,目前已难觅成片天然林,被列为国家Ⅱ级重点保护野生植物[3-4]。
江香梅等[5]利用随机扩增多态性(random amplified polymorphic DNA, RAPD)分子标记分析了闽楠天然群体的遗传多样性和遗传分化,认为闽楠天然群体间存在强烈的遗传分化,其遗传变异主要存在于群体内,来自福建省的天然群体具有较高的遗传多样性,说明在现有天然群体中进行优树选择可以获得较大的遗传增益。课题组已在闽楠天然群体中进行了优树选择,并开展了优树半同胞子代的遗传测定,选出了一批优良家系和优良单株[6]。这些优良家系已经在生产实践中进行了推广并取得了较大的经济效益。
简单重复序列(simple sequence repeat,SSR)标记是随机分布在基因组中的一种中性的分子标记[7]。SSR分子标记技术呈共显性,具有稳定性高、特异性强等优点,已被广泛应用于群体遗传多样性及种质资源亲缘关系的研究中[8-11]。本文利用开发的基因组SSR分子标记检测闽楠优良基因型的遗传差异,分析其亲缘关系,为进一步的优良无性系选育及杂交育种的亲本选配提供参考。
1 材料与方法 1.1 试验材料闽楠优良基因型种植于福建农林大学西芹教学林场(表 1)。课题组以材积为主要育种目标,在闽楠天然群体中选择了一批优树,并将其中部分优树的种子进行繁殖获得半同胞子代群体,在这些子代群体中进行优良单株的选择。本研究所用到的39个优良基因型包括在天然群体中选择的优树和在半同胞子代群体中选择的优良单株。
| 编号No. | 名称Name | 来源地Origin site |
| 1 | Jo20 | 福建省建瓯市Jian′ou, Fujian |
| 2 | Jo21 | 福建省建瓯市Jian′ou, Fujian |
| 3 | Jo23 | 福建省建瓯市Jian′ou, Fujian |
| 4 | Jo25 | 福建省建瓯市Jian′ou, Fujian |
| 5 | Jo26 | 福建省建瓯市Jian′ou, Fujian |
| 6 | Jo27 | 福建省建瓯市Jian′ou, Fujian |
| 7 | Jo28 | 福建省建瓯市Jian′ou, Fujian |
| 8 | Np601 | 福建省南平市Nanping, Fujian |
| 9 | Np602 | 福建省南平市Nanping, Fujian |
| 10 | Np603 | 福建省南平市Nanping, Fujian |
| 11 | Np604 | 福建省南平市Nanping, Fujian |
| 12 | Np605 | 福建省南平市Nanping, Fujian |
| 13 | Np606 | 福建省南平市Nanping, Fujian |
| 14 | Np607 | 福建省南平市Nanping, Fujian |
| 15 | Np615 | 福建省南平市Nanping, Fujian |
| 16 | Np616 | 福建省南平市Nanping, Fujian |
| 17 | Np617 | 福建省南平市Nanping, Fujian |
| 18 | Np618 | 福建省南平市Nanping, Fujian |
| 19 | Np619 | 福建省南平市Nanping, Fujian |
| 20 | Np620 | 福建省南平市Nanping, Fujian |
| 21 | Np621 | 福建省南平市Nanping, Fujian |
| 22 | Np622 | 福建省南平市Nanping, Fujian |
| 23 | Sx02 | 福建省沙县Shaxian, Fujian |
| 24 | Sx04 | 福建省沙县Shaxian, Fujian |
| 25 | Sx05 | 福建省沙县Shaxian, Fujian |
| 26 | Mx601 | 福建省明溪县Mingxi, Fujian |
| 27 | Mx602 | 福建省明溪县Mingxi, Fujian |
| 28 | Mx603 | 福建省明溪县Mingxi, Fujian |
| 29 | Mx604 | 福建省明溪县Mingxi, Fujian |
| 30 | Mx605 | 福建省明溪县Mingxi, Fujian |
| 31 | Mx606 | 福建省明溪县Mingxi, Fujian |
| 32 | Yp601 | 福建省南平市延平区Yanping, Nanping, Fujian |
| 33 | Yp602 | 福建省南平市延平区Yanping, Nanping, Fujian |
| 34 | Yp603 | 福建省南平市延平区Yanping, Nanping, Fujian |
| 35 | Yp604 | 福建省南平市延平区Yanping, Nanping, Fujian |
| 36 | Yx602 | 福建省尤溪县Youxi, Fujian |
| 37 | Yx603 | 福建省尤溪县Youxi, Fujian |
| 38 | Yx604 | 福建省尤溪县Youxi, Fujian |
| 39 | Sm1 | 福建省三明市Sanming, Fujian |
于2018年6月采集各个植株的枝条,带回实验室,摘取嫩叶保存在-80 ℃冰箱备用。利用试剂盒(上海生工生物工程股份有限公司)法提取DNA,1.5%的琼脂糖凝胶电泳检测DNA质量,Thermo NanoDrop 2000超微量分光光度计检测DNA的浓度,稀释浓度至40 ng · μL-1,置于-20 ℃保存备用。
所用到的基因组SSR引物为课题组前期开发(表 2)。PCR反应体系共25 μL:包括上游引物和下游引物各1 μL,DNA模版1 μL,2×Taq PCR Mix混合液12.5 μL,9.5 μL ddH2O。PCR反应程序:95 ℃预变性5 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸50 s,共32个循环;72 ℃延伸10 min;4 ℃保存。使用5 μL PCR扩增产物,通过6%变性聚丙烯酰胺凝胶电泳分离DNA片段。
| 编号 No. |
引物名称 Name |
引物序列(5′→3′) Primer sequences |
重复类型 SSR motif sequence |
预期片段大小 Predicted size/bp |
退火温度 θm/℃ |
| 1 | MN-g3 | F: TTGAGGGGGAATGTTGAGAG;R: TGAAGGCAACTGATCACAAGA | (TC)34 | 248 | 59 |
| 2 | MN-g5 | F: AAATGGTTGGGTCAAGTTCG;R: GTGCCCATATACCCGATGAC | (GA)35 | 325 | 59 |
| 3 | MN-g9 | F: ATTGCCTAAAGGCACGCAC;R: AAAATCTCAGCAAATTGGGC | (CA)27 | 139 | 60 |
| 4 | MN-g10 | F: CCTTGAGCCAATTCCAACAT;R: GGAAGCAGACAAGACCAAGC | (AG)26 | 115 | 60 |
| 5 | MN-g14 | F: ACGTCGCCACTTGCTAATCT;R: AAGCTGGAGCAAGAAAATCG | (GA)34 | 214 | 59 |
| 6 | MN-g18 | F: GAAGGTCCTCCTATCCTGCC;R: AATCCGGCTGATACTTCTGC | (TC)30 | 153 | 59 |
| 7 | MN-g26 | F: TGCATCATTTGCCTCCATAA;R: GACAGAGGTGTCCAAAACCAA | (AG)25 | 151 | 60 |
| 8 | MN-g27 | F: ATCCTGTGTAGATGGCCTGG;R: AATCCATGCCTTGTCCAAAT | (GA)17 | 174 | 59 |
| 9 | MN-g30 | F: AGAGATGTACCTCGCTGCGT;R: GAGAGCCCATCATGACCAAT | (GA)30 | 156 | 60 |
| 10 | MN-g52 | F: GCCTTGATTGTTGCCTCATT;R: ATGCGCATCACTGAAAACAA | (CA)16 | 249 | 60 |
采用人工读电泳图的方式,将有清晰条带的记为“1”,无条带的记为“0”,形成“0”“1”二元矩阵,通过读取的扩增条带数计算总扩增带数(total number of bands,TNB)、多态性带数(number of polymorphic bands,NPB)、多态性条带百分比(percentage of polymorphic bands,PPB)、多态性信息量(polymorphism information content,PIC)、条带信息指数(bands information index,BI)、条带分辨力(resolving power,RP)、标记指数(marker index,MI)。使用POPGEN软件[12]计算以下参数:观测基因数(Na)、有效等位基因数(Ne)、Nei′s遗传多样性指数(H)、Shannon信息指数(I)。利用MEGA4.0软件[13]进行UPGMA聚类。
2 研究结果 2.1 遗传多样性分析利用筛选的10对SSR引物对39个闽楠优良基因型进行扩增,扩增片段大小主要集中在50~2 000 bp,10对引物组合共扩增出了65个条带,多态性条带有50个,多态性条带占比76.9%。引物MN-g2和MN-g3的多态百分率均为100%,而引物MN-g6的多态性比率最小,为42.86%。单个引物的PIC值变幅为0.11~0.43,平均值为0.27;引物的条带信息指数变幅为0.12~0.72,平均值为0.39;引物的条带分辨力的变幅为0.36~5.38,平均值为1.939;标记指数的变幅为0.33~3.67,平均值为1.373(表 3)。闽楠39个优良基因型的观测基因数Na为1.769 2,有效等位基因数Ne为1.433 3,Nei′s遗传多样性指数H为0.258 9,Shannon信息指数I为0.390 8,说明这39个优良基因型具有一定的遗传多样性。
| 引物名称 Name of primer |
总扩增带数 TNB |
多态性带数 NPB |
多态性条带百分比 PPB/% |
多态性信息量 PIC |
条带信息指数 BI |
条带分辨力 RP |
标记指数 MI |
| MN-g1 | 6 | 5 | 83.33 | 0.19 | 0.22 | 1.08 | 0.93 |
| MN-g2 | 5 | 5 | 100.00 | 0.28 | 0.35 | 1.74 | 1.39 |
| MN-g3 | 11 | 11 | 100.00 | 0.33 | 0.49 | 5.38 | 3.67 |
| MN-g4 | 8 | 6 | 75.00 | 0.31 | 0.45 | 2.67 | 1.87 |
| MN-g5 | 8 | 5 | 62.50 | 0.4 | 0.63 | 3.13 | 1.99 |
| MN-g6 | 7 | 3 | 42.86 | 0.11 | 0.12 | 0.36 | 0.33 |
| MN-g7 | 4 | 3 | 75.00 | 0.34 | 0.46 | 1.38 | 1.01 |
| MN-g8 | 6 | 5 | 83.33 | 0.12 | 0.13 | 0.67 | 0.61 |
| MN-g9 | 7 | 5 | 71.43 | 0.22 | 0.31 | 1.54 | 1.08 |
| MN-g10 | 3 | 2 | 66.67 | 0.43 | 0.72 | 1.44 | 0.85 |
| 平均Mean | 6.5 | 5 | 76.01 | 0.27 | 0.39 | 1.94 | 1.37 |
| 总计Total | 65 | 50 |
通过POPGENE软件计算39个闽楠优良单株的遗传距离。基于遗传距离,对这39个基因型进行聚类分析,可以分为3类(图 1)。第1类包括7个基因型,分别为Jo26、Jo25、Jo21、Jo20、Jo27、Jo23、NP602;第2类包括31个基因型,又可分为3个亚类:第1亚类包括Np604、Np603、Np606、Np601、Np605、Np616,第2亚类包括Np622、Np621、Np619、Np620、Np618、Np615、Np607、Np617、Yp602、Jo28,第3亚类包括Mx605、Mx603、Mx602、Mx606、Mx601、Mx604、Sm1、Yx604、Sx05、Sx04、Sx02、Yp603、Yp604、Yx603、Yx602;第3类仅有1个基因型,为Yp601。
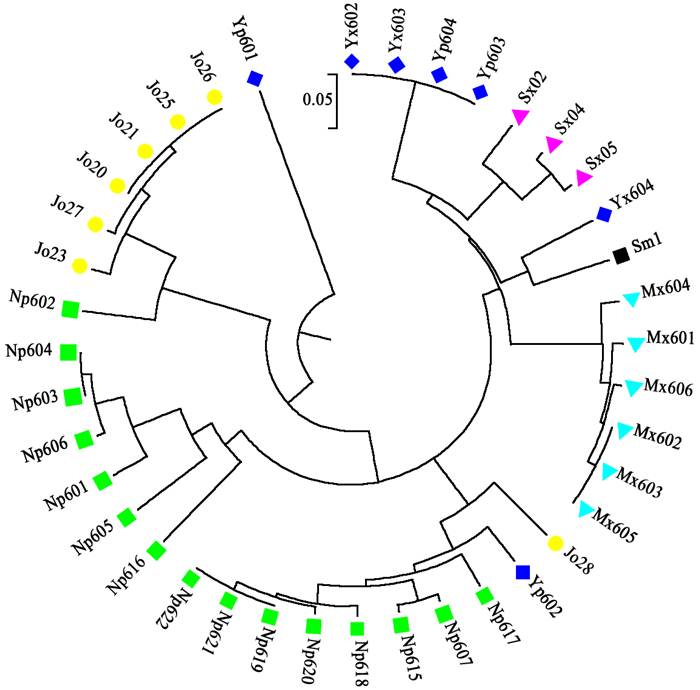
|
图 1 39个优良基因型的聚类图 Fig. 1 Dendrogram of the 39 genotypes |
SSR分子标记技术被广泛地应用于林木群体遗传多样性分析、种质资源鉴定、遗传连锁图谱构建和分子标记辅助选择等方面[14-18]。由于SSR分子标记的引物为特异引物,扩增的为基因组中的特异片段,所以其稳定性好且可重复性较高。大部分的SSR位点可以呈现出共显性的遗传方式,这在遗传连锁图谱构建和分子标记辅助选择中可以提供更多的遗传信息。通过对SSR富集文库的测序,利用获得的序列搜索SSR位点,本文前期随机选择了100对设计的SSR引物进行验证,并最终筛选出了多态性高、稳定性好的10对基因组SSR引物。利用这10对SSR对39个闽楠基因型进行了PCR扩增,可以获得多态性高、稳定性好且可重复的SSR位点,说明这10对SSR可以用于闽楠的群体遗传学和数量遗传学研究,也进一步说明利用高通量测序可以较为快速地开发可用的SSR标记。
目前,闽楠的遗传育种研究主要集中在优树选择和半同胞家系子代测定两方面。何子立等[19]采用优势木对比法,对湖南省8个县(市)区的闽楠林进行了优树选择,共初选出优树41株,初步确定了优树的选择标准:树高超过优势木平均树高的6%,胸径超过优势木平均胸径的16%,材积超过优势木平均材积的41%。李荣生等[20]对广东省乐昌市营造的5年生福建种源的闽楠进行了生长调查,选出的优良种源有福建永安、政和、尤溪等,其平均单位面积蓄积量均超过3.00 m3 · hm-2;以高于入选种源材积平均值为入选条件,从福建永安、政和和尤溪3个种源中共选择出优良家系17个。欧建德[21]对20个闽楠家系7年生子代林进行测定,选出4个优良家系,树高、胸径和单株材积分别比家系平均值提高了22.85%、29.56%和89.27%,从优良家系选出的11棵优良单株,其树高、胸径和单株材积较家系平均值增加了53.16%、84.96%和341.16%。魏永平[22]对8年生闽楠优树自由授粉子代测定林生长性状的遗传分析发现,不同家系间树高、胸径和冠幅性状存在显著性差异,变异系数都大于7.5%。通过综合评价选择了14个生长表现好的家系,平均树高6.88 m,平均遗传增益14.64%,平均胸径8.88 cm,平均遗传增益为47.83%。这些选出的优树、优良家系和优良单株等可以作为进一步遗传改良的基因资源。本文利用SSR分子标记技术检测了39个闽楠优良基因型的遗传多样性,这些优良基因型部分为天然群体中选择的优树,部分为优树自由授粉后代中选择出的优良单株。这39个优良基因型具有一定的遗传差异,如果将其无性系化并开展无性系选育,可以选出生长较快、材质较好的新品种,进一步在生产实践中推广,将产生较大的经济价值。关于闽楠的无性繁殖技术已有研究[23],但在生产中进行广泛应用还有一定的差距,今后应进一步开展嫁接、扦插、组织培养等无性繁殖技术的研究,进一步提高繁殖系数,为无性系的选育及良种推广提供技术支持。
闽楠种质资源破坏严重,若仅在自然群体中选择优良基因型,获得的遗传增益有限,而开展杂交育种将创造较大的变异,也是获得遗传改良的一种方法。本文基于遗传距离,将闽楠39个基因型分为3类,比较清楚地呈现出了39个基因型间的亲缘关系,为杂交育种的亲本选择奠定基础。但是,如果要进行良种繁育,建立种子园和采穗圃,还要在这39个基因型的基础上增加建园材料,维持较高的遗传多样性。
| [1] |
曹苜, 刘刚. 闽楠研究进展[J]. 长江大学学报(自然科学版), 2016, 13(27): 1-3, 27. DOI:10.3969/j.issn.1673-1409(s).2016.27.001 |
| [2] |
刘玉香, 宋晓琛, 江香梅. 白楠和闽楠染色体核型分析[J]. 南京林业大学学报(自然科学版), 2013, 37(5): 157-160. |
| [3] |
傅立国, 金鉴明. 中国植物红皮书:稀有濒危植物[M]. 北京: 科学出版社, 1991.
|
| [4] |
刘宝, 吴文峰, 何盛强, 等. 不同林龄闽楠林土壤呼吸与碳储量研究[J]. 森林与环境学报, 2018, 38(4): 431-438. |
| [5] |
江香梅, 温强, 叶金山, 等. 闽楠天然种群遗传多样性的RAPD分析[J]. 生态学报, 2009, 29(1): 438-444. DOI:10.3321/j.issn:1000-0933.2009.01.052 |
| [6] |
陈倩颖.闽楠种源家系生长差异及初步选择的研究[D].福州: 福建农林大学, 2018.
|
| [7] |
ZIETKIEWICZ E, RAFALSKI A, LABUDA D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 1994, 20(2): 176-183. DOI:10.1006/geno.1994.1151 |
| [8] |
LIAO G L, LI Z Y, HUANG C H, et al. Genetic diversity of inner quality and SSR association analysis of wild kiwifruit (Actinidia eriantha)[J]. Scientia Horticulturae, 2019, 248: 241-247. DOI:10.1016/j.scienta.2019.01.021 |
| [9] |
TAN L Q, LIU Q L, ZHOU B, et al. Paternity analysis using SSR markers reveals that the anthocyanin-rich tea cultivar 'Ziyan' is self-compatible[J]. Scientia Horticulturae, 2019, 245: 258-262. DOI:10.1016/j.scienta.2018.10.034 |
| [10] |
TOROKELDIEV N, ZIEHE M, GAILING O, et al. Genetic diversity and structure of natural Juglans regia L. populations in the southern Kyrgyz Republic revealed by nuclear SSR and EST-SSR markers[J]. Tree Genetics & Genomes, 2019, 15(1): 5. |
| [11] |
ALI A, PAN Y B, WANG Q N, et al. Genetic diversity and population structure analysis of Saccharum and Erianthus genera using microsatellite (SSR) markers[J]. Scientific Reports, 2019, 9(1): 395. |
| [12] |
YEH C F, BOULE T.POPGENE-1.32: a free program for the analysis of genetic variation among and within populations using co-dominant and dominant markers[R].Alberta: Department of Renewable Resources at the University of Alberta, 2000.
|
| [13] |
TAMURA K, DUDLEY J, NEI M, et al. MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24(8): 1 596-1 599. DOI:10.1093/molbev/msm092 |
| [14] |
LI Y, WANG D W, LI Z Q, et al. A molecular genetic linkage map of Eucommia ulmoides and quantitative trait loci (QTL) analysis for growth traits[J]. International Journal of Molecular Sciences, 2014, 15(2): 2 053-2 074. DOI:10.3390/ijms15022053 |
| [15] |
JIANG X B, TANG D, GONG B C. Genetic diversity and association analysis of Chinese chestnut (Castanea mollissima Blume) cultivars based on SSR markers[J]. Brazilian Journal of Botany, 2017, 40(1): 235-246. DOI:10.1007/s40415-016-0321-8 |
| [16] |
NISHIO S, TERAKAMI S, MATSUMOTO T, et al. Identification of QTLs for agronomic traits in the Japanese chestnut (Castanea crenata Sieb. et Zucc.) breeding[J]. The Horticulture Journal, 2018, 87(1): 43-54. DOI:10.2503/hortj.OKD-093 |
| [17] |
SANTOS C, NELSON C D, ZHEBENTYAYEVA T, et al. First interspecific genetic linkage map for Castanea sativa × Castanea crenata revealed QTLs for resistance to Phytophthora cinnamomi[J]. PLoS One, 2017, 12(9): e0 184 381. DOI:10.1371/journal.pone.0184381 |
| [18] |
UKOSKIT K, POSUDSAVANG G, PONGSIRIPAT N, et al. Detection and validation of EST-SSR markers associated with sugar-related traits in sugarcane using linkage and association mapping[J]. Genomics, 2019, 111(1): 1-9. DOI:10.1016/j.ygeno.2018.03.019 |
| [19] |
何子立, 杨华, 李铁华, 等. 湖南省闽楠优树选择[J]. 福建林业科技, 2018, 45(2): 34-39. |
| [20] |
李荣生, 尹光天, 杨锦昌, 等. 闽楠福建种源家系在广东的生长表现及其选择[J]. 中南林业科技大学学报, 2017, 37(6): 7-13. |
| [21] |
欧建德. 闽楠优良家系和单株的早期综合选择研究[J]. 西南林业大学学报, 2015, 35(1): 33-37. |
| [22] |
魏永平. 闽楠优树自由授粉子代测定及优良家系选择[J]. 林业勘察设计, 2018, 38(4): 31-35. |
| [23] |
申展.闽楠无性繁殖技术研究[D].长沙: 中南林业科技大学, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10538-1013212680.htm
|

 2019, Vol. 39
2019, Vol. 39


