文章信息
- 雷阿娜, 李煜, 李周岐, 周正君, 刘闵豪, 魏军坤
- LEI Ana, LI Yu, LI Zhouqi, ZHOU Zhengjun, LIU Minhao, WEI Junkun
- 黄帝手植柏DNA指纹图谱的构建
- DNA fingerprint of the Chinese arborvitae planted by Huang Di
- 森林与环境学报,2019, 39(3): 310-314.
- Journal of Forest and Environment,2019, 39(3): 310-314.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.03.013
-
文章历史
- 收稿日期: 2018-07-05
- 修回日期: 2018-10-23
2. 福建农林大学林学院, 福建 福州 350002
2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
黄帝陵地处黄土高原中南部陕西省黄陵县城西北的桥山上,陵内侧柏有8万余株,是覆盖陵园的主要树种,其中千年以上的古柏有3万余株。树王位于黄帝陵轩辕庙院内(35°40′N, 108°46′E),高约20 m,胸围11 m,苍劲挺拔,冠盖蔽空,相传为轩辕黄帝亲手所植,距今5 000多年,是世界上最古老的柏树(以下称为黄帝手植柏),被誉为“世界柏树之父”,具有重要的科学和文化价值。
近年来,对于黄帝陵古侧柏的保护备受重视,有关学者就黄帝陵古侧柏的衰老机理及保护策略开展了一系列研究[1-5]。徐龙光等[6]以黄帝手植柏幼嫩枝条为外植体,开展了组织培养研究,以期能够通过克隆手段对其进行繁殖和保护。但是,利用表型性状对黄帝手植柏的克隆植株进行鉴定较为困难。分子标记技术广泛应用于林木遗传多样性分析和种质资源鉴定,某个基因型特有标记可以对其进行准确的鉴定。本文将利用序列相关扩增多态性(sequence-related amplified polymorphism, SRAP)分子标记技术对侧柏群体及黄帝手植柏进行分析,构建黄帝手植柏DNA指纹图谱,为黄帝手植柏及其克隆植株的分子鉴定提供依据。
1 材料与方法 1.1 试验材料黄帝手植柏位于黄帝陵轩辕庙院内(35°35′01″ N,109°16′09″ E);在桥山古侧柏群中随机选取50株古侧柏;在侧柏天然分布区选取另外7个居群,分别为辽宁省建昌县(41°00′32″ N,120°01′38″ E)、北京市密云县(40°25′26″ N,117°07′49″ E)、山西省中阳县(37°19′21″ N,111°14′18″ E)、陕西省府谷县(39°07′09″ N,110°51′03″ E)、陕西省黄龙县(35°36′38″ N,109°50′09″ E)、陕西省麟游县(34°40′43″ N,107°47′05″ E)、云南省德钦县(28°35′03″ N,98°52′42″ E),每个居群内分别选取30株侧柏。采集侧柏新生的嫩芽(每个植株之间相隔100 m),保存在装有变色硅胶的密封袋中,带回实验室备用。
1.2 试验方法取侧柏叶片100 mg左右加入液氮,液氮散尽时迅速加入少许PVP粉,充分研磨(3~4次),将研磨后的粉末收集到离心管中,加入1 000 μL Buffer(100 mmol·L-1 Tris-HCl、1.4 mol·L-1 NaCl、50 mmol·L-1 EDTA,65 ℃预热)。加入β-巯基乙醇20 μL,充分震荡混匀,放入65 ℃水浴锅中保温10 min,期间摇匀3次。14 000 r·min-1离心10 min,弃上清液。获得的样品采用试剂盒(SurePlant DNA Kit,北京康为试剂生物科技有限公司)法提取,-80 ℃保存。用0.8%琼脂糖凝胶电泳检测DNA的质量,用NanoDrop ND-1000核酸蛋白检测仪(NanoDrop Technologies Inc.,美国)检测DNA的浓度。
SRAP分析基本采用LI et al[7]的方法,并针对侧柏样品特质,进行了一些改良,使用的SRAP引物由英潍捷基(上海)贸易有限公司合成,序列见表 1。反应体系:约50 ng DNA,2.5 mmol·L-1 MgCl2,0.2 mmol·L-1 dNTPs,0.4 mmol·L-1上游引物,0.4 mmol·L-1下游引物,1× PCR buffer,1.5 U Taq DNA聚合酶,补水至25 μL。PCR反应程序:94 ℃变性5 min;第一次5个循环(94 ℃变性1 min, 35 ℃退火1 min,72 ℃延伸1.5 min);第二次30个循环(94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1.5 min);最后72 ℃延伸10 min。PCR仪为S1000 Thermal Cycler(BioRad Laboratories, Inc.,美国)。用8%非变性聚丙烯酰胺凝胶电泳分离DNA片段。
| 名称Name | 正向引物(5′- 3′)Forward primers |
| Me1 | TGAGTCCAAACCGGATA |
| Me2 | TGAGTCCAAACCGGAGC |
| Me3 | TGAGTCCAAACCGGAAT |
| Me4 | TGAGTCCAAACCGGACC |
| Me5 | TGAGTCCAAACCGGAAG |
| Me6 | TGAGTCCAAACCGGACA |
| Me7 | TGAGTCCAAACCGGACG |
| Me8 | TGAGTCCAAACCGGACT |
| Me9 | TGAGTCCAAACCGGAGG |
| Me10 | TGAGTCCAAACCGGAAA |
| Me11 | TGAGTCCAAACCGGAAC |
| Me12 | TGAGTCCAAACCGGAGA |
| Me13 | TGAGTCCAAACCGGATG |
| Me14 | TGAGTCCAAACCGGATC |
| Me15 | TGAGTCCAAACCGGATT |
| Me16 | TGAGTCCAAACCGGAGT |
| Em1 | GACTGCGTACGAATTAAT |
| Em2 | GACTGCGTACGAATTTGC |
| Em3 | GACTGCGTACGAATTGAC |
| Em4 | GACTGCGTACGAATTTGA |
| Em5 | GACTGCGTACGAATT AAC |
| Em6 | GACTGCGTACGAATTGCA |
| Em7 | GACTGCGTACGAATT CAA |
| Em8 | GACTGCGTACGAATT CAC |
| Em9 | GACTGCGTACGAATTCAG |
| Em10 | GACTGCGTACGAATTCAT |
| Em11 | GACTGCGTACGAATT CTA |
| Em12 | GACTGCGTACGAATTCTC |
| Em13 | GACTGCGTACGAATTCTG |
| Em14 | GACTGCGTACGAATTCTT |
| Em15 | GACTGCGTACGAATTGAT |
| Em16 | GACTGCGTACGAATTGTC |
以黄帝手植柏和采自桥山的另一株侧柏为材料,对合成的256对SRAP引物组合进行筛选;利用筛选出的引物组合对采自桥山的30株古侧柏及黄帝手植柏进行分析,找到黄帝手植柏特异标记;对其它7个居群的210株样品及采自桥山的另外20株样品进行分析,对黄帝手植柏特异标记进行验证,构建黄帝手植柏DNA指纹图谱。
2 结果与分析 2.1 引物筛选以黄帝手植柏和采自桥山的另一株古侧柏为材料,对合成的256对SRAP引物组合进行了筛选,筛选出条带清晰,多态性高的引物组合22对(表 2)。这22对引物组合共扩增出272个条带,其中多态性条带188个,多态性比率为69.12%;引物组合Me3Em15和Me4Em9的多态性比率高达100%,多态性比率最小的为引物组合Me4Em10,为52.94%;每个引物组合平均扩增出多态性条带8个。
| 序号 No. |
引物 Primers |
多态性片段数 Polymorphic fragments |
扩增片段数 Amplified fragments |
多态性比率 Polymorphism rate/% |
| 1 | Me1Em3 | 7 | 16 | 43.75 |
| 2 | Me1Em6 | 7 | 13 | 53.85 |
| 3 | Me1Em10 | 12 | 20 | 60.00 |
| 4 | Me1Em11 | 8 | 11 | 72.72 |
| 5 | Me1Em15 | 7 | 10 | 70.00 |
| 6 | Me3Em4 | 7 | 13 | 53.85 |
| 7 | Me3Em6 | 9 | 14 | 64.29 |
| 8 | Me3Em8 | 7 | 8 | 87.50 |
| 9 | Me3Em10 | 9 | 14 | 64.29 |
| 10 | Me3Em11 | 8 | 11 | 72.73 |
| 11 | Me3Em13 | 9 | 15 | 60.00 |
| 12 | Me3Em15 | 8 | 8 | 100.00 |
| 13 | Me4Em9 | 12 | 12 | 100.00 |
| 14 | Me4Em10 | 9 | 17 | 52.94 |
| 15 | Me6Em2 | 8 | 12 | 66.67 |
| 16 | Me6Em14 | 12 | 14 | 85.71 |
| 17 | Me10Em4 | 8 | 9 | 88.89 |
| 18 | Me10Em6 | 8 | 10 | 80.00 |
| 19 | Me10Em11 | 7 | 11 | 63.64 |
| 20 | Me10Em14 | 8 | 10 | 80.00 |
| 21 | Me10Em15 | 8 | 10 | 80.00 |
| 22 | Me11Em14 | 10 | 14 | 71.43 |
| 总计 | 188 | 272 | 69.12 |
利用筛选出的22对引物组合对采自桥山的30株古侧柏及黄帝手植柏进行了分析,找到黄帝手植柏特异标记6个,分别为:Me4Em9-150、Me4Em9-1600、Me4Em9-1700、Me10Em4-80、Me10Em4-90、Me10Em4-120(图 1、图 2)。
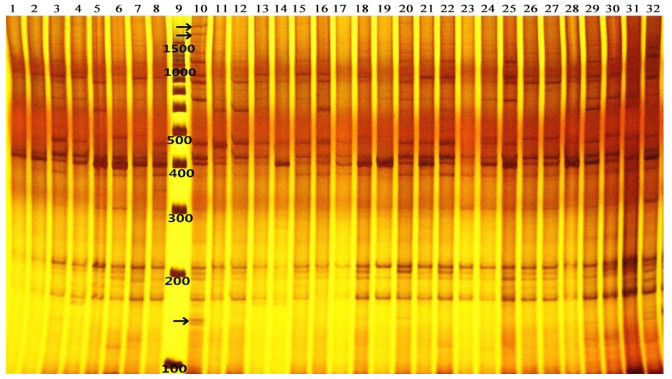
|
注:泳道9为Marker,泳道10为黄帝手植柏,其它泳道为采自桥山的古侧柏。箭头所示为黄帝手植柏特异标记Me4Em9-150、Me4Em9-1600、Me4Em9-1700。Note: Lane 9 is molecular weight marker. Lane 10 is DNA fingerprint of the old Chinese arborvitae planted by Huang Di. The rest lanes are DNA fingerprints of old trees from the Qiao mountain. The arrows indicate the rare DNA fragments of the old Chinese arborvitae planted by Huang Di (Me4Em9-150, Me4Em9-1600, Me4Em9-1700). 图 1 引物组合Me4Em9的扩增结果 Fig. 1 Fingerprint of primer combination Me4Em9 |

|
注:泳道5为Marker,泳道6为黄帝手植柏,其它泳道为采自桥山的古侧柏。箭头所示为黄帝手植柏的特异标记Me10Em4-80、Me10Em4-90、Me10Em4-120。Note: Lane 5 is molecular weight marker. Lane 6 is DNA fingerprint of the old Chinese arborvitae planted by Huang Di. The rest lanes are DNA fingerprints of old trees from the Qiao mountain. The arrows indicate the rare DNA fragments of the old Chinese arborvitae planted by Huang Di (Me10Em4-80, Me10Em4-90, Me10Em4-120). 图 2 引物组合Me10Em4的扩增结果 Fig. 2 Fingerprint of primer combination Me10Em4 |
对其它7个居群的210样品及采自桥山的另外20株样品进行分析,对黄帝手植柏特异标记进行验证。在全部的260株样品中,具有标记Me4Em9-150的样品占6.15%,具有标记Me4Em9-1600的样品占1.92%,具有标记Me4Em9-1700的样品占3.08%,具有标记Me10Em4-80的样品占1.15%,具有标记Me10Em4-90的样品占3.08%,具有标记Me10Em4-120的样品占4.23%(表 3)。利用这6个标记对黄帝手植柏进行鉴定,发生错误的概率为5.4×10-8%,正确的概率为99.999 999 9%。
| 引物组合 Primers |
标记名称 Markers |
样品总数 Number of trees |
具有该标记样品数 Number of trees with the marker |
具有该标记比率 Percentages/% |
| Me4Em9 | Me4Em9-150 | 260 | 16 | 6.15 |
| Me4Em9-1600 | 260 | 5 | 1.92 | |
| Me4Em9-1700 | 260 | 8 | 3.08 | |
| Me10Em4 | Me10Em4-80 | 260 | 3 | 1.15 |
| Me10Em4-90 | 260 | 8 | 3.08 | |
| Me10Em4-120 | 260 | 11 | 4.23 |
本文找到了6个黄帝手植柏特异标记,分别为Me4Em9-150、Me4Em9-1600、Me4Em9-1700、Me10Em4-80、Me10Em4-90、Me10Em4-120。同时利用这6个标记对黄帝手植柏进行鉴定,发生错误的概率为5.4×10-8%,正确的概率为99.999 999 9%;说明这6个标记可以作为黄帝手植柏的DNA指纹图谱,对黄帝手植柏进行分子鉴定。黄帝手植柏具有重要的科学价值,研究其生命周期可以为古树名木的保护提供基础,本文为古树名木及其克隆植株的鉴定提供方法;黄帝手植柏又具有重要的文化价值,如果可以将黄帝手植柏的克隆植株广泛传播,对于凝聚中华民族精神具有重要意义,本文鉴定的6个标记将对生产的黄帝手植柏克隆植株进行分子鉴定。
SRAP分子标记技术具有诸如操作简单、标记产率高、部分标记为共显性、对开放阅读框进行扩增、DNA片段测序容易等优点,已广泛的应用于林木群体遗传多样性分析、种质资源遗传差异及分子鉴定、遗传连锁图谱构建及数量性状基因定位等研究[8-11]。本文以黄帝手植柏和采自桥山的另一株古侧柏为材料筛选出多态性较高的引物组合22对,建立了侧柏SRAP分子标记分析方法,为进一步开展侧柏群体遗传多样性及基因资源保护研究提供了技术支持。
| [1] | 刘铭汤, 戴建昌. 黄帝陵古侧柏死亡原因调查[J]. 西北林学院学报, 1995, 10(2): 109–111. |
| [2] | 梅秀英, 姜在民, 杨茂生, 等. 黄帝陵与药王山侧柏叶污染状况的比较研究[J]. 西北林学院学报, 1997, 12(1): 57–60. |
| [3] | 李方民, 王勋陵, 岳明, 等. 人为扰动对黄帝陵侧柏生理生态学特性的影响[J]. 西北植物学报, 2003, 23(2): 239–241. DOI:10.3321/j.issn:1000-4025.2003.02.008 |
| [4] | 常二梅, 史胜青, 刘建锋, 等. 古侧柏针叶活性氧产生及其清除机制[J]. 东北林业大学学报, 2011, 39(11): 8–11. DOI:10.3969/j.issn.1000-5382.2011.11.003 |
| [5] | 杨玲, 康永祥, 李小军, 等. 黄帝陵古侧柏健康评价[J]. 浙江农林大学学报, 2014, 31(5): 779–784. |
| [6] | 徐龙光, 郭军战, 严婷. 古侧柏组织培养研究[J]. 西北林学院学报, 2014, 30(5): 92–95. DOI:10.3969/j.issn.1001-7461.2014.05.17 |
| [7] | LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2/3): 455–461. |
| [8] | LI M, ZHAO Z, MIAO X J, et al. Genetic diversity and population structure of Siberian apricot(Prunus sibirica L.) in China[J]. International Journal of Molecular Sciences, 2014, 15(1): 377–400. |
| [9] | LI Y, WANG S H, LI Z Q, et al. Genetic diversity and relationships among Chinese Eucommia ulmoides cultivars revealed by sequence-related amplified polymorphism, amplified fragment length polymorphism, and inter-simple sequence repeat markers[J]. Genetics and Molecular Research:GMR, 2014, 13(4): 8704–8713. DOI:10.4238/2014.October.27.11 |
| [10] | CHEN W X, CAO M, WANG Y X, et al. A genetic linkage map of Pinus massoniana based on SRAP, SSR and ESTP markers[J]. Silvae Genetica, 2014, 63(1/2/3/4/5/6): 1–8. |
| [11] | LI Y, WANG D W, LI Z Q, et al. A molecular genetic linkage map of Eucommia ulmoides and quantitative trait loci (QTL) analysis for growth traits[J]. International Journal of Molecular Sciences, 2014, 15(2): 2053–2074. |

 2019, Vol. 39
2019, Vol. 39


