文章信息
- 吴妙丽, 陈世品, 陈辉
- WU Miaoli, CHEN Shipin, CHEN Hui
- 竹亚科叶绿体基因组的密码子使用偏性分析
- Condon preference of chloroplast genome of Bambusoideae
- 森林与环境学报,2019, 39(1): 9-14.
- Journal of Forest and Environment,2019, 39(1): 9-14.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2019.01.002
-
文章历史
- 收稿日期: 2018-05-03
- 修回日期: 2018-05-31
2. 福建农林大学林学院, 福建 福州 350002
2. College of Forestry, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
由通用的61种有义遗传密码决定的氨基酸中,除了甲硫氨酸和色氨酸外,其余氨基酸可由2~6个同义密码子共同编码。在遗传信息的转译过程中,同义密码子的使用并不是完全均匀的,而是存在密码子偏性使用现象,其偏嗜程度因物种、组织或基因的不同呈现出丰富的多样性。一般认为,这是物种或基因在长期进化的过程中受选择、突变和漂变共同作用的结果[1-2]。除了受选择和突变影响外,密码子的使用还与诸多因素关联,如基因组大小[3]、内含子数[4]、tRNA的丰度[5-6]和基因的表达水平[7]等。密码子使用偏性是生物体的进化特征之一,分析不同物种的密码子使用模式及影响因素,将有助于理解生物与环境适应的分子机制、探讨物种间的进化关系,还能为基于密码子同义突变率的进化模型的建立提供参考数据。
叶绿体基因组作为核外遗传因子,编码与光合作用有关的蛋白。由于叶绿体基因组具有相对保守的分子结构、序列获得容易和进化速率适中等优点,被广泛用于系统发育、分子进化和遗传表达等领域的研究。对密码子使用模式的比较基因组学分析,可进一步为揭示叶绿体分子的进化特征提供参考。目前,关于叶绿体基因组密码子使用模式的研究,已在普通油茶[8]、糜子[9]和柿属植物[10]等多种经济植物上开展。
竹亚科(Bambusoideae)是禾本科唯一具有类似木质茎结构的类群,种数超过1 400种[8],是重要的食用和材用植物。竹亚科被分为3大支,即北美箭竹族(Arundinarieae,温带木本竹)、箣竹族(Bambuseae,热带木本竹)和莪利竹族(Olyreae,热带草本竹)。由于族内大部分种类的叶绿体DNA序列多样性水平较低[11],为了得到可靠的种间进化关系,近年来竹亚科的叶绿体基因组数据不断增长。目前在密码子水平上对竹亚科植物的叶绿体分子进化分析还仅限于北美箭竹族的少数种类[12],本文以NCBI中登录的竹亚科3个族的代表植物的叶绿体基因组为研究对象,分析各竹种编码基因的密码子使用模式的相似性及其影响因素,旨在为研究竹亚科植物的系统发育关系、叶绿体基因组的遗传变异及基因功能研究提供数据支持。
1 材料与方法 1.1 数据来源从NCBI数据库获取竹亚科3个族的代表种巴山木竹(Arundinaria fargesii E. G. Camus, AF, JX235347)、印度簕竹[Bambusa bambos (L.) Voss, BB, KJ870988]、Olyra latifolia L. (OL, KF515509)的CDS序列。为减少取样误差[13],分析时删除长度小于300 bp的CDS序列,最终每种保留51条数据用于偏性分析。
1.2 研究方法 1.2.1 碱基组成和同义密码子的偏好性分析用CodonW 1.4.2程序(http://codonw.sourceforge.net)计算以下4个反映密码子使用情况的参数。两个唯一密码子(ATG和TGG)和3个终止密码子(TAA、TAG和TGA)不存在简并性,分析前从数据中删除。(1)编码区GCall、GCcds含量和密码子第1位GC1、第2位GC2、第3位GC3的GC含量;(2)有效密码子数(effective number of codon, ENC),该值范围为20~61,当每种氨基酸仅对应使用1种密码子时,其值最小、偏性最强[13];(3)同义密码子相对使用度(relative synonymous codon usage, RSCU)为某一密码子的观测频率与编码相同氨基酸的同义密码子无偏使用时预期频率的比值。RSCU<1表明该密码子的偏性使用程度较低;RSCU>1表示该密码子为偏好性密码子;(4)密码子适应性指数(codon adaption index, CAI)反映的是编码某一蛋白的所有密码子与该蛋白在完全由最优密码子编码时的接近程度,可用来预测基因的表达水平。CAI值在0~1之间,偏好程度越低则该值越接近0。参数间的相关性分析使用SPSS 18.0完成。
1.2.2 影响密码子使用的因素分析 1.2.2.1 中性绘图以GC1和GC2的平均值(GC12)为纵坐标、GC3为横坐标分析密码子三个位置碱基组成的差异性[14]。若二者呈显著相关关系,说明碱基的变异模式相同,密码子的使用受突变的影响;反之则表示基因有较高的保守性,密码子使用受选择压力的影响较大。
1.2.2.2 ENC-plot分析用GC3和ENC分别作X和Y轴,衡量GC3含量对各基因同义密码子使用的影响[13]。密码子使用在无选择压力时,ENC和GC3的关系可由公式ENC=2.5-GC3+29.5/[GC32+(1-GC3)2]获得。当基因的密码子使用仅受GC3突变影响时,其ENC值将分布于期望值曲线上或附近;受到较强选择作用基因则出现在曲线下方较远的位置。
1.2.2.3 最优密码子分析根据ΔRSCU法[15]预测最优密码子,将基因按ENC值大小排序,取两端10%的基因(5个基因)组成高(低ENC值)、低样本组。比较2组密码子的RSCU值:若差异ΔRSCU> 0.08,且该密码子的RSCU值在高样本组中大于1而在低样本组中小于1,即确定为最优密码子。
1.2.2.4 奇偶偏差分析为评估AT或GC组成的偏差是否对密码子使用偏性造成影响,以密码子第3位的碱基含量作图分析,其中以A3/ (A3+T3)和G3/ (G3+C3)为纵坐标和横坐标,图的中心(0.5,0.5)表示A=T且C=G [16]。
1.2.2.5 对应分析根据每个基因所包含的59个密码子的RSCU值将基因分布于一个59维向量空间中,通过检测基因在主向量轴上分布的相对位置,推测出影响密码子使用变异的主要因素。
2 结果与分析 2.1 3种竹子叶绿体编码基因密码子的使用特性3种竹子在整个编码区的碱基组成十分相似(表 1),CAI值均为0.167。比较同义密码子3个位置的碱基组成发现,GC含量并非均匀分布:第1位的碱基含量最高,约50%;第2位的含量GC2与GCall、GCcds含量较一致;第3个位置GC3最低,仅为28%左右。统计得出,2个木本竹的有效密码子数接近,ENC值分别在38.02~61和39.41~61之间,草本竹O. latifolia则表现出较大的偏性(34.22~59.18)。除O. latifolia中rps18基因的ENC值小于35外,大部分基因的ENC值接近61,说明叶绿体基因的密码子使用偏性较弱。各氨基酸同义密码子的使用频数分析结果如表 2所示, 3个竹种对29个密码子的使用具有共同的偏好(RSCU>1), 其中以T或A结尾的密码子居多,分别为16个和11个,说明竹亚科植物偏爱结尾为T的密码子。此外,2个密码子的使用偏性在物种间有差异:相比其它2种竹子,A. fargessii对UCA的使用度大于1;O. latifolia在编码脯氨酸时较少使用密码子CCC。
| 物种Species | GCall/% | GCcds/% | GC1/% | GC2/% | GC3/% |
| A. fargesii | 38.9 | 39.2 | 50 | 39.4 | 28.1 |
| B. bambos | 38.9 | 39.1 | 50 | 39.3 | 28.1 |
| O. latifolia | 38.9 | 39.0 | 49.8 | 39.4 | 27.8 |
| 密码子 Codons |
AF | BB | OL | 密码子 Codons |
AF | BB | OL | 密码子 Codons |
AF | BB | OL | 密码子 Codons |
AF | BB | OL | |||
| AAA(K) | 1.44 | 1.44 | 1.45 | AUU(I) | 1.49 | 1.49 | 1.50 | CUC(L) | 0.40 | 0.40 | 0.37 | GGU(G) | 1.30 | 1.24 | 1.25 | |||
| AAA(K) | 1.44 | 1.44 | 1.45 | CAA(Q) | 1.53 | 1.52 | 1.52 | CUG(L) | 0.32 | 0.32 | 0.31 | GGU(G) | 1.30 | 1.24 | 1.25 | |||
| AAC(N) | 0.51 | 0.50 | 0.55 | CAA(Q) | 1.53 | 1.52 | 1.52 | CUU(L) | 1.28 | 1.29 | 1.30 | GUA(V) | 1.50 | 1.50 | 1.49 | |||
| AAC(N) | 0.51 | 0.50 | 0.55 | CAC(H) | 0.53 | 0.54 | 0.52 | GAA(E) | 1.46 | 1.46 | 1.48 | GUC(V) | 0.50 | 0.48 | 0.47 | |||
| AAG(K) | 0.56 | 0.56 | 0.55 | CAC(H) | 0.53 | 0.54 | 0.52 | GAA(E) | 1.46 | 1.46 | 1.48 | GUG(V) | 0.50 | 0.54 | 0.55 | |||
| AAG(K) | 0.56 | 0.56 | 0.55 | CAG(Q) | 0.47 | 0.48 | 0.48 | GAC(D) | 0.47 | 0.47 | 0.46 | GUU(V) | 1.50 | 1.48 | 1.49 | |||
| AAU(N) | 1.49 | 1.50 | 1.45 | CAG(Q) | 0.47 | 0.48 | 0.48 | GAC(D) | 0.47 | 0.47 | 0.46 | UAC(Y) | 0.40 | 0.41 | 0.42 | |||
| AAU(N) | 1.49 | 1.50 | 1.45 | CAU(H) | 1.47 | 1.46 | 1.48 | GAG(E) | 0.54 | 0.54 | 0.52 | UAC(Y) | 0.40 | 0.41 | 0.42 | |||
| ACA(T) | 1.11 | 1.10 | 1.09 | CAU(H) | 1.47 | 1.46 | 1.48 | GAG(E) | 0.54 | 0.54 | 0.52 | UAU(Y) | 1.60 | 1.59 | 1.58 | |||
| ACC(T) | 0.74 | 0.76 | 0.78 | CCA(P) | 1.07 | 1.07 | 1.07 | GAU(D) | 1.53 | 1.53 | 1.54 | UAU(Y) | 1.60 | 1.59 | 1.58 | |||
| ACG(T) | 0.46 | 0.47 | 0.46 | CCC(P) | 1.01 | 1.02 | 0.99 | GAU(D) | 1.53 | 1.53 | 1.54 | UCA(S) | 1.00 | 1.00 | 1.00 | |||
| ACU(T) | 1.69 | 1.67 | 1.67 | CCG(P) | 0.45 | 0.45 | 0.46 | GCA(A) | 1.17 | 1.19 | 1.18 | UCC(S) | 1.20 | 1.18 | 1.19 | |||
| AGA(R) | 1.69 | 1.68 | 1.71 | CCU(P) | 1.47 | 1.46 | 1.49 | GCC(A) | 0.60 | 0.59 | 0.57 | UCG(S) | 0.60 | 0.56 | 0.54 | |||
| AGA(R) | 1.69 | 1.68 | 1.71 | CGA(R) | 1.31 | 1.30 | 1.31 | GCG(A) | 0.48 | 0.48 | 0.47 | UCU(S) | 1.60 | 1.58 | 1.58 | |||
| AGC(S) | 0.43 | 0.40 | 0.42 | CGA(R) | 1.31 | 1.30 | 1.31 | GCG(A) | 0.48 | 0.48 | 0.47 | UGC(C) | 0.50 | 0.49 | 0.48 | |||
| AGC(S) | 0.43 | 0.40 | 0.42 | CGC(R) | 0.55 | 0.53 | 0.55 | GCU(A) | 1.75 | 1.75 | 1.78 | UGC(C) | 0.50 | 0.49 | 0.48 | |||
| AGG(R) | 0.64 | 0.68 | 0.63 | CGC(R) | 0.55 | 0.53 | 0.55 | GGA(G) | 1.57 | 1.57 | 1.59 | UGU(C) | 1.50 | 1.51 | 1.52 | |||
| AGG(R) | 0.64 | 0.68 | 0.63 | CGG(R) | 0.43 | 0.42 | 0.43 | GGA(G) | 1.57 | 1.57 | 1.59 | UGU(C) | 1.50 | 1.51 | 1.52 | |||
| AGU(S) | 1.25 | 1.28 | 1.27 | CGG(R) | 0.43 | 0.42 | 0.43 | GGC(G) | 0.43 | 0.44 | 0.43 | UUA(L) | 2.00 | 1.97 | 2.02 | |||
| AGU(S) | 1.25 | 1.28 | 1.27 | CGU(R) | 1.37 | 1.39 | 1.36 | GGC(G) | 0.43 | 0.44 | 0.43 | UUC(F) | 0.70 | 0.70 | 0.68 | |||
| AUA(I) | 0.92 | 0.92 | 0.93 | CGU(R) | 1.37 | 1.39 | 1.36 | GGG(G) | 0.74 | 0.74 | 0.73 | UUG(L) | 1.10 | 1.14 | 1.13 | |||
| AUC(I) | 0.59 | 0.59 | 0.57 | CUA(L) | 0.89 | 0.88 | 0.88 | GGG(G) | 0.74 | 0.74 | 0.73 | UUU(F) | 1.30 | 1.30 | 1.32 |
3种竹子的中性分析散点图显示,各基因均分布于对角线上部,GC3值的分布范围较小(图 1),在0.2~0.4之间。GC3与GC12的回归曲线近平行于X轴,其相关系数分别为0.147 4、0.079 7和0.098 9,双尾检验未达到显著水平(P>0.05)。GC3与GC12的相关性不高,说明密码子第1、2位碱基的突变模式不同于第3位,基因序列的突变偏性较小而更多地受到选择压力的约束。
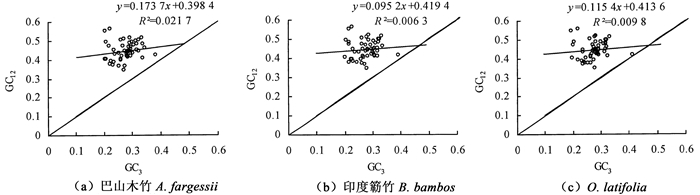
|
图 1 中性绘图 Fig. 1 Neutrality plots |
按功能的不同将基因分为5组:基因表达相关基因(19个)、光合作用代谢相关基因(6个)、跨膜运输相关基因(4个)、光合作用体系编码基因(11个)和编码NADH脱氢酶亚基的基因(11个),分析了基因间的GC3与ENC的关系。从图 2可看出,基因的ENC值分布比较分散,基因间偏好程度差别较大。一部分基因落在期望曲线上或附近,表明密码子第3位的碱基组成GC3对这些基因密码子用法的影响要大于其它基因。一些倾向于随机使用密码子的基因则落在曲线上方;另一部分基因,如rpl16、rps18和psbA,偏离了期望值并且位于期望曲线下方较远的位置,表明该基因受到选择压力。分析结果提示,3种竹子的叶绿体基因密码子使用受到GC3组成的影响,同时自然选择等其它因素亦决定了一些基因的密码子使用方式。
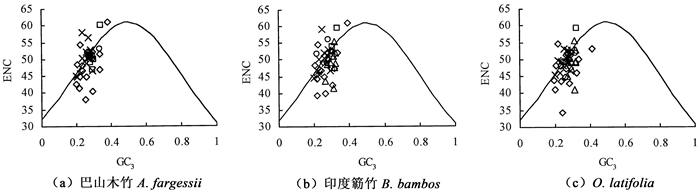
|
注:◇表示基因表达相关基因;□表示光合代谢相关基因;○表示跨膜运输相关基因;×表示编码NADH脱氢酶亚基的基因;△表示光合作用体系编码基因。Note:◇ gene expression related gene;□ photosynthesis metabolism gene;○ transmembrane transportation related gene;× coding NADH dehydrogenase subunit gene;△ photosynthesis system coding gene. 图 2 ENC值散点图 Fig. 2 ENC-plots |
根据ΔRSCU法确定了A. fargessii的最优密码子6个(CCU、CGU、AAC、GCU、GUA、GAG)、B. bambos有2个(UUA、ACU)、O. latifolia有3个(UUA、ACU、GUC)。可以看出,在这些最优密码子中,仍以A或T结尾的密码子占多数,此结果与GCall和GC3的高A、T含量一致。
2.4 密码子使用的PR2分析和对应分析为进一步反映整个叶绿体编码序列4种碱基组成的偏差程度,对密码子第3位的嘧啶和嘌呤的关系进行了奇偶偏好分析。总体上,密码子第3位的嘧啶含量(C+T)大于嘌呤含量(A+G) (图 3)。除了在B. bambos中G=C外,其余2种竹子的C的含量都略大于G (A. fargesii的平均G3/(G3+C3)值为0.493,O.latifolia为0.491);而3种竹子的T含量均一致高于A (A. fargesii, B. bambos和O.latifolia的A3/(T3+T3)平均值依次为0.485,0.485为0.486)。一般认为,当基因仅受到碱基组成的影响时,密码子第3位置上应有相同的G和C (或A和T)含量[17]。4种碱基并非均匀地分布在密码子的第3位上,说明选择在此过程中起到了重要的作用,而突变等其他因素密码子偏好性影响较弱。
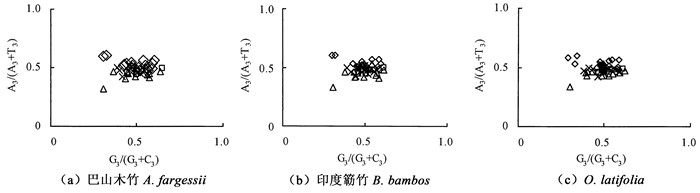
|
图 3 奇偶偏好分析 Fig. 3 Analysis of parity rule 2 |
3种竹子基于各基因的RSCU值进行的对应分析结果中,第1个主要因子轴占总变异量的比值分别为9.68%、10.35%和10.52%(图 4),前4个主要因子总贡献量合计为34.31%、34.64%和34.61%。第一轴变异度所占贡献量最大,其余各因子轴的贡献量依次递减,表明3种竹子叶绿体基因的密码子偏性使用模式的形成过程复杂,受到了多种因素的共同作用。将轴1~4与CAI、ENC和GC3进行简单相关(表 3),发现3种竹子的第2轴对应值与CAI呈显著负相关关系(P < 0.05);轴4与GC3、ENC呈极显著相关(P < 0.01)。此外,CAI和GC3也检测到显著的正相关(P < 0.05)。这些结果说明基因的表达水平(选择)是密码子偏性形成的主要因素。图 4中由各基因落在第1、2因子轴组成的平面位置,可显示各基因密码子使用性的变异趋势:大部分基因集聚在0点附近,表示这些基因偏性较低且有相似的密码子使用性。一些与基因表达相关的基因、光合作用代谢相关基因和编码NADH脱氢酶亚基的基因分散度较高,这些基因在3种竹子中受到的影响程度不同而分散程度不同,说明尽管3种竹子有着整体相似的密码子使用模式,但不同种间的基因仍有各自特殊的密码子使用演化特征。
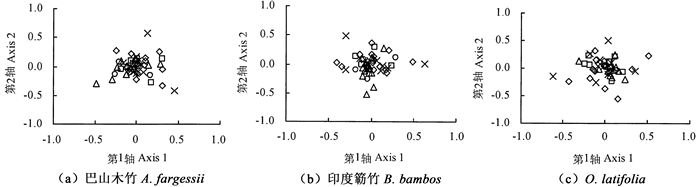
|
图 4 基于同义密码子相对使用度RSCU的对应性分析 Fig. 4 Correspondence analysis based on the relative synonymous codon usage |
| 轴Axis | 巴山木竹A. fargesii | 印度簕竹B. bambos | O. latifolia | ||||||||
| CAI | ENC | GC3 | CAI | ENC | GC3 | CAI | ENC | GC3 | |||
| 第1轴Axis1 | -0.52** | 0.31* | -0.02 | -0.03 | 0.11 | -0.01 | 0.20 | -0.36** | 0.06 | ||
| 第2轴Axis2 | -0.37** | -0.15 | -0.20 | -0.64** | 0.11 | -0.03 | -0.31* | 0.04 | -0.32* | ||
| 第3轴Axis3 | 0.14 | 0.07 | 0.02 | 0.10 | -0.19 | -0.24 | 0.54** | 0.02 | -0.06 | ||
| 第4轴Axis4 | 0.26 | 0.37** | 0.45** | -0.257 | -0.38** | -0.45** | -0.04 | -0.41** | -0.41** | ||
| 注:*表示显著相关,P<0.05,* *表示极显著相关,P<0.01。Note:* indicates significant difference,P<0.05,* *indicates highly significant difference,P<0.01. | |||||||||||
同义密码子非随机使用现象普遍存在于原核生物和真核生物中,被认为是自然选择和突变压力平衡调节的结果。本研究对竹亚科3个族的代表种的同义密码子使用偏性分析,发现3种竹子的叶绿体编码基因的密码子使用几乎相同。从ENC和CAI值来看,偏好程度都较低,其密码子使用偏性受自然选择和突变压力共同影响。这些影响因素对不同基因的影响强度不同从而造成了基因间的密码子偏性大小存在差异。
3种竹子所偏好的密码子与禾本科其它植物相似[12, 18],偏爱以A或T结尾的密码子,密码子3个位置的GC含量不同且都不高于AT含量,并以第3位的GC3含量为最低。已有研究表明,GC3与基因组的总GC含量间存在明显的线性关系[19],正是由于碱基突变偏好GC或AT造成了基因组内同义密码子的使用偏性[20-21]。银白杨[22]、小果咖啡[23]、北美箭族的竹种[12]的叶绿体基因分析中也表明,GC含量尤其是GC3的含量强烈影响了同义密码子的使用,但对水稻、高粱和小麦的叶绿体基因序列的分析则揭示出选择在密码子使用偏性形成过程中起着主导作用[24]。KAWABE et al[17]认为,密码子使用若受到自然选择的影响,GC3值倾向于分布在较小的范围内并且GC12和GC3无显著相关性。本研究发现3种竹子的叶绿体编码基因的GC3值分布都较窄,在0.2~0.4之间,且GC3和GC12的相关性弱(图 1),意味着密码子使用偏性受自然选择压力的影响更大。对应分析中轴2与CAI的关联和PR2分析中4种碱基在密码子第3位上的非均衡分布,也支持了选择的主导作用,这一结果与北美箭竹族的竹种[9]的结论并不一致,可能与不同物种有关。
GC3碱基组成也在一定程度上影响基因同义密码子的使用偏性。从ENC-plot (图 2)看出,除了一些与基因表达相关基因和光合作用体系编码基因(如rpl16、rps18和psbA等)受强烈的选择而具有较低的ENC值外,仍有部分基因落在预期曲线上。同样地,对应分析中检测到第4主轴与GC3的相关性,这说明了GC3碱基组成是影响密码子使用的部分因素。综合以上分析结果,自然选择和基因的碱基组成是竹亚科植物叶绿体基因组密码子使用的主要因素,结果符合突变-选择-漂变理论[1-2]的观点,但2个主要因素作用的强弱在种间具有差异性,其内在机制还有待深入研究。
| [1] | BULMER M. The selection-mutation-drift theory of synonymous codon usage[J]. Genetics, 1991, 129(3): 897–907. |
| [2] | Hershberg R, Petrov D A. Selection on codon bias[J]. Annual Review of Genetics, 2008, 42: 287–299. DOI:10.1146/annurev.genet.42.110807.091442 |
| [3] | DOS REIS M, SAVVA R, WERNISCH L. Solving the riddle of codon usage preferences:a test for translational selection[J]. Nucleic Acids Research, 2004, 32(17): 5036–5044. DOI:10.1093/nar/gkh834 |
| [4] | QIN Z, CAI Z Q, XIA G M, et al. Synonymous codon usage bias is correlative to intron number and shows disequilibrium among exons in plants[J]. BMC Genomics, 2013, 14: 56. DOI:10.1186/1471-2164-14-56 |
| [5] | DURET L. tRNA gene number and codon usage in the C. elegans genome are co-adapted for optimal translation of highly expressed genes[J]. Trends in Genetics, 2000, 16(7): 287–289. DOI:10.1016/S0168-9525(00)02041-2 |
| [6] | OLEJNICZAK M, UHLENBECK O C. tRNA residues that have coevolved with their anticodon to ensure uniform and accurate codon recognition[J]. Biochimie, 2006, 88(8): 943–950. DOI:10.1016/j.biochi.2006.06.005 |
| [7] | HIRAOKA Y, KAWAMATA K, HARAGUCHI T, et al. Codon usage bias is correlated with gene expression levels in the fission yeast Schizosaccharomyces pombe[J]. Genes to Cells, 2009, 14(4): 499–509. DOI:10.1111/gtc.2009.14.issue-4 |
| [8] | 王鹏良, 杨利平, 吴红英, 等. 普通油茶叶绿体基因组密码子偏好性分析[J]. 广西植物, 2018, 38(2): 135–144. |
| [9] | 刘慧, 王梦醒, 岳文杰, 等. 糜子叶绿体基因组密码子使用偏性的分析[J]. 植物科学学报, 2017, 35(3): 362–371. |
| [10] | 傅建敏, 索玉静, 刘慧敏, 等. 柿属植物叶绿体蛋白质编码基因密码子用法[J]. 经济林研究, 2017, 35(2): 38–44. |
| [11] | MA P F, ZHANG Y X, ZENG C X, et al. Chloroplast phylogenomic analyses resolve deep-level relationships of an intractable bamboo tribe Arundinarieae (Poaceae)[J]. Systematic Biology, 2014, 63(6): 933–950. DOI:10.1093/sysbio/syu054 |
| [12] | ZHANG Y R, NIE X J, JIA X O, et al. Analysis of codon usage patterns of the chloroplast genomes in the Poaceae family[J]. Australian Journal of Botany, 2012, 60(5): 461–470. DOI:10.1071/BT12073 |
| [13] | LIU X E. A more accurate relationship between 'effective number of codons' and GC3s under assumptions of no selection[J]. Computational Biology and Chemistry, 2013, 42: 35–39. DOI:10.1016/j.compbiolchem.2012.11.003 |
| [14] | SUEOKA N. Directional mutation pressure and neutral molecular evolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8): 2653–2657. DOI:10.1073/pnas.85.8.2653 |
| [15] | 尚明照, 刘方, 华金平, 等. 陆地棉叶绿体基因组密码子使用偏性的分析[J]. 中国农业科学, 2011, 44(2): 245–253. DOI:10.3864/j.issn.0578-1752.2011.02.003 |
| [16] | 宋辉, 王鹏飞, 马登超, 等. 蒺藜苜蓿WRKY转录因子密码子使用偏好性分析[J]. 农业生物技术学报, 2015, 23(2): 203–212. DOI:10.3969/j.issn.1674-7968.2015.02.007 |
| [17] | KAWABE A, MIYASHITA N T. Patterns of codon usage bias in three dicot and four monocot plant species[J]. Genes & Genetic Systems, 2003, 78(5): 343–352. |
| [18] | SABLOK G, NAYAK K C, VAZQUEZ F, et al. Synonymous codon usage, GC3, and evolutionary patterns across plastomes of three pooid model species:emerging grass genome models for monocots[J]. Molecular Biotechnology, 2011, 49(2): 116–128. DOI:10.1007/s12033-011-9383-9 |
| [19] | KUSUMI J, TACHIDA H. Compositional properties of green-plant plastid genomes[J]. Journal of Molecular Evolution, 2005, 60(4): 417–425. DOI:10.1007/s00239-004-0086-8 |
| [20] | KNIGHT R D, FREELAND S J, LANDWEBER L F. A simple model based on mutation and selection explains trends in codon and amino-acid usage and GC composition within and across genomes[J]. Genome Biology, 2001, 2(4): research 0010.1–0010.13. |
| [21] | MORTON B R, LEVIN J A. The atypical codon usage of the plant psbA gene may be the remnant of an ancestral bias[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(21): 11434–11438. DOI:10.1073/pnas.94.21.11434 |
| [22] | ZHOU M, LONG W, LI X. Analysis of synonymous codon usage in chloroplast genome of Populus alba[J]. Journal of Forestry Research, 2008, 19(4): 293–297. DOI:10.1007/s11676-008-0052-1 |
| [23] | NAIR R R, NANDHINI M B, MONALISHA E, et al. Synonymous codon usage in chloroplast genome of Coffea arabica[J]. Bioinformation, 2012, 8(22): 1096–1104. DOI:10.6026/bioinformation |
| [24] | LIU Q P, XUE Q Z. Comparative studies on codon usage pattern of chloroplasts and their host nuclear genes in four plant species[J]. Journal of Genetics, 2005, 84(1): 55–62. DOI:10.1007/BF02715890 |

 2019, Vol. 39
2019, Vol. 39

