文章信息
- 张庆波, 胡尚连, 徐刚, 曹颖, 卢学琴, 龙治坚
- ZHANG Qingbo, HU Shanglian, XU Gang, CAO Ying, LU Xueqin, LONG Zhijian
- 毛竹bZIP转录因子的基因结构与进化分析
- Analysis on gene structure and evolution of bZIP transcription factor of Phyllostachys edulis
- 森林与环境学报, 2016, 36(01): 54-61
- Journal of Forest and Environment, 2016, 36(01): 54-61.
- http://dx.doi.org/10.13324/j.cnki.jfcf.2016.01.009
-
文章历史
- 收稿日期: 2015-03-24
- 修订日期: 2015-07-02
2. 四川省生物质资源利用与改性工程技术研究中心, 四川 绵阳 621010
2. Engineering Research Center for Biomass Resource Utilizaiton and Modification of Sichuan Province, Mianyang, Sichuan 621010, China
碱性亮氨酸拉链(basic leucine zipper,bZIP)转录因子是真核生物转录因子中分布最广泛、最保守的一类转录因子,其保守的bZIP结构域由结合DNA的碱性区域和一个亮氨酸拉链的二聚化基序构成,根据其结构特点和功能可以将bZIP转录因子家族划分为10个亚族,分为A、B、C、D、E、F、G、H、I和S亚族,在同一亚族的bZIP转录因子往往有相似的功能[1]。bZIP转录因子在植物生长发育过程中参与多种生理过程,其中A和S亚族的成员主要参与非生物胁迫应答,包括干旱、冷、高盐和脱落酸(abscisic acid,ABA)的转录调控。其中,高盐和干旱胁迫会激活一个丝裂原活化蛋白激酶(mitogen-activated protein kinase,MAPK)级联信号途径,使细胞内Ca2+浓度升高,引发MAPK磷酸化或去磷酸化反应来激活bZIP和MYB转录因子,进而调控抗盐基因的表达,如慈竹(Bambusa emeiensis Chia et H. L. Fung ‘Viridiflava’)的BeMYB1和BeMYB2基因对模拟干旱和高盐环境的聚乙二醇(polyethylene glycol,PEG)6000和NaCl溶液敏感[2, 3, 4]。拟南芥(Arabidopsis thaliana L.)A亚族的AtbZIP36(ABF2/AREB1)、AtbZIP38(ABF4/AREB2)和AtbZIP37(ABF3)基因在响应脱落酸(abscisic acid,ABA)信号、干旱和盐胁迫过程中表现上调[5];水稻(Oryza sativa L.)S亚族的OsbZIP12(OsABF1)和A亚族的OsbZIP46(OsABF2)对干旱、盐和冷胁迫以及ABA诱导更敏感[6, 7]。D亚族的bZIP基因参与植物病原体防御和生长发育过程,在烟草(Nicotiana tabacum L.)和拟南芥中它们通过TGACG序列结合蛋白(TGACG sequence binding protein,TGA)/章鱼碱合成酶元件结合因子(octopine synthase element-binding factor,OBF)参与发育反应[8, 9, 10],并与非表达病程相关基因1(non-expression of PR1,NPR1)相互作用,是水杨酸(salicylic acid,SA)防御信号途径的一个关键组成部分[11, 12, 13]。
毛竹[Phyllostachys edulis (Carr.) H. de Lehaie]是中国分布最广、面积最大、经济价值最高的竹种之一,然而对毛竹bZIP转录因子的相关研究却鲜见报道。目前,毛竹高质量的基因组草图测序及毛竹5种组织的高通量测序工作已经完成[14],为识别和鉴定毛竹中bZIP转录因子家族整体轮廓奠定了基础。本课题组检索得到毛竹基因组数据库中107个编码bZIP转录因子的基因,采用生物信息学的分析方法,对毛竹中涉及到抗逆、病原体防御和生长发育相关且各自成员数量均大于10的A、D和S亚族bZIP转录因子在系统发育、基因结构、适应性进化、基因对间进化分歧时间以及同源建模等方面进行分析,为毛竹bZIP转录因子的进一步功能研究提供理论依据。
1 材料与方法 1.1 数据来源与获取文中所需要的数据,包括拟南芥,水稻和毛竹bZIP氨基酸序列和相应的核苷酸序列以及基因组序列,从PlantTFDB(http://planttfdb.cbi.pku.edu.cn/),BambooGDB(http://www.bamboogdb.org/)和TAIR(http://www.arabidopsis.org/)等数据库中下载。
1.2 毛竹bZIP基因的系统发育分析使用MEGA 5.10(http://gsds.cbi.pku.edu.cn/)软件,将毛竹全部bZIP氨基酸序列与拟南芥A、D和S亚族bZIP氨基酸序列分别进行比对,构建进化树,选用最大似然法,自举检测1 000次,筛选得到毛竹A、D和S亚族bZIP蛋白质序列。将分析得到的毛竹A、D和S亚族bZIP蛋白质序列,全部用于构建进化树,同样采用最大似然法,自举检测1 000次,观察毛竹3个亚族之间的基因进化情况。
1.3 毛竹bZIP基因的内含子—外显子分析为获得绘制毛竹bZIP基因内含子—外显子结构图的核苷酸编码序列和相应基因组序列,分别从PlantTFDB和BambooGDB数据库中得到相应的序列,再将毛竹bZIP基因序列和相应的基因组序列一起输入到GSDS v2.0(=http://gsds.cbi.pku.edu.cn/)中,来界定内含子剪切位点,从而得到内含子—外显子结构图。
1.4 毛竹bZIP基因的适应性进化分析将毛竹A、D和S亚族bZIP基因的全部核苷酸编码序列提交至Selecton程序[15](http://selecton.tau.ac.il/),设置模型为力学—经验型模型(mechanistic-empirical model,MEC)。Selecton程序会计算每个蛋白位点的非同义替换率(Ka)和同义替换率(Ks),及两者比率ω(ω=Ka/Ks),Ka和Ks分别代表每个非同义和同义替换位点的数目,并通过统计学方法对某个位点的预测结果进行验证,将每个位点根据ω值的不同,用不同颜色标记在蛋白质的一级结构上,以显示特定位点上受到的选择压力(阳性选择、净化选择或中性突变),进而得到3个亚族基因之间的选择压力。
1.5 毛竹与水稻同源bZIP基因分歧时间的计算从BambooGDB数据库中检索得到毛竹和水稻间的bZIP同源基因对,然后将基因对中氨基酸序列及其相应的cDNA序列一并输入到PAL2NAL(http://www.bork.embl.de/pal2nal/)软件中,去除空位(gap)序列,最后将去除gap的cDNA比对序列输入到KaKs_Calculator 2.0软件[16](http://sourceforge.net/projects/kakscalculator2/)中,计算Ka、Ks以及二者的比值ω,选用KaKs_Calculator 2.0软件中的gYN方法。这些参数被用于划分目标序列的进化选择情况:净化选择时,Ka < Ks(ω < 1);阳性选择时,Ka > Ks(ω > 1);中性突变时,Ka=Ks(ω=1)。bZIP基因对间的分歧时间以百万年(million years ago,Mya)表示,分歧时间的计算公式为T=Ks/2λ(λ=6.5×10-9)[17],其中λ表示每年每个同义位点的同义突变替换率。
1.6 毛竹bZIP蛋白质的同源建模蛋白质三级结构是在二级结构的基础上进一步盘绕折叠形成的。通过对毛竹3个亚族47条氨基酸序列利用Swiss-Model(http://swissmodel.expasy.org/)进行三维结构同源建模,获得相应的PDB文件,用Antheprot 3D(http://antheprot-pbil.ibcp.fr/)软件对获得的PDB文件进行编辑,进而得出三级结构图。
2 结果与分析 2.1 毛竹bZIP基因的系统发育分析查询得到毛竹全部bZIP氨基酸序列,经过与拟南芥的A、D和S亚族bZIP基因编码的氨基酸序列比对,获得了毛竹A、D和S亚族bZIP基因序列共计47条,占毛竹总bZIP基因的43.9%,比重较大;其中A亚族18条(16.8%)、D亚族16条(15.0%)、S亚族13条(12.1%)。将得到的毛竹A、D和S亚族bZIP氨基酸序列,构建进化树后发现同亚族的bZIP成员各自聚为一支[图 1(a)]。
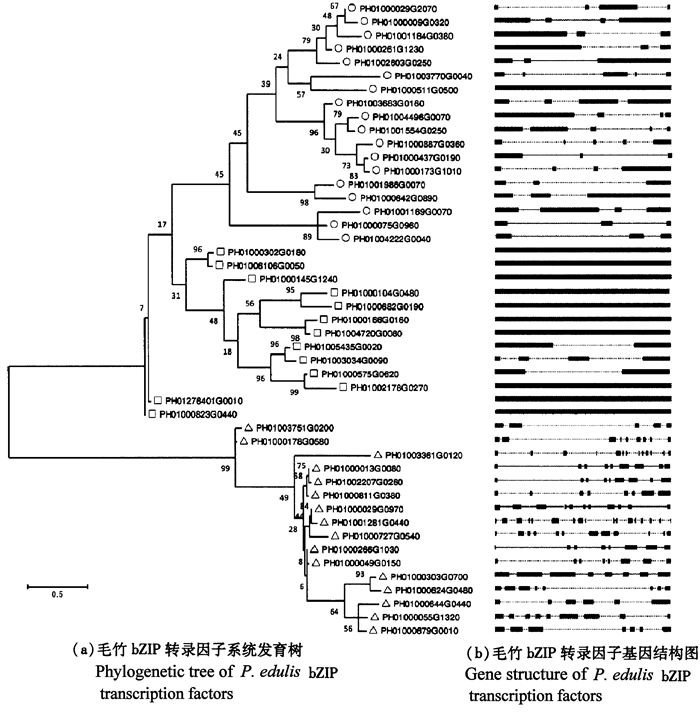 |
注:○,△和□分别代表毛竹A、D、S亚族bZIP基因,图中标尺为距离标尺代表遗传距离;━表示毛竹bZIP基因的外显子,┈表示内含子。Notes: ○, △ and □ represent A, D and S subgroups of bZIP genes of P. edulis, respectively. The scale represents the genetic distance. ━ and ┈ represent exon and intron of P. edulis bZIP genes, respectively.图 1 毛竹bZIP转录因子的进化树及基因结构 Figure 1 Phylogenetic tree and gene structure of P. edulis bZIP transcription factors |
每个基因的基因组结构,特别是内含子—外显子的数量和分布,可能是记录进化发生的关键,并且为某个特定基因的出现以及进化提供更深入的了解[18]。毛竹A、D和S亚族bZIP基因外显子—内含子的分析结果表明,毛竹bZIP基因内含子结构高度保守的基因亚族一般聚集为一类,且有相似的基因结构,特别是外显子的数量和长度。D亚族的基因结构明显复杂于A和S亚族,而S亚族的基因结构最为简单,内含子数量最少,仅为0到3个,D亚族均含有内含子结构,而A亚族仅有1条序列无内含子。
2.3 毛竹bZIP基因的适应性进化分析解析相关基因进化过程中选择的作用,是进化生物学研究的重要内容[19]。运用Selecton程序中的MEC模型在毛竹A、D和S亚族bZIP序列上探测正选择位点。蛋白质一级结构位点的标注颜色大部分为白色到紫色之间,尤以紫色和深紫色居多,表明这些位点为经历了强的净化选择的位点,而黄色位点在1-170位置上相对比较集中,其余的零星分布在序列中,且颜色多为浅黄,说明这些位点受到阳性选择压力(图 2)。非同义替换的速率远远低于同义替换,且发生速率随着基因的不同而大相径庭,这被认为是净化选择的结果[20]。由此可见在MEC模型下存在很多位点处于强烈的净化选择压力下,在毛竹A、D和S亚族bZIP基因中净化选择压力主导3个亚族之间基因的进化。
 |
图 2 基于MEC模型的毛竹A、D和S亚族bZIP基因选择压力 Figure 2 Selection pressure on the bZIP gene sequences of A, D and S groups of P. edulis using the MEC model |
生物氨基酸或核苷酸替代速率在进化过程中是近似地保持恒定。分子进化改变量(替换数或替换百分率)与分子进化时间呈正比,可用它来推断未知进化事件发生的可能时间。因而对毛竹和水稻同源基因对之间进行了Ka、Ks、ω以及进化分歧时间的计算。毛竹与水稻bZIP同源基因对之间的ω计算结果表明,毛竹A亚族ω值从0.35到1.52之间变化,D亚族从0.36到1.60之间变化,S亚族从0.29到2.05之间变化(表 1)。3个亚族ω平均值分别为0.65,0.79和0.77,均小于1,但三者相差不大,可见毛竹与水稻同源bZIP基因之间非同义替换的速率要远远低于同义替换的速率,即Ka远远小于Ks。另外,毛竹A、D和S亚族bZIP基因与水稻同源基因对的平均进化分歧时间分别为279.0、265.6和202.9 Mya(表 1)。
| 亚族Subgroup | 毛竹基因ID P. edulis gene ID | 水稻基因ID O. sative gene ID | Ks | Ka | ω | 分歧时间divergence time/Mya |
| A | PH01001184G0380 | LOC_Os07g48660.1 | 3.58 | 1.25 | 0.35 | 275.4 |
| PH01002603G0250 | LOC_Os05g41070.1 | 4.04 | 6.14 | 1.52 | 310.8 | |
| PH01000261G1230 | LOC_Os05g41070.1 | 2.34 | 2.50 | 1.07 | 180.0 | |
| PH01000029G2070 | LOC_Os01g59760.1 | 4.02 | 2.88 | 0.72 | 309.2 | |
| PH01000009G0320 | LOC_Os01g59760.1 | 3.99 | 3.50 | 0.88 | 306.9 | |
| PH01003770G0040 | - | - | - | - | - | |
| PH01000511G0500 | LOC_Os09g36910.1 | 3.15 | 1.09 | 0.35 | 242.3 | |
| PH01001986G0070 | LOC_Os06g50830.1 | 3.32 | 2.24 | 0.67 | 255.4 | |
| PH01000642G0890 | - | - | - | - | - | |
| PH01004496G0070 | LOC_Os09g28310.1 | 4.02 | 2.57 | 0.64 | 309.2 | |
| PH01001554G0250 | LOC_Os09g28310.1 | 3.98 | 2.31 | 0.58 | 306.2 | |
| PH01003683G0160 | LOC_Os08g36790.1 | 3.90 | 1.64 | 0.42 | 300.0 | |
| PH01000887G0360 | - | - | - | - | - | |
| PH01000437G0190 | LOC_Os02g52780.2 | 3.91 | 1.43 | 0.37 | 300.8 | |
| PH01000173G1010 | LOC_Os02g52780.2 | 3.90 | 1.47 | 0.38 | 300.0 | |
| PH01000075G0960 | - | - | - | - | - | |
| PH01004222G0040 | - | - | - | - | - | |
| PH01001169G0070 | LOC_Os01g07880.1 | 3.00 | 1.31 | 0.44 | 230.8 | |
| 平均值Mean | 3.63 | 2.33 | 0.65 | 279.0 | ||
| D | PH01000644G0440 | LOC_Os02g10140.1 | 3.77 | 1.71 | 0.45 | 290.0 |
| PH01000178G0580 | LOC_Os06g41100.1 | 2.61 | 4.16 | 1.60 | 200.8 | |
| PH01003751G0200 | - | - | - | - | - | |
| PH01000266G1030 | LOC_Os01g17260.2 | 5.00 | 4.93 | 0.98 | 384.6 | |
| PH01000055G1320 | - | - | - | - | - | |
| PH01000679G0010 | LOC_Os04g54474.3 | 4.01 | 1.48 | 0.37 | 308.5 | |
| PH01000303G0700 | LOC_Os08g07970.1 | 3.89 | 1.92 | 0.49 | 299.2 | |
| PH01000624G0480 | LOC_Os08g07970.1 | 3.87 | 2.09 | 0.54 | 297.7 | |
| PH01000727G0540 | LOC_Os06g15480.2 | 3.94 | 2.17 | 0.55 | 303.1 | |
| PH01003361G0120 | LOC_Os09g10840.2 | 3.60 | 1.31 | 0.36 | 276.9 | |
| PH01000049G0150 | LOC_Os01g17260.2 | 3.89 | 1.97 | 0.51 | 299.2 | |
| PH01000811G0380 | LOC_Os07g48820.1 | 2.25 | 3.01 | 1.34 | 173.1 | |
| PH01000029G0970 | LOC_Os01g59350.1 | 1.98 | 2.87 | 1.45 | 152.3 | |
| PH01001281G0440 | LOC_Os01g59350.1 | 2.75 | 3.01 | 1.09 | 211.5 | |
| PH01000013G0080 | LOC_Os03g20310.3 | 2.75 | 2.44 | 0.89 | 211.5 | |
| PH01002207G0260 | LOC_Os03g20310.3 | 4.03 | 2.03 | 0.50 | 310.0 | |
| 平均值Mean | 3.45 | 2.51 | 0.79 | 265.6 | ||
| S | PH01005435G0020 | LOC_Os09g13575.1 | 3.33 | 1.58 | 0.47 | 256.2 |
| PH01003034G0090 | LOC_Os09g13575.1 | 3.14 | 1.94 | 0.62 | 241.5 | |
| PH01000575G0620 | LOC_Os08g26880.1 | 1.33 | 0.38 | 0.29 | 102.3 | |
| PH01002178G0270 | - | - | - | - | - | |
| PH01000145G1240 | LOC_Os02g03960.1 | 1.93 | 1.14 | 0.59 | 148.5 | |
| PH01000166G0160 | - | - | - | - | - | |
| PH01004720G0080 | - | - | - | - | - | |
| PH01000302G0180 | LOC_Os12g37415.1 | 2.85 | 1.37 | 0.48 | 219.2 | |
| PH01006106G0050 | LOC_Os12g37415.1 | 1.06 | 1.69 | 1.60 | 81.5 | |
| PH01000823G0440 | LOC_Os05g03865.1 | 3.01 | 1.36 | 0.45 | 231.5 | |
| PH01276401G0010 | LOC_Os05g03865.1 | 4.21 | 1.40 | 0.33 | 323.8 | |
| PH01000104G0480 | LOC_Os08g38020.1 | 3.47 | 2.85 | 0.82 | 266.9 | |
| PH01000682G0190 | LOC_Os02g49560.1 | 2.05 | 4.21 | 2.05 | 157.7 | |
| 平均值Mean | 2.64 | 1.79 | 0.77 | 202.9 |
通过对毛竹3个亚族47条氨基酸序列利用Swiss-Model进行了三维结构同源建模,图 3为毛竹bZIP转录因子A、D和S亚族中9个具有代表性的蛋白质三级结构模型。结果显示,A和S亚族bZIP蛋白质的三级结构类似,它们的三级结构主要由α-螺旋组成。一些A和S亚族bZIP蛋白质,例如A亚族的PH01001184G0380、PH01003770G0040和PH01000075G0960以及S亚族的PH01005435G0020和PH01000823G0440除了有α-螺旋之外,还存在少许的无规卷曲结构,而S亚族PH01276401G0010只由一个α-螺旋组成。毛竹D亚族bZIP蛋白质的三维结构具有多样性,虽然同样主要是由α-螺旋组成,但是α-螺旋数量明显增多并且含有不同数目的β-折叠,如D亚族的PH01000727G0540和PH01003361G0120均含有4个α-螺旋结构,PH01000055G1320含有5个α-螺旋结构,同时,PH01000055G1320和PH01003361G0120则分别包含有1个和3个β-折叠,明显比A和S亚族复杂。同时发现同一亚族的不同成员之间的三维结构也有差异,如D亚族各个成员间的α-螺旋和β-折叠数目不尽相同,这些差异可能使得它们在功能上有所区别。
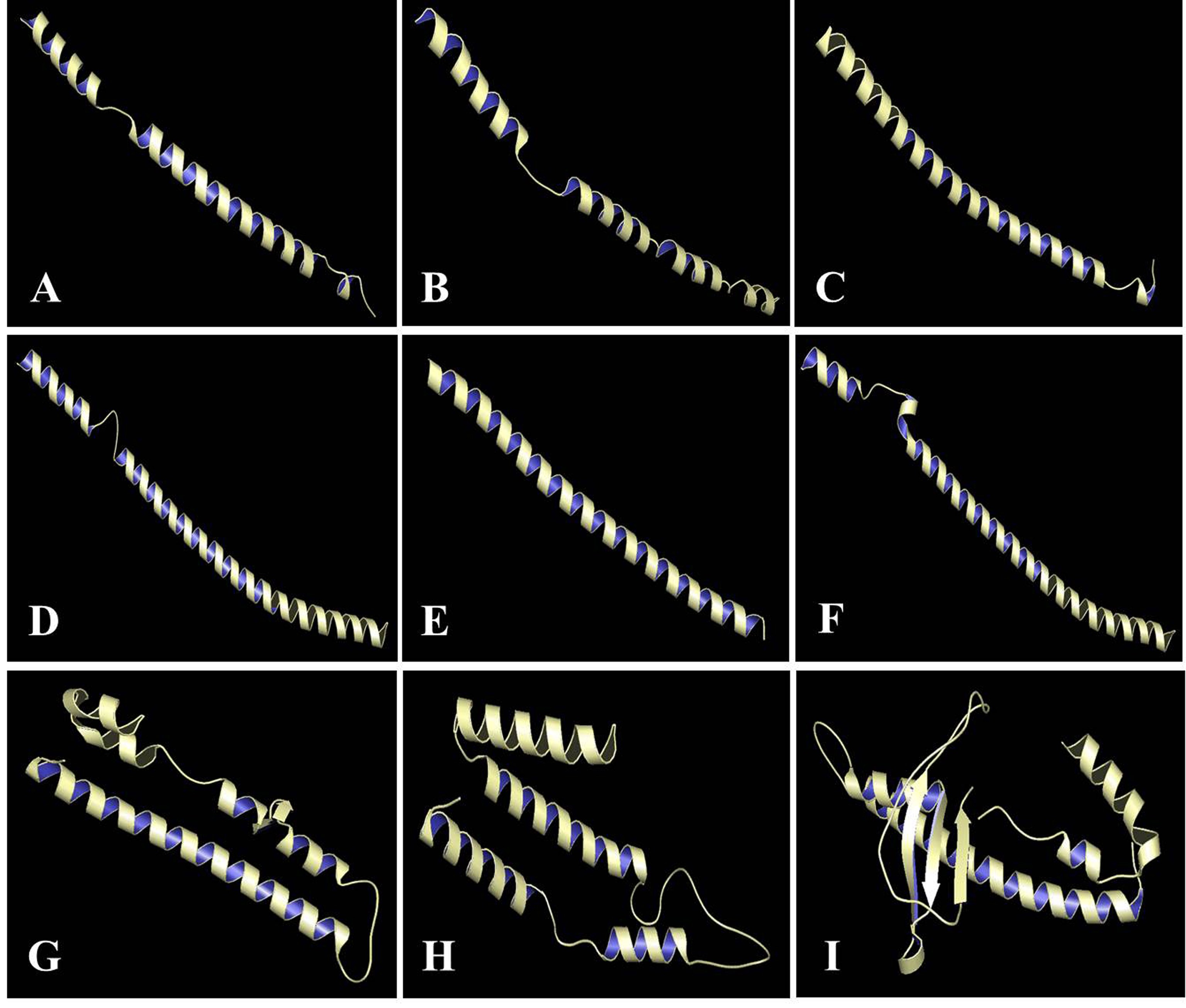 |
注:A为毛竹A亚族PH01001184G0380 bZIP蛋白质的三级结构;B为毛竹A亚族PH01003770G0040 bZIP蛋白质的三级结构; C为毛竹A亚族PH01000075G0960 bZIP蛋白质的三级结构;D为毛竹S亚族PH01005435G0020 bZIP蛋白质的三级结构; E为毛竹S亚族PH01276401G0010 bZIP蛋白质的三级结构;F为毛竹S亚族PH01000823G0440 bZIP蛋白质的三级结构; G为毛竹D亚族PH01000055G1320 bZIP蛋白质的三级结构;H为毛竹D亚族PH01000727G0540 bZIP蛋白质的三级结构; I为毛竹D亚族PH01003361G0120 bZIP蛋白质的三级结构。Notes: A represents tertiary structure of PH01001184G0380 bZIP protein of A subgroup in P. edulis; B represents tertiary structure of PH01003770G0040 bZIP protein of A subgroup in P. edulis; C represents tertiary structure of PH01000075G0960 bZIP protein of A subgroup in P. edulis; D represents tertiary structure of PH01005435G0020 bZIP protein of S subgroup in P. edulis; E represents tertiary structure of PH01276401G0010 bZIP protein of S subgroup in P. edulis; F represents tertiary structure of PH01000823G0440 bZIP protein of S subgroup in P. edulis; G represents tertiary structure of PH01000055G1320 bZIP protein of D subgroup in P. edulis; H represents tertiary structure of PH01000727G0540 bZIP protein of D subgroup in P. edulis; I represents tertiary structure of PH01003361G0120 bZIP protein of D subgroup in P. edulis. 图 3 毛竹9个代表性的bZIP蛋白质三级结构 Figure 3 Tertiary structure of nine representative bZIP proteins of P. edulis |
bZIP转录因子家族在整个真核生物界中扮演着重要角色。在植物中,bZIP可以调控包括光形态建成、叶和种子的发育、能量平衡、以及生物和非生物胁迫响应等一系列重要的生物学过程[21]。毛竹基因组中bZIP转录因子家族中涉及到植物逆境胁迫以及病原体防御和生长发育相关的A、D和S亚族的47个成员,占毛竹总bZIP转录因子的43.9%。在拟南芥中,A、D和S3个亚族bZIP转录因子的总和为40条,占拟南芥全部bZIP转录因子(75条)的53.3%[1]。在玉米(Zea mays L.)[22]和高粱(Sorghum bicolor L.)[23]中则分别占67.2%和53.3%,说明这3个亚族的bZIP转录因子成员总数占比较大,因而推测它们在植物中起到重要的作用。
进化树分析结果表明,这47个毛竹bZIP转录因子各自亚族聚到了一起,同时研究发现,同一亚族内有些基因内含子—外显子结构高度相似,这可能与毛竹bZIP基因在进化过程中经历了基因组范围内的复制和染色体区域内复制等事件有关,使毛竹bZIP基因家族中各亚族基因呈一定程度的重复性和冗余性,从而行使其功能。在选择性进化压力方面,运用MEC模型计算的结果显示,毛竹A、D和S3个亚族bZIP蛋白质一级结构位点的标注颜色大部分为白色到紫色之间,表明净化选择压力主导了毛竹3个亚族之间bZIP基因的进化。有研究表明,结合2个已知的化石节点和Li-Tanimura法估计,认为单子叶植物从双子叶植物分歧开始于140-150 Mya,同时对拟南芥、水稻和毛果杨bZIP基因的进化研究表明,bZIP基因家族的祖先在单双子叶植物分化之前就已经存在[21, 24]。因此可以说,通过计算得到的毛竹A、D和S亚族与水稻同源bZIP基因对的平均进化分歧时间279.0、265.6和202.9 Mya,均早于单双子叶植物的进化分歧时间。此外,从蛋白质的三级结构上看,A和S亚族bZIP蛋白质的三级结构比较简单且高度类似,主要由α-螺旋组成,个别蛋白质还存在少许的无规卷曲结构,而D亚族的三级结构明显比A和S亚族复杂,除了α-螺旋外,有些还具有β-折叠结构,推测D亚族进化过程比A和S亚族更复杂,从而在对抗更为复杂多变的环境过程中行使更多的生物学功能,在黄瓜(Cucumis sativus L.)[17]中也有类似的报道。本课题组对毛竹bZIP基因家族进行了初步分析,为深入研究该基因家族的表达调控、结构和功能等提供参考数据;为从整体水平上弄清bZIP转录因子调控机制,寻找bZIP上游关键调控因子奠定基础,通过调控它们的表达来提高毛竹的抗逆性,进而达到提高竹材产量和质量的目的。
| [1] | JAKOBY M, WEISSHAAR B, DROGE-LASER W, et al. bZIP transcription factors in Arabidopsis[J]. Trends in Plant Science, 2002, 7(3):106-111. |
| [2] | 王玉成,李红艳,杨传平,等.cDNA微阵列技术研究干旱胁迫下柽柳基因的表达[J].植物研究,2007,27(2):186-194. |
| [3] | 沈元月,黄丛林,张绣海,等.植物抗旱分子机制[J].中国生态农业学报,2002,10(1):30-34. |
| [4] | 杨传凤,曹颖,胡尚连,等.基于慈竹转录组MYB基因的克隆及胁迫诱导表达[J].森林与环境学报,2015,35(1):60-66. |
| [5] | UNO Y, FURIHATA T, ABE H, et al. Arabidopsis basic leucine zipper transcription factors involved in an abscisic acid-dependent signal transduction pathway under drought and high-salinity conditions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(21):11 632-11 637. |
| [6] | HOSSAIN M A, CHO J I, HAN M, et al. The ABRE-binding bZIP transcription factor OsABF2 is a positive regulator of abiotic stress and ABA signaling in rice[J]. Journal of Plant Physiology, 2010, 167(17):1 512-1 520. |
| [7] | HOSSAIN M A, LEE Y, CHO J I, et al. The bZIP transcription factor OsABF1 is an ABA responsive element binding factor that enhances abiotic stress signaling in rice[J]. Plant Molecular Biology, 2010, 72(4/5):557-566. |
| [8] | KATAGIRI F, LAM E, CHUA N H. Two tobacco DNA binding proteins with homology to the nuclear factor CREB[J]. Nature, 1989, 340(6 236):727-730. |
| [9] | SINGH K B, FOLEY R C, OÁATE-SÁNCHEZ L. Transcription factors in plant defense and stress responses[J]. Current Opinion in Plant Biology, 2002, 5(5):430-436. |
| [10] | DESPRÉS C, DELONG C, GLAZE S, et al. The Arabidopsis NPR1/NIM1 protein enhances the DNA binding activity of a group of the TGA family of bZIP transcription factors[J]. The Plant Cell Online, 2000, 12(2):279-290. |
| [11] | ZHOU J M, TRIFA Y, SILVA H, et al. NPR1 differentially interacts with members of the TGA/OBF family of transcription factors that bind an element of the PR-1 gene required for induction by salicylic acid[J]. Molecular Plant-Microbe Interactions, 2000, 13(2):191-202. |
| [12] | ZHANG Y, CHENG Y T, QU N, et al. Negative regulation of defense responses in Arabidopsis by two NPR1 paralogs[J]. Plant Journal, 2006, 48(5):647-656. |
| [13] | BLANCO F, SALINAS P. Early genomic responses to salicylic acid in Arabidopsis[J]. Plant Molecular Biology, 2009, 70(1):79-102. |
| [14] | PENG Z, LU Y, LI L, et al. The draft genome of the fast-growing non-timber forest species moso bamboo (Phyllostachys heterocycla)[J]. Nature Genetics, 2013, 45(4):456-461. |
| [15] | STERN A, DORON-FAIGENBOIM A, EREZ E, et al. Selecton 2007: advanced models for detecting positive and purifying selection using a Bayesian inference approach[J]. Nucleic Acids Research, 2007, 35(Web Server Issue): W506-W511. |
| [16] | WANG D, ZHANG Y, ZHANG Z, et al. KaKs_Calculator 2.0: a toolkit incorporating gamma-series methods and sliding window strategies[J]. Genomics Proteomics Bioinformatics, 2010, 8(1):77-80. |
| [17] | BALOGLU M C, ELDEM V, HAJYZADEH M, et al. Genome-wide analysis of the bZIP transcription factors in cucumber[J]. Public Library of Science One, 2014, 9(4):e96 014. |
| [18] | BETTS M J, GUIG R, AGARWAL P, et al. Exon structure conservation despite low sequence similarity: a relic of dramatic events in evolution[J]. The European Molecular Biology Organization Journal, 2001, 20(19):5 354-5 360. |
| [19] | NEI M. Selectionism and neutralism in molecular evolution[J]. Molecular Biology and Evolution, 2005, 22(10):2 318-2 342. |
| [20] | KIMURA M. Preponderance of synonymous changes as evidence for the neutral theory of molecular evolution[J]. Nature, 1977, 267(5 608):275-276. |
| [21] | CORREA L G, RIANO-PACHON D M, SCHRAGO C G, et al. The role of bZIP transcription factors in green plant evolution: adaptive features emerging from four founder genes[J]. Public Library of Science One, 2008, 3(8):e2 944. |
| [22] | WEI K, CHEN J, WANG Y, et al. Genome-wide analysis of bZIP-encoding genes in maize[J]. DNA Research, 2012, 19(6):463-476. |
| [23] | WANG J, ZHOU J, ZHANG B, et al. Genome wide expansion and expression divergence of the basic leucine zipper transcription factors in higher plants with an emphasis on sorghum[J]. Journal of Integrative Plant Biology, 2011, 53(3):212-231. |
| [24] | CHAW S M, CHANG C C, CHEN H L, et al. Dating the monocot-dicot divergence and the origin of core eudicots using whole chloroplast genome[J]. Journal of Molecular Evolution, 2004, 58(4):424-441. |

 2016, Vol. 36
2016, Vol. 36


