2. 上海海洋大学 海洋动物系统分类与进化上海高校重点实验室, 上海 201306
 (Acheilognathus macropterus),表明崇明岛内河鱼类群落结构由淡水鱼类主导。主坐标分析和PERMANOVA检验结果显示东-西部群落鱼类组成存在差异(R2=0.101 2,P=0.027 6)。研究结果亦表明eDNA技术可以作为传统鱼类资源调查的一个重要辅助手段。本研究为崇明岛内河鱼类资源现存状况、合理利用及保护提供了基础资料。
(Acheilognathus macropterus),表明崇明岛内河鱼类群落结构由淡水鱼类主导。主坐标分析和PERMANOVA检验结果显示东-西部群落鱼类组成存在差异(R2=0.101 2,P=0.027 6)。研究结果亦表明eDNA技术可以作为传统鱼类资源调查的一个重要辅助手段。本研究为崇明岛内河鱼类资源现存状况、合理利用及保护提供了基础资料。上海崇明岛地处长江入海口,岛内河网密布,蕴含丰富的鱼类资源。过去已有研究围绕崇明岛鱼类多样性这一方向开展[1-3],但这些研究中,都仅利用撒网、拖网等传统捕捞方法进行。某些鱼类具有隐蔽性强、昼伏夜出等生活习性,使得其难以被捕获。因此,单一利用传统捕捞方法调查鱼类组成具有一定的局限性,不一定能充分揭示研究区域的种类组成。
环境DNA(eDNA)是指从环境样本中提取到的DNA片段,是来自微生物、动物、植物等不同物种DNA的混合物[4]。通过不同的DNA宏条形码,扩增环境样本中特异的DNA片段,用以检测环境中物种种类和数量的技术称为eDNA技术。近年来,eDNA技术以其灵敏度高、省时高效等特性在物种监测方面得到广泛应用:王晨等[5]利用eDNA技术调查了秦淮河鱼类资源;ALAM等[6]在韩国4条河流发现了多种入侵鱼类;徐念等[7]在长江中下游检测出长江江豚序列。还有其他水生生物的研究[8-14]应用eDNA技术,得到了传统方法未能获得的信息。目前,已有较多研究将eDNA技术应用于长江流域鱼类多样性研究中[5, 7-8, 15-16],但尚无关于崇明岛鱼类多样性的研究。另外,崇明岛内河存在违规捕捞现象,破坏了鱼类资源[17]。为实现“长江十年禁渔计划”及“崇明世界级生态岛”发展目标,在崇明岛开展全面的鱼类多样性调查十分有必要。本研究以崇明岛内河为调查区域,利用eDNA宏条形码技术结合传统捕捞技术,探究崇明岛内河鱼类资源及多样性,以期为“长江十年禁渔计划”及“崇明世界级生态岛”建设的水环境治理和鱼类多样性保护提供参考。
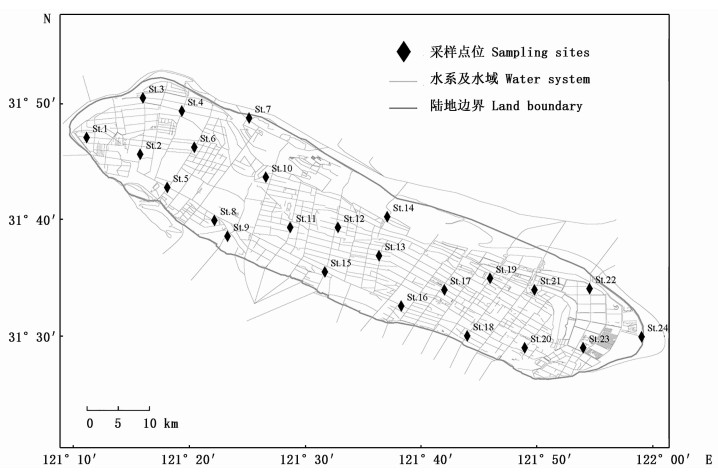
1 材料与方法 1.1 样品采集及处理在崇明岛2条市级河道(南横引河、北横引河)、数条县级河道、镇级河道、村级河道设置24个采样位点(图 1)。于2020年6月至2021年5月期间,春季(2021年5月25日至28日)、夏季(2020年6月28日至7月4日)、秋季(2020年10月6日至8日)、冬季(2021年1月30日至2月2日)各进行一次采样工作。对于传统捕捞方法采样:每个位点放置2张刺网(100 m长2指宽、100 m长4指宽)及2个地笼,放置时间为2 h。在获得位点鱼类标本后,首先拍摄样本照片,随后将样本装入密封袋并写上采样点信息,放入便携式冰箱,带回实验室鉴定。eDNA调查方法:每个位点采集河道表层、中层、底层(每个水层断面左、中、右)水样共10 L,混匀后取3 L水样进行现场真空抽滤。抽滤使用3 μm孔径的聚碳酸酯滤膜(GVS公司)对eDNA进行截留,每张滤膜过滤1 L水样,同时每个采样点均抽取1 L蒸馏水作为空白对照。为避免交叉污染,过滤器和手套均为一次性,在每个样点采样后丢弃。抽滤前,抽滤装置均用84消毒液润洗1次,再用采样点的水样润洗3次。抽滤完成后将滤膜保存至装有700 μL CTAB溶液的无菌无酶的2 mL离心管中,放至便携式冰箱储存(冰箱温度控制在0~4 ℃),直至带回实验室进行DNA提取。
|
图 1 崇明岛内河24个采样点 Fig. 1 24 sampling sites in the rivers of the Chongming Island |
鱼类样品在鉴定前用自来水清洗掉表面泥沙污垢,然后根据《中国动物志:硬骨鱼纲鲈形目-虾虎鱼亚目》[18]、《中国动物志: 硬骨鱼纲鲤形目(中卷)》[19]、《长江口鱼类》[20]对样品进行鉴定、分类,记录各物种样本数。
1.3 eDNA提取、扩增及测序eDNA的提取基于酚氯仿DNA提取法原理,作出了一些修改。首先将离心管在65 ℃下加热10 min,随后向离心管内加入氯仿-异戊醇混合溶液(24∶1),涡旋振荡5分钟后在15 000 g下离心15 min,将上清液移入新的无菌无酶的2 mL离心管中,再依次加入500 μL冰镇异丙醇、250 μL浓度为5摩尔质量的氯化钠溶液,放入-20 ℃冰箱冷冻1小时。冷冻后取出,在15 000 g下离心15分钟,轻轻倒出废液。加入150 μL 70%乙醇,在15 000 g下离心5 min,轻轻倒出废液,再次加入150 μL 70%酒精及在15 000 g下离心5 min。倒出废液后,在真空离心空干机中以45 ℃空干10 min。最后加入100 μL TE缓冲液,在55 ℃下加热10 min,得到eDNA溶液。
对于同一样点的3份水样,首先将提取出的eDNA溶液混成1管,然后分出3个平行管,再而利用MiFish鱼类通用引物[21-22](表 1)进行PCR扩增。扩增体系与测序文库构建方法一致遵循MIYA等[22]所描述,同一样点的3个平行采用同一组测序接头索引序列以方便后续总结每物种的序列数。空白对照的实验操作与样品一致。测序平台为Illumina NovaSeq PE150(北京诺禾致源科技股份有限公司)。

|
表 1 本研究中使用的MiFish引物 Tab.1 MiFish primers used in this study |
将原始测序数据去除测序接头,上传至MiFish Pipeline[23]网站(http://mitofish.aori.u-tokyo.ac.jp/mifish)进行一站式分析,过程包括序列组装、引物去除、数据库序列比对。使用的序列比对数据库为MitoFish数据库[24],eDNA序列与数据库序列相似度阈值设置为97%,即eDNA序列与数据库序列有≥97%相似度时,记录该eDNA序列及数据库序列对应的物种名称,以此得到利用eDNA技术检测出的物种信息。将各位点同种鱼类序列数相加得到该种的总序列数。最后,分别基于捕获鱼类个体数、eDNA检出鱼类对应的总序列数,计算4个季节和全年的香农-威纳(Shannon-Wiener)多样性指数,并采用McNaughton优势度指数确定优势种。指数计算公式如下:
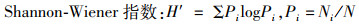 (1)
(1)
 (2)
(2)
式中:N为所捕获鱼类总个体数或序列数,Ni为第i种鱼类的个体数或序列数,Yi为第i种鱼类的优势度指数(Yi> 0.02的物种为优势种),fi为第i种鱼类出现的频率。
基于eDNA检出结果,使用R语言中的vegan、ape包对24个采样位点进行主坐标分析(PCoA)。然后根据24个采样位点的采样位置进行分组,一组为南支(位点1、5、8、9、15、16、18、20)、北支(位点3、4、7、14、22、24)、竖河(位点2、6、10、11、12、13、17、19、21、23),另一组为西部(位点1、2、3、4、5、6、7)、中部(位点8、9、10、11、12、13、14、15)、东部(位点16、17、18、19、20、21、22、23、24),分析南支、北支、竖河间以及西部、中部、东部河道间物种组成差异,并利用PERMANOVA检验进行显著性检验。
2 结果 2.1 鱼类组成和多样性2020年至2021年4次调查,共采集鱼类样本2 095尾,鉴定出38种鱼类(表 2);eDNA技术检出75种鱼类(表 3)。综合渔网捕获样本种类与eDNA技术检出种类,在崇明岛内河共检出78种鱼类。其中:鲤形目鱼类最多,有34种,占检出物种数的44%;鲈形目次之,有24种,占检出物种数的31%;鲇形目有4种,占检出物种数的5%;鲻形目、鲀形目都各有3种,各占检出物种数的4%;鲱形目、鲑形目各有2种,各占检出物种数的3%;鳗鲡目、鲹形目、鲽形目、颌针鱼目、鳉形目、合鳃鱼目都仅有1种,各占检出物种数的1%。

|
表 2 基于传统捕捞法在崇明岛内河检出的鱼类物种个体数、物种优势度 Tab.2 Number of individuals and dominance index (Y) of fish detected in the river of Chongming Island based on traditional fishing method |

|
表 3 基于eDNA技术在崇明岛内河检出的鱼类物种序列数、物种优势度 Tab.3 Number of sequences and dominance index (Y) of fish detected in the river of Chongming Island based on eDNA method |
基于捕获的鱼类样本种类和数目,计算得到春季的Shannon-Wiener多样性指数为2.33,夏季为1.82,秋季为2.34,冬季为2.49,全年为2.08。全年优势种为子陵吻虾虎鱼、似鳊、鳊、麦穗鱼、大鳍鱊、中华鳑鲏(表 2)。基于eDNA检出的鱼类序列数,计算得到春季的Shannon-Wiener多样性指数为2.78,夏季为2.45,秋季为3.17,冬季为2.71,全年为2.96。全年优势种为刀鲚、子陵吻虾虎鱼、斑尾刺虾虎鱼、鲫、鲢、鳙、细鳞鲴、似鳊、鳊、达氏鲌、蒙古鲌、麦穗鱼、大鳍
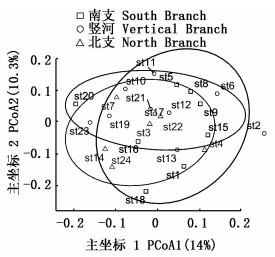
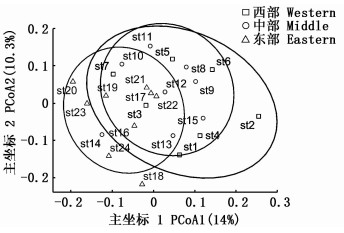
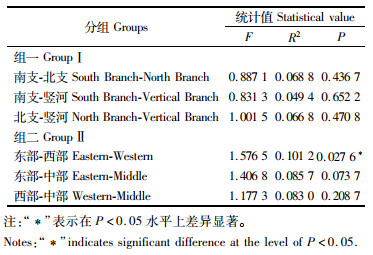
使用PCoA将数据降为二维,PCo1和PCo2两个主坐标共解释24.3%的差异。在PCoA图中,无论在南支、北支、竖河组(图 2)或西部、中部、东部组(图 3)中,组内各小组重叠较多,没显著分开。PERMANOVA检验结果(表 4)表明:南支、北支、竖河组间可解释部分(R2)较小,无显著差异;东部与西部组间R2最大,两组间鱼类种类组成存在差异;其余组间鱼类种类组成无显著差异。
|
图 2 南支、北支、竖河组主坐标分析 Fig. 2 Principal coordinates analysis of group of the south branch, north branch, and vertical branch |
|
图 3 西部、中部、东部组主坐标分析 Fig. 3 Principal coordinates analysis of group of western, middle, and eastern |
|
表 4 两大组内各小组间PERMANOVA检验 Tab.4 PERMANOVA test between subgroups in two main groups |
在α多样性上,本调查除了检出常见的淡水鱼类,如鲫、草、鲤等,也检出了洄游鱼类,如鳗鲡(降海洄游)、刀鲚(溯河洄游)等;近海鱼类,如鲻、
优势种是指对群落结构和群落环境的形成有明显控制作用的物种[30]。两种方法计算出的共同优势种为鳊、似鳊、麦穗鱼、子陵吻虾虎鱼、大鳍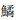
本调查中,刺网/地笼捕获种类与eDNA技术检出种类相差43种,eDNA未检测到传统捕捞法获得的3种鱼类:弹涂鱼、波氏吻虾虎鱼、蛇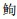
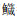
eDNA技术检测鱼类多样性的原理是利用特定引物扩增鱼类序列,然后根据扩增的序列再比对到数据库上已有的序列,得出检出鱼类物种。但由于引物特性,某些鱼类物种序列无法被扩增,或者数据库的不完整性,扩增出的序列无法对比数据库已知序列中,这两种原因都会导致eDNA结果的“假阴性”。目前,已有针对eDNA采样与分析流程优化的研究[38]。本研究中eDNA技术亦无法检出由传统方法捕获的弹涂鱼、波氏吻虾虎鱼、蛇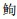
综上,本调查结合传统捕捞法和eDNA技术,揭示了崇明岛内河鱼类种类组成、鱼类物种多样性现状,也侧面反映崇明河道水环境现状。同时,eDNA技术能检测出传统鱼类多样性调查手段难以捕获的物种,可以作为鱼类资源调查的一个重要补充手段。
| [1] |
刘凯, 张敏莹, 徐东坡, 等. 长江春季禁渔对崇明北滩渔业群落的影响[J]. 中国水产科学, 2006, 13(5): 834-840. LIU K, ZHANG M Y, XU D P, et al. Effects of spring closed season on fishery community of the north beach of Chongming, the Yangtze River[J]. Journal of Fishery Sciences of China, 2006, 13(5): 834-840. DOI:10.3321/j.issn:1005-8737.2006.05.022 |
| [2] |
张涛, 庄平, 刘健, 等. 长江口崇明东滩鱼类群落组成和生物多样性[J]. 生态学杂志, 2009, 28(10): 2056-2062. ZHANG T, ZHUANG P, LIU J, et al. Species composition and biodiversity of fish community in Chongming Dongtan of Yangtze River estuary[J]. Chinese Journal of Ecology, 2009, 28(10): 2056-2062. |
| [3] |
从婷婷, 童春富, 赵成建, 等. 崇明岛内河夏季鱼类群落组成及分布特征[J]. 生态学报, 2021, 41(5): 2067-2076. CONG T T, TONG C F, ZHAO C J, et al. Community composition and distribution characteristics of the fish assemblages in the rivers of Chongming Island in summer[J]. Acta Ecologica Sinica, 2021, 41(5): 2067-2076. |
| [4] |
TABERLET P, COISSAC E, HAJIBABAEI M, et al. Environmental DNA[J]. Molecular Ecology, 2012, 21(8): 1789-1793. DOI:10.1111/j.1365-294X.2012.05542.x |
| [5] |
王晨, 陶孟, 李爱民, 等. 基于环境DNA宏条形码技术的秦淮河生物多样性研究[J]. 生态学报, 2022, 42(2): 611-624. WANG C, TAO M, LI A M, et al. Research on the biodiversity of Qinhuai River based on environmental DNA metabacroding[J]. Acta Ecologica Sinica, 2022, 42(2): 611-624. |
| [6] |
ALAM M J, KIM N K, ANDRIYONO S, et al. Assessment of fish biodiversity in four Korean rivers using environmental DNA metabarcoding[J]. PeerJ, 2020, 8(7): e9508. |
| [7] |
徐念, 熊美华, 邵科, 等. 长江中下游环境DNA宏条形码生物多样性检测技术初步研究[J]. 环境科学研究, 2020, 33(5): 1187-1196. XU N, XIONG M H, SHAO K, et al. Preliminary study on environmental DNA metabarcoding for detecting biodiversity in the middle and lower reaches of the Yangtze River[J]. Research of Environmental Sciences, 2020, 33(5): 1187-1196. DOI:10.13198/j.issn.1001-6929.2020.03.06 |
| [8] |
YU D, SHEN Z Y, CHANG T, et al. Using environmental DNA methods to improve detectability in an endangered sturgeon (Acipenser sinensis) monitoring program[J]. BMC Ecology and Evolution, 2021, 21(1): 216. DOI:10.1186/s12862-021-01948-w |
| [9] |
EUCLIDE P T, LOR Y, SPEAR M J, et al. Environmental DNA metabarcoding as a tool for biodiversity assessment and monitoring: reconstructing established fish communities of north-temperate lakes and rivers[J]. Diversity and Distributions, 2021, 27(10): 1966-1980. DOI:10.1111/ddi.13253 |
| [10] |
VENCES M, LYRA M L, PERL R G B, et al. Freshwater vertebrate metabarcoding on Illumina platforms using double-indexed primers of the mitochondrial 16S rRNA gene[J]. Conservation Genetics Resources, 2016, 8(3): 323-327. DOI:10.1007/s12686-016-0550-y |
| [11] |
PLANTE F, BOURGAULT P, DUBOIS Y, et al. Environmental DNA as a detection and quantitative tool for stream-dwelling salamanders: A comparison with the traditional active search method[J]. Environmental DNA, 2021, 3(6): 1128-1141. DOI:10.1002/edn3.233 |
| [12] |
XU N, ZHU B, SHI F, et al. Monitoring seasonal distribution of an endangered anadromous sturgeon in a large river using environmental DNA[J]. The Science of Nature, 2018, 105(11/12): 62. |
| [13] |
吴昀晟, 唐永凯, 李建林, 等. 环境DNA在长江江豚监测中的应用[J]. 中国水产科学, 2019, 26(1): 124-132. WU Y S, TANG Y K, LI J L, et al. The application of environmental DNA in the monitoring of the Yangtze finless porpoise, Neophocaena phocaenoides asaeorientalis[J]. Journal of Fishery Sciences of China, 2019, 26(1): 124-132. |
| [14] |
闫卉果, 董智玲, 马婷婷, 等. 基于环境DNA的岩原鲤检测及生物量评估[J]. 水产学报, 2022, 26(6): 1018-1026. YAN H G, DONG Z L, MA T T, et al. Detection and biomass assessment of Procypris rabaudi based on environmental DNA[J]. Journal of Fisheries of China, 2022, 26(6): 1018-1026. |
| [15] |
周春花, 钟伟翔, 陈金萍, 等. 利用沉积物和水体的环境DNA检测鱼类物种多样性的差异[J]. 南昌大学学报(理科版), 2021, 45(6): 565-570, 577. ZHOU C H, ZHONG W X, CHEN J P, et al. Difference in fish species diversity detected by environmental DNA from sediments and water[J]. Journal of Nanchang University (Natural Science), 2021, 45(6): 565-570, 577. |
| [16] |
王梦, 杨鑫, 王维, 等. 基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究[J]. 水生生物学报, 2022, 46(1): 2-16. WANG M, YANG X, WANG W, et al. Fish diversity in Chongqing section of the national nature reserve for rare and endemic fish in the upper Yangtze River based on eDNA technology[J]. Acta Hydrobiologica Sinica, 2022, 46(1): 2-16. |
| [17] |
史树明. 上海市崇明区捕捞渔业存在的问题及相关建议[J]. 上海农业科技, 2019(1): 8-9. SHI S M. The problems of fisheries of Chongming district, Shanghai, and the management recommendations[J]. Shanghai Agricultural Science and Technology, 2019(1): 8-9. |
| [18] |
伍汉霖, 钟俊生. 中国动物志: 硬骨鱼纲鲈形目-虾虎鱼亚目[M]. 北京: 科学出版社, 2008. WU H L, ZHONG J S. Fauna Sinica: Osteichthyes, Perciformes V[M]. Beijing: Science Press, 2008. |
| [19] |
陈宜瑜. 中国动物志: 硬骨鱼纲鲤形目(中卷)[M]. 北京: 科学出版社, 1998. CHEN Y Y. Fauna Sinica: Osteichthyes, Cypriniformes Ⅱ[M]. Beijing: Science Press, 1998. |
| [20] |
庄平. 长江口鱼类[M]. 上海: 上海科学技术出版社, 2006. ZHUANG P. Fishes of the Yangtze estuary[M]. Shanghai: Shanghai Scientific & Technical Publishers, 2006. |
| [21] |
MIYA M, SATO Y, FUKUNAGA T, et al. MiFish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species[J]. Royal Society Open Science, 2015, 2(7): 150088. |
| [22] |
MIYA M, SADO T. Environmental DNA sampling and experiment manual[M]. Otsu: The eDNA Society, 2019: 30-66.
|
| [23] |
SATO Y, MIYA M, FUKUNAGA T, et al. MitoFish and MiFish Pipeline: a mitochondrial genome database of fish with an analysis pipeline for environmental DNA metabarcoding[J]. Molecular Biology and Evolution, 2018, 35(6): 1553-1555. |
| [24] |
IWASAKI W, FUKUNAGA T, ISAGOZAWA R, et al. MitoFish and MitoAnnotator: a mitochondrial genome database of fish with an accurate and automatic annotation pipeline[J]. Molecular Biology and Evolution, 2013, 30(11): 2531-2540. |
| [25] |
张颖纯, 姜德刚, 李建华. 崇明岛"闸控型"河网水体富营养化特征及其影响因素[J]. 湖泊科学, 2013, 25(3): 366-372. ZHANG Y C, JIANG D G, LI J H. Characteristics of eutrophication and its affecting factors in gate-controlled river network system of Chongming Island[J]. Journal of Lake Sciences, 2013, 25(3): 366-372. |
| [26] |
吴逢润, 童春富. 近30年长江口北支演变及其对物种多样性的影响[J]. 海洋学报, 2017, 39(2): 72-85. WU F R, TONG C F. Evolution of the North Branch of Yangtze Estuary in last 30 years and corresponding effects on species diversity[J]. Haiyang Xuebao, 2017, 39(2): 72-85. |
| [27] |
李娣, 李旭文, 牛志春, 等. 江苏省不同营养状况湖泊底栖动物群落结构与多样性比较[J]. 生态毒理学报, 2017, 12(1): 163-172. LI D, LI X W, NIU Z C, et al. A comparative study on macrobenthic community structure and diversity in different trophic status lakes of Jiangsu province[J]. Asian Journal of Ecotoxicology, 2017, 12(1): 163-172. |
| [28] |
余文娟, 沈建忠, 龚江, 等. 长江中游贝氏 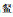 繁殖生物学研究[J]. 淡水渔业, 2018, 48(3): 53-60. 繁殖生物学研究[J]. 淡水渔业, 2018, 48(3): 53-60.YU W J, SHEN J Z, GONG J, et al. Reproductive biology of Hemiculter bleekeri in the middle reaches of the Yangtze River[J]. Freshwater Fisheries, 2018, 48(3): 53-60. |
| [29] |
乐观. 崇明岛河道水质理化指标及其与三种主要浮游动物分布的相关性研究[D]. 上海: 华东师范大学, 2008: 9-15. LE G. Studies on physico-chemical parameter and its correlation with the distribution of three main zooplanktons collected from rivers in Chongming Island[D]. Shanghai: East China Normal University, 2008: 9-15. |
| [30] |
牛翠娟, 娄安如, 孙儒泳, 等. 基础生态学[M]. 3版. 北京: 高等教育出版社, 2015. NIU C J, LOU A R, SUN R Y, et al. Foundations in ecology[M]. 3rd ed. Beijing: Higher Education Press, 2015. |
| [31] |
徐晓丽, 张彩军, 吴剑峰, 等. 麦穗鱼对盐度和酸碱度的耐受性[J]. 湖北农业科学, 2012, 51(7): 1423-1425. XU X L, ZHANG C J, WU J F, et al. Study on the tolerance of salinity and pH in the fish, Pseudorasbora parva[J]. Hubei Agricultural Sciences, 2012, 51(7): 1423-1425. |
| [32] |
夏曼莉. 大鳍  (鳑鲏)对水质和沉水植物生长的影响及其对浅水湖泊生态修复的启示[D]. 广州: 暨南大学, 2020: 38-39. (鳑鲏)对水质和沉水植物生长的影响及其对浅水湖泊生态修复的启示[D]. 广州: 暨南大学, 2020: 38-39. XIA M L. Effects of Acheilognathus macropterus (bitterling fish) on water quality and growth of submerged macrophytes: implications for shallow lake restoration[D]. Guangzhou: Jinan University, 2020: 38-39. |
| [33] |
钟爱华, 李明云. 中华乌塘鳢的生物学特性及人工育苗研究进展[J]. 浙江海洋学院学报(自然科学版), 2002, 21(3): 269-272. ZHONG A H, LI M Y. The advancement of the biological characters and the artificial propagation of Bostrichtys sinesis[J]. Journal of Zhejiang Ocean University (Nature Science), 2002, 21(3): 269-272. |
| [34] |
赵永军, 徐文彦, 赵慧. 鳜、鳢、鳡、鲶的生态习性[J]. 水产科学, 2004, 23(6): 26-27. ZHAO Y J, XU W Y, ZHANG H. Ecology of Siniperperca chuatsi, Ophicephalus argu, Elopichthys bambusa, and Silurus asotus[J]. Fisheries Science, 2004, 23(6): 26-27. |
| [35] |
刘孝华. 泥鳅的生物学特性及养殖技术[J]. 湖北农业科学, 2008, 47(1): 93-95. LIU X H. Biological characteristics and cultivation of Loach[J]. Hubei Agricultural Sciences, 2008, 47(1): 93-95. |
| [36] |
赵子明, 刘美剑. 我国刺鳅生物学研究进展[J]. 江苏农业科学, 2017, 45(4): 9-12. ZHAO Z M, LIU M J. Recent progress in the biology of the family Mastacembelidae in China[J]. Jiangsu Agricultural Sciences, 2017, 45(4): 9-12. |
| [37] |
DEINER K, FRONHOFER E A, MÄCHLER E, et al. Environmental DNA reveals that rivers are conveyer belts of biodiversity information[J]. Nature Communications, 2016, 7(1): 12544. |
| [38] |
BUXTON A, MATECHOU E, GRIFFIN J, et al. Optimising sampling and analysis protocols in environmental DNA studies[J]. Scientific Reports, 2021, 11(1): 11637. |
2. Shanghai Universities Key Laboratory of Marine Animal Taxonomy and Evolution, Shanghai Ocean University, Shanghai 201306, China
 2022,
Vol. 31
2022,
Vol. 31


