2. 上海海洋大学 海洋科学学院, 上海 201306;
3. 华南农业大学 海洋学院, 广东 广州 510642;
4. 大连海洋大学 水产与生命学院, 辽宁 大连 116023;
5. 南方海洋科学与工程广东省实验室, 广东 广州 511458
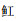 (Taeniura meyeni)。通过与珠江河口不同海域的两项类似的研究对比发现,研究区域、样品采集策略和采用的条形码标记的不同使得检测到的鱼类种类的数量和组成上有明显差异。研究表明,环境DNA宏条形码技术适用于珠江河口伶仃洋鱼类种类组成调查,可以作为传统调查方法的一种补充手段,用于河口区鱼类多样性调查和研究。
(Taeniura meyeni)。通过与珠江河口不同海域的两项类似的研究对比发现,研究区域、样品采集策略和采用的条形码标记的不同使得检测到的鱼类种类的数量和组成上有明显差异。研究表明,环境DNA宏条形码技术适用于珠江河口伶仃洋鱼类种类组成调查,可以作为传统调查方法的一种补充手段,用于河口区鱼类多样性调查和研究。河口水域生态系统是海洋生态系统的重要组成部分,也是多种海洋性鱼类索饵、产卵和育肥的重要场所,对海洋生态系统中鱼类资源的补充及可持续利用具有重要作用[1]。鱼类多样性是水生生态系统健康监测的关键指标,研究鱼类多样性可以有效了解水生生态系统的变化和状态,为资源保护、生态系统恢复、渔业资源的可持续利用和科学管理提供重要的参考[2]。以往鱼类多样性的研究主要是通过捕捞的方式进行采样的,然后进行形态学鉴定和计数称重,以此识别鱼类种类并统计鱼类丰度与生物量[3-4]。传统的调查采样方法普遍存在调查费用高、所需时间久、采样效率低、对生物的破坏性大以及严格依赖鱼类分类学专家等缺点,使鱼类多样性的研究受到一定的限制。
环境DNA(environmental DNA, eDNA)宏条形码(metabarcoding)技术通过直接提取环境样品(如水、沉积物、土壤等)中的DNA,运用高通量测序(high-throughput sequencing, HTS)技术,可同时对环境样本中存在的多个物种进行识别[5]。环境DNA宏条形码技术具有操作简单、灵敏度高、对生物体以及生态系统无破坏性、不受水域以及休渔制度的影响等一系列优势,能够弥补传统鱼类资源调查方法的不足,非常适合用于河口等生态系统鱼类多样性的研究,是调查和评估鱼类多样性的一个潜在的强大工具。目前,基于环境DNA宏条形码技术的鱼类多样性的研究中,常用的分子标记有12S基因[6-8]、COI基因[9-11]以及Cyt b基因[12-14],其中MIYA[15]等设计的引物MiFish-U使用最为广泛,该引物以鱼类12S rRNA基因高变区(163~185 bp)为靶标,该区域包含了足够的信息,可用于鱼类在科、属和种水平上的分类鉴定,且非常适合读长短的高通量测序[16]。
近年来,环境DNA宏条形码技术发展迅速,已在多个类型水域得到应用,如淡水河流[17]、湖泊[18]和海洋[19]等。该技术因其具备的众多优势逐渐被科研工作者重视,目前国内运用环境DNA宏条形码技术进行河口鱼类多样性的研究非常有限,在珠江河口海域,仅有ZOU等[20]和CHEANG等[21]分别对内河口的南沙湿地附近水域和香港西部海域(图 1)进行过研究,所涉及的样品采集范围较小。本研究选取珠江河口伶仃洋相对靠外的海域(图 1)为研究区域,以12S rRNA基因短序列为标记,运用环境DNA宏条形码技术对鱼类种类组成进行初步探究,并与上述两个不同采样范围的研究进行对比,分析珠江河口不同区域的鱼类种类组成差异,为珠江河口伶仃洋鱼类多样性监测以及空间变化的研究提供新的思路。
|
红色虚线框代表本文样品采集区域,蓝色虚线框代表ZOU等的样品采集区域,绿色虚线框代表CHEANG等的样品采集区域。 The study area of this paper is indicated by the red rectangle, the study area of ZOU, et al is indicated by the blue rectangle, the study area of CHEANG, et al is indicated by the green rectangle. 图 1 珠江口伶仃洋环境DNA水样采集位点示意图 Fig. 1 eDNA sampling locations in Lingding Bay of the Pearl River Estuary |
水体样本于2017年8月采集于珠江口伶仃洋海域,共采集了7个位点(图 1)。每个位点利用采水器采集近表层和近底层水样各1 L,混合保存于无菌塑料瓶中带回实验室。使用直径47 mm、孔径0.45 μm的硝酸纤维滤膜(WhatmanTM,美国GE Healthcare Life Sciences)进行真空过滤。每张滤膜过滤一个采样点的水样(共2 L),用无水乙醇浸透并装入5 mL无菌冻存管中,置于- 40 ℃低温保存箱保存。为了避免交叉污染,过滤装置依次经过次氯酸钠浸洗、蒸馏水冲洗和水样润洗。每个采样点均以相同的方式过滤2 L蒸馏水作为阴性对照。
1.2 环境DNA的提取使用血液与组织基因组DNA提取试剂盒(Qiagen)提取滤膜上的环境DNA。提取步骤依据试剂盒说明书并稍作修改,具体步骤如下:(1)取1张滤膜置于2 mL的离心管中,剪碎;(2)加入800 μL Buffer ATL和80 μL蛋白酶K后,加入2颗直径5 mm的小钢珠,使用快速组织细胞破碎仪(Bullet Blender STORM, 美国)研磨15 s后,置于56 ℃恒温水浴2.5 h,每30 min旋涡震荡15 s;(3)加入400 μL Buffer AL,混匀,56 ℃恒温水浴20 min,12 000 r/min离心30 s,取900 μL上清液转移至新的2 mL离心管中。(4)加入400 μL无水乙醇,混匀后分两次将混合物转移至吸附柱中,12 000 r/min离心30 s,弃去滤液和收集管。(5)将吸附柱放入新的收集管中,加入500 μL Buffer AW1,12 000 r/min离心30 s,弃去滤出液及收集管。(6)将吸附柱放入新的收集管中,加入500μL Buffer AW2,12 000 r/min离心3 min,弃去滤液及收集管。(7)将吸附柱转移至新的1.5 mL离心管中,加入100 μL Buffer AE以洗脱DNA,室温(15~25 ℃)放置2 min,12 000 r/min离心2 min。重复洗脱2~3次以提高DNA产率。
所有试剂和耗材自购置后分装成小份,每个样本的提取均使用独立的1份,以防止交叉污染,提取过程在超净工作台中进行。并使用同样的方法提取蒸馏水滤膜样本作为空白对照组。提取产物用微量分光光度计检测浓度,并用2%琼脂糖凝胶电泳检测对照组有无污染。
1.3 目的序列的获取环境总DNA使用引物MiFish-U[15](正向:5′-GTCGGTAAAACTCGTGCCAGC-3′,反向:5′-CATAGTGGGGTATCTAATCCCAGTTTG-3′)进行扩增。为提高目的序列的浓度,对提取的DNA产物进行两次扩增。扩增体系如下:TaKaRa Ex Taq(5U/L)0.25 μL,10 x Ex Taq buffer(20 mmol/L Mg2+ plus)2.5 μL,dNTP mixture 2 μL,上游引物(5′端,10 pmol/μL)0.7 μL,下游引物(3′端,10 pmol/μL)0.7 μL,DNA模板2 μL,补充灭菌去离子水至总体系25 μL。扩增程序如下:94 ℃预变性3 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸20 s,共进行35个循环;最后72 ℃延伸2 min。将第一次PCR扩增产物稀释10倍并作为第二次PCR扩增的模板,在原始引物的5′端(上游和下游)添加6个碱基的barcode序列作为第二次PCR扩增引物,其余组分保持不变。两次PCR扩增程序不变。PCR产物均经1%琼脂糖凝胶电泳检测,最终产物送至华大基因公司(青岛)进行测序。
1.4 数据处理下机数据经过数据过滤,滤除低质量的读长(Reads),剩余高质量的净数据(Clean datas)可用于后期分析;通过读长之间的重叠关系将读长拼接成序列标签(Tags);在给定的相似度下将序列标签聚成可操作分类单元(Operational taxonomic units, OTU),然后通过OTU与数据库比对,对OTU进行物种注释;基于OTU和物种注释结果进行样品物种组成和组间物种差异分析。
为了获得高质量的净数据,对原始数据进行过滤处理,以消除质量低、接头污染、含N以及复杂度低的读长。使用软件FLASH(v1.2.11)[22]通过序列之间的重叠关系将序列拼接成原始标签数据(Raw Tags),拼接得到的原始标签经过严格的过滤处理得到高质量的标签数据。为剔除重复序列获取物种的OTU,利用软件USEARCH(v7.0.1090)[23]对有效序列进行聚类(相似性≥97%),选取丰度最高的序列作为OTU,OTUs序列通过之前实验室建立的珠江口鱼类12S序列库及NCBI数据库的BLASTn功能进行比对和物种注释。若序列全长与NT和NCBI数据库的序列相似度≥96%,则该序列被指定为一个物种;若同一属内仅有一个物种的序列可供比较(且相似率<96%),并且该物种的分布与珠江口的公开记录相匹配,则也认定该序列为一个物种[24],若一条序列可以匹配多个物种(匹配率≥99%),并且物种属于同一属,则分类学分辨率下降到属水平[20]。若序列不符合以上原则或未注释到物种,则舍去该序列。
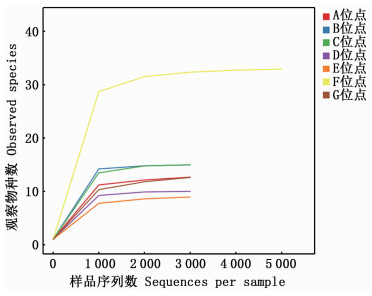
2 结果 2.1 OTU划分与分类地位鉴定本次下机数据经筛选、过滤和拼接处理后,7个采样点共获得有效拼接序列26 777条,有效拼接序列数目最少的是E点,共3 227条,最多的站位是F点,共5 287条,平均序列数为(3 883±721)条。按照97%相似性聚类后得到35个OTU代表序列。图 2为本次测序所得的鱼类物种稀疏曲线(Rarefaction curve),由图可见曲线逐渐趋于平缓,则认为测序深度已经基本覆盖到样品中所有的物种,测序量足以反映样本多样性,可以用于后续数据分析。
|
图 2 珠江口伶仃洋鱼类物种稀释曲线 Fig. 2 Rarefaction curve of the observed fish species in Lingding bay of the Pearl river estuary |
OTU序列经比对,共检测到35种鱼类,隶属于9目20科29属,有5个物种仅鉴定到属水平,分别为叫姑鱼属(Johnius)、

|
表 1 基于环境DNA宏条形码的珠江河口7个采样点检测到的鱼类物种 Tab.1 List of fish species detected by eDNA metabarcoding at 7 sampling sites in the Pearl River estuary |
|
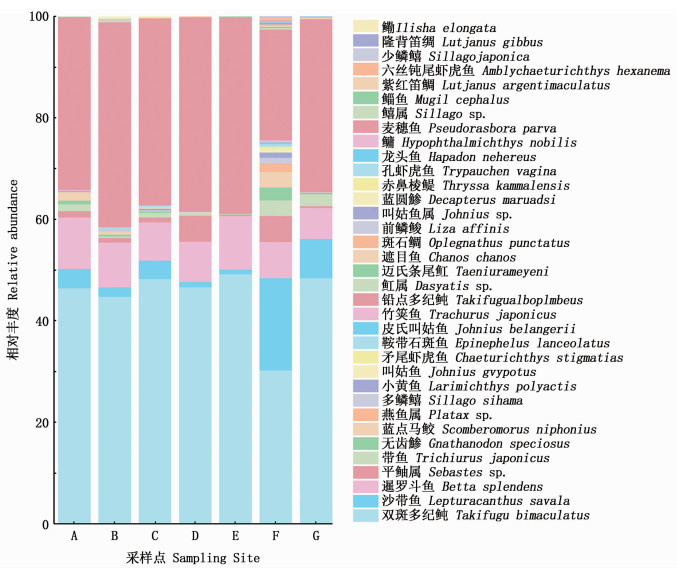
图 3 各采样点的鱼类物种组成 Fig. 3 Composition of fish species at each sampling site |
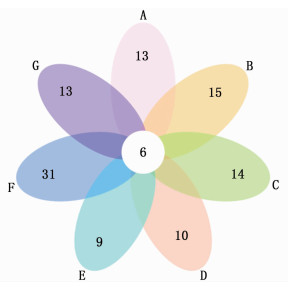
不同采样点间检测到的鱼类种类数和共有种类数如图 4所示。所有采样点共同检测到的鱼类共有6种,分别是双斑多纪鲀(Takifugu bimaculatus)、沙带鱼(Lepturacanthus savala)、暹罗斗鱼(Betta splendens)、小黄鱼(Larimichthys polyactis)、铅点多纪鲀(Takifugu alboplumbeus)和平鲉属(Sebastes sp.)鱼类。7个采样点中,采样点E检测到的鱼类种类数最少(9种),F点检测到的种类数最多(31种)。其余采样点分别检测到的鱼类种类数为10~15种。有9种鱼类仅在F点被检测到,分别是孔虾虎鱼(Trypauchen vagina)、六丝钝尾虾虎鱼(Amblychaeturichthys hexanema)、少鳞
|
图 4 不同采样点间的物种数量差异 Fig. 4 Variations of the number of species in different sampling sites |
|
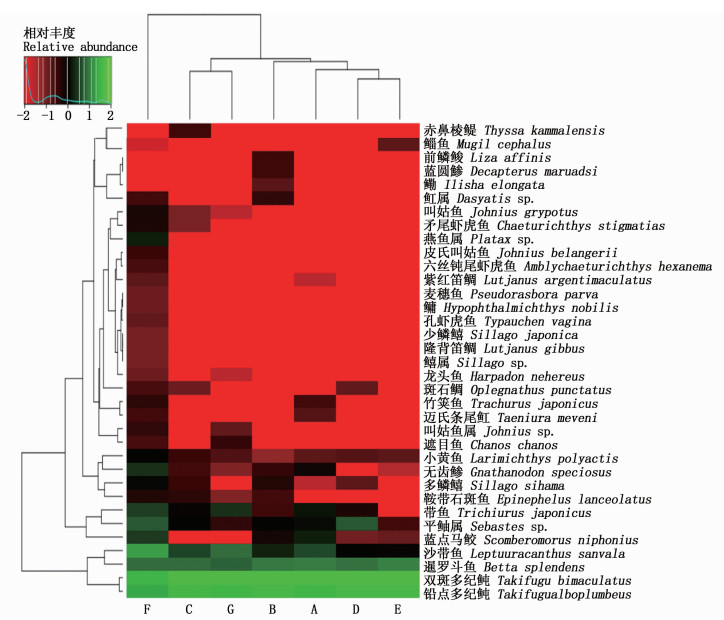
图 5 采样点间的物种分布热图 Fig. 5 The distribution heatmap of species between sampling sites |
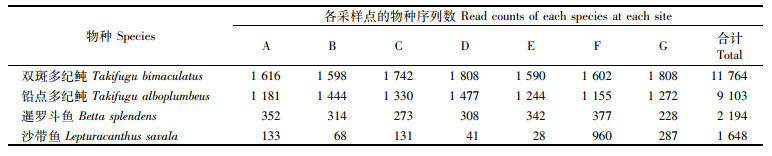
7个采样点共获得有效序列26 777条。其中: 鲀形目鱼类的序列数量最多,共20 867条,占比77.9%;其次是鲈形目,共5 177条,占比19.3%;其余目的鱼类仅占2.6%。在35种鱼类中,序列数排名前4位的物种为双斑多纪鲀、铅点多纪鲀、暹罗斗鱼和沙带鱼,序列数占比分别为43.9%、34%、8%和6%(表 2),采样点间物种分布热图(图 5)也显示,这4种鱼类的序列在各个采样点的丰度都很高。
|
表 2 不同采样点4种优势物种的序列数 Tab.2 The number of sequences of 4 dominant species at different sampling sites |
ZOU等[20]在珠江口南沙湿地附近水域6个采样点中共检测到24科57种鱼类,CHEANG等[21]在香港西部海域5个采样点中共检测到了17科22种鱼类。本研究在与上述2个研究区域均不重叠的珠江口伶仃洋海域7个采样点共检测到20科35种鱼类。对比3个研究区域检测到的鱼类种类(图 6)发现,南沙湿地附近水域的研究中有14种鱼类在本研究中也被检测到,分别是鞍带石斑鱼(Epinephelus lanceolatus)、双斑多纪鲀、铅点多纪鲀、沙带鱼、蓝圆鲹、六丝钝尾虾虎鱼、少鳞
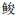
|
A为本研究海域;B为珠江口南沙湿地;C为香港西部海域。 A indicates this study; B indicates Nansha District Coast of Pearl River estuary; C indicates Hong Kong's waters. 图 6 珠江口不同研究区域的鱼类物种数量比较 Fig. 6 The comparison of fish species numbers in different study areas of Pearl River estuary |
本研究基于环境DNA宏条形码技术在珠江口伶仃洋7个采样点共检测到35种鱼类,绝大部分种类都是传统调查中出现过的种类,或是区域指南、文献以及数据库中已有记载的种类[25-26]。在检测到的鱼类中,鲈形目鱼类在物种数量上占绝对优势,但总的序列数仅占总序列数的19.3%。鲀形目鱼类仅有2种,但鲀形目鱼类的序列数占总序列数的77.9%。一方面,物种序列的相对丰度在一定程度上受物种数量的影响,序列相对丰度体现了物种在环境中可能具有的丰度[7];另一方面,检测结果中的序列相对丰度受多种因素的影响,如模板DNA的来源、检测物种的分布、生活史和生态位、引物的扩增偏向性等[27-28]。因此推测, 出现上述结果的原因可能与采样时这些物种在采样点的种群大小、释放的DNA总量和速度或所处生态位有关。
本研究从海水样本中检测出3种淡水鱼类:鳙、麦穗鱼和暹罗斗鱼。原因可能是带有这些鱼类DNA的介质通过水流或者其他途径的传播进入采样区域而被检测到。相关研究[29]发现鲤的DNA会通过多种环境介质传播,如:人类排放的生活污水、鸟类误食鱼体产生的粪便、丢弃入河的鲤残尸以及船舶运输的间接污染。由于鳙和麦穗鱼是常见的经济食用鱼类[30],渔民在船上食用后将残渣倒入海里或者食用后排泄到海水里,因此可能产生污染。也可能由珠江的淡水将这些鱼的组织或死亡个体带入到海洋中,因而被检测出来。暹罗斗鱼是一种观赏鱼,有商贩会在海边、海岛上进行售卖,死亡个体和暂养水体可能得以进入海水中。另外,周边水族市场的排污也可能将带有其DNA的水体带入采样区域,从而被检测出来。
此次在A和F两个采样点检测到1种易危物种——迈氏条尾
在进行物种注释时,有5种鱼类仅能鉴定到属水平,包括:(1)鉴定到叫姑鱼属的物种序列与卡氏叫姑鱼最为匹配,但匹配度仅有87%而未能定种。(2)鉴定到
在检测到的鱼类种类的数量和组成上,本研究与珠江口其他2个区域的研究有较大的差异。在物种数量上,本研究检测到的鱼类种类(35种)低于南沙湿地附近水域(57种),高于香港西部海域(22种)。在种类组成上,本研究与南沙湿地周围水域检测到的共同种类较多,而与更为邻近的香港西部海域的共同种类则很少。这与研究区域、样品采集策略和采用的条形码标记有很大的关系。南沙湿地周围水域为咸淡水交汇处,同时是淡水鱼类和海水鱼类的栖息地,鱼类种类可能本身就比海水海域要丰富;同时,在样品采集上,本研究仅在雨季采集了水体样本,而南沙湿地周围水域的研究则在旱季和雨季均采集了水样,其研究结果表明两个时间段的物种组成有明显的不同,旱季的鱼类多样性大于雨季[20]。因此也可能是检测到的鱼类种类数要多于本研究的原因。本研究的鱼类种类组成与地理距离较远、生境差别较大的南沙湿地周围水域更为接近,而与地理距离上邻近的香港西部海域差别明显,原因很可能是本研究和南沙湿地周围水域的研究采用了相同的线粒体12S序列作为条形码标记,而香港西部海域的研究采用的标记是线粒体COI序列。不同引物对模板的匹配程度和扩增效率等都可以影响目标序列的产出,从而影响种类组成[35-37]。这可能也是香港西部海域检测到的种类数偏少的原因。综合以上分析得知,在利用环境DNA宏条形码研究鱼类多样性时,选取扩增的序列种类多的条形码标记引物、增加采样的时间跨度可以提高检出率,从而更真实地反应研究区域的多样性指标。
4 结论本研究使用环境DNA宏条形码技术对珠江河口伶仃洋的鱼类组成进行了初步探究,在7个采样点共检测出9目20科29属35种鱼类,证实了环境DNA宏条形码技术在珠江河口鱼类种类组成和分布调查研究应用上的可行性,并通过与其他两项研究比较了解了珠江河口不同区域的鱼类种类组成差异。虽然环境DNA宏条形码技术具有对数据库的依赖性、无法评估鱼类的个体状况、无法进行生物学测量等缺点。不能完全替代传统的鱼类调查方法,但该技术具有对生物体以及生态环境无害、操作简单、高灵敏度以及高检测率等诸多优势。随着数据库的持续完善,该技术的适用性会越来越强。环境DNA宏条形码技术可以作为一种对传统调查方法的补充,用于河口鱼类多样性的调查研究,为资源调查评估的开展提供有力的技术支持。
| [1] |
郑亮, 吕振波, 李凡, 等. 黄河口、长江口和珠江口水域鱼类分类学多样性的对比研究[J]. 大连海洋大学学报, 2014, 29(5): 530-535. ZHENG L, LYU Z B, LI F, et al. Comparison of ontaxonomic diversity of fish community among the Yellow River estuary, Yangtze River estuary, Pearl River estuary and their adjacent waters[J]. Journal of Dalian Ocean University, 2014, 29(5): 530-535. DOI:10.3969/J.ISSN.2095-1388.2014.05.020 |
| [2] |
CAHILL A E, PEARMAN J K, BORJA A, et al. A comparative analysis of metabarcoding and morphology-based identification of benthic communities across different regional seas[J]. Ecology and Evolution, 2018, 8(17): 8908-8920. DOI:10.1002/ece3.4283 |
| [3] |
BONAR S A, MERCADO-SILVA N, HUBERT W A, et al. Standard methods for sampling freshwater fishes: opportunities for international collaboration[J]. Fisheries, 2017, 42(3): 150-156. DOI:10.1080/03632415.2017.1276352 |
| [4] |
BAYLEY P B, PETERSON J T. An approach to estimate probability of presence and richness of fish species[J]. Transactions of the American Fisheries Society, 2001, 130(4): 620-633. DOI:10.1577/1548-8659(2001)130<0620:AATEPO>2.0.CO;2 |
| [5] |
OLDS B P, JERDE C L, RENSHAW M A, et al. Estimating species richness using environmental DNA[J]. Ecology and Evolution, 2016, 6(12): 4214-4226. DOI:10.1002/ece3.2186 |
| [6] |
YAMAMOTO S, MASUDA R, SATO Y, et al. Environmental DNA metabarcoding reveals local fish communities in a species-rich coastal sea[J]. Scientific Reports, 2017, 7: 40368. DOI:10.1038/srep40368 |
| [7] |
USHIO M, MURAKAMI H, MASUDA R, et al. Quantitative monitoring of multispecies fish environmental DNA using high-throughput sequencing[J]. Metabarcoding and Metagenomics, 2018, 2: e23297. |
| [8] |
BLABOLIL P, HARPER L R, ŘÍČANOVÁ Š, et al. Environmental DNA metabarcoding uncovers environmental correlates of fish communities in spatially heterogeneous freshwater habitats[J]. Ecological Indicators, 2021, 126: 107698. DOI:10.1016/j.ecolind.2021.107698 |
| [9] |
SINGER G A C, FAHNER N A, BARNES J G, et al. Comprehensive biodiversity analysis via ultra-deep patterned flow cell technology: a case study of eDNA metabarcoding seawater[J]. Scientific Reports, 2019, 9(1): 5991. DOI:10.1038/s41598-019-42455-9 |
| [10] |
KELLY R P, CLOSEK C J, O'DONNELL J L, et al. Genetic and manual survey methods yield different and complementary views of an ecosystem[J]. Frontiers in Marine Science, 2017, 3: 283. |
| [11] |
BAKKER J, WANGENSTEEN O S, CHAPMAN D D, et al. Environmental DNA reveals tropical shark diversity in contrasting levels of anthropogenic impact[J]. Scientific Reports, 2017, 7(1): 16886. DOI:10.1038/s41598-017-17150-2 |
| [12] |
MINAMOTO T, YAMANAKA H, TAKAHARA T, et al. Surveillance of fish species composition using environmental DNA[J]. Limnology, 2012, 13(2): 193-197. DOI:10.1007/s10201-011-0362-4 |
| [13] |
THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Detection of a diverse marine fish fauna using environmental DNA from seawater samples[J]. PLoS One, 2012, 7(8): e41732. DOI:10.1371/journal.pone.0041732 |
| [14] |
THOMSEN P F, KIELGAST J, IVERSEN L L, et al. Monitoring endangered freshwater biodiversity using environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2565-2573. DOI:10.1111/j.1365-294X.2011.05418.x |
| [15] |
MIYA M, SATO Y, FUKUNAGA T, et al. Mifish, a set of universal PCR primers for metabarcoding environmental DNA from fishes: detection of more than 230 subtropical marine species[J]. Royal Society Open Science, 2015, 2(7): 150088. DOI:10.1098/rsos.150088 |
| [16] |
GOLD Z, CURD E E, GOODWIN K D, et al. Improving metabarcoding taxonomic assignment: A case study of fishes in a large marine ecosystem[J]. Molecular Ecology Resources, 2021, 21(7): 2546-2564. DOI:10.1111/1755-0998.13450 |
| [17] |
CIVADE R, DEJEAN T, VALENTINI A, et al. Spatial representativeness of environmental DNA metabarcoding signal for fish biodiversity assessment in a natural freshwater system[J]. PLoS One, 2016, 11(6): e0157366. DOI:10.1371/journal.pone.0157366 |
| [18] |
HÄNFLING B, HANDLEY L L, READ D S, et al. Environmental DNA metabarcoding of lake fish communities reflects long-term data from established survey methods[J]. Molecular Ecology, 2016, 25(13): 3101-3119. DOI:10.1111/mec.13660 |
| [19] |
SIGSGAARD E E, NIELSEN I B, CARL H, et al. Seawater environmental DNA reflects seasonality of a coastal fish community[J]. Marine Biology, 2017, 164(6): 128. DOI:10.1007/s00227-017-3147-4 |
| [20] |
ZOU K S, CHEN J W, RUAN H T, et al. eDNA metabarcoding as a promising conservation tool for monitoring fish diversity in a coastal wetland of the Pearl River Estuary compared to bottom trawling[J]. Science of the Total Environment, 2020, 702: 134704. DOI:10.1016/j.scitotenv.2019.134704 |
| [21] |
CHEANG C C, LEE B Y, IP B H Y, et al. Fish and crustacean biodiversity in an outer maritime estuary of the Pearl River Delta revealed by environmental DNA[J]. Marine Pollution Bulletin, 2020, 161: 111707. DOI:10.1016/j.marpolbul.2020.111707 |
| [22] |
MAGO Ŏ T, SALZBERG S L. FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [23] |
EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [24] |
李桂峰, 庄平. 珠江口鱼类多样性与资源保护[M]. 北京: 中国农业出版社, 2018. LI G F, ZHUANG P. Conservations of fish diversity and resource in the Pearl River estuary[M]. Beijing: China Agriculture Press, 2018. |
| [25] |
何国民. 珠江口水域水生生物生态研究文集[M]. 北京: 科学出版社, 2004. HE G M. A collection of ecological studies on aquatic organisms in the Pearl River estuary[M]. Beijing: Science Press, 2004. |
| [26] |
詹海刚. 珠江口及邻近水域鱼类群落结构研究[J]. 海洋学报, 1998, 20(3): 91-97. ZHAN H G. Study on fish community structure in the Zhujiang estuary and adjacent waters[J]. Acta Oceanologica Sinica, 1998, 20(3): 91-97. DOI:10.3321/j.issn:0253-4193.1998.03.013 |
| [27] |
李晗溪, 黄雪娜, 李世国, 等. 基于环境DNA-宏条形码技术的水生生态系统入侵生物的早期监测与预警[J]. 生物多样性, 2019, 27(5): 491-504. LI H X, HUANG X N, LI S G, et al. Environmental DNA (eDNA)-metabarcoding-based early monitoring and warning for invasive species in aquatic ecosystems[J]. Biodiversity Science, 2019, 27(5): 491-504. |
| [28] |
徐念, 熊美华, 邵科, 等. 长江中下游环境DNA宏条形码生物多样性检测技术初步研究[J]. 环境科学研究, 2020, 33(5): 1187-1196. XU N, XIONG M H, SHAO K, et al. Preliminary study on environmental DNA metabarcoding for detecting biodiversity in the middle and lower reaches of the Yangtze River[J]. Research of Environmental Sciences, 2020, 33(5): 1187-1196. |
| [29] |
LODGE D M, TURNER C R, JERDE C L, et al. Conservation in a cup of water: estimating biodiversity and population abundance from environmental DNA[J]. Molecular Ecology, 2012, 21(11): 2555-2558. DOI:10.1111/j.1365-294X.2012.05600.x |
| [30] |
帅方敏, 李新辉, 李跃飞, 等. 珠江东塔产卵场鳙繁殖的生态水文需求[J]. 生态学报, 2016, 36(19): 6071-6078. SHUAI F M, LI X H, LI Y F, et al. Analysis of the ecohydrological demands of bighead carp breeding in the Dongta spawning grounds of the Pearl River, via cross-wavelet analysis[J]. Acta Ecologica Sinica, 2016, 36(9): 6071-6078. |
| [31] |
KYNE P M, WHITE W T. Taeniurops meyeni. The IUCN red list of threatened species 2015: e. T60162A68646736[EB/OL]. https://www.iucnredlist.org/species/60162/68646736.
|
| [32] |
SCARPONI P, CORO G, PAGANO P. A collection of Aquamaps native layers in NetCDF format[J]. Data in Brief, 2018, 17: 292-296. DOI:10.1016/j.dib.2018.01.026 |
| [33] |
O'DONNELL J L, KELLY R P, SHELTON A O, et al. Spatial distribution of environmental DNA in a nearshore marine habitat[J]. PeerJ, 2017, 5: e3044. DOI:10.7717/peerj.3044 |
| [34] |
PORT J A, O'DONNELL J L, ROMERO-MARACCINI O C, et al. Assessing vertebrate biodiversity in a kelp forest ecosystem using environmental DNA[J]. Molecular Ecology, 2016, 25(2): 527-541. DOI:10.1111/mec.13481 |
| [35] |
MIYA M, GOTOH R O, SADO T. MiFish metabarcoding: a high-throughput approach for simultaneous detection of multiple fish species from environmental DNA and other samples[J]. Fisheries Science, 2020, 86(6): 939-970. |
| [36] |
COLLINS R A, BAKKER J, WANGENSTEEN O S, et al. Non-specific amplification compromises environmental DNA metabarcoding with COI[J]. Methods in Ecology and Evolution, 2019, 10(11): 1985-2001. DOI:10.1111/2041-210X.13276 |
| [37] |
DEINER K, BIK H M, MÄCHLER E, et al. Environmental DNA metabarcoding: transforming how we survey animal and plant communities[J]. Molecular Ecology, 2017, 26(21): 5872-5895. |
2. College of Marine Sciences, Shanghai Ocean University, Shanghai 201306, China;
3. College of Marine Science, South China Agricultural University, Guangzhou 510642, Guangdong, China;
4. College of Fisheries and Life Science, Dalian Ocean University, Dalian 116023, Laoning, China;
5. Southern Marine Science and Engineering Guangdong Laboratory, Guangzhou 511458, Guangdong, China
 2022,
Vol. 31
2022,
Vol. 31


