2. 上海海洋大学 农业农村部鱼类营养与环境生态研究中心, 上海 201306;
3. 上海海洋大学 上海市水产养殖工程技术研究中心, 上海 201306
青田田鱼(Cyprinus carpio var. qingtianensis),又名瓯江彩鲤,隶属于鲤科(Cyprinidae)鲤属(Cyprinus),是中国浙江省青田县和永嘉县一带特有的一种稻田养殖鲤鱼品种[1]。稻田生境和传统池塘养殖环境不同,其水域农作物和土壤耗氧量大[2]、水位低,致使稻田生境溶氧低且昼夜变化大[3],同时存在干涸的风险并且无法进行人工辅助增氧。稻田生境一天中存在长时间低氧期(DO < 4 mg/L,6~10 h),而当水体溶氧量维持在4 mg/L以上时,鱼类才能正常生长发育[4]。溶氧(Dissolved oxygen,DO)是水体中最重要的环境因子,对鱼类的生长、繁育、代谢等生理活动都有重要影响[5]。传统水产养殖过程中,水体缺氧问题一直难以处理,除了依靠人工增氧外,更多地依赖鱼类自身的适应性调节[6]。在急性低氧环境中,鱼类更易产生氧化应激,尤其是在低氧之后的复氧恢复过程中,氧化应激通常会表现得更剧烈[7]。脑作为对氧敏感型器官,缺氧对鱼类脑组织的影响不容忽视。鱼类暴露于轻度低氧环境可以诱导大脑中各种神经元细胞的抗氧化反应来促进机体细胞保护机制,使其免受氧化应激,例如通过增加星形胶质细胞中的PI3K-Akt信号和促红细胞生成素表达、增加MAPK信号和大脑皮层神经元中的衍生神经营养因子表达[8]。同时低氧环境会增加大脑中活性氧(ROS)的生成,导致氧化还原失衡;而ROS水平的整体提升也会促进神经元凋亡,并最终形成脑损伤[9-10]。此外糖原作为脊椎动物最重要的储能物质,对维持机体的能量代谢以及其他功能物质的合成有重要作用。在一般认知中,脑糖原的含量远不如肌糖原和肝糖原,因而常常被忽视[11],但是脑糖原含量却是脑葡萄糖的好几倍[12]。
目前关于青田田鱼如何适应稻田生境的研究,仍未得到系统的阐释。本研究通过模拟稻田溶氧变化环境,对青田田鱼进行急性低氧胁迫(6 h)后的复氧恢复(6 h)实验处理,为青田田鱼对稻田溶氧环境的适应性提供理论依据,也为其他鱼类在急性溶氧变化过程中的生理调节机制提供参考。
1 材料与方法 1.1 实验用鱼与暂养管理实验用鱼购自浙江省青田县愚公生态农业有限公司同一批次繁育的青田田鱼幼鱼,选取活力好,健康无损伤,体长为(16.93±1.61)cm,体质量为(80.67±4.67)g的幼鱼运送至上海海洋大学生态与渔业实验室进行2周暂养,正式实验前1周随机选取27尾平均放入3个容量为150 L(实验水体为100 L)的实验桶中。
1.2 实验设计在25 ℃条件下进行预试验,测定青田田鱼幼鱼(50~70 g)低氧耐受参数:浮头时溶氧为0.31 mg/L,低氧翻转点的溶氧为0.26 mg/L,窒息点的溶氧为0.17 mg/L。根据预试验结果并结合实际监测到的稻田溶氧变化情况,设定溶氧为0.5 mg/L作为低氧胁迫阈值, 低氧胁迫和复氧恢复时长定为6 h。
为减少其他应激干扰,正式实验直接在3个实验桶中进行。正式低氧胁迫实验前,完成常氧组的取样,此时的溶氧为(7.07±0.39)mg/L,水温为(25.37±0.76)℃,样品编号记为CB。随后往水中注入N2,待水中溶氧下降到0.5 mg/L时,调节氮气和空气的注入流速来控制水中的溶氧,待低氧胁迫6 h时进行低氧胁迫6 h组实验样品取样,胁迫期间水中的溶氧为(0.57±0.16)mg/L,水温为(25.65±0.76)℃,样品编号记为HB。待低氧胁迫实验取样结束后,停止氮气注入并增大空气注入流速,待水中溶解氧含量升至7 mg/L时,调节空气注入量维持水中的溶氧含量,待复氧恢复6 h后进行样品取样,恢复期间水中的溶氧为(7.21±0.53)mg/L,水温为(25.78±0.58)℃,样品编号记为RB。整个实验期间,每10 min监测1次水中的溶氧和水温。
1.3 样品采集每个实验桶每次取3尾幼鱼,共9尾。将其置于质量浓度为300 mg/L的MS-222溶液中快速麻醉,之后将幼鱼搁置在冰板上进行解剖,取下全脑并用滤纸吸干表面血液后,每个样品单独置于2 mL的RNA-free管中,经过液氮预冷后放置在-80 ℃冰箱保存以备后续实验使用。
1.4 总RNA提取、文库构建和转录组测序从-80 ℃冰箱中取出样品,在提前预冷的研钵中加入液氮研磨成粉末,使用Trizol法提取总RNA。运用琼脂糖凝胶电泳(1%琼脂糖)和NanoDrop 2000对提取的总RNA进行完整性、浓度和纯度等方面的质量检测。根据LIU等[13]将组织样品进行混合的方法来消除个体间差异,本实验将各组3尾鱼的全脑组织样品进行等量混合,每组取其中的1 μg RNA用于cDNA文库构建及Illumina测序。本次研究中的文库构建和Illumina测序均由美吉生物科技有限公司完成,建库试剂为TruseqTM RNA sample prep Kit(Illumina,CA,USA);测序系统为Illumina Novaseq 6000,测序模式为2 × 150 bp。
1.5 筛选差异表达基因和富集分析使用RSEM1.3.1软件,采用TPM(Transcripts Per Million)定量标准,先对基因长度进行均一化再对测序深度均一化,统一不同样本的总表达量获得read counts分析样本之间的基因差异表达情况。使用DEGseq 2 v. 1.24.0软件[14]进行read counts的标准化处理并筛选差异表达基因(Differentially expressed genes,DEGs),以P < 0.05与| log2(FoldChange) |≥1作为差异表达的阈值。利用GO(Gene ontology)[15]、KEGG(Kyoto encyclopedia of genes and genomes)[16]等数据库对差异表达基因进行功能注释。使用Goatools v. 0.6.5软件[17]和KOBAS v. 2.1软件[18]运用Fisher检验分别对DEGs进行GO功能富集分析和KEGG Pathway代谢通路进行富集分析。当P < 0.05,认为差异表达基因在GO功能和KEGG Pathway代谢通路存在显著富集。
2 结果与分析 2.1 转录组测序结果检测9个样本质控序列的碱基分布和测序质量,GC含量为45.57%~46.47%,Q20(测序精确率99%)为98.48%~98.63%,Q30(测序准确率99.9%)为95.34%~95.70%,错配率为0.023%~0.024%,可以进行后续的分析与研究。比对结果统计如表 1所示。本实验转录组测序结果已上传至NCBI(National center for biotechnology information),注册号为SRP251334。
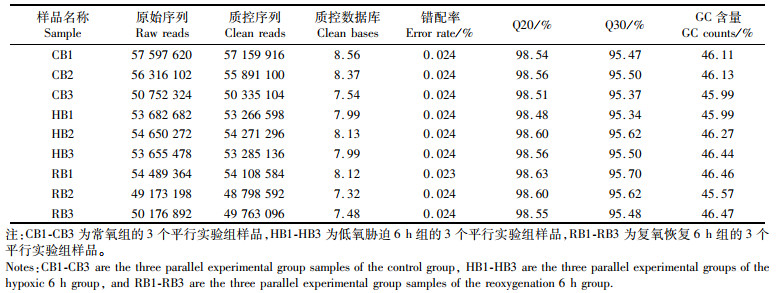
|
表 1 青田田鱼脑组织RNA-seq文库原始序列和质控序列的统计 Tab.1 Statistical of the raw and clean sequencing of the RNA-seq library of brain samples in Cyprinus carpio var. qingtianensis |
对样本数据进行主成分分析,发现从横坐标PC1看,样本的按照处理条件排布,CB组聚在一起,HB组聚在一起,RB组聚在一起,说明本次实验数据可靠,样本选择合理,可进行后续分析(图 1)。

|
图 1 主成分分析 Fig. 1 Principal component analysis of RNA-seq of 9 samples DEGs |
对HB vs CB组、RB vs HB组和RB vs CB 3个比较组进行DEGs的筛选。HB vs CB组筛选出1 094个DEGs,其中,上调表达730个,下调表达364个;RB vs HB组筛选出2 288个DEGs,其中,上调表达803个,下调表达1 485个;RB vs CB筛选出201个DEGs,其中,上调表达80个,下调表达121个(图 2)。
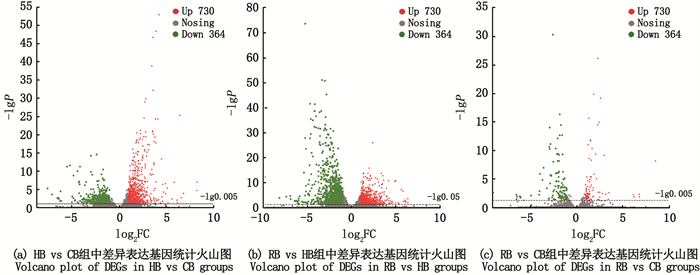
|
图 2 各比较组中差异表达基因统计图 Fig. 2 Statistics figure of DEGs among each comparison group |
为确定青田田鱼脑组织在适应急性溶氧变化过程中差异表达基因所涉及的生理调控,对DEGs进行GO功能注释分析,发现DEGs分布于生物学(BP)、细胞组分(CC)和分子功能(MF),其中富集到生物学功能的DEGs数目最多。在HB vs CB组中主要以糖酵解过程(Glycolytic process,GO: 0006096)、ATP再生(ATP generation from ADP,GO: 0006757)、氧化还原过程(Oxidation-reduction process,GO: 0055114)、免疫过程(Immune system process, GO: 0002376)和应激反应(Response to stimulus, GO: 0050896)等功能为主;在RB vs HB组中主要以免疫反应过程(Immune system process,GO: 0002376)、ADP代谢过程(ADP metabolic process,GO: 0046031)、生理调控(Circadian regulation of gene expression,GO: 0032922)、蛋白质羟化(Protein hydroxylation,GO: 0018126)和碳水化合物合成过程(Carbohydrate biosynthetic process,GO: 0016051)等功能为主;在RB vs CB组中主要以生理节律(Circadian rhythm,GO: 0007623)、单糖代谢过程(Circadian regulation of gene expression,GO: 0005996)和DNA结合转录因子活性(DNA-binding transcription factor activity,GO: 0003700)等功能为主。在低氧胁迫与复氧恢复过程中,能量代谢、应激反应等调控过程中差异基因富集较为显著,这表明青田田鱼脑组织产生了大量参与调控代谢等相关的活动过程(图 3)。

|
圆圈代表一个GO二级条目,圆圈颜色代表P值,颜色越红代表显著富集性越可靠,圆圈越大代表富集的基因数目越多。 Ordinate represents the name of the GO term, the color of the circle represents P-adjust. The deeper the red color, the more reliable the significant enrichment, and the larger the circle, the greater the number of enriched genes. 图 3 各比较组中差异表达基因GO富集分析(富集排名前20) Fig. 3 GO function enrichment analysis of DEGs in each comparison group(top 20) |
为了进一步探究在持续性高温胁迫下差异表达基因的生物学功能,对DEGs进行KEGG pathway富集分析,发现HB vs CB组中的DEGs主要富集在新陈代谢与环境信息处理过程,其中HIF-1信号通路(HIF-1 signaling pathway,ko04066,44个)富集DEGs数量较多,糖酵解/糖异生(Glycolysis / Gluconeogenesis,ko00010,21个),果糖和甘露糖代谢途径(Fructose and mannose metabolism,ko00051,16个),NOD-like受体信号通路(NOD-like receptor signaling pathway,ko04621,22个)及造血细胞调控(Hematopoietic cell lineage,ko04640,11个)等通路也有不同程度富集;在RB vs HB组中,DEGs主要富集在新陈代谢和机体系统,包括mTOR信号通路(mTOR signaling pathway,ko04150),VEGF信号通路(VEGF signaling pathway,ko04370), 果糖和甘露糖代谢途径(Fructose and mannose metabolism,ko00051)及蛋白质消化吸收通路(Protein digestion and absorption,ko04974)等;在RB vs CB组中,仅富集了5个通路,且仅生理节律(Circadian rhythm,ko04710)差异性显著,其余通路虽有少数DEGs富集,但无显著性差异(图 4)。

|
图 4 各比较组中差异表达基因KEGG pathway富集分析(富集排名前20) Fig. 4 KEGG pathway function enrichment analysis of DEGs in each comparison groups (top 20) |
为验证RNA-seq的准确性,随机选取了hif-1a (hypoxia inducible factor-1,低氧诱导因子)、hk(hexokinase,己糖激酶)、pdk3 (pyruvate dehydrogenase kinase isozyme 3,丙酮酸脱氢酶激酶同工酶3)、pfka(6-phosphofructokinase,6磷酸果糖激酶)、ldh(lactate dehydrogenase,乳酸脱氢酶)和vegf-a(vascular endothelial growth factor A,血管内皮生长因子)等6个DEGs(HB vs CB组:6个上调;RB vs HB组:6个下调)进行RT-qPCR定量验证。结果显示,在HB vs CB组中上调DEGs和RB vs HB组中下调DEGs的RT-qPCR定量和RNA-seq表达虽然存在表达量上的不同,但其上下调变化趋势一致,证明了RNA-seq的结果准确可靠,可以进行后续研究分析(图 5)。
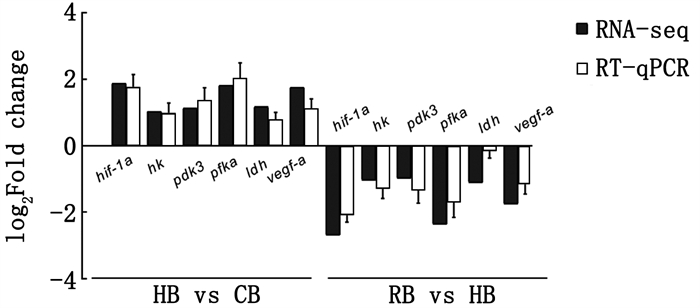
|
图 5 RT-qPCR验证RNA-seq结果图 Fig. 5 Validation of RNA-seq results by RT-qPCR |
在青田田鱼(瓯江彩鲤)转录组的研究中:陈晓雯等[19]探究不同转录组拼接方法对瓯江彩鲤的适用性,结果表明基于序列比对优先策略注释77 601个转录本,基于从头拼接共69 406个;钱永生[20]在对4种不同体色瓯江彩鲤的脾脏被嗜水气单胞菌侵染实验中,共获得173 570条转录本。原始数据经过质控检测处理后,获得的质控后序列与鲤鱼基因组进行序列比对分析的总比对率为82.92%~83.5%,这是由于青田田鱼长期生活在山区稻田环境,加上自然和人工双重选择压力会造成基因遗传突变[21-22]。本研究根据转录本和基因外显子对应关系,共筛选出65 021个表达基因,再与鲤鱼基因组中已注释的69 494个表达基因进行比对,比对注释率达到93.68%,这能较好说明测序结果中筛选得到表达基因的功能。根据筛选的6个候选DEGs基因表达量,在HB vs CB组中显示各基因在RT-PCR和RNA-seq检测中皆呈上调趋势,且两个方法得出的表达量基本一致,除了ldh基因和vegf-a基因;在RB vs CB组中各基因的表达趋势皆为下调趋势,表达量基本一致除了ldh基因和vegf-a基因,这可能是因为基因在不同生理状态下会对自身的转录体表达量产生影响[23]。虽然个别基因的表达量有些许差异,但总体基因表达量及变化趋势一致,验证了RNA-seq准确性。
3.2 基因表达差异分析脑组织中上调表达DEGs数目(730)多于下调表达DEGs基因(364),表明脑组织通过上调更多的基因表达来应对急性低氧环境刺激。花斑裸鲤(Gymncypris eckloni)[24]、团头鲂(Megalobrama amblycephala)[25]、大黄鱼(Larimichthys crocea)[26]等在低氧胁迫中也有类似结果,不同点在于差异基因表达数量不同,这可能是由胁迫处理时间、溶氧质量浓度及靶器官的敏感性的差异所致。经过6 h复氧恢复后(RB vs HB),上调表达的DEGs数量(803)明显少于下调表达的DEGs数量(1 485),表明脑组织通过下调更多的基因表达来缓解复氧对机体造成的应激响应。对复氧恢复6 h组和常氧组进行比较(RB vs CB), 上调表达的DEGs数量(80)略少于下调表达的DEGs数量(121),而两组之间的DEGs数量(201)远少于整个测序得到的DEGs数量(65 021),这说明经过6 h的复氧恢复,脑组织的基因表达水平已基本恢复到常氧组水平,这与沙尖鱼(Sillago sihama)鳃组织[27]低氧后再恢复比较的转录实验的结果类似,只是由于恢复时间和鱼类本身恢复能力的差异会存在一定的DEGs数目区别。
3.3 差异表达基因调控功能分析通过GO功能及KEGG Pathway富集分析得出DEGs大多集中于能量代谢和响应环境信号两大通路上,因此我们着重从这两个角度分析青田田鱼在应对急性溶氧环境变化的适应情况。
3.3.1 能量代谢鱼类在应对急性低氧环境时,通常会有提高血液循环、增强血氧亲和力、提高呼吸频率和增强能量代谢等相关策略进行适应性调整[28]。若上述调控措施无法满足自身的能量需求,就会开启无氧糖酵解通路,通过碳水化合物代谢提供能量[29-30]。本研究中在急性低氧胁迫过程(HB vs CB)中发现,ldh(乳酸脱氢酶)、hk(己糖激酶)和pfka(6-磷酸果糖激酶)等基因均上调表达,这与急性低氧胁迫尼罗罗非鱼(Oreochromis niloticus)会增强其能量代谢的研究[31]相似。hk和pfka基因作为调控己糖激酶和6-磷酸果糖激酶表达的主效基因,参与调控葡萄糖的糖酵解过程,其表达量的上调促进葡糖糖的磷酸化过程并生成大量的丙酮酸和NADH,加速脑组织的供能代谢,以应对低氧胁迫带来的氧化应激。在缺氧环境下丙酮酸会在乳酸脱氢酶的催化下生成乳酸,ldh基因表达量上调也印证了丙酮酸转化为乳酸的速率增强。pfk、hk和ldh基因的上调表达以增强糖酵解和乳酸代谢途径,而acly基因下调则抑制TCA循环,从而导致脑细胞有氧呼吸和无氧呼吸呈“此消彼长”的效应[32]。在低氧胁迫的研究中,鲤科鱼类糖酵解的主要终产物为乙醇[33],而催化乙醇合成的最后环节需要乙醇脱氢酶(Alcohol dehydrogenase,ADH)的参与,但本研究并未发现ADH基因的低氧诱导表达,此结果与低氧胁迫花斑裸鲤(Gymncypris eckloni)[24]重要组织(肝胰脏、脑、心脏、肌肉和血液组织)转录结果一致。在复氧恢复过程(RB vs HB)中, ldh、hk和pfk基因表达量均显著下调,表明青田田鱼在复氧恢复过程中其脑细胞中的糖酵解途径受到抑制。研究发现,在HB vs CB组和RB vs HB组中,磷酸戊糖途径只在低氧胁迫过程中显著被激活,一方面磷酸戊糖途径可以提供大量的还原力NADPH,可以减少氧化应激带来的损伤;另一方面,磷酸戊糖途径的中间产物存在五碳糖,可以为基因转录上调表达提供充足的碳骨架[34]。在低氧胁迫过程中,氨基酸和脂类代谢为糖异生途径提供底物以及保障供能代谢[35],同时脂质和氨基酸合成也受到抑制[36]。在复氧恢复过程中,丙酮酸和乳酸作为能量代谢主要原料生成的糖类既维持循环中的血糖水平[37],也是大脑发挥正常功能的必需品[38];其他糖类利用减少也增强脂质和氨基酸的合成[37]。
3.3.2 响应环境信号hif-1α (缺氧诱导因子1-α)是HIF-1家族重要成员之一,对维持调节机体内氧稳态具有重要作用[39]。hif-1α基因能在缺氧环境下高度表达,并以其为主体串联下游信号通路。具有血管生成功能的vegf-a基因在hif-1α基因诱导下启动VEGF转录,使表达量大幅增加。本次研究发现青田田鱼经过6 h急性低氧胁迫后(HB vs CB),hif-1α基因和vegf-a基因显著上调,而在复氧恢复6 h处理后,hif-1α基因和vegf-a基因表达量都显著下调,这表明hif-1α基因受到低氧环境刺激后通过其表达量上下调节来应对机体应激反应,而连带下游vegf-a基因受到hif-1α基因诱导使其表达趋势与hif-1α基因一致。当鱼类受到低氧胁迫时,低氧诱导因子会与细胞膜上特异性受体结合,从而引发细胞内信号传递,触发级联反应影响功能代谢[40]。如mTOR(哺乳动物雷帕霉素靶蛋白)通路得到增强,其在细胞生长和诱发细胞凋亡中发挥重要作用,SRINIVAS等[41]发现mTOR和AMPK(AMP激活的蛋白激酶)依赖hif-1α基因对细胞自噬起活化调节作用来调控细胞的成熟和分化来完成细胞内能量代谢的适应性调节。
综上所述,本研究为了解青田田鱼在应对急性溶氧变化过程中的生理调节机制,从转录组层面对常氧组(CB)、低氧胁迫6 h组(HB)及复氧恢复6 h组(RB)的青田田鱼脑组织进行研究分析。在低氧胁迫6 h后(HB vs CB),上调表达的DEGs数多余下调表达,说明青田田鱼在应对低氧胁迫过程中增强更多的代谢通道来应对氧化应激,如增强糖酵解和磷酸戊糖途径为机体提供能量和还原力;经过6 h复氧恢复(RB vs CB)后下调表达的DEGs更多,说明脑通过抑制更多的代谢过程来完成适应性恢复,如糖异生途径加强,恢复期间合成大量糖源补充低氧消耗的糖量。在响应信号方面hif-1α基因调控VEGF和mTOR通路来调节细胞的增殖和分化以达到适应细胞内的能量代谢。特别是经过复氧恢复(RB vs CB)后青田田鱼脑组织95%以上的基因表达量基本恢复到常氧组水平(CB),表明青田田鱼已基本适应稻田中急性溶氧变化的环境。
| [1] |
REN W Z, HU L L, GUO L, et al. Preservation of the genetic diversity of a local common carp in the agricultural heritage rice-fish system[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(3): E546-E554. |
| [2] |
FAGERIA N K, CARVALHO G D, SANTOS A B, et al. Chemistry of lowland rice soils and nutrient availability[J]. Communications in Soil Science & Plant Analysis, 2011, 42(16): 1913-1933. |
| [3] |
张云杰. 稻蟹共生对稻田水质影响的研究[D]. 上海: 上海海洋大学, 2013. ZHANG Y J. Study on the effect of water quality in rice-crab culture model[D]. Shanghai: Shanghai Ocean University, 2013. |
| [4] |
张国松. 瓦氏黄颡鱼(Pelteobagrus vachelli)应对低氧胁迫的分子机制研究[D]. 南京: 南京师范大学, 2017 ZHANG G S. Study on the molecular mechanism of Pelteobagrus vachelli in response to hypoxia stress[D]. Nanjing: Nanjing Agricultural University, 2017. |
| [5] |
MALL K, KOPF R K, WATTS R J, et al. Hypoxia, blackwater and fish kills: experimental lethal oxygen thresholds in juvenile predatory lowland river fishes[J]. PLoS One, 2014, 9(4): e94524. DOI:10.1371/journal.pone.0094524 |
| [6] |
张敬旺. 草鱼养殖池塘溶氧收支平衡及关键影响因子的研究[D]. 上海: 上海海洋大学, 2012. ZHANG J W. Study on the oxygen budgets of grass carp ponds and its critical impact factors[D]. Shanghai: Shanghai Ocean University, 2012. |
| [7] |
LI C Y, JACKSON R M. Reactive species mechanisms of cellular hypoxia-reoxygenation injury[J]. American Journal of Physiology-Cell Physiology, 2002, 282(2): C227-C241. DOI:10.1152/ajpcell.00112.2001 |
| [8] |
TERRANEO L, SAMAJA M. Comparative response of brain to chronic hypoxia and hyperoxia[J]. International Journal of Molecular Sciences, 2017, 18(9): 1914. DOI:10.3390/ijms18091914 |
| [9] |
TURRENS J F. Mitochondrial formation of reactive oxygen species[J]. Journal of Physiology-London Then Cambridge-, 2003. |
| [10] |
COIMBRA C D, ALVA N, DURAN M, et al. Oxidative stress and apoptosis after acute respiratory hypoxia and reoxygenation in rat brain[J]. Redox Biology, 2017, 12: 216-225. DOI:10.1016/j.redox.2017.02.014 |
| [11] |
张绘宇, 赵玉男, 王中立, 等. 脑糖原在脑能量代谢中的作用研究进展[J]. 中国药理学通报, 2013, 29(7): 908-913. ZHANG H Y, ZHAO Y N, WANG Z L, et al. The function research progress of cerebral glycogen in brain energy metabolism[J]. Chinese Pharmacological Bulletin, 2013, 29(7): 908-913. DOI:10.3969/j.issn.1001-1978.2013.07.006 |
| [12] |
BROWN A M. Brain glycogen re-awakened[J]. Journal of Neurochemistry, 2004, 89(3): 537-552. DOI:10.1111/j.1471-4159.2004.02421.x |
| [13] |
LIU S K, WANG X L, SUN F Y, et al. RNA-Seq reveals expression signatures of genes involved in oxygen transport, protein synthesis, folding, and degradation in response to heat stress in cat fish[J]. Physiological Genomics, 2013, 45(12): 462-476. DOI:10.1152/physiolgenomics.00026.2013 |
| [14] |
ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: a bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 |
| [15] |
RICHARDS J G. Physiological, behavioral and biochemical adaptations of intertidal fishes to hypoxia[J]. Journal of Experimental Biology, 2011, 214(2): 191-199. DOI:10.1242/jeb.047951 |
| [16] |
ZITTIER Z M C, HIRSE T, POERTNER H O. The synergistic effects of increasing temperature and CO2 levels on activity capacity and acid-base balance in the spider crab, Hyas araneus[J]. Marine Biology, 2013, 160(8): 2049-2062. DOI:10.1007/s00227-012-2073-8 |
| [17] |
KLOPFENSTEIN D V, ZHANG L, PEDERSEN B S, et al. GOATOOLS: A Python library for gene ontology analyses[J]. Scientific reports, 2018, 8(1): 10872. DOI:10.1038/s41598-018-28948-z |
| [18] |
MAO J D, WU P, XIA X H, et al. Correlation between expression of gastrin, somatostatin and cell apoptosis regulation gene bcl-2/bax in large intestine carcinoma[J]. World J Gastroenterol, 2005, 11(5): 721-725. DOI:10.3748/wjg.v11.i5.721 |
| [19] |
陈晓雯, 王军, 岳武成, 等. 两种拼接方法在瓯江彩鲤转录组研究中的适用性比较[J]. 上海海洋大学学报, 2017, 26(5): 666-673. CHEN X W, WANG J, YUE W C, et al. Comparison of two transcriptome assembly strategies on Oujiang color common carp(Cyprinus carpio var. color)[J]. Journal of Shanghai Ocean University, 2017, 26(5): 666-673. |
| [20] |
钱永生. 四种体色瓯江彩鲤的摄食和呼吸特性差异及其感病转录组学分析[D]. 上海: 上海海洋大学, 2019. QIAN Y S. Comparison of feeding and respiration characteristics and infected transcriptomics analysis among the four color patterns of Oujiang color common carp (Cyprinus carpio var. color)[D]. Shanghai: Shanghai Ocean University, 2019. |
| [21] |
胡文静, 苏超群, 刘韬, 等. 青田田鱼和金华田鱼肌肉营养成分的比较[J]. 上海海洋大学学报, 2020, 29(4): 552-558. HU W J, SU C Q, LIU T, et al. Analysis of muscle nutrient composition of Cyprinus carpio var. color in Qingtian and Jinhua[J]. Journal of Shanghai Ocean University, 2020, 29(4): 552-558. |
| [22] |
严瑞芬. 秀丽线虫缺氧相关基因可变剪接变体的研究[D]. 杨凌: 西北农林科技大学, 2011. YAN R F, et al. A study on alternative splicing variants of the genes associated with hypoxia in C. elegans[D]. Yangling: Northwest A&F University, 2011. |
| [23] |
肖武汉. 低氧信号传导途径与鱼类低氧适应[J]. 中国科学: 生命科学, 2014, 44(12): 1227-1235. XIAO W H. The hypoxia signaling pathway and hypoxic adaptation in fishes[J]. Science China(Life Sciences), 2014, 44(12): 1227-1235. |
| [24] |
赵永丽. 花斑裸鲤低氧胁迫转录组学及其主要差异基因表达调控研究[D]. 西宁: 青海大学, 2018. ZHAO Y L. Transcriptomics of Gymncypris eckloni under hypoxia and the expression regulation of major differentially genes[D]. Xining: Qinghai university, 2018. |
| [25] |
陈柏湘. 团头鲂(Megalobrama amblycephala)低氧相关分子标记的开发及应用[D]. 武汉: 华中农业大学, 2016. CHEN B X. Identification and application of hypoxia-associated molecular markers in Megalobrama amblycephala[D]. Wuhan: Huazhong Agricultural University, 2016. |
| [26] |
MU Y, LI W, WEI Z, et al. Transcriptome analysis reveals molecular strategies in gills and heart of large yellow croaker (Larimichthys crocea) under hypoxia stress[J]. Fish & Shellfish Immunology, 2020, 104: 304-313. DOI:10.3969/j.issn.1004-2490.2020.03.006 |
| [27] |
SAETAN W, TIAN C X, YU J W, et al. Comparative transcriptome analysis of gill tissue in response to hypoxia in silver Sillago (Sillago sihama)[J]. Animals, 2020, 10(4): 628. DOI:10.3390/ani10040628 |
| [28] |
COSSINS A R, CRAWFORD D L. Fish as models for environmental genomics[J]. Nature Reviews Genetics, 2005, 6(4): 324-333. DOI:10.1038/nrg1590 |
| [29] |
SMITH R W, HOULIHAN D F, NILSSON G E, et al. Tissue-specific changes in protein synthesis rates in vivo during anoxia in crucian carp[J]. American Journal of Physiology, 1996, 40(4): R897-R904. |
| [30] |
CAMERON J S, DEWITT J P, NGO T T, et al. Cardiac KATP channel alterations associated with acclimation to hypoxia in goldfish (Carassius auratus L.)[J]. Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology, 2013, 164(4): 554-564. |
| [31] |
LI M X, WANG X D, QI C L, et al. Metabolic response of Nile tilapia (Oreochromis niloticus) to acute and chronic hypoxia stress[J]. Aquaculture, 2018, 495: 187-195. DOI:10.1016/j.aquaculture.2018.05.031 |
| [32] |
NILSSON G E. Surviving anoxia with the brain turned on[J]. News in Physiological Sciences, 2001, 16(5): 217-221. |
| [33] |
DING J, LIU C, LUO S Y, et al. Transcriptome and physiology analysis identify key metabolic changes in the liver of the large yellow croaker (Larimichthys crocea) in response to acute hypoxia[J]. Ecotoxicology and Environmental Safety, 2020, 189: 109957. DOI:10.1016/j.ecoenv.2019.109957 |
| [34] |
HU W, ZHI L, ZHUO M Q, et al. Purification and characterization of glucose 6-phosphate dehydrogenase (G6PD) from grass carp (Ctenopharyngodon idella) and inhibition effects of several metal ions on G6PD activity in vitro[J]. Fish Physiology and Biochemistry, 2013, 39(3): 637-647. DOI:10.1007/s10695-012-9726-x |
| [35] |
WRIGHT P A, PERRY S F, MOON T W. Regulation of hepatic gluconeogenesis and glycogenolysis by catecholamines in rainbow trout during environmental hypoxia[J]. The Journal of Experimental Biology, 1989, 147(1): 169-188. DOI:10.1242/jeb.147.1.169 |
| [36] |
ZHANG G S, ZHANG J J, WEN X, et al. Comparative iTRAQ-based quantitative proteomic analysis of Pelteobagrus vachelli liver under acute hypoxia: implications in metabolic responses[J]. Proteomics, 2017, 17(17/18): 1700140. |
| [37] |
ZHONG X P, WANG D, ZHANG Y B, et al. Identification and characterization of hypoxia-induced genes in Carassius auratus blastulae embryonic cells using suppression subtractive hybridization[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2009, 152(2): 161-170. DOI:10.1016/j.cbpb.2008.10.013 |
| [38] |
PUGH C W, RATCLIFFE P J. New horizons in hypoxia signaling pathways[J]. Experimental Cell Research, 2017, 356(2): 116-121. DOI:10.1016/j.yexcr.2017.03.008 |
| [39] |
GODA N, KANAI M. Hypoxia-inducible factors and their roles in energy metabolism[J]. International Journal of Hematology, 2012, 95(5): 457-463. DOI:10.1007/s12185-012-1069-y |
| [40] |
LI L H, QU I, LI J H, et al. Relationship between HIF-1α expression and neuronal apoptosis in neonatal rats with hypoxia-ischemia brain injury[J]. Brain Research, 2007, 1180: 133-139. DOI:10.1016/j.brainres.2007.08.059 |
| [41] |
SRINIVAS V, BOHENSKY J, SHAPIRO I M. Autophagy: a new phase in the maturation of growth plate chondrocytes is regulated by HIF, mTOR and AMP kinase[J]. Cells Tissues Organs, 2008, 189(1/4): 88-92. |
2. Research Center for Fish Nutrition and Environmental Ecology, Ministry of Agriculture and Rural Affairs, Shanghai Ocean University, Shanghai 201306, China;
3. Shanghai Aquaculture Engineering Technology Research Center, Shanghai Ocean University, Shanghai 201306, China
 2023,
Vol. 32
2023,
Vol. 32


