2. 信阳农林学院水产学院 河南省渔业生物工程技术研究中心, 河南 信阳 464000;
3. 上海海洋大学 海洋动物系统分类与进化上海高校重点实验室, 上海 201306
瘤背石磺(Onchidium reevesii)隶属于软体动物门(Mollusca)腹足纲(Gastropoda)肺螺亚纲(Pulmonata)柄眼目(Stylommatophora)石磺科(Onchidiidae)[1],在中国主要分布在江苏、上海、浙江等沿海地区[2]。相对于加州海兔(Aplysia californica),瘤背石磺在我国更加容易获得,并且其中枢神经结构简单,容易分离,是进行神经功能研究的良好素材[3]。
生活在潮间带的生物,由于潮水的涨落,往往会形成特有的潮汐节律,如帽贝(Patella caerulea)[4]和红树林蟋蟀(Apteronemobius asahinai)[5]。同样生活在潮间带的瘤背石磺,能够在涨潮前及时进入潮上带洞穴中或者爬至芦苇枝上躲避海水,这种能够感知潮汐及时归穴的行为早在100多年前就已经被报道[6],刘杰等[7]报道了瘤背石磺其产卵规律趋势、产卵周期与潮高相关。在实验室长期投饵暂养后的瘤背石磺仍能记忆潮汛期,对潮汐有节律性,能记住当地的潮汐规律,具有潮汐记忆和感知能力。
海洋环境产生的声音对海洋动物的行为、生理等都有影响[8]。SOLÉ等[9]发现水下的噪声会破坏水母(Cotylorhiza tuberculata和Rhizostoma pulmo)的平衡囊结构,导致水母的听力产生损伤,KAIFU等[10]发现章鱼(Octopus ocellatus)利用平衡囊感知水下声音,梁威等[11]发现外界的声波对瘤背石磺的钙调素蛋白和类钙调素蛋白表达产生影响。瘤背石磺潜在的对外界声波的感知功能可能是瘤背石磺进行潮汐感知的关键。
磷脂酶C(Phospholipase C, PLC)是一类与膜蛋白相关的酶,可在磷酸二酯键甘油侧选择性催化磷脂酰肌醇4, 5双磷酸酯(PIP2)的水解,产生二酰基甘油(DAG)和肌醇1, 4, 5-三磷酸(IP3)两个第二信使,IP3与内质网外膜上的Ca2+通道结合后释放Ca2+进入胞浆,胞浆内Ca2+浓度增加使Ca2+与细胞内钙调蛋白(CAM)结合,激活蛋白激酶,促进蛋白质酶磷酸化,从而发挥信号转导的功能[12-14]。PENG等[15]发现人类噪声对缢蛏(Sinonovacula constricta) Ca2+ / Mg2+ -ATPase的活性产生影响,从而导致其下潜行为产生变化,WONG等[16]发现在小鼠的耳蜗毛细胞中Ca2+对声音的传导和听觉的神经传递起作用。在瘤背石磺中,钙调蛋白的相关基因已经有了报道[17-18],而PLC作为一种可以调控钙调蛋白的酶,推测其在瘤背石磺的低频感知中可能起到作用。
本实验通过瘤背石磺神经转录组,利用RACE-PCR技术获得瘤背石磺的PLC基因的cDNA全长,利用荧光定量PCR技术检测不同频率刺激对瘤背石磺PLC基因的表达情况,为今后深入研究瘤背石磺潮汐感知功能奠定科学基础。
1 材料与方法 1.1 实验材料与样品处理瘤背石磺采集于上海市崇明岛滩涂区的芦苇丛中,选择质量为(30±2)g、体长为(7.0±0.5)cm的400只瘤背石磺置于若干个塑料培养箱暂养,塑料培养箱斜倾并放扁平砖块,在倾斜的低端加水,模拟海边的环境,暂养期间每天晚上投喂玉米粉并且清理粪便,如果有个体死亡及时清除,保持塑料培养箱的低端有水,维持塑料培养箱的土壤处于湿润的状态。
将暂养2周以上的瘤背石磺分别放置到声波频率为0 (对照组)、10、40、70、100、130、160、190、220、250、280和310 Hz的处理条件下,连续刺激0、0.5、1、2、4、8、12和24 h后取样。
1.2 实验方法 1.2.1 瘤背石磺神经环总RNA的提取和cDNA的合成每个处理选取3只瘤背石磺成体,取其神经环,将剪取的神经环放置到一个冻存管中,并立即放置到液氮中进行保存备用,或者立即进行RNA的提取。在超净工作台中使用TaKaRa公司生产的RNAiso Plus试剂,按照说明书的标准流程进行瘤背石磺神经环RNA的提取。实验过程中,试剂与样品放置到冰盒上,确保提取RNA的完整性。使用Nanodrop 2000 (Thermo Scientific,美国)检测提取的总RNA浓度和核酸吸收峰,使用1.2 %的琼脂糖凝胶电泳检测提取的RNA完整性。使用南京诺唯赞生物公司的HiScript Ⅱ Q Select RT SuperMix for qPCR (+gDNA wiper)产品试剂盒,以RNA模板量为1 000 ng的统一标准,按照该试剂盒说明书进行操作,反转录成cDNA。
1.2.2 瘤背石磺OrPLC基因的全长转录本克隆与生物信息学分析在瘤背石磺神经环PacBio转录组数据中,筛选出PLC基因的转录本(cb16156_c767/f3p0/2572),并将其在本转录本中的片段命名为OrPLC(GenBank登录号MT862760)。使用Primer premier 5.0工具软件设计特异性引物(表 1)验证本序列的准确性。PCR反应体系为50 μL: 2×Taq Master Mix 25 μL (TaKaRa,上海),上下游引物各1.5 μL,ddH2O 20 μL,cDNA 2 μL。PCR反应程序: 94 ℃预变性3 min; 94 ℃ 30 s,60 ℃ 30 s,72 ℃ 60 s,38个循环; 72 ℃延伸10 min。
|
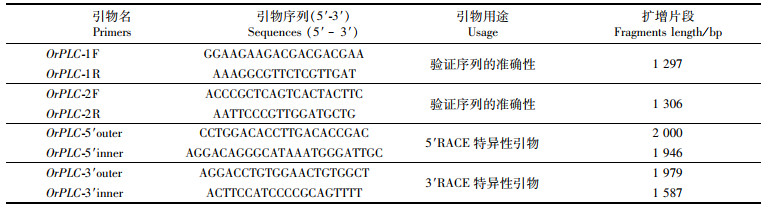
表 1 基因片段验证及RACE PCR克隆中所用到的引物序列 Tab.1 Primer sequences used in gene fragment verification and RACE PCR |
OrPLC基因cDNA全长5′端和3′端的克隆采用RACE(rapid-amplification of cDNA ends)技术,按照SMARTER®RACE5′/3′Kit User Manual (TaKaRa,Japan)进行,克隆过程中所用的引物见表 1。割胶回收试剂盒(天根生化科技北京有限公司)回收目的条带,连接到pGEM-T vector(Promega,美国),反应体系:PCR反应产物1 μL,T4 DNA Ligase 1 μL,ddH2O 2 μL。16 ℃孵育后转化到感受态DH5α大肠杆菌细胞(天根生化科技北京有限公司)后,涂布于含有氨苄的选择性培养基,37 ℃培养16~24 h,LB平板蓝白斑筛选阳性克隆,挑取出白斑菌落,进行菌液PCR反应检测,随后挑取目的菌液送生工生物工程(上海)有限公司测序。测序成功的序列利用DNAman软件进行拼接,获得OrPLC基因的cDNA全长。
将组装之后获得的OrPLC基因的cDNA序列,使用NCBI在线工具orfFinder(https://www.ncbi.nlm.nih.gov/orffinder/)预测转录本基因的开放阅读框;使用SMART在线分析网站(http://smart.emblheidelberg.de/)预测转录本所翻译的蛋白质功能结构域;使用Phyre2(http://www.sbg.bio.ic.ac.uk/phyre/)预测所翻译氨基酸序列的二级结构;使用SwissPdb Viewer软件分析所翻译的氨基酸序列的蛋白质三级结构;使用DNAman软件对氨基酸序列进行同源性序列比对,MEGA 6.0和MrBayes软件构建系统发育进化树。
1.2.3 OrPLC基因的荧光定量根据已获得的OrPLC的转录本序列在其CDS区域设计qRT-PCR特异性引物,依据转录组数据,从中选择常作为内参基因的转录本。本次实验所使用的内参基因为18S rRNA[19]。使用诺唯赞生物公司的产品ChamQ Universal SYBR qPCR Master Mix对1.1节中的不同样本进行荧光定量PCR检测,每个样品进行3次重复,使用的定量检测仪器为ABIQ6荧光定量PCR仪(Thermo Fisher,美国)。PCR的总反应体系采用说明书推荐的20 μL体系:2×ChamQ Universal SYBR qPCR Master Mix 10 μL,上下游引物各0.4 μL,cDNA模板1.5 μL,ddH2O 7.7 μL;按照说明书推荐的流程条件进行检测,使用的引物序列如表 2所示。
|
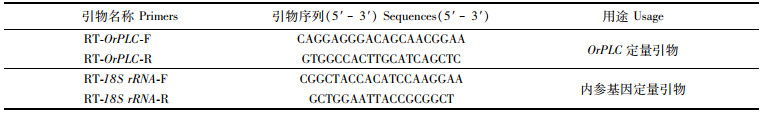
表 2 实时荧光定量PCR实验所用的特异性引物 Tab.2 Specific primers for qRTPCR experiments |
对1.2.3节得到的数据采用相对定量算法2-ΔΔCt法[20]计算OrPLC基因的相对表达量;使用SPSS 18.0进行单因素方差分析(One-way ANOVA), 采用Turky法进行差异显著性分析, P < 0.05表示具有显著性差异;使用Rstudio作图。
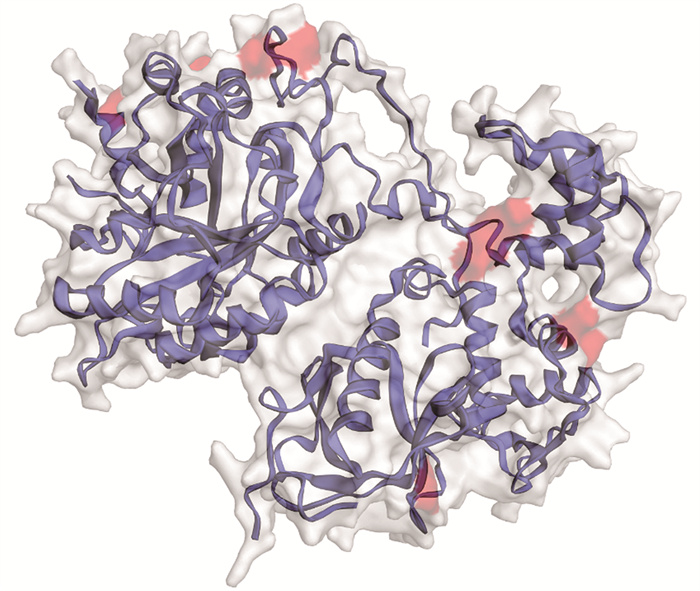
2 结果 2.1 OrPLC的序列分析经过DNAman拼接后得到的OrPLC基因的全长转录本序列长度为2 578 bp,其中ORF框长度为1 881 bp,共编码627个氨基酸,5′端有337 bp的UTR区,3′端有340 bp的UTR区,并且3′末端存在AACAA加尾信号(图 1)。所编码的蛋白质的相对分子量71.473 ku,理论等电点为6.52,氨基酸链中占比最高的氨基酸为丝氨酸(Ser,9.4%)和亮氨酸(Leu,9.1%)。二级结构预测结果显示,OrPLC蛋白的二级结构以α螺旋为主,占比达到了整个氨基酸序列的34%。在预测结果中,OrPLC氨基酸链中分别拥有磷脂酶C典型的PH和EF hand结构域。三级结构预测显示OrPLC蛋白的三维结构为左右对称中间带有夹沟的构象(图 2);相邻两个α螺旋的拐角位置为Ca2+结合位点(图 2中的红色区域)。

|
单下划线部分为PH超家族结构域,波浪线部分为EF hand结构域,方框内是加尾信号。 The single underlined part is the PH superfamily domain, the wave line part is the EF hand domain, and the square box part is the tail signal. 图 1 瘤背石磺OrPLC基因的cDNA序列及其编码的氨基酸序列 Fig. 1 OrPLC cDNA sequence and its encoded amino acid sequence |
|
红色区域预测为钙离子结合位置。 Red area is predicted as the calcium ion binding site. 图 2 瘤背石磺OrPLC蛋白的三级结构模型预测 Fig. 2 Model prediction of the tertiary structure of OrPLC protein |
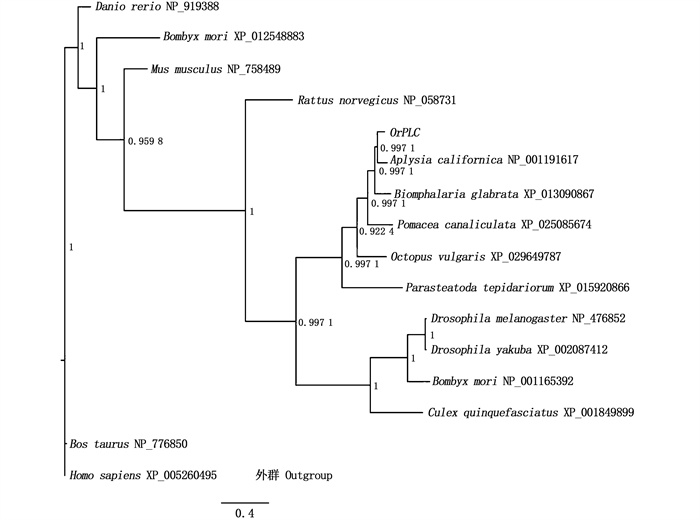
OrPLC基因翻译的氨基酸序列在NCBI数据库中进行BLAST比对分析结果显示,其与福寿螺(Pomacea canaliculata)的磷脂酶C样蛋白2的氨基酸序列(XP_025085674)匹配度最高,一致性达到88%,其次是美洲牡蛎(Crassostrea virginica)的磷脂酶C样蛋白2的氨基酸序列(XP_022329093)一致性为86%,通过DNAman对匹配度较高的蛋白质序列的同源性比较见图 3。根据OrPLC的氨基酸序列,选取了包括瘤背石磺在内的15个物种的15条氨基酸序列,并选择人类的磷脂酶C氨基酸序列作为外类群,利用MEGA 6.0和贝叶斯软件构建系统进化树(图 4);在进化树形中,该蛋白对应的最近的亲缘关系物种为加州海兔(Aplysia californica)。

|
图 3 OrPLC的氨基酸序列与同源性较高的PLC氨基酸序列的多重序列比对 Fig. 3 Multiple sequence alignment of OrPLC amino acid sequence with PLC protein |
|
图 4 基于15个物种的PLC氨基酸序列构建的系统进化树 Fig. 4 Phylogenetic tree constructed based on PLC amino acid sequences of 15 species |
瘤背石磺在不同声波刺激下的OrPLC基因的荧光定量PCR分析结果如图 5,通过和对照组比较,从OrPLC的定量分析图可以发现:在刺激声波频率为10、100、160和310 Hz的时候,OrPLC基因的表达量较低;当刺激频率为40、70、130、190和250 Hz时,OrPLC基因表达的水平较为稳定;在声波频率为220 Hz和280 Hz时,OrPLC基因表达较高。
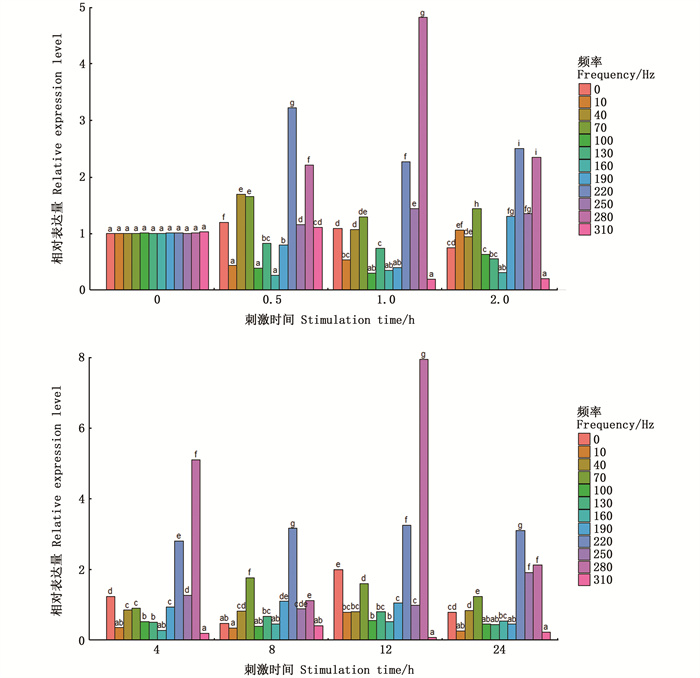
|
不同字母表示差异显著(P < 0.05)。 Different letters indicate significant difference (P < 0.05). 图 5 OrPLC基因在瘤背石磺神经环中不同声波频率下的表达量 Fig. 5 Expression of OrPLC at different acoustic frequency in O.reveesii ganglion |
本次实验通过克隆技术得到了瘤背石磺神经环组织的OrPLC基因的全长转录本序列,并对序列进行了生物信息学分析。预测的二级结构中,该基因所编码的氨基酸链大部分组装成为α螺旋结构,这为构成三维蛋白模型中与Ca2+结合的活性中心提供了结构基础,此外OrPLC氨基酸链中存在PH超家族和EF hand结构域区域,与其他物种磷脂酶C的结构研究相吻合[21]。三维结构模型预测发现OrPLC蛋白中存在着多个能够结合Ca2+的构象,这与其基于Ca2+催化特性是密不可分的[22]。运用OrPLC氨基酸序列比对构建系统进化树,结果显示瘤背石磺与加州海兔(A. californica)聚为一支,与吴容宇等[23]研究一致,说明瘤背石磺OrPLC与软体动物门腹足纲贝类的亲缘关系非常接近,与传统的形态学分类相吻合,体现了OrPLC在进化上的保守性;OrPLC的进化关系的分析能为研究物种的进化提供参考。
在不同声波频率刺激下OrPLC基因在瘤背石磺神经环组织中的表达情况结果显示,OrPLC基因对不同声波频率刺激的反应有所差别,例如在声波频率较低(40 Hz和70 Hz)时该基因在所有取样时间有一个较为稳定的表达,而在用310 Hz的声波频率处理0.5 h以上时其表达受到抑制,这种急剧性的变化可能是暴露于外界噪音时间过长,导致其平衡囊受损,从而使其失去了对外界声波感知能力。本次实验中,声波刺激频率为220 Hz和280 Hz时,OrPLC基因表达量有显著提升,可见瘤背石磺可以感知到空气中的声波变化并且对不同频率有分辨能力。在潮汐的整个涨退潮的过程中,海浪对海岸的冲击会产生一系列的声波[24],而涨退潮这种现象是有规律变化的[25],因此潮汐的声波应该也是有规律变化的,由此推测瘤背石磺可以感知海水涨潮时的声波,进而能够及时回归巢穴躲避潮水。
栖息潮间带的瘤背石磺的归家行为取决于潮汐周期,而1个半日潮的潮汐周期约为12.4 h[25],SCHNYTZER等[26]研究了齿轮帽贝(Cellana rota) 12.4 h的潮汐节律变化,发现了相关调控基因的变化与潮汐节律相互吻合,所以尽管没有低频声音的刺激,对照组的OrPLC的表达量在12 h依旧较高,而当受到外部低频刺激时,可能会影响原有的长期记忆从而影响了OrPLC的表达,另外目前已有实验表明在长期记忆的形成过程中,长时间昼夜节律会影响瘤背石磺的记忆形成[27],而本次实验中,不同时间组未有长时间的昼夜变化,因此在不同时间的维度下OrPLC的变化没有明显规律。
综上所述,本实验成功克隆出瘤背石磺体内的OrPLC基因全长转录本序列,并进行了生物信息学的预测分析,其OrPLC蛋白质结构与其他物种的磷脂酶C的结构类似,进化比较保守;荧光定量PCR结果显示瘤背石磺可以感知空气中声波刺激,将为进一步研究瘤背石磺的潮汐感知功能提供基础。
| [1] |
BOUCHET P, ROCROI J P, FRDA J, et al. Classification and nomenclator of gastropod families[J]. Malacologia, 2005, 47(1/2): 1-368. |
| [2] |
吴旭峰, 沈和定, 吴文健, 等. 我国华东沿海4种石磺形态学比较[J]. 动物学杂志, 2010, 45(6): 92-100. WU X F, SHEN H D, WU W J, et al. Comparison on morphology of onchidiidae in eastern coast of China[J]. Chinese Journal of Zoology, 2010, 45(6): 92-100. |
| [3] |
张坤霞. 我国石磺科形态结构再描述及同工酶分析[D]. 上海: 上海海洋大学, 2013. ZHANG K X. Study of morphology and isozymes of Onchidiidae in China[D]. Shanghai: Shanghai Ocean University, 2013. |
| [4] |
PARPAGNOLI D, PECCHIOLI S, SANTINI G. Temporal determinants of grazing activity in the Mediterranean limpet Patella caerulea[J]. Ethology Ecology & Evolution, 2013, 25(4): 388-399. |
| [5] |
SATOH A, YOSHIOKA E, NUMATA H. Circatidal activity rhythm in the mangrove cricket Apteronemobius asahinai[J]. Biology Letters, 2008, 4(3): 233-236. DOI:10.1098/rsbl.2008.0036 |
| [6] |
AREY L B, CROZIER W J. The 'Homing Habits' of the pulmonate mollusk Onchidium[J]. Proceedings of the National Academy of Sciences of the United States of America, 1918, 4(11): 319-321. DOI:10.1073/pnas.4.11.319 |
| [7] |
刘杰, 黄金田. 瘤背石磺产卵规律与天文潮汐关系[J]. 上海海洋大学学报, 2018, 27(1): 73-78. LIU J, HUANG J T. The relationship between Onchidium struma's spawning regularity and astronomical tides[J]. Journal of Shanghai Ocean University, 2018, 27(1): 73-78. |
| [8] |
张国胜, 顾晓晓, 邢彬彬, 等. 海洋环境噪声的分类及其对海洋动物的影响[J]. 大连海洋大学学报, 2012, 27(1): 89-94. ZHANG G S, GU X X, XING B B, et al. The classification and the impact of marine environment noise on marine animals[J]. Journal of Dalian Ocean University, 2012, 27(1): 89-94. DOI:10.3969/j.issn.1000-9957.2012.01.018 |
| [9] |
SOLÉ M, LENOIR M, FORTUÑO J M, et al. Evidence of Cnidarians sensitivity to sound after exposure to low frequency noise underwater sources[J]. Scientific Reports, 2016, 6: 37979. DOI:10.1038/srep37979 |
| [10] |
KAIFU K, AKAMATSU T, SEGAWA S. Underwater sound detection by cephalopod statocyst[J]. Fisheries Science, 2008, 74(4): 781-786. DOI:10.1111/j.1444-2906.2008.01589.x |
| [11] |
梁威, 吴容宇, 严彩瑞, 等. 低频声波频率下瘤背石磺CaM-like、CaMKII基因的表达及功能[J]. 中国水产科学, 2020, 27(2): 166-176. LIANG W, WU R Y, YAN C R, et al. Expression of CaM-like and CaMKII genes in Onchidium reevesii un-der low frequency sound wave stimulation[J]. Journal of Fishery Sciences of China, 2020, 27(2): 166-176. |
| [12] |
KADAMUR G, ROSS E M. Mammalian phospholipase C[J]. Annual Review of Physiology, 2012, 75(1): 127-154. |
| [13] |
ESSEN L O, PERISIC O, KATAN M, et al. Structural mapping of the catalytic mechanism for a mammalian phosphoinositide-specific phospholipase C[J]. Biochemistry, 1997, 36(7): 1704-1718. DOI:10.1021/bi962512p |
| [14] |
CHIN D, MEANS A R. Calmodulin: a prototypical calcium sensor[J]. Trends in Cell Biology, 2000, 10(8): 322-328. DOI:10.1016/S0962-8924(00)01800-6 |
| [15] |
PENG C, ZHAO X G, LIU S X, et al. Effects of anthropogenic sound on digging behavior, metabolism, Ca2+/Mg2+ ATPase activity, and metabolism-related gene expression of the bivalve Sinonovacula constricta[J]. Scientific Reports, 2016, 6: 24266. DOI:10.1038/srep24266 |
| [16] |
WONG A C Y, BIRNBAUMER L, HOUSLEY G D. Canonical transient receptor potential channel subtype 3-mediated hair cell Ca2+ entry regulates sound transduction and auditory neurotransmission[J]. European Journal of Neuroscience, 2013, 37(9): 1478-1486. DOI:10.1111/ejn.12158 |
| [17] |
梁威, 杨铁柱, 沈和定, 等. 瘤背石磺钙/钙调蛋白依赖性蛋白激酶Ⅳ(CaMK Ⅳ)基因的克隆及组织表达[J]. 水产学报, 2019, 43(11): 2336-2346. LIAN W, YANG T Z, SHEN H D, et al. Cloning and tissue expression analysis of the calcium/calmodulin dependent protein kinase Ⅳ gene (CaMK Ⅳ) in Onchidium struma[J]. Journal of Fisheries of China, 2019, 43(11): 2336-2346. |
| [18] |
李杰, 许国绿, 沈和定, 等. 石磺钙调蛋白Os-IP3R和Os-RyR基因的克隆、相对表达量及进化关系[J]. 水产学报, 2018, 42(12): 1857-1868. LI J, XU G L, SHEN H D, et al. Clone and relative expression of Os-IP3R and Os-RyR to discuss the evolutionary relationship in Onchidiidae[J]. Journal of Fisheries of China, 2018, 42(12): 1857-1868. |
| [19] |
YANG T Z, GU B N, XU G L, et al. Identification of candidate reference genes for qRT-PCR normalization studies of salinity stress and injury in Onchidium reevesii[J]. PeerJ, 2019, 7: e6834. DOI:10.7717/peerj.6834 |
| [20] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [21] |
刘丽荣, 夏时海. 磷脂酶C结构及活化机制的研究进展[J]. 武警医学院学报, 2006, 15(2): 175-178. LIU L R, XIA S H. Progress in the structure of phospholipase C and mechanisms of its activation[J]. Acta Academiae Medicinae CPAPF, 2006, 15(2): 175-178. DOI:10.3969/j.issn.1008-5041.2006.02.042 |
| [22] |
BRUZIK K S, TSAI M D. Toward the mechanism of phosphoinositide-specific phospholipases C[J]. Bioorganic & Medicinal Chemistry, 1994, 2(2): 49-72. |
| [23] |
吴容宇, 梁威, 土志涵, 等. 瘤背石磺Egr-1基因的克隆、分析及低频声音刺激对其表达的影响[J]. 基因组学与应用生物学, 2021, 40(2): 622-630. WU R Y, LIAN W, TU Z H, et al. Cloning and expression analysis of the Egr-1 gene of Onchidium reevesii and low frequence sound stimulaton on gene expression[J]. Genomics and Applied Biology, 2021, 40(2): 622-630. |
| [24] |
BONDAR' L F, BUGAEVA L K, RUTENKO A N. Effect of tide on sound propagation in the shelf zone of the Sea of Japan[J]. Acoustical Physics, 2000, 46(5): 534-543. DOI:10.1134/1.1310377 |
| [25] |
张凯宁. 潮汐成因探讨[J]. 大学物理, 2014, 33(10): 31-34. ZHANG K N. Discussion on the cause of tide[J]. College Physics, 2014, 33(10): 31-34. |
| [26] |
SCHNYTZER Y, SIMON-BLECHER N, LI J, et al. Tidal and diel orchestration of behaviour and gene expression in an intertidal mollusc[J]. Scientific Reports, 2018, 8: 4917. DOI:10.1038/s41598-018-23167-y |
| [27] |
XU G L, YANG T Z, SHEN H D. Effect of circadian clock and light-dark cycles in Onchidium reevesii: possible implications for long-term memory[J]. Genes (Basel), 2019, 10(7): 488. DOI:10.3390/genes10070488 |
2. Fishery Biological Engineering Technology Research Center of Henan Province, College of Fisheries, Xinyang Agriculture and Forestry University, Xinyang 464000, Henan, China;
3. Marine Animal System Classification and Evolution Shanghai University Key Laboratory, Shanghai Ocean University, Shanghai 201306, China
 2022,
Vol. 31
2022,
Vol. 31


