2. 大洋渔业资源可持续开发教育部重点实验室, 上海 201306;
3. 国家远洋渔业工程技术研究中心, 上海 201306;
4. 农业农村部大洋渔业开发重点实验室, 上海 201306;
5. 农业农村部大洋渔业资源环境科学观测实验站, 上海 201306
太平洋褶柔鱼(Todarodes pacificus)又名东洋鱿、日本鱿等,属头足纲(Cephalopoda)枪形目(Teuthida)柔鱼科(Ommastrephidae)褶柔鱼亚科(Todarodinae)褶柔鱼属(Todarodes)[1],主要分布在太平洋21°N~50°N海域,分为冬生群、秋生群和夏生群,为一年生暖温性长距离洄游种类[2-3]。太平洋褶柔鱼是世界上最早被大规模开发利用的头足类种类之一,20世纪70年代后期由于过度捕捞其产量急剧下降,随后产量慢慢回升,但1997—1998年经历了强厄尔尼诺后,其产量锐减至历史最高产量的一半[4]。作为一种高度洄游生物,其生长、繁殖受环境影响较大[5],进而影响该种群资源丰度,需引起广泛重视,合理开发利用并加强管理太平洋褶柔鱼资源亟待需要。
线粒体是真核细胞内为细胞活动提供能量的重要细胞器,为共价闭合的环状超螺旋结构,作为细胞内核外唯一存在的遗传物质[6-7],包括线粒体细胞色素C氧化酶Ⅰ基因(COⅠ)、ND2、线粒体r DNA等众多基因,与核DNA相比,具有分子量小、结构简单且保守紧凑、进化速率快并遵循母系遗传、无组织特异性等特点,是一个相对独立的复制单位[8-9]。刘连为等[10]利用COⅠ和Cytb基因分析了北太平洋柔鱼遗传结构,因此Mt DNA标记法广泛应用于头足类种群遗传结构研究中,近些年来也逐渐应用到柔鱼类的研究中,成为研究太平洋褶柔鱼种群遗传结构的不可替代的技术手段[11]。
目前对太平洋褶柔鱼研究主要集中于资源丰度及其评估,基础生物学和渔场时空分布等方面[4-5, 11-13]。KATUGIN[14]采用蛋白质电泳方法对太平洋褶柔鱼进行了遗传多样性分析,发现太平洋褶柔鱼存在2个亚种,并且依据其繁殖习性分为3或4个种下群体,为该种群遗传分析提供了重要的基础数据;WAKABAYASHI等[15]利用COⅠ和16S rRNA重新推断了柔鱼科的系统发育关系,认为褶柔鱼亚科中,太平洋褶柔鱼与双柔鱼(Nototodarus)的种类关系比南极褶柔鱼(Todarodes filippovae)的关系更为密切。太平洋褶柔鱼经历被开发利用、过度捕捞再到产量逐渐回升以及环境不断变化等,该种群遗传结构是否已经产生变异, 发生变异的主要原因是什么,目前相关研究较少。因此,为进一步规范和提升太平洋褶柔鱼的渔业资源管理,该研究利用COⅠ和Cytb分子标记法综合分析了太平洋褶柔鱼的遗传结构和变异,以期为太平洋褶柔鱼渔业资源的开发和管理提供理论依据。
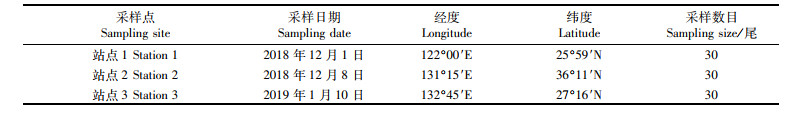
1 材料与方法 1.1 材料来源样本由中国鱿钓船于2018年12月—2019年1月在日本海、东海和西北太平洋生产调查期间采集,采样点、采样时间及样本数量见表 1。
|
表 1 太平洋褶柔鱼样本采集信息 Tab.1 Collecting information of Todarodes pacificus samples |
取太平洋褶柔鱼肌肉50 mg左右,利用酚-氯仿法提取基因组DNA[16],用NanoDrop 2 000 C超微量分光光度计测量DNA浓度后于-20 ℃冰箱保存备用。该研究获得的COⅠ(GenBank: KY771096.1)和Cytb(GenBank: EF423147.1)序列于NCBI下载,然后用Primer 5.0软件分别设计其引物,由生工生物工程(上海)有限公司合成,具体引物序列如表 2所示。PCR反应体系总体积为25 μL,其中:引物各0.5μL,2xEasy TaqpcrSuperMix酶12.5 μL,模板DNA 1 μL,加灭菌超纯水至25 μL。PCR反应在S 1 000型双48孔PCR仪上进行。COⅠ和Cytb基因的扩增程序一致,其扩增反应条件为95 ℃预变性3 min,然后进行34个循环,每个循环包括95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸1 min,最后72 ℃终延伸10 min。PCR扩增结束之后跑胶,若条带单一且明亮,随即送生工生物工程(上海)股份有限公司测序,并分析测序结果。

|
表 2 太平洋褶柔鱼COⅠ和Cytb基因序列引物设计 Tab.2 Design of primers for COⅠ and Cytb gene sequences of Todarodes pacificus |
测序完成后,首先利用MEGA 7.0软件进行序列比对[17],将各条不同的碱基序列首尾适当删除得到可用的序列长度,并利用邻接法(NJ)[18]构建分子进化树;通过DnaSP 6.12软件分析[19]单倍型数、单倍型多样性指数(h)、核苷酸多样性指数(π)以及核苷酸错配分布曲线。然后通过软件Arequin 3.1[20]计算遗传分化系数(Fst)并进行分子生物学方差分析(AMOVA)以及Tajima’s D和Fu’s Fs中性检验。最后利用软件Network 4.0[21]构建太平洋褶柔鱼COⅠ和Cytb单倍型网络图。
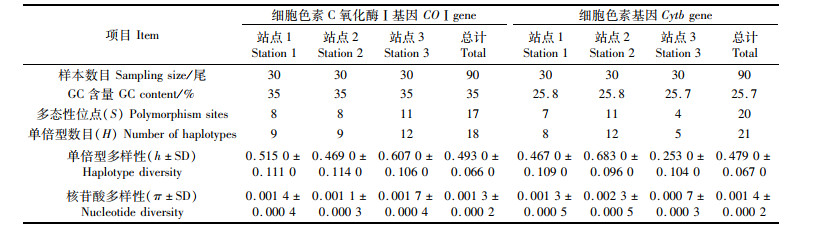
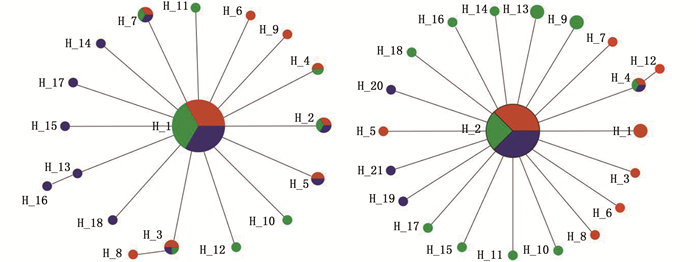
2 结果 2.1 太平洋褶柔鱼种群遗传多样性该研究得到太平洋褶柔鱼COⅠ基因序列长度为465 bp,3个地理群体90尾样本共检测到17个可变异位点,共定义了18个单倍型(表 3),有且仅有一个核心单倍型H_1(图 1)为其他群体所共有。3个站点太平洋褶柔鱼COⅠ序列遗传多样性指数并无显著差异(表 3),与总体处于同一水平。样本总体表现出较低的单倍型多样性(0.493 0±0.066 0)和较低的核苷酸多样性(0.001 3±0.000 2)。
|
表 3 基于COⅠ和Cytb基因序列的遗传多样性 Tab.3 Genetic diversity based on COⅠ and Cytb gene sequences |
|
图 1 基于太平洋褶柔鱼COⅠ基因(左)和Cytb基因(右)标记的单倍型网络图 Fig. 1 Haplotype network diagram based on COⅠ gene (left) and Cytb gene (right) markers of Todarodes pacificus |
该种群Cytb基因序列长度为404 bp,3个地理群体90尾样本共检测到20个可变异位点,其中20个为多态性信息位点,共定义了21个单倍型(表 3),唯一核心单倍型为H_2(图 1)。与COⅠ基因所表现的结果相同,3个站点太平洋褶柔鱼拥有较低的单倍型多样性(0.479 0±0.067 0)和核苷酸多样性(0.001 4±0.000 2)。
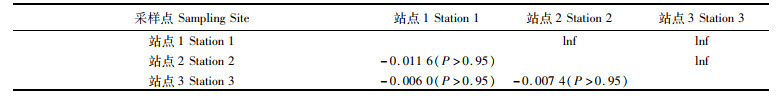
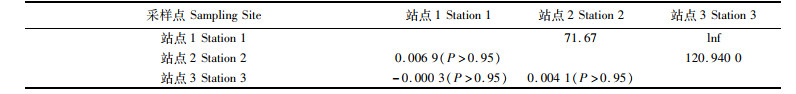
2.2 种群遗传结构变异分析太平洋褶柔鱼COⅠ序列的分子方差分析(AMOVA)表明几乎全部的遗传变异来自于该种群内部(表 4),遗传分化系数均为负值,基因流Nm为无穷大inf(表 5),表明各个群体间基因交流非常频繁群体间保持着较高的遗传同质性。该种群Cytb序列AMOVA分析也表明了变异几乎来源于该群体内部,只有极少数(0.43%)变异来自群体间(表 4)。群体间遗传分化系数(Fst)绝对值接近于0,差异不显著(P>0.95),同时群体间基因流数值较大(表 6),与COⅠ序列结果类似。

|
表 4 基于太平洋褶柔鱼COⅠ和Cytb基因的AMOVA分析 Tab.4 AMOVA analysis results based on COⅠ and Cytb genes of Todarodes pacificus |
|
表 5 基于CO I基因序列的Fst值(对角线下方)和基因流值(对角线上方) Tab.5 Fst value (below diagonal) and gene flow value (above diagonal) based on COⅠ gene sequences |
|
表 6 基于Cytb基因序列的Fst值(对角线下方)和基因流值(对角线上方) Tab.6 Fst value (below diagonal) and gene flow value (Nm diagonal) based on Cytb gene sequences |
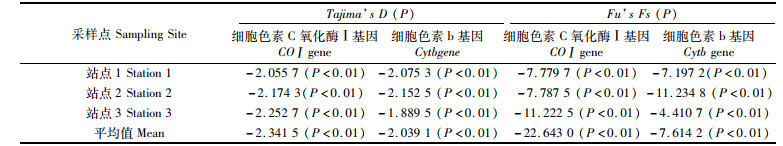
该研究中太平洋褶柔鱼单倍型核苷酸错配分布分析(图 2)中都出现了明显的单峰,说明该群体可能经历过种群扩张事件,而Tajima’s D和Fu’s Fs的中性检验值(表 7)均为负值且统计检验极显著(P<0.01),进一步佐证该群体很可能发生过历史扩张事件。Cytb基因序列常被用来估算种群扩张时间,通过Arlequin 3.1软件计算得到基于Cytb基因序列的太平洋褶柔鱼群体的Tau为0.567,Cytb基因的进化速率为(2.15%~2.60%)/百万年[22],根据公式T=t×Tau/4kμ(其中k为文中分析基因序列的碱基数,μ为分析基因序列每一代的进化速率),进一步推算群体扩张时间,得到太平洋褶柔鱼群体扩张大概发生在1.2万~1.5万年前。

|
图 2 基于太平洋褶柔鱼COⅠ(a)和Cytb(b)基因序列的Mismatch错配分布图 Fig. 2 Mismatch distribution diagram based on COⅠ gene(a)and Cytb gene (b) sequences of T.pacificus |
|
表 7 基于COⅠ和Cytb基因序列的中性检验结果 Tab.7 Neutral test results based on COⅠ and Cytb gene sequences |
遗传多样性是生物多样性的重要组成部分,研究太平洋褶柔鱼遗传多样性不仅能够判断该物种对环境的适应能力并揭示其起源与进化历史,而且能为遗传资源的保存提供重要的理论支持[22]。该研究中太平洋褶柔鱼G+C含量明显低于A+T含量,具有明显AT碱基偏好性,在北太平洋柔鱼(Ommastrephes bartramii)[10]以及中国沿海长蛸(Octopus variabilis)[23]等大洋性头足类也有类似的结论,该特性使头足类动物具有更易变异的潜力;且太平洋褶柔鱼生命周期短、性成熟快、繁殖群体时空范围广和多季度等[24],这些生活史特征可能使该种群能够快速增长,保存新突变,产生高的单倍型指数h,然而该研究中太平洋褶柔鱼的h并不高。与同样基于COⅠ和Cytb序列标记的大洋性头足类相比,如北太平洋柔鱼(hCOⅠ = 0.773±0.105、hCytb= 0.868±0.095)、阿根廷滑柔鱼(Illex argentinus,hCOⅠ=0.535±0.066、hCytb= 0.528±0.058)、茎柔鱼(Dosidicus gigas,hCOⅠ= 0.743±0.055、hCytb= 0.767±0.047)都具有较高的单倍型多样性指数h[10]。目前已有研究[25]证实过度捕捞会导致海洋鱼类遗传多样性衰退,而太平洋褶柔鱼在19世纪七八十年代曾遭受过度开发[4],使得该种群资源量急剧减少,这有可能是导致太平洋褶柔鱼单倍型多样性不高,遗传多样性总体上偏低的原因。另外也存在太平洋褶柔鱼本身遗传多样性就低的可能,但由于缺乏历史研究数据而无从考证。因此,该研究可作为一个基线资料,供后续种质检测提供参考依据。
AMOVA分析结果表明这3个地理群体之间并未出现明显的遗传分化,从COⅠ和Cytb基因单倍型网络图中也可从侧面反映该种群有且仅有一个核心单倍型,其他单倍型之间几乎无交集。由此推断这3个站点群体可能属于同一种群。此外,东海和日本海在地理环境上不存在明显的阻隔,且亲潮-黑潮分支的入侵以及季风作用下环流的存在[4]使得西北太平洋全年受洋流影响较大,洋流运输作用明显,同时太平洋褶柔鱼可在不同季节分批产卵,且产卵时间长、产卵量大[24],大量的褶柔鱼卵和幼体则可能随洋流扩散到太平洋各个区域,使得东海和日本海这3个采样群体间的基因交流十分频繁,从而与极大的基因流(Nm)结果相呼应。
另外,太平洋褶柔鱼种群扩张时间大约发生在1.2万~1.5万年前,这个时期正处于晚更新世末期。GORBARENKO等[26]利用日本海中部和南部5个高分辨率岩芯的浮游有孔虫δ18O记录、岩性变化,发现15 kaBP前后,水体盐度逐渐恢复正常,海平面逐渐上升,大洋水进入日本海,此后,浮游有孔虫δ18O值分别在14.5 kaBP和12 kaBP前后发生2次快速变轻,水温迅速升高,太平洋褶柔鱼的卵和幼鱼可能借助海平面上升而扩散,并且该种群结构、性腺发育、分布等受环境变化,特别是水温变化的影响较大[11],这能够给太平洋褶柔鱼在内的其他生物的生长、繁殖及扩张提供非常有利的条件。
种群是渔业资源利用和保护的基本单位,该研究利用COⅠ和Cytb两种线粒体基因标记方法,探究了西北太平洋褶柔鱼群体遗传结构及变异分析,结果表明太平褶柔鱼遗传多样性较低且不存在显著分化,为该种群遗传分析提供了重要的基础数据。因此,可以考虑将太平洋褶柔鱼划分为一个管理单元,以便该渔业资源更加合理的保护与开发利用。
| [1] |
解明阳, 陈新军, 汪金涛. 基于灰色系统的太平洋褶柔鱼冬春生群资源丰度变化研究[J]. 海洋渔业, 2018, 40(6): 641-648. XIE M Y, CHEN X J, WANG J T. On changes of abundance index of winter-spring cohort of Todarodes pacificus in the Northwest Pacific based on grey system theory[J]. Marine Fisheries, 2018, 40(6): 641-648. DOI:10.3969/j.issn.1004-2490.2018.06.001 |
| [2] |
张硕, 李莉, 陈新军. 基于海表温因子的太平洋褶柔鱼冬生群资源丰度预测模型比较[J]. 水产学报, 2018, 42(5): 704-710. ZHANG S, LI L, CHEN X J. A comparative study on forecasting model of the stock abundance index for the winter-spawning cohort of Todarodes pacificus in the Pacific Ocean based on the factor of SST[J]. Journal of Fisheries of China, 2018, 42(5): 704-710. |
| [3] |
董正之. 黄海太平洋褶柔鱼资源现状与开发[J]. 海洋科学, 1996, 20(6): 34-38. DONG Z Z. On the present status and exploratory problems of resource of Todarodes pacificus in the Yellow sea[J]. Marine Sciences, 1996, 20(6): 34-38. |
| [4] |
方舟, 陈新军. 太平洋褶柔鱼渔业资源及渔场学研究进展[J]. 海洋渔业, 2018, 40(1): 102-116. FANG Z, CHEN X J. Review on fishery of Japanese flying squid Todarodes pacificus[J]. Marine Fisheries, 2018, 40(1): 102-116. DOI:10.3969/j.issn.1004-2490.2018.01.012 |
| [5] |
武胜男, 余为, 陈新军. 太平洋褶柔鱼秋生群产卵场环境变化及对资源丰度的影响[J]. 海洋渔业, 2018, 40(2): 129-138. WU S N, YU W, CHEN X J. Variations in environmental conditions on the spawning grounds of autumn cohort of Todarodes pacificus and their impacts on squid abundance[J]. Marine Fisheries, 2018, 40(2): 129-138. DOI:10.3969/j.issn.1004-2490.2018.02.001 |
| [6] |
赵群. 分子亲缘地理学及遗传标记技术的应用[J]. 生物技术世界, 2016(3): 297-298. ZHAO Q. Application of molecular genetic geography and genetic[J]. Biotech World, 2016(3): 297-298. |
| [7] |
刘丽丽, 李建辉, 程海翔. 分子标记在种群历史动态研究中的应用[J]. 农业科技通讯, 2014(7): 207-211. LIU L L, LI J H, CHEN H X. Research and application of molecular markers in population history[J]. Bulletin of Agricultural Science and Technology, 2014(7): 207-211. DOI:10.3969/j.issn.1000-6400.2014.07.075 |
| [8] |
白云, 王妍, 胡莹, 等. 5种类型鲤鱼线粒体DNA D-loop及邻近区段的遗传多样性分析[J]. 水产科学, 2019, 38(4): 498-505. BAI Y, WANG Y, HU Y, et al. Genetic diversity analysis of mitochondrial DNA D-loop and its adjacent region in five types of common carp[J]. Fisheries Science, 2019, 38(4): 498-505. |
| [9] |
任衍钢, 白冠军, 宋玉奇, 等. 线粒体DNA的发现及其基因组的揭示过程[J]. 生物学通报, 2019, 54(6): 55-58. REN Y G, BAI G J, SONG Y Q, et al. Discovery of mitochondrial DNA and the process of genome discovery[J]. Bulletin of Biology, 2019, 54(6): 55-58. DOI:10.3969/j.issn.0006-3193.2019.06.020 |
| [10] |
刘连为, 许强华, 陈新军. 基于线粒体COⅠ和Cytb基因序列的北太平洋柔鱼种群遗传结构研究[J]. 水产学报, 2012, 36(11): 1675-1684. LIU L W, XU Q H, CHEN X J. Population genetic structure of Ommastrephes bartramii in the North Pacific Ocean based on the COⅠ and Cytb gene sequences analysis[J]. Journal of Fisheries of China, 2012, 36(11): 1675-1684. |
| [11] |
刘连为. 三种大洋性柔鱼类种群遗传结构及分子系统地理学研究[D]. 上海: 上海海洋大学, 2014: 47-115. LIU L W. Population genetics structure and molecular phylogeography of three oceanic Ommatrephidae species[D]. Shanghai: Shanghai Ocean University, 2014: 47-115. |
| [12] |
吴梦瑶, 田思泉. 基于海表面温度SST的剩余产量模型评估太平洋褶柔鱼秋生群资源[J]. 上海海洋大学学报, 2019, 28(6): 933-942. WU M Y, TIAN S Q. Stock assessment for autumn cohort of Todarodes pacificus based on surplus yield model with sea surface temperature[J]. Journal of Shanghai Ocean University, 2019, 28(6): 933-942. |
| [13] |
陈新军, 陆化杰, 刘必林, 等. 大洋性柔鱼类资源开发现状及可持续利用的科学问题[J]. 上海海洋大学学报, 2012, 21(5): 831-840. CHEN X J, LU H J, LIU B L, et al. Current exploitation and some scientific issues in the sustainable utilization of Ommastrephidae[J]. Journal of Shanghai Ocean University, 2012, 21(5): 831-840. |
| [14] |
KATUGIN O N. Patterns of genetic variability and population structure in the North Pacific squids Ommastrephes bartramii, Todarodes pacificus, and Berryteuthis magister[J]. Bulletin of Marine Science, 2002, 71(1): 383-420. |
| [15] |
WAKABAYASHI T, SUZUKI N, SAKAI M, et al. Phylogenetic relationships among the family Ommastrephidae (Mollusca: Cephalopoda) inferred from two mitochondrial DNA gene sequences[J]. Marine Genomics, 2012, 7: 11-16. DOI:10.1016/j.margen.2012.04.005 |
| [16] |
顾铭悦, 许强华. 贝氏肩孔南极鱼基因组DNA提取方法改进的初步研究[J]. 上海海洋大学学报, 2013, 22(2): 168-172. GU M Y, XU Q H. Preliminary study on improved genomic DNA extraction method from Trematomus bernacchii[J]. Journal of Shanghai Ocean University, 2013, 22(2): 168-172. |
| [17] |
KUMAR S, STECHER G, TAMURA K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [18] |
ROZAS J, FERRER-MATA A, SÁNCHEZ-DELBARRIO J C, et al. DnaSP 6:DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution, 2017, 34(12): 3299-3302. DOI:10.1093/molbev/msx248 |
| [19] |
ROGERS A R, HARPENDING H. Population growth makes waves in the distribution of pairwise genetic differences[J]. Molecular Biology and Evolution, 1992, 9(3): 552-569. |
| [20] |
EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5:a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564-567. DOI:10.1111/j.1755-0998.2010.02847.x |
| [21] |
TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585-595. DOI:10.1093/genetics/123.3.585 |
| [22] |
崔朝霞, 张峘, 宋林生, 等. 中国重要海洋动物遗传多样性的研究进展[J]. 生物多样性, 2011, 19(6): 815-833. CUI Z X, ZHANG H, SONG L S, et al. Genetic diversity of marine animals in China: a summary and prospectiveness[J]. Biodiversity Science, 2011, 19(6): 815-833. |
| [23] |
孙宝超, 杨建敏, 孙国华, 等. 中国沿海长蛸(Octopus variabilis)自然群体线粒体COⅠ基因遗传多样性研究[J]. 海洋与湖沼, 2010, 41(2): 259-265. SUN B C, YANG J M, SUN G H, et al. Sequence and molecular phylogeny of mitochondrial COⅠ gene fragment in five populations of Octopus variabilis in China[J]. Oceanologia et Limnologia Sinica, 2010, 41(2): 259-265. |
| [24] |
杨林林, 姜亚洲, 程家骅. 东海太平洋褶柔鱼生殖群体的空间分布及其与环境因子的关系[J]. 生态学报, 2010, 30(7): 1825-1833. YANG L L, JIANG Y Z, CHENG J Y. Spatial distribution of mature stock of Pacific squid, Todarodes pacificus and its relation to environmental factors in the East China Sea[J]. Acta Ecologica Sinica, 2010, 30(7): 1825-1833. |
| [25] |
PINSKY M L, PALUMBI S R. Meta-analysis reveals lower genetic diversity in overfished populations[J]. Molecular Ecology, 2014, 23(1): 29-39. DOI:10.1111/mec.12509 |
| [26] |
GORBARENKO S A, SOUTHON J R. Detailed Japan Sea paleoceanography during the last 25 kyr: constraints from AMS dating and δ18O of planktonic foraminifera[J]. Palaeogeography, Palaeoclimatology, Palaeoecology, 2000, 156(3/4): 177-193. |
2. Key Laboratory of Sustainable Exploitation of Oceanic Fisheries Resources, Ministry of Education, Shanghai 201306, China;
3. National Distant-water Fisheries Engineering Research Center, Shanghai 201306, China;
4. Key Laboratory of Ocean Fisheries Exploitation, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China;
5. Scientific Observing and Experimental Station of Oceanic Fishery Resources, Ministry of Agriculture and Rural Affairs, Shanghai 201306, China
 2021,
Vol. 30
2021,
Vol. 30


