2. 上海海洋大学 水产种质资源发掘与利用教育部重点实验室, 上海 201306;
3. 上海海洋大学 水产科学国家级实验教学示范中心, 上海 201306;
4. 密歇根州立大学 渔业与野生动物系, 密歇根 东兰辛 48824
糖原是一种高分子量多糖物质,在动物组织中作为葡萄糖的储藏库用于能量代谢,对于生命活动的维持具有重要作用[1]。糖原磷酸化酶(glycogen phosphorylase, GP)是糖原分解过程中的限速酶,主要作用是将糖原水解成1-磷酸葡萄糖,继而转化成6-磷酸葡萄糖,再经过糖酵解过程转化为丙酮酸,在有氧情况下,丙酮酸进入柠檬酸循环,产生大量三磷酸腺苷(adenosine triphosphate, ATP)[2],为机体提供大量能量。糖原磷酸化酶是一种大分子蛋白聚合体,其活化状态时的结构为两个相同的亚单位形成的二聚体,并具备多个功能结构域,包括催化域、糖原结合域、变构域和丝氨酸残基磷酸化域。在人体,糖原磷酸化酶具有3种亚型,即肝型(liver-type glycogen phosphorylase, PYGL)、脑型(brain-type glycogen phosphorylase, PYGB)和肌型糖原磷酸化酶(muscle-type glycogen phosphorylase, PYGM)[3]。
糖原贮积症(glycogen storage disease, GSD)是一组遗传性糖原代谢障碍引起的疾病[4],主要涉及心脏、肝脏、肌肉和脑中糖原代谢异常[5]。糖原贮积症有很多类型,其中Ⅰ、Ⅲ、Ⅵ和Ⅸ型以肝脏病变为主,Ⅱ、Ⅴ和Ⅶ型以肌肉组织受损为主[4-5]。GSDⅤ型又名McArdle氏病,该病的致病原因是肌型糖元磷酸化酶PYGM缺乏导致糖原大量累积在肌纤维内[5-6],该病的主要临床特征为运动耐受不良[7],在严重的病例中,可伴有横纹肌溶解、肌红蛋白尿和致命的肾功能衰竭的表型[8]。患有McArdle氏病的老年患者由于缺乏锻炼,也更容易受到Ⅱ型糖尿病和心脏病等继发性病症的影响[9]。
动物疾病模型的构建对于在生理学、病理学和治疗学方面开展疾病研究具有重要意义。目前,NOGALES-GADEA等[10]通过将携带突变位点的Pygm(R50X)敲入小鼠体内,造成小鼠体内糖原磷酸化酶遭到破坏,成功构建了McArdle氏病小鼠模型;纯合突变体小鼠活检实验显示肌肉中有大量糖原累积,行为学实验测试显示,纯合突变体小鼠运动能力很差,体内出现高尿酸血症,这些特征与人类McArdle氏病患者特征一致[10]。
斑马鱼(Danio rerio)因其具有生殖周期短、产卵量大、胚胎透明、养殖成本低等优点, 已经成为现代遗传学和发育生物学研究的重要模式动物。近年来,斑马鱼也被广泛应用于人类疾病模型构建和药物筛选平台,已成为继小鼠之后的另一种重要的动物疾病模型[11-12]。McArdle氏病致病基因PYGM在斑马鱼中有2个同源基因,分别是pygma和pygmb,其编码的蛋白与人类PYGM蛋白相似性高达90%以上,因此斑马鱼McArdle氏病模型可能与小鼠疾病模型的表型相似,但又可能存在差别。利用CRISPR/Cas9技术成功构建了斑马鱼pygma和pygmb纯合突变体,表型分析证实突变体具有糖原贮积症肌肉糖原累积的典型特征。该疾病模型的建立,为糖原贮积症的发病进程、详细机制和药物筛选等治疗策略提供基础数据。
1 材料与方法 1.1 材料与试剂野生型斑马鱼AB品系购自中国科学院生物化学与细胞生物学研究所斑马鱼平台。gRNA质粒由北京大学分子医学研究所熊敬维教授惠赠;DH5α菌株购自天根生化科技(北京)有限公司;gRNA体外转录试剂盒MAXIscript ® T7 in Vitro Transcription Kit(AM1314)购自Ambion公司;Cas9蛋白NLS-Cas9-NLS(Z03389-100)购自金斯瑞生物科技公司;DNA Clean & Concentrator ®-5(D4014)购自Zymo Research公司;Phusion ® High-Fidelity PCR Master Mix(M0531L)和T7 Endonuclease Ⅰ(M0302L)购自NEB公司;2×EasyTaq ® PCR SuperMix for PAGE(AS112)购自北京全式金生物技术有限公司;TRIzol(15596-018)购自Invitrogen公司;反转录试剂盒PrimeScriptTM RT reagent Kit with gDNA Eraser(RR047A)购自TaKaRa公司;LightCycler ® 480 SYBR Green Ⅰ Master(Roche, 04887352001)和LightCycler ® 480Ⅱ(Roche)实时荧光定量PCR系统均购自罗氏公司;糖原PAS染色液试剂盒(G1281)购自北京索莱宝生物科技有限公司。
1.2 方法 1.2.1 基因敲除靶点设计及gRNA合成从Ensembl数据库(http://asia.ensembl.org/index.html)获得斑马鱼pygma和pygmb基因组序列。pygma基因包含20个外显子,pygmb基因包含21个外显子。靶点位置选择尽量位于靠近起始密码子的外显子序列,通过zifit网站(http://zifit.partners.org/ZiFiT/)查找靶点,靶点序列通过NCBI网站与斑马鱼基因组序列进行BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对,确定靶点的特异性。
利用T7启动子合成靶点gRNA。分别利用pygma上游引物:TAATACGACTCACTATAGGACTGGCTGCCTACGGTTAGTTTTAGAGCTAGAAATAGC,pygmb上游引物:TAATACGACTCACTATAGGTCGCGGACGGTGTGCGCCGTTTTAGAGCTAGAAATAG C(下划线碱基为靶点序列)和下游通用引物:5′-AAAAAAAGCACCGACTCGGTGCCAC-3′扩增gRNA质粒,然后利用PCR产物作为模板,体外转录合成gRNA。
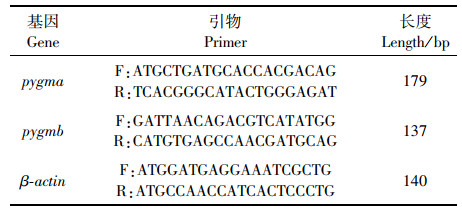
1.2.2 显微注射制备基因敲除F0嵌合体首先,进行亲本纯合子筛选。选取生长良好的性成熟野生型AB品系斑马鱼雌雄各10尾,剪取尾鳍碱裂解法提取基因组DNA,利用靶点上下游引物(表 1)扩增获得PCR产物,并送至生工生物有限公司进行测序。如果所有个体PCR产物序列一致,则认为该亲本为纯合子,其胚胎可以用于显微注射。

|
表 1 靶点及检测引物序列信息 Tab.1 Information of target sites and detecting primer sequences |
其次,对受精卵进行显微注射并检测基因敲除效率。注射液Cas9蛋白质量浓度为800 ng/μL, gRNA质量浓度为100 ng/μL, 注射剂量为1 nL。将已注射和阴性对照胚胎置于28.5 ℃培养箱中进行孵化,并定期换水。取注射后2 dpf(days post fertilization)的胚胎(5枚胚胎/管,3管)进行敲除效率检测。向每管样品中加入50 μL 50 mmol/L NaOH,95 ℃加热10 min,涡旋振荡;重复加热振荡;加入5 μL pH 8.0,1 mol/L Tris-HCl,10 000 r/min,离心5 min,上清即为基因组。根据表 1所列引物进行PCR扩增。PCR反应体系包括2.0 μL DNA模板,1.0 μL上游引物,1.0 μL下游引物,8.5 μL ddH2O,12.5 μL 2×EasyTaq ® PCR SuperMix for PAGE。PCR反应程序:94 ℃ 3 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 40 s,共33个循环;72 ℃ 5 min。
利用T7E1酶切法和测序法进行基因敲除效率检测。将PCR产物进行T7E1酶切检测,计算基因编辑效率[13],具体公式如下:
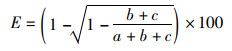 (1)
(1)
式中:E为基因编辑效率,%;a为未切开电泳条带的灰度值,b和c分别为切开电泳条带的灰度值。
将酶切成功的PCR产物测序,若敲除成功,靶点附近及其以后出现套峰。将敲除成功的F0嵌合体胚胎饲养至性成熟。
1.2.3 纯合突变体的筛选及表型分析将性成熟的F0嵌合体与显微注射时留作对照的野生型成鱼外交获得F1,然后进行基因突变类型的筛选,F1包含多种突变类型。待F1长大后,剪取尾鳍提取基因组,进行PCR扩增和产物T7E1酶切。酶切成功的PCR产物纯化后进行TA克隆,每尾F1挑选5个单克隆进行测序,确定基因突变类型。
将相同突变类型的F1杂合子成鱼分别内交,获得不同突变类型的F2。对F2成鱼剪尾鳍提取基因组,同基因编辑效率检测方法,进行PCR扩增和产物T7E1酶切检测。PCR产物切开对应的个体为杂合子;PCR产物未切开对应的个体为WT或突变体纯合子,通过PCR产物测序及序列比对,确定F2为WT或纯合突变体。F2纯合突变体可以继续内交获得F3,观察表型。
1.2.4 pygma和pygmb纯合突变体糖原累积情况检测通过PAS染色法(periodic acid Schiff stain)检测糖原在组织中的累积情况。分别收集野生型、pygma-/-和pygmb-/-成鱼的心脏和肌肉组织,用体积分数为10%的甲醛固定液固定24 h后进行常规石蜡切片。PAS染色步骤按照试剂盒使用说明操作。染色封片后,在光学显微镜下观察并拍照。
1.2.5 pygma和pygmb在不同发育时期和成鱼不同组织的表达对pygma和pygmb基因在早期不同发育时期和成鱼不同组织的表达水平进行检测。选取斑马鱼15个早期发育时间点[1细胞期(1-cell), 4细胞期(4-cell), 16细胞期(16-cell), 1 k细胞期(1 k-cell), 球形期(sphere stage), 胚盾期(shield stage), 尾芽期(bud stage),12 hpf(hours post-fertilization, hpf), 24 hpf, 36 hpf, 48 hpf, 60 hpf, 72 hpf, 100 hpf和125 hpf],每个时期的胚胎取3组生物学重复。选取野生斑马鱼成鱼的12个组织,分别为心脏(heart)、肌肉(muscle)、皮肤(skin)、脾脏(slpeen)、脑(brain)、鳃(gill)、肠(intestine)、肾脏(kidney)、卵巢(ovary)、精巢(testis)、眼睛(eye)和肝脏(liver),每个组织取3个生物学重复。所有样品采用TRIzol法提取总RNA。
使用PrimeScriptTM RT reagent Kit with gDNA Eraser试剂盒进行总RNA反转录,获得cDNA;使用NCBI在线程序(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)设计pygma和pygmb qRT-PCR引物(表 2),使用罗氏SYBR/Green和LightCycler ® 480Ⅱ实时荧光定量PCR系统进行荧光定量检测。以斑马鱼β-actin基因作为内参对照,使用2-ΔΔCt法计算基因相对表达量,使用软件Graphpad prism 5绘制柱状图。
|
表 2 qRT-PCR引物序列 Tab.2 Primer sequences for qRT-PCR |
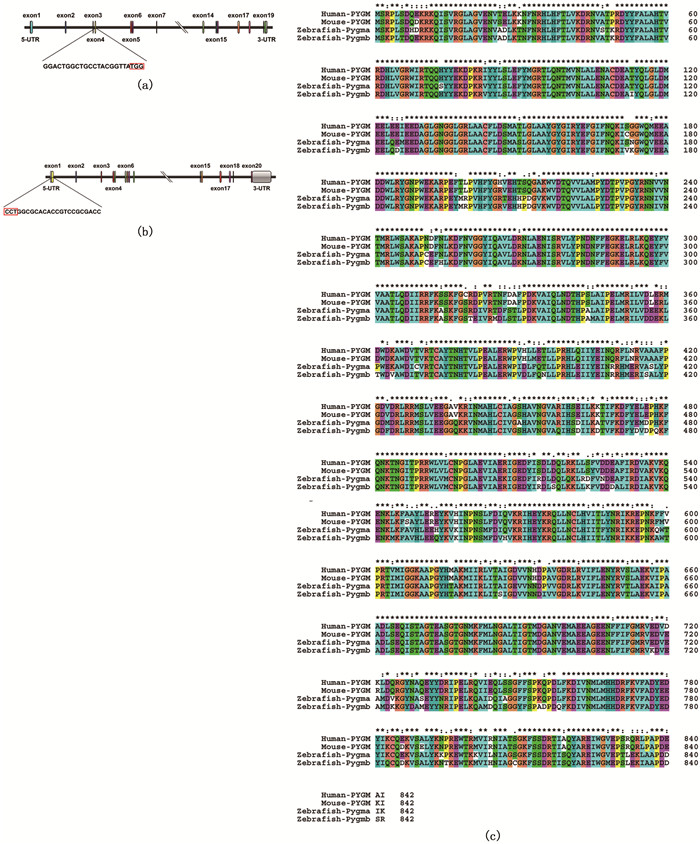
在pygma基因的第4个外显子上设计1个正向靶点(图 1a),在pygmb基因的第1个外显子上设计1个反向靶点(图 1b),并将斑马鱼pygma和pygmb基因编码蛋白序列与小鼠pygm以及人类PYGM基因编码蛋白序列进行相似性分析。结果显示,PYGM蛋白在人类、小鼠和斑马鱼中高度保守(图 1c),斑马鱼Pygma与小鼠和人类PYGM蛋白的序列相似性都是93%,斑马鱼Pygmb与小鼠和人类PYGM蛋白相似性都是92%。
|
(a) pygma基因结构及其敲除靶点的序列和位置;(b)pygmb基因结构及其敲除靶点的序列和位置;(c)人类、小鼠和斑马鱼之间PYGM蛋白序列相似性比对。红色方框表示Cas蛋白识别的PAM(protospacer adjacent motif)区。 (a) The gene structure of pygma and the sequence and location of the target site; (b) The gene structure of pygmb and the sequence and location of the target site; (c)The alignment of amino acid sequences of PYGM between human, mouse, and zebrafish.The red box indicates the Cas9 protein recognized PAM region. 图 1 斑马鱼pygma和pygmb基因敲除靶点示意图及编码蛋白序列相似性分析 Fig. 1 Schematic diagram of target sites of pygma and pygmb and the sequence alignment of PYGM between human, mouse and zebrafish |
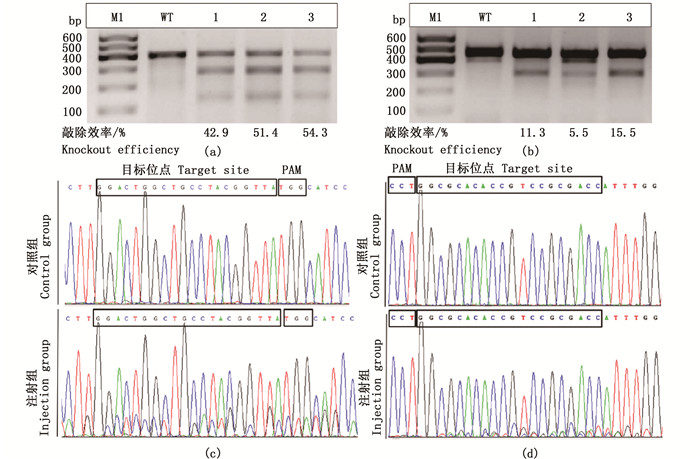
对显微注射的5枚混合胚胎F0嵌合体基因敲除效率进行了T7E1酶切检测。根据酶切片段条带的灰度值,利用公式计算得出pygma基因3组平行实验的敲除效率分别为42.9%、51.4%和54.3%(图 2a);pygmb基因3组平行实验的敲除效率分别为11.3%、5.5%和15.5%(图 2b)。将酶切成功对应的PCR产物进行测序,发现在靶点附近及其后出现套峰现象,表明基因编辑成功(图 2c, d)。
|
(a) T7E1酶切法检测pygma基因编辑效率;(b) T7E1酶切法检测pygmb基因编辑效率;(c) Sanger测序法检测pygma基因编辑成功;(d) Sanger测序法检测pygmb基因编辑成功; 方框中的碱基分别代表靶点和Cas蛋白识别的位点。 (a) The gene editing efficiency of pygma detected by T7E1 enzyme digestion; (b) The gene editing efficiency of pygmb detected by T7E1 enzyme digestion; (c) The gene editing of pygma detected by Sanger sequencing; (d) The gene editing of pygmb detected by Sanger sequencing; The bases in the black boxes indicate the target sites and PAM sites, respectively. 图 2 斑马鱼pygma和pygmb基因敲除T7E1酶切检测和Sanger测序法检测 Fig. 2 Gene editing of pygma and pygmb was detected with T7E1 digestion and Sanger sequencing in zebrafish |
突变可遗传的F0嵌合体与野生型外交获得的后代F1生长至合适大小,通过T7E1酶切检测方法筛选杂合子F1。在52尾pygma F1中筛选到31尾杂合子(图 3a),在54尾pygmb F1中筛选到22尾杂合子(图 3b)。F1杂合子基因组PCR产物通过TA克隆测序检测,pygma筛选出2种有效突变类型(图 3c),pygmb也筛选出2种有效突变类型(图 3d)。
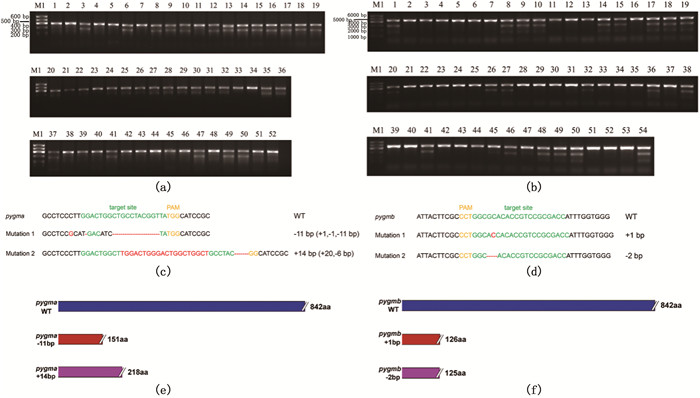
|
(a) T7E1酶切法筛选pygma的F1杂合子;(b) T7E1酶切法筛选pygmb的F1杂合子;(c) Sanger法测序确定pygma的F1杂合子突变类型;(d)Sanger法测序确定pygmb的F1杂合子突变类型;(e)pygma突变基因编码截短蛋白;(f)pygmb突变基因编码截短蛋白。 (a) T7E1 screening of pygma F1 heterozygotes; (b) T7E1 screening of pygmb F1 heterozygotes; (c) The mutation type of pygma detected by Sanger sequencing; (d) The mutation type of pygmb detected by Sanger sequencing; (e) The truncated proteins of two mutation types of pygma gene; (f) The truncated proteins of two mutation types of pygmb gene. 图 3 斑马鱼pygma和pygmb突变体F1筛选及突变类型确定 Fig. 3 Screening of heterozygous F1 of pygma and pygmb and the representation of mutation types and truncated proteins |
分别将pygma两种突变类型(-11 bp和+14 bp)和pygmb两种突变类型(+1 bp和-2 bp)F1内交获得F2。待F2长至2个月大小,对其剪尾进行基因型鉴定,并对F2个体基因型和数目进行统计分析,具体基因型及其数目见表 3。
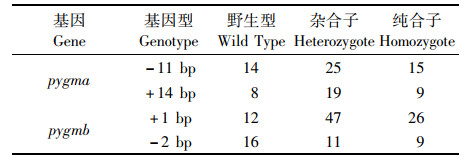
|
表 3 pygma和pygmb不同基因型F2个体数目统计 Tab.3 Statistics on the number of different genotypes of pygma and pygmb F2 |
pygma和pygmb基因最长转录本编码的氨基酸数目为842。pygma-/-的两种突变类型:-11 bp和+14 bp分别编码的氨基酸长度为151和218。pygmb-/-的两种突变类型:+1 bp和-2 bp分别编码的氨基酸长度为126和125。
2.4 纯合子F2及其后代F3表型分析与野生型相比,pygma和pygmb两种突变类型的F2纯合子在生长发育过程中未发现异常表型。pygma-/-和pygmb-/- F2自交后代F3发育正常无异样。
利用PAS糖原染色法对野生型、pygma-/-和pygmb-/-斑马鱼心脏和肌肉组织中糖原含量进行检测,结果显示:与野生型相比较,pygma-/-和pygmb-/-的心脏和肌肉组织中出现明显的糖原累积(图版)。
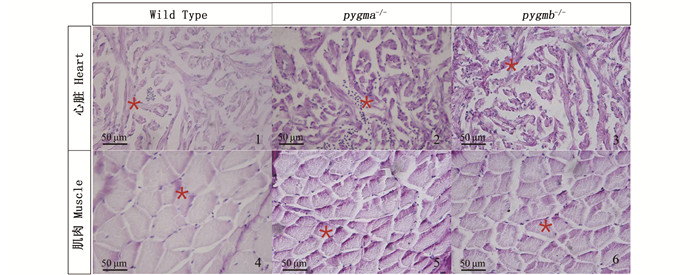
|
*表示组织中累积的糖原。 * indicates the accumulation of glycogen. 图版 pygma-/-和pygmb-/-骨骼肌和心肌细胞内糖原累积情况 Plate Glycogen accumulation in myocytes and myocardia of pygma-/- and pygmb-/- |
对野生型斑马鱼15个早期发育时期和成鱼12个组织pygma和pygmb基因表达进行定量分析。早期发育时期基因表达分析结果显示:pygma和pygmb斑马鱼早期发育阶段表达模式相似,pygma在24 hpf前几乎不表达,pygmb表达稍早一些,在胚盾期(shield stage)开始表达;从24 hpf到125 hpf,两基因表达量总体呈上升趋势(图 4a, b), 且pygmb的上升幅度大于pygma的上升幅度,pygmb的表达量高于pygma。
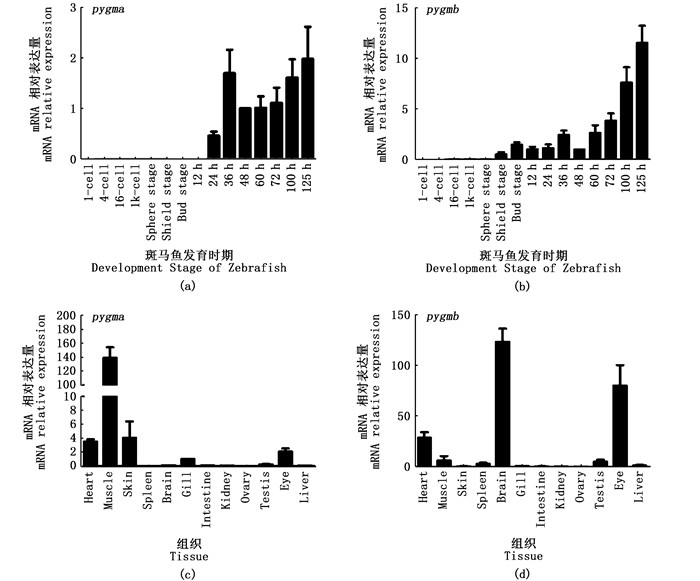
|
(a, b)pygma和pygmb在野生型斑马鱼15个早期发育时期的表达模式;(c, d) pygma和pygmb在野生斑马鱼成鱼12个组织中的表达模式。 (a, b) The expression pattern of pygma and pygmb in early embryos and larvae at 15 development stages; (c, d) The expression pattern of pygma and pygmb in 12 different adult tissues. 图 4 pygma和pygmb基因在野生型斑马鱼胚胎和幼鱼时期表达谱和成鱼组织表达谱 Fig. 4 Expression pattern of pygma and pygmb in embryos and larvae at early different stages and in different adult tissues of WT zebrafish |
斑马鱼成鱼组织基因表达分析结果显示,在检测的12个组织中,pygma和pygmb只在少数几个组织中有明显表达。pygma在斑马鱼肌肉组织中表达最高,其次为心脏和皮肤组织,在眼睛中也有表达(图 4c);pygmb在心脏、脑、眼睛这3个组织中高表达,在脑组织中表达含量最高;在肌肉组织中也有表达,但表达量相对较低(图 4d)。
3 讨论糖原贮积症是一组单基因遗传病,基因突变导致其编码的糖原转化相关酶缺陷,代谢底物堆积、供能障碍继而引发多器官受累的代谢性疾病。1928年首次报道[14],发病率在1/20 000~1/43 000[15];根据缺陷酶的种类不同,目前发现的糖原贮积症类型已经超过16型[16]。以骨骼肌损害受累为主的称为肌糖原贮积症,主要包括Ⅱ型、Ⅲ型、Ⅴ型和Ⅶ型等,其中以Ⅴ型最为常见。1951年苏格兰医生Brian McArdle首次报道糖原贮积症Ⅴ型疾病,因此该病也常称为McArdle氏病,该病是肌型磷酸化酶PYGM基因突变导致的[17]。目前,全世界范围内有大量关于该基因遗传突变的报道;迄今为止已报道147个致病性突变和39个多态性位点;外显子1和17是PYGM基因的突变热点区域,50%的突变是错义突变;其中c.148C>T(通常称为p.R50X)是大多数研究人群中最常见的突变,占报道突变人群的比例高达31%~81%[18]。
动物疾病模型对于疾病在生理学、病理学和治疗学方面开展研究具有重要意义。在家畜群体中发现自然发生的McArdle病动物模型,1995年在夏洛莱牛中发现了该病的第一个动物模型[19],此后又在美利奴绵羊[20]中报道了该病的动物模型。NOGALES-GADEA等[10, 17]将携带McArdle氏病最常见突变位点的Pygm(R50X)敲入小鼠体内,成功地构建了第一个人工McArdle氏病动物模型。尽管自然发生的牛羊疾病模型都很好地模拟了人类疾病的表型,但由于其体形较大,很多实验操作困难;人工构建的小鼠模型不仅模拟了人类疾病的表型,也较好地解决了实验操作的困难。
斑马鱼因其具有生殖周期短、产卵量大、胚胎透明、养殖成本低等优点, 已经成为现代遗传学和发育生物学研究的重要模式动物。近年来,斑马鱼也被广泛应用于人类疾病模型构建和药物筛选。CRISPR/Cas9基因编辑系统目前可用于多种基因组的编辑,已在实验动物中构建出大量疾病模型,包括癌症模型[21]、神经疾病模型[22]、心血管疾病模型[23]和传染性疾病模型等[24]。目前,McArdle氏病还没有满意的治疗方法,因此利用斑马鱼疾病模型作为一个良好的疾病模型和药物筛选模型,为该病的治疗提供新的策略。
McArdle氏病致病基因PYGM在斑马鱼中有两个同源基因,分别是pygma和pygmb,其编码的蛋白与人类PYGM蛋白相似性分别是93%和92%(图 1c),因此,斑马鱼pygma和pygmb突变体能很好地模拟人类PYGM基因突变导致的McArdle疾病。笔者分别在pygma和pygmb的第4个和第1个外显子上设计靶点(图 1a, b),利用CRISPR/Cas9技术成功敲除了斑马鱼pygma和pygmb基因。在获得的pygma(-11bp)-/-和pygma(+14bp)-/-两种纯合突变体中,突变基因编码的截短蛋白长度分别为151和218个氨基酸(图 3e);在pygmb(+1bp)-/-和pygmb(-2bp)-/-纯合突变体中,突变基因编码的截短蛋白长度分别为126和125个氨基酸(图 3f);与NOGALES-GADEA等构建的Pygm(R50X)基因敲入小鼠模型截短蛋白长度50个氨基酸相比,笔者构建的斑马鱼突变体中截短蛋白长度更长一些,但是截短蛋白也完全缺失了蛋白的结构功能域。
PAS糖原染色结果显示,pygma-/-和pygmb-/-纯合突变体心脏和肌肉组织糖原累积明显增多,这与McArdle氏病患者肌肉活检典型症状相一致,与小鼠模型的PAS染色体结果也一致[10]。编码肌型糖原磷酸化酶PYGM基因在人和小鼠中只有1个拷贝,在斑马鱼中有2个拷贝pygma和pygmb,且两者都在心脏和肌肉中高表达(图 4c, d);在小鼠模型中,杂合突变体未出现McArdle氏病肌肉糖原累积症状[10],而斑马鱼中不论是pygma-/-纯合突变体,还是pygmb-/-纯合突变体在心脏和肌肉中都出现糖原累积的症状,可能的原因是一个基因突变导致的代谢异常并不能由另一个基因通过调控代谢进行补偿。下一步,将检测斑马鱼单基因杂合子和双突变体心脏和肌肉糖原的累积情况。
除肌肉糖原累积的典型症状外,McArdle氏病的病患和小鼠模型都具有早期疲劳、肌肉僵硬和挛缩等运动不耐受现象,有时伴有明显的肌肉损伤或横纹肌溶解导致肌间蛋白如肌酸激酶(creatine kinase, CK)流出至血液[25]。下一步,将对斑马鱼模型进行行为学研究和蛋白酶检测,以进一步确定斑马鱼模型与人类患者和小鼠模型的异同。
| [1] |
张学庆. 运动后补充肉碱可提升骨骼肌糖原合成代谢[J]. 基因组学与应用生物学, 2019, 38(1): 342-348. ZHANG X Q. Carnitine supplementation could promote skeletal muscle glycogenanabolism after exercise[J]. Genomics and Applied Biology, 2019, 38(1): 342-348. |
| [2] |
吴剑, 胡扬, 李燕春, 等. PYGM基因SNP及单体型多态性与女子长跑运动员生理表型的关联性[J]. 首都体育学院学报, 2014, 26(4): 376-379. WU J, HU Y, LI Y C, et al. Association study of PYGM gene and haplotypes with women endurance athletes physiological phenotypes[J]. Journal of Capital University of Physical Education and Sports, 2014, 26(4): 376-379. DOI:10.3969/j.issn.1009-783X.2014.04.019 |
| [3] |
赵勇, 徐晓, 董光龙. 糖原磷酸化酶与肿瘤的研究进展[J]. 中华临床医师杂志(电子版), 2017, 11(6): 1020-1023. ZHAO Y, XU X, DONG G L. Research progress of glycogen phosphorylase in cancer[J]. Chinese Journal of Clinicians(Electronic Edition), 2017, 11(6): 1020-1023. DOI:10.3877/cma.j.issn.1674-0785.2017.06.028 |
| [4] |
王利群, 李焰生, 黄坚. 糖原贮积症一例报告[J]. 第二军医大学学报, 2003, 24(1): 43. WANG L Q, LI Y S, HUANG J. Glycogenosis type V:a case report[J]. Academic Journal of Second Military Medical University, 2003, 24(1): 43. DOI:10.3321/j.issn:0258-879X.2003.01.040 |
| [5] |
曾召琼, 易帆. 糖原累积病研究进展[J]. 检验医学与临床, 2018, 15(22): 3458-3461. ZENG Z Q, YI F. Progress in research on glycogen accumulation disease[J]. Laboratory Medicine and Clinic, 2018, 15(22): 3458-3461. DOI:10.3969/j.issn.1672-9455.2018.22.043 |
| [6] |
DELMONT E, SACCONI S, BERGE-LEFRANC J L, et al. McArdle disease (gycogenosis type V):analysis of clinical, biological and genetic features of five French patients[J]. Revue Neurologique, 2008, 164(11): 912-916. DOI:10.1016/j.neurol.2008.03.020 |
| [7] |
NOGALES-GADEA G, SANTALLA A, BALLESTER-LOPEZ A, et al. Exercise and preexercise nutrition as treatment for McArdle disease[J]. Medicine and Science in Sports and Exercise, 2016, 48(4): 673-679. DOI:10.1249/MSS.0000000000000812 |
| [8] |
FIUZA-LUCES C, NOGALES-GADEA G, GARCíA-CONSUEGRA I, et al. Muscle signaling in exercise intolerance:insights from the McArdle mouse model[J]. Medicine and Science in Sports and Exercise, 2016, 48(8): 1448-1458. DOI:10.1249/MSS.0000000000000931 |
| [9] |
MUNGUÍA-IZQUIERDO D, SANTALLA A, LUCIA A. Cardiorespiratory fitness, physical activity, and quality of life in patients with McArdle disease[J]. Medicine and Science in Sports and Exercise, 2015, 47(4): 799-808. DOI:10.1249/MSS.0000000000000458 |
| [10] |
NOGALES-GADEA G, PINÓS T, LUCIA A, et al. Knock-in mice for the R50X mutation in the PYGM gene present with McArdle disease[J]. Brain, 2012, 135(7): 2048-2057. DOI:10.1093/brain/aws141 |
| [11] |
李辉辉, 黄萍, 董巍, 等. 斑马鱼研究走向生物医学[J]. 遗传, 2013, 35(4): 410-420. LI H H, HUANG P, DONG W, et al. A brief history of zebrafish research:toward biomedicine[J]. Hereditas (Beijing), 2013, 35(4): 410-420. DOI:10.3760/cma.j.issn.1003-9406.2013.04.007 |
| [12] |
BOOTORABI F, MANOUCHEHRI H, CHANGIZI R, et al. Zebrafish as a model organism for the development of drugs for skin cancer[J]. International Journal of Molecular Sciences, 2017, 18(7): 1550. DOI:10.3390/ijms18071550 |
| [13] |
ZETSCHE B, GOOTENBERG J S, ABUDAYYEH O O, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [14] |
VAN CREVELD S. The clinical course of glycogen disease[J]. Canadian Medical Association Journal, 1963, 88(1): 1-15. |
| [15] |
ÖZEN H. Glycogen storage diseases:new perspectives[J]. World Journal of Gastroenterology, 2007, 13(18): 2541-2553. |
| [16] |
KANUNGO S, WELLS K, TRIBETT T, et al. Glycogen metabolism and glycogen storage disorders[J]. Annals of Translational Medicine, 2018, 6(24): 474. DOI:10.21037/atm.2018.10.59 |
| [17] |
NOGALES-GADEA G, SANTALLA A, BRULL A, et al. The pathogenomics of McArdle disease-genes, enzymes, models, and therapeutic implications[J]. Journal of Inherited Metabolic Disease, 2015, 38(2): 221-230. DOI:10.1007/s10545-014-9743-2 |
| [18] |
NOGALES-GADEA G, BRULL A, SANTALLA A, et al. McArdle disease:update of reported mutations and polymorphisms in the PYGM gene[J]. Human Mutation, 2015, 36(7): 669-678. DOI:10.1002/humu.22806 |
| [19] |
ANGELOS S, VALBERG S J, SMITH B P, et al. Myophosphorylase deficiency associated with rhabdomyolysis and exercise intolerance in 6 related Charolais cattle[J]. Muscle & Nerve, 1995, 18(7): 736-740. |
| [20] |
TAN P, ALLEN J G, WILTON S D, et al. A splice-site mutation causing ovine McArdle's disease[J]. Neuromuscular Disorders, 1997, 7(5): 336-342. DOI:10.1016/S0960-8966(97)00062-X |
| [21] |
TORRES-RUIZ R, RODRIGUEZ-PERALES S. CRISPR-Cas9:a revolutionary tool for cancer modelling[J]. International Journal of Molecular Sciences, 2015, 16(9): 22151-22168. DOI:10.3390/ijms160922151 |
| [22] |
ZHU P, WU F R, MOSENSON J, et al. CRISPR/Cas9-mediated genome editing corrects dystrophin mutation in skeletal muscle stem cells in a mouse model of muscle dystrophy[J]. Molecular Therapy-Nucleic Acids, 2017, 7: 31-41. DOI:10.1016/j.omtn.2017.02.007 |
| [23] |
CHADWICK A C, MUSUNURU K. Treatment of dyslipidemia using CRISPR/Cas9 genome editing[J]. Current Atherosclerosis Reports, 2017, 19(7): 32. DOI:10.1007/s11883-017-0668-8 |
| [24] |
刘赛宝, 李亚芳, 王辉, 等. 利用CRISPR/Cas9技术构建流感病毒高产细胞系MDCK-Tpl2-/-[J]. 中国生物工程杂志, 2019, 39(1): 46-54. LIU S B, LI Y F, WANG H, et al. Construction of influenza virus high-producing cell line MDCK-Tpl2-/- with CRISPR/Cas9[J]. China Biotechnology, 2019, 39(1): 46-54. |
| [25] |
BRULL A, DE LUNA N, BLANCO-GRAU A, et al. Phenotype consequences of myophosphorylase dysfunction:insights from the McArdle mouse model[J]. The Journal of Physiology, 2015, 593(12): 2693-2706. DOI:10.1113/JP270085 |
2. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China;
3. National Demonstration Center for Experimental Fisheries Science Education, Shanghai Ocean University, Shanghai 201306, China;
4. Department of Fisheries and Wildlife, Michigan State University, East Lansing 48824, USA
 2021,
Vol. 30
2021,
Vol. 30


