2. 苏州市申航生态科技发展股份有限公司农业农村部大宗淡水鱼繁育与养殖技术重点实验室, 江苏 苏州 215221;
3. 江西省水产技术推广站, 江西 南昌 330046;
4. 杭州市农业科学研究院, 浙江 杭州 310024
草鱼(Ctenopharyngodon idellus)是我国最重要的大宗淡水鱼养殖品种,目前其产值、产量在世界淡水养殖鱼类中位居第一。但近些年来由于不注意亲鱼的选育与更新,甚至进行逆向选育或近亲繁殖,其种质资源出现逐步衰退现象,亲鱼后代主要经济性状严重退化。因此,对草鱼种质资源的保护和开发工作亟待开展。微卫星(microsatellite)标记是均匀分布于真核生物基因组中的简单重复序列,由2~6个核苷酸的串联重复片段构成,由于重复单位的重复次数在个体间呈高度变异性并且数量丰富,因此微卫星标记的应用非常广泛[1]。目前微卫星标记已广泛应用于多个物种的种质评价和遗传结构的分析。袁吉贵等[2]采用10对微卫星引物对广东省化州光辉养殖场有限公司新选育的吉富罗非鱼第21代群体进行遗传多样性研究。傅建军等[3]利用微卫星标记分析了草鱼长江和珠江群体及其人工杂交后代的遗传结构,探究杂交后代与其亲本间的遗传结构及多样性,旨在选育草鱼优良品种。本研究于2017年开展了对筛选获得的长江、珠江和黑龙江三水系的草鱼优秀群体完全双列杂交试验,在初步获得9个组合的基础上,采用本课题组自主研发的多重SSR-PCR体系对9个草鱼组合群体的遗传多样性和遗传结构进行分析,协助草鱼的繁殖和遗传改良,获得优良性状的草鱼苗对促进水产养殖高产、优质、可持续健康发展具有重要意义。
1 材料与方法 1.1 实验材料与DNA提取本实验选用亲本分别来自于长江、珠江、黑龙江筛选F1群体[4-5]。本实验繁育所使用草鱼亲本均在农业农村部草鱼遗传育种中心苏州申航生态科技发展股份有限公司培育,并于2016年底进行亲本分塘管理,以上草鱼优良亲本都进行了PIT电子标记进行个体识别。2017年5月初,采用完全双列杂交方法人工配组繁殖产生了6组杂交后代和3组自交后代,分别为珠江♀×黑龙江♂杂交组合(ZH)、黑龙江♀×珠江♂杂交组合(HZ)、长江♀×珠江♂杂交组合(CZ)、长江♀×黑龙江♂杂交组合(CH)、珠江♀×长江♂杂交组合(ZC)、黑龙江♀×长江♂杂交组合(HC)、黑龙江♀×黑龙江♂自繁组合(HH)、长江♀×长江♂自繁组合(CC)及珠江♀×珠江♂自繁组合(ZZ),共9个草鱼组合群体。经过60日的饲养管理后,对本实验中的所需样品剪取草鱼尾鳍组织,每个群体各随机剪取32尾鱼鳍,共计288尾,保存于无水乙醇中,随后使用海洋动物组织基因组DNA提取试剂盒提取基因组DNA,通过1%的琼脂糖凝胶电泳检测其完整性,经NanoDrop 2000C分光光度计检测其纯度及浓度,并将DNA样品稀释成50 ng/μL,于-20 ℃保存备用。
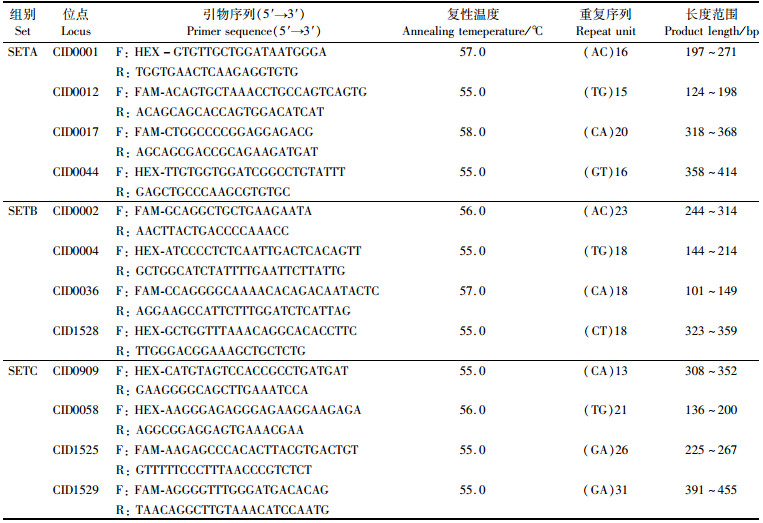
1.2 微卫星引物与PCR扩增本实验所用的12对微卫星标记来自于本课题组自行开发的多重SSR-PCR体系。引物由上海迈浦生物科技有限公司合成,分别在上游引物5'端加上FAM和HEX荧光标记(表 1)。参照傅建军等[6]反应体系PCR扩增体系为20 μL,包括10 μL Taq PCR MasterMix (2×),上下游引物(10 mmol/L)各1 μL,50 ng/μLDNA模板3 μL,补充ddH2O 5 μL。所需试剂购于天根生化科技(北京)有限公司。PCR反应程序:94 ℃预变性2 min;94 ℃变性30 s,55℃-58 ℃复性30 s(各引物反应复性温度见表),72 ℃延伸30 s,扩增35个循环;最后72 ℃延伸10 min,4 ℃保温。扩增反应在Eppendorf 5345 PCR仪上进行。反应产物通过ABI3730XL全自动DNA测序仪分析,以LIZ500为分子量内标,以期基因型读数更加准确[7]。
|
表 1 12对微卫星引物信息 Tab.1 Detailed information for 12 pairs of microsatellite primers |
采用Genemapper Version 3.5软件读取微卫星扩增产物的分子量数据,利用DataFormater软件[7],根据分子量数据确定个体各位点基因型,采用POPGENE (Version 1.32)软件包[8],计算各个群体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、近交系数(inbreeding coefficient, Fis)、群体间奈氏标准遗传距离(standard genetic distance, Dn)[9]。采用Cervus 3.0软件计算多态性信息含量(polymorphism information content,PIC)[10]。采用Mega 7软件根据群体间奈氏标准遗传距离构建UPGMA系统发育树[11]。
2 结果 2.1 微卫星位点多态性采用12对微卫星引物对9个草鱼群体共288个个体进行了遗传变异分析,共得到366个等位基因,平均每个位点得到31个等位基因。
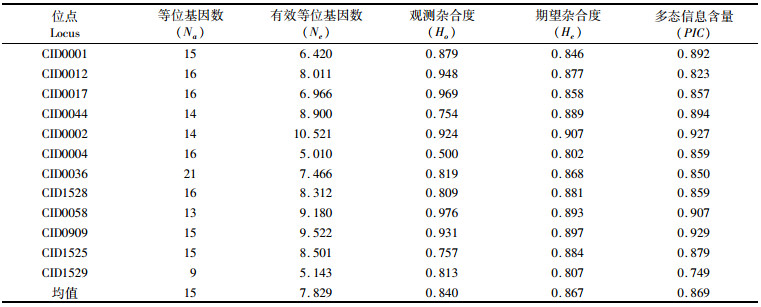
平均每个位点的Na为15,Ne为7.829,Ho为0.840,He为0.867,PIC为0.869 (表 2),均属于高度多态位点(PIC>0.5), 12个微卫星位点的Shannon多样性指数平均值为2.234 3,其中位点CID0002的Shannon多样性指数最高(I=2.4287),位点CID1529的Shannon多样性指数最低(I=1.8304)。
|
表 2 12个微卫星位点的等位基因数、有效等位基因数、杂合度及多态信息含量 Tab.2 Number of alleles (Na) and the polymorphism information content (PIC) of 12 microsatellite loci |
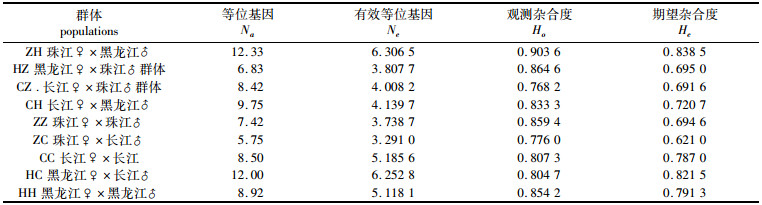
9个草鱼组合群体的遗传多样性显示,6个草鱼杂交群体各位点平均范围Na为5.75~12.33(表 3),Ne为3.291 0~6.306 5(表 4),Ho为0.768 2~0.903 6(表 5),He为0.621 0~0.838 5(表 6),3个草鱼自繁群体各位点平均范围Na为7.42~8.92,Ne为3.738 7 ~5.185 6,Ho为0.807 3~0.859 4,He为0.694 6 ~0.791 3。9个群体中,珠江♀×黑龙江♂ (ZH)组合的平均等位基因数最多(Na=12.33),珠江♀×长江♂ (ZC)群体最少(Na=5.75);珠江♀×黑龙江♂ (ZH)组合的平均有效等位基因数最多(Ne=6.306 5),珠江♀×长江♂ (ZC)群体最少(Ne=3.291 0);珠江♀×黑龙江♂ (ZH)组合的观测杂合度最高(Ho=0.903 6),CZ组合最低(Ho=0.768 2);珠江♀×黑龙江♂ (ZH)组合的期望杂合度最高(He=0.838 5),珠江♀×长江♂ (ZC)群体最低(He=0.621 0)。总体上,9个草鱼组合群体均具有较高的遗传多样性,能直观从分子角度体现各个草鱼群体间的遗传信息,遗传多样性以珠江♀×黑龙江♂ (ZH)组合最高,珠江♀×长江♂ (ZC)群体最低(表 3)。
|
表 3 草鱼9个组合的遗传多样性参数 Tab.3 The Genetic Diversity Parameters of Nine C. idella Populations |
|
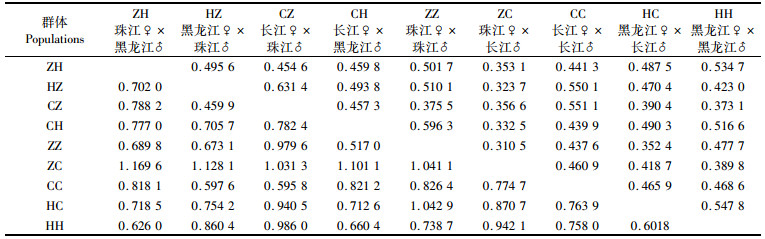
表 4 不同群体的Nei’s遗传相似性(对角线上方)和遗传距离(对角线下方) Tab.4 Nei's genetic identity (above diagonal) and genetic distance (below diagonal) |
9个群体间的遗传距离和Nei’s遗传相似性如表 4所示。利用Popgene(Version1.32)软件[12]进行分析,9个群体间的Nei’s遗传相似系数范围为0.310 5~0.631 4,珠江♀×长江♂ (ZC)与珠江♀×7珠江♂ (ZZ)的遗传相似系数最低为0.310 5,黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ)的遗传相似系数最高为0.631 4;遗传距离变化范围为0.459 9~0.969 6,遗传距离最小的是黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ),珠江♀×黑龙江♂ (ZH)与黑龙江♀×长江♂ (HC)组合的遗传距离最大。
根据奈式标准遗传距离为基础构建的9个群体间的UPGMA系统发育树(图 1),基于9个草鱼群体间奈式遗传距离,构建9个群体的UPGMA系统发育树,结果表明,黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ)首先聚类,表明两个组合亲缘关系更近,然后与长江♀×长江♂ (CC)聚为一支,其次长江♀×黑龙江♂ (CH)与珠江♀×珠江♂ (ZZ)聚类,黑龙江♀×长江♂ (HC)与黑龙江♀×黑龙江♂ (HH)聚类形成一支再与珠江♀×黑龙江♂ (ZH)聚为一类,其中9个草鱼群体中,珠江♀×长江♂ (ZC)与黑龙江♀×长江♂ (HC)组合遗传距离最远。
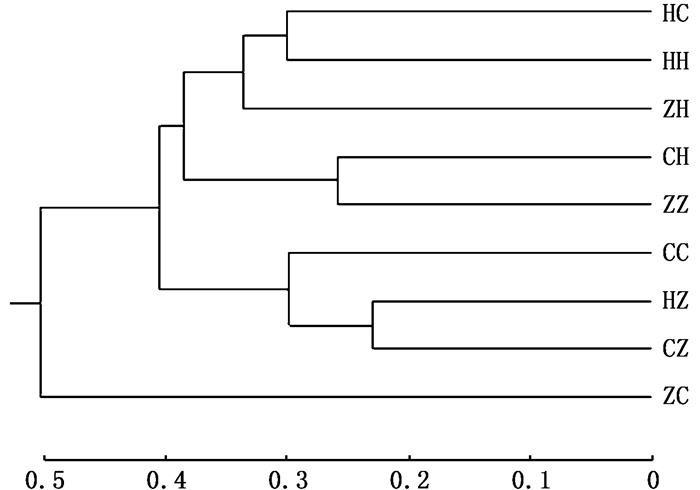
|
图 1 基于Nei’s标准遗传距离构建的9个草鱼群体UPGMA系统发育树 Fig. 1 The UPGMA phylogenetic tree of the nine C.idella populations based on Nei's genetic distance |
杂交育种手段能够显著提升水生生物的经济性状,尤其是经人工繁育的多代物种[13];通过多方位评估杂交后代的生长性状,能够对选育方案提供参考依据[14]。经杂交选育产生的后代通常更具优势[15]。双列杂交设计(diallel cross design)育种作为水产育种工作者广泛应用的近交系杂交实验方案[16],即1套品种既做父本又做母本,彼此进行所有可能的交配组合,可以产生4种后代。这种育种手段所产生的后代具备完备整齐的遗传信息,较适合分子结构遗传分析,广泛受到研究人员的青睐。对双列杂交后代遗传多样性和遗传结构的系统分析是进行杂交育种的重要手段。苏胜彦等[17]针对完全双列杂交选育手段产生的17月龄鲤的主要经济性状进行分析,并结合其遗传信息研究表明,建鲤与黄河鲤、黑龙江野鲤产生的杂交后代生长优势更显著。生长性状关联的基因差异表达在一定程度上影响各组合间的生长状况[18-19]。由此可见,鱼类的完全双列杂交具有科学的遗传育种价值,有助于解决目前我国草鱼种质退化,种质混杂等现象,有良好的应用前景,通过草鱼的完全双列杂交技术手段,以期得到更多杂种优势的良种[17]。
目前,微卫星技术已被广泛应用于水产动物的杂交研究[20-21]。多态性信息含量、等位基因数、杂合度等遗产信息通常作为群体遗传多样性的评价标准[22]。李思发等研究表明杂交后代相比于亲本更显杂种优势,且遗传信息更具多态性[23]。本实验课题中,9个群体均提取样本数量相同的个体进行微卫星分析,结果显示,9个群体平均等位基因数(Na)为5.75~12.33,平均有效等位基因数(Ne)为3.807 7~6.306 5,参考Barker对微卫星的筛选标准,平均具有4个以上的等位基因能够更好的评估遗传多样性[24]。
本实验课题中9个组合群体的平均等位基因数(Na)均高于界定标准,此结果显示,经过完全双列杂交得出的9个草鱼群体均表现显著遗传多样性,另一方面也表明本实验室自行研发的多重SSR-PCR体系内的各微卫星标记具有多态性[3]。衡量遗传变异的另一重要参数是杂合度[25],其能较直观体现群体的遗传结构变异程度。9个草鱼群体的平均观测杂合度(Ho)为0.768 2~0.903 6,9个草鱼组合群体均显示出较高的遗传多样性水平,平均期望杂合度(He)为0.838 5~0.621 0,高杂合度说明所选用的微卫星可以提供丰富的多态信息,可以作为探究遗传多样性的有力参考。6个草鱼杂交组合的平均期望杂合度普遍高于3个自繁组合,体现出一定的杂交优势,说明杂交手段能够有利的改善草鱼遗传结构变异,丰富遗传多样性[3]。12个草鱼微卫星位点的多态信息含量(PIC)分别为0.892、0.823、0.857、0.894、0.927、0.859、0.850、0.859、0.907、0.929、0.879和0.749,均表现为高度多态位点(PIC>0.5000),进一步说明遗传多样性的有效提高。综上,6个杂交草鱼群体相较于3个自繁群体在遗传多样性上体现出一定的优势。根据杂种优势的理论基础,杂交手段能够改善后代的遗传组合,丰富基因多样性。基因型与环境的有机结合能够改善杂交后代的生长性状等重要的遗传特性[26]。
根据等位基因频率得出的群体间遗传距离能够直观的体现群体间遗传关系。通常使用微卫星分析群体间的遗传距离能够得出可靠结果[3]。本实验课题使用9个草鱼组合群体的奈式遗传距离构建聚类UPMGA系统发育树,其结果显示:9个群体间的Nei’s遗传相似系数范围为0.310 5~0.631 4,珠江♀×长江♂ (ZC)与珠江♀×珠江♂ (ZZ)的遗传相似系数最低为0.310 5,黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ)的遗传相似系数最高为0.631 4;遗传距离变化范围为0.459 9~0.969 6,遗传距离最小的是黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ),珠江♀×长江♂ (ZC)与黑龙江♀×长江♂ (HC)组合的遗传距离最大。以奈式标准遗传距离为基础构建的9个群体间的UPGMA系统发育树(图 1),以9个草鱼群体间奈式遗传距离为基础,构建9个群体的UPGMA系统发育树。结果表明:黑龙江♀×珠江♂ (HZ)与长江♀×珠江♂ (CZ)首先聚类,表明以珠江水系草鱼作为母本的亲缘关系更近,然后与长江♀×长江♂ (CC)聚为一支;其次长江♀×黑龙江♂ (CH)与珠江♀×珠江♂ (ZZ)聚类,黑龙江♀×长江♂ (HC)与黑龙江♀×黑龙江♂ (HH)聚类形成一支再与珠江♀×黑龙江♂ (ZH)聚为一类,其中9个草鱼群体中,珠江♀×长江♂ (ZC)与黑龙江♀×长江♂ (HC)组合遗传距离最远。综上,草鱼长江水系、珠江水系、黑龙江水系遗传距离与地理距离存在微小差异,其原因可能是不同水系的亲本选择关系与选育世代较短,同时不同水系间的人工迁移也对研究结果有影响[27]。分化结果处于中等水平,说明此次完全双列杂交获得的9个草鱼群体仍具备进一步选育价值[7]。
本研究采用多重PCR技术对产生的6组杂交后代(珠江♀×黑龙江♂ (ZH)、黑龙江♀×珠江♂ (HZ)、长江♀×珠江♂ (CZ)、长江♀×黑龙江♂ (CH)、珠江♀×长江♂ (ZC)、黑龙江♀×长江♂ (HC))、和3组自繁后代(黑龙江♀×黑龙江♂ (HH)、长江♀×长江♂ (CC)及珠江♀×珠江♂ (ZZ))共9个草鱼群体进行了微卫星序列遗传变异分析。研究结果显示,采用完全双列杂交方案得出的9个草鱼组合群体,其中杂交群体遗传多样性与遗传分化信息普遍高于自交群体,可以说明总体的选育效果较为显著,目前阶段筛选的草鱼优秀群体具有一定意义的选育价值,不同地理来源的草鱼群体杂交能够更好地体现杂交优势,这为今后草鱼选育工作提供参考价值,为进一步草鱼的品种更新和遗传改良打下基础,同时为草鱼目前种质退化现象提供解决思路,为草鱼优良品系的选育提供理论依据。
| [1] |
KIM H S, LEE B L, WOO D K, et al. Assessment of markers for the identification of microsatellite instability phenotype in gastric neoplasms[J]. Cancer Letters, 2001, 164(1): 61-68. DOI:10.1016/S0304-3835(00)00719-9 |
| [2] |
袁吉贵, 刘丽, 陈增祥, 等. 吉富罗非鱼第二十一代选育群体微卫星标记研究[J]. 基因组学与应用生物学, 2017, 36(4): 1498-1504. YUAN J G, LIU L, CHEN Z X, et al. Microsatellite marker research of the twenty-one generation breeding population of GIFT strains of Oreochromisniloticus[J]. Genomics and Applied Biology, 2017, 36(4): 1498-1504. |
| [3] |
傅建军, 王荣泉, 刘峰, 等. 草鱼长江和珠江群体及长江♀×珠江♂杂交组合遗传差异的微卫星分析[J]. 上海海洋大学学报, 2010, 19(4): 433-439. FU J J, WANG R Q, LIU F, et al. Microsatellite analysis of genetic variation in Yangtze River, Zhujiang River, and Yangtze River ♀×Zhujiang River ♂ groups of Ctenopharyngodonidella[J]. Journal of Shanghai Ocean University, 2010, 19(4): 433-439. |
| [4] |
LI D, WANG S T, SHEN Y B, et al. A multiplex microsatellite PCR method for evaluating genetic diversity in grass carp (Ctenopharyngodonidellus)[J]. Aquaculture and Fisheries, 2018, 3(6): 238-245. DOI:10.1016/j.aaf.2018.09.001 |
| [5] |
FU JJ, SHEN Y B, XU X Y, et al. Genetic parameter estimates for growth of grass carp, Ctenopharyngodonidella, at 10 and 18 months of age[J]. Aquaculture, 2016, 450: 342-348. DOI:10.1016/j.aquaculture.2015.08.018 |
| [6] |
FU JJ, SHEN Y B, XU X Y, et al. Multiplex microsatellite PCR sets for parentage assignment of grass carp(Ctenopharyngodonidella)[J]. Aquaculture International, 2013, 21(6): 1195-1207. DOI:10.1007/s10499-013-9623-z |
| [7] |
王沈同, 沈玉帮, 孟新展, 等. 草鱼野生与选育群体遗传变异微卫星分析[J]. 水产学报, 2018, 42(8): 1273-1284. WANG S T, SHEN Y B, MENG X Z, et al. Genetic variability in wild and selected populations of Ctenopharyngodonidella using microsatellite markers[J]. Journal of Fisheries of China, 2018, 42(8): 1273-1284. |
| [8] |
MIR R R, KUMAR J, BALYAN H S, et al. A study of genetic diversity among Indian bread wheat (Triticumaestivum L.) cultivars released during last 100? years[J]. Genetic Resources andCrop Evolution, 2012, 59(5): 717-726. DOI:10.1007/s10722-011-9713-6 |
| [9] |
NEI M. Mathematical models of speciation and genetic distance[M]//KARLIN S, NEVO E. Population Genetics and Ecology. New York: Academic Press, 1976: 723-765.
|
| [10] |
李明, 王小明, 盛和林, 等. 四种鹿属动物的线粒体DNA差异和系统进化关系研究[J]. 动物学报, 1999, 45(1): 99-105. LI M, WANG X M, SHENG H L, et al. Mitochondrial DNA divergence and phylogeny of four species of deer of the genus Cervus[J]. Acta Zoologica Sinica, 1999, 45(1): 99-105. |
| [11] |
HALL B G. Building phylogenetic trees from molecular data with MEGA[J]. Molecular Biology and Evolution, 2013, 30(5): 1229-1235. DOI:10.1093/molbev/mst012 |
| [12] |
GHARGHANI A, ZAMANI Z, TALAIE A, et al. Genetic identity and relationships of Iranian apple (Malus×domestica Borkh.) cultivars and landraces, wild Malus species and representative old apple cultivars based on simple sequence repeat (SSR) marker analysis[J]. Genetic Resources andCrop Evolution, 2009, 56(6): 829-842. DOI:10.1007/s10722-008-9404-0 |
| [13] |
MULLIKEN R S. Electronic population analysis on LCAO-MO molecular wave functions. Ⅲ. Effects of hybridization on overlap and gross AO populations[J]. Journal of Chemical Physics, 1955, 23(12): 2338-2342. DOI:10.1063/1.1741876 |
| [14] |
季士治, 雷霁霖, 王伟继, 等. 双列杂交法分析2个大菱鲆养殖群体的杂交效果[J]. 中国水产科学, 2006, 13(6): 1001-1005. JI S Z, LEI J L, WANG W J, et al. Analysis of hybridization effects on complete diallel crosses in two stocks of turbot, -Scophthalmusmaximus-[J]. Journal of Fishery Sciences of China, 2006, 13(6): 1001-1005. DOI:10.3321/j.issn:1005-8737.2006.06.020 |
| [15] |
李思发, 王成辉, 刘志国, 等. 三种红鲤生长性状的杂种优势与遗传相关分析[J]. 水产学报, 2006, 30(2): 175-180. LI S F, WANG C H, LIU Z G, et al. Analysis of heterosis and genetic correlation of growth traits in three variants of red common carp[J]. Journal of Fisheries of China, 2006, 30(2): 175-180. |
| [16] |
CHRISTIE B R, SHATTUCK V I. The diallel cross: design, analysis, and use for plant breeders[M]//JANICK J. Plant Breeding Reviews.Hoboken: John Wiley & Sons, Inc., 1992.
|
| [17] |
苏胜彦, 董在杰, 袁新华, 等. 3×3完全双列杂交F1不同阶段生长特点的分析[J]. 水生生物学报, 2012, 36(4): 618-625. SU S Y, DONG Z J, YUAN X H, et al. Characters of different growth stage on the f1 progeny of 3×3 full diallel cross in common carp (Cyprinuscarpio L.)[J]. ActaHydrobiologicaSinica, 2012, 36(4): 618-625. |
| [18] |
张建社, 夏新界, 褚武英, 等. 基于异源cDNA基因芯片杂交的鳜鱼肌肉组织基因表达谱初步分析[J]. 水生生物学报, 2009, 33(1): 46-53. ZHANG J S, XIA X J, CHU W Y, et al. Gene expression profiles of the muscle tissues of the mandarin fish, Sinipercachuatsi L. with zebrafishcDNAmicroarray[J]. ActaHydrobiologicaSinica, 2009, 33(1): 46-53. |
| [19] |
CHU W Y, FU G H, CHEN J, et al. Gene expression profiling in muscle tissues of the commercially important teleost, Sinipercachuatsi L.[J]. Aquaculture International, 2010, 18(4): 667-678. DOI:10.1007/s10499-009-9289-8 |
| [20] |
POTEAUX C, GUYOMARD R, BERREBI P. Single and joint gene segregation in intraspecific hybrids of brown trout (Salmotrutta L.) lineages[J]. Aquaculture, 2000, 186(1/2): 1-12. |
| [21] |
李思发, 颜标, 蔡完其, 等. 尼罗罗非鱼与萨罗罗非鱼正反杂交后代耐盐性能的杂种优势及其与遗传的相关性的SSR分析[J]. 中国水产科学, 2008, 15(2): 189-197. LI S F, YAN B, CAI W Q, et al. Heterosis and related genetic analysis by SSR for the salt tolerance of reciplrocal hybrids between Nile tilapia (Oreochromisniloticus) and blackchin tilapia(Sarotherodonmelanotheron)[J]. Journal of Fishery Sciences of China, 2008, 15(2): 189-197. DOI:10.3321/j.issn:1005-8737.2008.02.001 |
| [22] |
BEARDMORE J A, MAIR G C, LEWIS R I. Biodiversity in aquatic systems in relation to aquaculture[J]. Aquaculture Research, 1997, 28(10): 829-839. DOI:10.1111/j.1365-2109.1997.tb01007.x |
| [23] |
陈林, 李思发, 简伟业, 等. 吉奥罗非鱼(新吉富罗非鱼♀×奥利亚罗非鱼♂)生长性能的评估[J]. 上海水产大学学报, 2008, 17(3): 257-262. CEHN L, LI S F, JIAN W Y, et al. Evaluation of growth performance of JA tilapia(NEW GIFT strain O. niloticus♀×O.aureus♂)[J]. Journal of Shanghai Fisheries University, 2008, 17(3): 257-262. |
| [24] |
WAJID A, WASIM M, YAQUB T, et al. Assessment of genetic diversity in Balochi and Rakhshani sheep breeds of Balochistan using microsatellite DNA markers[J]. The Journal of Animal & Plant Sciences, 2014, 24(5): 1348-1354. |
| [25] |
TINA K G, BHADRA R, SRINIVASAN N. PIC:protein interactions calculator[J]. Nucleic Acids Research, 2007, 35(s2): 473-476. |
| [26] |
张志伟, 曹哲明, 杨弘, 等. 草鱼野生和养殖群体间遗传变异的微卫星分析[J]. 动物学研究, 2006, 27(2): 189-196. ZHANG Z W, CAO Z M, YANG H, et al. Microsatellites analysis on genetic variation between wild and cultured populations of Ctenopharyngodonidella[J]. Zoological Research, 2006, 27(2): 189-196. DOI:10.3321/j.issn:0254-5853.2006.02.010 |
| [27] |
郑国栋, 陈杰, 蒋霞云, 等. 长江草鱼不同群体EST-SSR多态性标记的筛选及其遗传结构分析[J]. 水生生物学报, 2015, 39(5): 1003-1011. ZHENG G D, CHEN J, JIANG X Y, et al. Detection of EST-SSRS markers and genetic structure of different populations of grass carp in Yangtze river system[J]. ActaHydrobiologicaSinica, 2015, 39(5): 1003-1011. |
2. Key Laboratory of Conventional Freshwater Fish Breeding and Health Culture Technology Germplasm Resources, Suzhou Shenhang Eco-technology Development Limited Company, Suzhou 215221, Jiangsu, China;
3. Jiangxi Aquatic Technology Promotion Station, Nanchang 330046, Jiangxi, China;
4. Hangzhou Academy of Agricultural Sciences, Hangzhou 310024, Zhejiang, China
 2019,
Vol. 28
2019,
Vol. 28


