副溶血弧菌(Vibrio parahaemolyticus,Vp)是一种革兰氏阴性、无芽孢的兼性厌氧杆菌,广泛存在于海洋及河口等水域环境及鱼、虾、贝类等水生生物的体内[1]。人们生食或食用不完全煮熟的被该致病菌污染的水产品可导致食物中毒,从而引发肠胃炎、败血症等疾病[2]。目前,该菌已成为我国首要的食源性致病菌[3]。副溶血弧菌不仅是引起人类急性食物中毒的重要致病菌, 同时也是多种水生动物的条件致病菌, 可引起大黄鱼(Pseudosciaena crocea)[4]、杂色鲍(Haliotis diversicolor)[5]、中华绒螯蟹(Eriocheir sinensis)[6]、斑节对虾(Penaeus monodon)[7]、凡纳滨对虾(Litopenaeus vannamei)[8]等水生动物发病。此外,携带有特异性质粒的副溶血弧菌还能导致对虾患急性肝胰腺坏死病(acute hepatopancreas necrosis disease,AHPND) [9],且发病后死亡率高达100%。由此可见,由副溶血弧菌引起的水生动物病害往往会给水产养殖业造成巨大的经济损失,需引起足够重视。
当前,国内外学者[3, 10]针对弧菌对人体及水生动物的致病机理开展了大量的研究工作。研究表明:弧菌的致病性主要取决于弧菌与宿主间的相互作用,主要致病过程包括:粘附、侵袭、增殖以及产生毒素等;在整个过程中,溶血性毒素、尿素酶、粘附相关因子、胞外蛋白酶、Ⅲ型分泌系统等诸多致病因子发挥着重要作用[3]。然而,还不太清楚弧菌对于水生动物的分子致病机理,它们的关键毒力基因还未被确定[10],但有研究[11]证实,高毒力副溶血弧菌的特异性pVA质粒上携带 AP1或AP2基因时,可导致对虾急性肝胰腺坏死病的发生。
快速、准确地对副溶血弧菌进行细致分型及比较菌株间亲缘关系,对副溶血弧菌流行病学调查、预防和控制具有重要意义[12]。传统的血清学分型方法分辨率不高、操作复杂、耗时长,且很多菌株不能用此法分型;而从基因水平进行分型可从根本上区分菌株的差异,且结果比血清学分型方法更具可靠性[13]。随着细菌基因分型方法的快速发展,利用肠杆菌基因间共有重复序列PCR技术(enterobacterial repetitive intergenic consensus-PCR, ERIC-PCR)开展副溶血弧菌的基因分型研究已有一些报道[14],目前国内外学者针对副溶血弧菌的相关致病基因及基因分型开展相关研究[15-16],但试验菌株是从海产品和环境等分离得到的,而针对分离自低盐度淡化养殖的虾源副溶血弧菌开展相关研究则未见报道。此外,针对携带特异性pVA质粒的副溶血弧菌的研究,目前国内仅见少量报道[17]。
本研究以从上海市郊区低盐度淡化养殖的对虾体内分离到的19株副溶血弧菌为研究对象,同时设置1株标准菌株作为参考菌株,对该菌的4种致病基因tdh、trh、ORF8、tox RS/new携带情况进行检测,并针对能够引起对虾AHPND的副溶血弧菌特异性pVA质粒基因AP1、AP2进行了筛查,同时运用ERIC-PCR分子分型方法对分离株基于基因片段大小及其分子多态性开展了分型研究,旨在了解上海地区虾源副溶血弧菌相关致病基因的携带及基因分型情况,分析其潜在致病性菌株的分布,为进一步开展该菌的流行趋势预测及同源性追踪等方面研究工作提供基础数据。
1 材料与方法 1.1 菌株来源及编号2014年7月—2015年9月,从上海地区19个池塘养殖虾类样品中陆续分离获得19株副溶血性弧菌,菌株保藏编号为B01901~B01919,并按分离时间顺序分别编号为Vp1~Vp19,1株副溶血弧菌标准株ATCC17802编号Vp20,购自中国科学院微生物研究所。19株分离株中Vp1~Vp10为2014年分离,Vp11~Vp19为2015年分离,所有菌株经生化鉴定和分子生物学16S rDNA鉴定,并采用甘油冷冻保存法将所有分离株保存于-80 ℃冰箱。其中:Vp1、Vp3、Vp4分离自同一养殖单位;Vp2、Vp6、Vp7、Vp9、Vp10、Vp14分离自同一养殖单位;Vp5、Vp8、Vp13、Vp17分离自同一养殖单位;菌株中Vp1~Vp18均分离自凡纳滨对虾(Litopenaeus vannamei), Vp19分离自罗氏沼虾(Macrobrachium rosenbergii)。
1.2 主要试剂胰蛋白胨大豆琼脂培养基(TSA)、胰蛋白胨大豆肉汤培养基(TSB)购自北京陆桥技术有限责任公司;Ezup柱式细菌基因组DNA抽提试剂盒、SanPrep柱式质粒DNA小量抽提试剂盒、Agarose B、4s Red plus核酸染色剂购自生工生物工程(上海)股份有限公司;PCR Premix Taq购自宝生物工程(大连)有限公司。
1.3 菌株活化将保存于-80 ℃冰箱的菌株冻存液室温解冻后,用接种环挑取1环菌液划线接种到TSA斜面培养基上,然后置于生化培养箱中,于30 ℃培养24 h后,挑取单个菌落接种到TSB液体培养基中,置于摇床培养箱,37 ℃、180 r/min振荡培养8~12 h,获得的菌悬液用于提取细菌基因组DNA和质粒DNA。
1.4 致病基因检测方法 1.4.1 基因组和质粒DNA的提取分别取适量上述经活化的菌悬液,置于离心管内,8 000 r/min离心2 min,收集菌体,分别按照细菌基因组DNA提取试剂盒和柱式细菌质粒DNA抽提试剂盒说明步骤进行操作,提取各菌株基因组和质粒DNA,保存于-20 ℃冰箱备用。
1.4.2 引物设计与合成参照茆丹[1]的方法设计合成基因组基因tdh、trh、ORF8、tox RS/new检测引物;参照LEE等[18]的方法设计合成质粒基因 AP1、AP2检测引物,引物序列见表 1,由生工生物工程(上海)股份有限公司合成。

|
表 1 6种致病基因的引物序列 Tab.1 Primer sequences of 6 pathogenic genes |
PCR反应体系(50 μL):PCR Premix 25 μL,上、下游引物各1 μL,DNA模板2 μL,补充无菌双蒸水至50 μL,实验同时设置无菌双蒸水作为阴性对照模板。
PCR反应程序为tdh、trh基因:94 ℃预变性5 min,94 ℃变性30 s,58 ℃退火30 s,72 ℃延伸30 s,30个循环,72 ℃延伸10 min。ORF8基因:94 ℃预变性5 min,94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸60 s,30个循环,72 ℃延伸10 min。tox RS/new基因:94 ℃预变性5 min,94 ℃变性60 s,46 ℃退火60 s,72 ℃延伸2 min,30个循环,72 ℃延伸10 min。AP1、AP2基因:94 ℃预变性5 min,94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸60 s,30个循环,72 ℃延伸10 min。
1.4.4 PCR产物检测采用100 μL/L 4s Red plus核酸染色剂染色,1.0%琼脂糖凝胶电泳进行PCR产物检测,用DL2 000 DNA标准分子质量作为对照。在5 V/cm电压条件下电泳30 min后,用紫外凝胶成像系统观察电泳结果并拍照。
1.5 分离株的ERIC-PCR分型 1.5.1 引物设计与合成参照马月姣等[19]方法设计分离株分型引物,引物序列为ERIC-F:5'-ATGTAAGCTCCTGGGGA TTCAC-3',ERIC-R:5'-AAGTAAGTGACTGGGGTG AGCG-3',由生工生物工程(上海)股份有限公司合成。
1.5.2 ERIC-PCR扩增PCR扩增反应体系为25.0 μL,体系中含有2.5 μL 10×PCR反应缓冲液,1.5 μL 2.5 mmol/L Mg2+,1.5 μL 2.0 mmol/L dNTP,1.0 μL正反向引物(10 μmol/L)混合液,1.0 U Taq聚合酶以及1.0 μL模板DNA,补充无菌水至25.0 μL,同时设置无菌双蒸水作为阴性对照模板。PCR反应程序为94 ℃预变性5 min,94 ℃变性45 s,52 ℃退火1 min,72 ℃延伸5 min,35个循环,72 ℃延伸10 min,4 ℃保存。
1.5.3 ERIC-PCR产物的电泳检测采用100 μL/L 4s Red plus核酸染色剂染色,1.5%琼脂糖凝胶电泳进行PCR产物检测,用DL10 000 DNA标准分子质量作为对照。在5 V/cm电压条件下电泳40 min后,用紫外凝胶成像系统观察电泳结果并拍照。
1.5.4 ERIC-PCR图谱分析ERIC-PCR电泳图谱首先采用Quantity One(Bio-Rad,USA)软件进行扩增条带分析,识别结果记录方法:针对某一条带,软件自动识别后“有”记做“1”,“无”记做“0”,并辅助相应的人工校对;再采用NTSYSpc 2.1软件计算得到Jaccard的遗传相似性系数矩阵和遗传距离矩阵,随后利用遗传距离矩阵采用非加权配对算术平均法(unweighted pair-group method with arithmetic mean,UPGMA)对分离株进行遗传聚类分析;最后根据聚类结果进行系统进化树的构建,比较不同来源菌株间的亲缘关系。
2 结果与分析 2.1 分离株致病基因检测结果所有菌株均未检测出tdh和ORF8致病基因;仅分离株Vp3和标准株Vp20检出trh基因,其余分离株均未检出(图 1),该基因在19株分离株中携带率为5.3%;除分离株Vp5、Vp10、Vp11、Vp13和Vp19及标准株Vp20未检出tox RS/new基因外,其余14株分离株均检出tox RS/new基因(图 2),在19株分离株中基因携带率为73.7%。

|
M为DL 2 000 DNA Marker;1~19分别代表Vp1~Vp19分离株,20代表Vp20标准株,NC代表阴性对照 M mesas DL 2 000 DNA Marker; 1 to 19 represent isolated strains Vp1 to Vp19, 20 represent standard strain Vp20, NC represent negative control 图 1 Trh基因1.0%琼脂糖凝胶电泳结果 Fig. 1 Result of 1.0% agarose gel electrophoresis of trh |

|
M为DL 2 000 DNA Marker;1~19分别代表Vp1~Vp19分离株,20代表Vp20标准株,NC代表阴性对照 M mesas DL 2 000 DNA Marker; 1 to 19 represent isolated strains Vp1 to Vp19, 20 represent standard strain Vp20, NC represent negative control 图 2 Tox RS/new基因1.0%琼脂糖凝胶电泳结果 Fig. 2 Result of 1.0% agarose gel electrophoresis of tox RS/new |
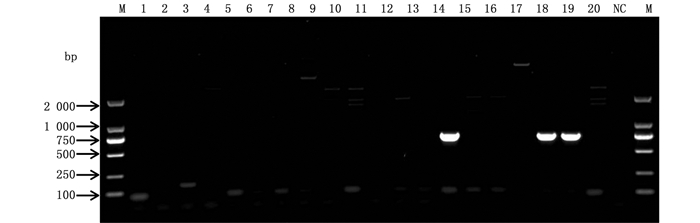
针对能够引起虾类AHPND的高毒力副溶血弧菌特异性质粒上携带 AP1或AP2致病基因的检测结果表明,Vp12、Vp14、Vp19分离株检出携带 AP1 致病基因(图 3),Vp14、Vp18、Vp19分离株检出携带 AP2 致病基因(图 4),两者的携带率均为15.8%。由图 3、图 4可知,AP1、AP2致病基因作为同一特异性质粒上的2个不同基因片段均可在Vp14、Vp19分离株中检出,而Vp12和Vp18分离株仅能检出 AP1或AP2。

|
M为DL 2 000 DNA Marker;1~19分别代表Vp1~Vp19分离株,20代表Vp20标准株,NC代表阴性对照 M mesas DL 2 000 DNA Marker; 1 to 19 represent isolated strains Vp1 to Vp19, 20 represents standard strain Vp20, NC represents negative control 图 3 AP1基因1.0%琼脂糖凝胶电泳结果 Fig. 3 Result of 1.0% agarose gel electrophoresis of AP1 |
|
M为DL 2 000 DNA Marker;1~19分别代表Vp1~Vp19分离株,20代表Vp20标准株,NC代表阴性对照 M mesas DL 2 000 DNA Marker; 1 to 19 represent isolated strains Vp1 to Vp19, 20 represents standard strain Vp20, NC represents negative control 图 4 AP2基因1.0%琼脂糖凝胶电泳结果 Fig. 4 Result of 1.0% agarose gel electrophoresis of AP2 |
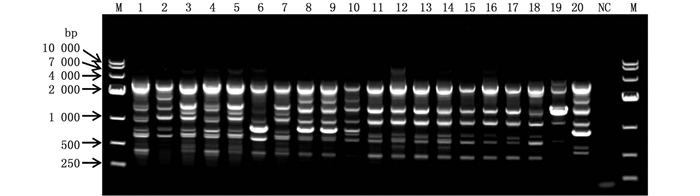
由图 5可知,运用ERIC-PCR法能扩增出较为清晰的ERIC图谱,每个分离株的ERIC条带大小都在250~4 000 bp之间,扩增条带数介于5~10之间。由图 5可知,Vp1和Vp4,Vp8和Vp9,Vp12和Vp14分离株的扩增图谱分别较为一致,Vp3、Vp5、Vp7分离株的扩增图谱也基本一致,此外Vp11~Vp18分离株的扩增图谱也较为相似。但Vp19分离株仅扩增出5个条带,明显区别于Vp11~Vp18分离株,而Vp2、Vp6、Vp10、Vp20分离株的扩增图谱亦分别与其他分离株存在一定差异。
|
M为DL 10 000 DNA Marker;1~19分别代表Vp1~Vp19分离株,20代表标准株Vp20,NC代表阴性对照 M mesas DL 10 000 DNA Marker, 1 to 19 represent isolated strains Vp1 to Vp19, 20 represents standard strain Vp20, NC represents negative control 图 5 20株副溶血弧菌的ERIC-PCR电泳图谱 Fig. 5 Results of ERIC-PCR electrophoresis of 20 V.parahaemolyticus strains |
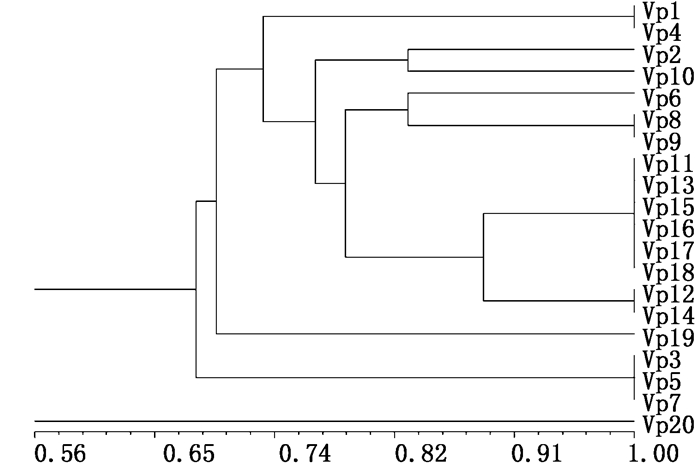
运用NTSYSpc2.1软件对上述ERIC电泳图谱进行聚类分析,使用非加权配对平均法(UPGMA)计算得到聚类树状图,从图 6中可以看到:Vp1和Vp4,Vp2和Vp10,Vp6、Vp8和Vp9,亲缘关系较近而分别聚为一支;扩增图谱带型较为一致的菌株Vp11~Vp18也基本聚在一起;Vp3、Vp5、Vp7和Vp19分别与其他菌株亲缘关系均较远, 标准株Vp20与实验中其他19株分离株关系均较远,单独聚为一支,分型结果发现Vp11~Vp18亲缘关系较近,且均分离自2015年。这说明该方法能够有效区分不同时间段获得的菌株,而对不同地理位置分离株间区分并不明显。
|
图 6 20株副溶血弧菌的ERIC-PCR聚类图 Fig. 6 ERIC-PCR clustering figure of 20 V. parahaemolyticus strains |
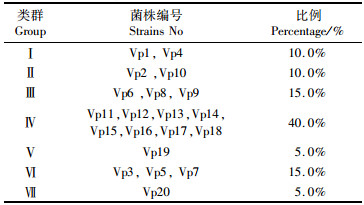
根据聚类图谱亲缘远近关系,在相似值80.0%的水平上,20株副溶血弧菌通过ERIC-PCR分型统计共分为7个不同的类群(表 2),其中:遗传多样性优势群为群体Ⅳ,菌株所占比例为40.0%;使用HUNTER等[20]的方法计算出此分型方法的分辨力指数DI值为0.811。这表明ERIC-PCR方法可以对该实验中副溶血弧菌进行较好的基因分型。
|
表 2 20株副溶血弧菌的ERIC-PCR分型聚类表 Tab.2 ERIC-PCR clustering table of 20 V. parahaemolyticus strains |
溶血性毒素是副溶血弧菌极其重要的致病因子,副溶血弧菌的耐热直接溶血毒素(thermostable direct hemolysin, tdh)和耐热相关溶血毒素(tdh-related hemolysin, trh)被认为是极为重要的两个致病因子。tdh和trh都具有溶血活性、细胞毒性、肠毒素活性[21],可以直接作用于红血球并产生神奈川现象(Kanagawa phenomenon,KP),即溶血现象[22]。tox RS/new是副溶血弧菌转座子tox RS变化之后的名称,tox RS上的tox R和tox S转座子能编码跨膜蛋白,增强副溶血弧菌的侵袭能力并且此致病基因被认为是1996年后的流行致病株特有[23],对于了解潜在新流行致病株具有重要意义。致病基因ORF8是丝状噬菌体f237的开放阅读框,该噬菌体是副溶血弧菌所特有,只拥有唯一的开放阅读框,并且被认为与副溶血弧菌的大流行株有关[24],ORF8能编码一种粘附蛋白,从而增加副溶血弧菌对人类肠道细胞的粘附能力,增强其致病性。本研究对从上海市不同单位养殖的对虾中分离得到的副溶血弧菌进行6种致病基因的检测筛查,结果发现:trh基因的携带率较低,19株分离株中仅有一株分离株Vp3检出阳性,说明该菌株存在一定的致病能力,对tdh和ORF8基因检测未发现阳性菌株,该结果与环境及海产品分离株中tdh基因检出率较低等研究结果基本一致[14];对tox RS/new基因的检出率较高,19株副溶血分离菌株中14株为阳性菌株,基因携带率为73.7%。以上结果表明,上海市6个对虾养殖单位中,从对虾中分离到的副溶血弧菌毒性并不强,但存在一定的流行性,这可能导致一些新的流行致病株的产生。
对能够引起对虾急性肝胰腺坏死病的特异性副溶血弧菌检测结果发现AP1基因携带菌株为Vp12、Vp14、Vp19,AP2 基因携带菌株为Vp14、Vp18、Vp19。AP1和AP2两个基因均是副溶血弧菌质粒基因,携带有这种质粒的副溶血弧菌理论上能够引起南美白对虾的急性肝胰腺坏死病(AHPND)[11, 18],本研究对这两种基因的检测结果并不完全一致,其原因可能是由于质粒基因具有转染能力,存在一定的基因丢失情况,也可能与这种特异性副溶血弧菌的质粒致病机理有关。有研究报道,不仅在副溶血弧菌中存在这种特异性质粒,在其他弧菌中也有该特异性质粒存在[25],表明该质粒基因存在地理范围及跨种扩张的趋势,应当引起重视,但该质粒基因的致病机理仍需进一步探索研究。
ERIC-PCR技术是一种简单快速易操作的分子分型技术,相对于传统血清学分型方法具有稳定性好和分辨率高等优点,也被越来越多地应用于菌株溯源和菌种分类等研究中。也有研究[26]表明,同一血清型的不同来源菌株通过ERIC-PCR分型技术仍有明显图谱差异,说明ERIC-PCR分型技术在种内分型溯源上更有优势。本研究采用ERIC-PCR技术对19株副溶血弧菌分离株进行基因分型,通过对指纹图谱的软件分析得到聚类树,初步探索不同菌株间的亲缘关系远近,并通过类群中的菌株分布了解了上海地区6家不同对虾养殖单位中副溶血弧菌的分子遗传差异,结果发现19株副溶血弧菌分离株中,从不同养殖单位取样分离得到的菌株,其分型结果并没有明显的地理关系,这可能与本研究样品数量有限而本身地理位置较近所致,可以后续结合传统血清学分型技术进行进一步研究探索;而从不同时间取样分离保存的菌株,结果发现2015年分离得到的Vp11~Vp18菌株间关系较近,而与2014年分离得到的菌株区分较明显,这可能是由于不同时间分离得到的这些菌株来源于1个相似的流行株[27],这也与本研究中这些菌株流行相关基因tox RS/new携带率较高结果相符合。通过对20株副溶血弧菌ERIC-PCR分型聚类,结果表明ERIC-PCR可以快速方便的分析致病菌背景,结合菌株的分离时间、地点,可以有效的对潜在的流行致病菌株进行流行病学溯源,为对虾养殖单位中副溶血弧菌的流行与预防控制提供相关参考。
| [1] |
茆丹.不同来源副溶血弧菌分子分型及其毒力基因筛查[D]. 上海: 上海交通大学, 2014. MAO D. Molecular typing and virulence genes screening of Vibrio parahaemolyticus from different sources[D]. Shanghai: Shanghai Jiao Tong University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10248-1015029004.htm |
| [2] |
陈志芸, 施春雷, 周秀娟, 等. 上海市售海产品中副溶血性弧菌的分布状况[J]. 中国食品学报, 2015, 15(8): 196-202. CHEN Z Y, SHI C L, ZHOU X J, et al. Distribution status of Vibrio parahaemolyticus in seafood markets of Shanghai[J]. Journal of Chinese Institute of Food Science and Technology, 2015, 15(8): 196-202. |
| [3] |
马立芝, 郭李平, 邱业峰. 副溶血弧菌的致病机制[J]. 生物技术通讯, 2013, 24(6): 871-876. MA L Z, GUO L P, QIU Y F. Pathogenesis of Vibrio parahaemolyticus[J]. Letters in Biotechnology, 2013, 24(6): 871-876. DOI:10.3969/j.issn.1009-0002.2013.06.029 |
| [4] |
葛明峰, 郑晓叶, 王国良. 大黄鱼感染致病弧菌的检测及其病害的预测预警[J]. 水产学报, 2014, 38(12): 2068-2074. GE M F, ZHENG X Y, WANG G L. Detection of pathogenic vibrios infection in Larimichthys crocea and its forecast and warning of disease[J]. Journal of Fisheries of China, 2014, 38(12): 2068-2074. |
| [5] |
陈蓉, 刘建勇, 曾碧健, 等. 杂色鲍消化道副溶血弧菌的分离与微生物学鉴定[J]. 湛江师范学院学报, 2012, 33(3): 100-104. CHEN R, LIU J Y, ZENG B J, et al. Isolation and identification of Vibrio parahaemolyticus in digestion guts of abalone, Haliotis diversicolor[J]. Journal of Zhanjiang Normal College, 2012, 33(3): 100-104. DOI:10.3969/j.issn.1006-4702.2012.03.019 |
| [6] |
王金霞, 李士虎, 王笃彩, 等. 中华绒螯蟹苗种生产中副溶血性弧菌病的诊断与防治[J]. 淮海工学院学报, 2003, 12(1): 52-54. WANG J X, LI S H, WANG D C, et al. Diagnosis, prevention and curing of Vibrio parahaemolyticuses in the production of river crab larvae[J]. Journal of Huaihai Institute of Technology, 2003, 12(1): 52-54. DOI:10.3969/j.issn.1672-6685.2003.01.016 |
| [7] |
陶保华, 胡超群, 吴蔚. 斑节对虾弧菌病的病原生物学研究[J]. 热带海洋学报, 2001, 20(2): 80-87. TAO B H, HU C Q, WU W. Studies on pathogenetic biology of vibriosis occurring in Penaeus monodon[J]. Journal of Tropical Oceanography, 2001, 20(2): 80-87. DOI:10.3969/j.issn.1009-5470.2001.02.014 |
| [8] |
张晓君, 陈翠珍, 阎斌伦, 等. 凡纳滨对虾(Litopenaeus vannamei)病原副溶血弧菌(Vibrio parahaemolyticus)的表型及分子特征[J]. 海洋与湖沼, 2009, 40(5): 654-662. ZHANG X J, CHEN C Z, YAN B L, et al. Phenotypic and molecular characterization of pathogenic Vibrio parahaemolyticus isolated from Penaeus vannamei[J]. Oceanologia et Limnologia Sinica, 2009, 40(5): 654-662. DOI:10.3321/j.issn:0029-814X.2009.05.021 |
| [9] |
文国樑, 曹煜成, 徐煜, 等. 养殖对虾肝胰腺坏死综合症研究进展[J]. 广东农业科学, 2015, 42(11): 118-123. WEN G L, CAO Y C, XU Y, et al. Review on hepatopancreas necrosis syndrome of shrimp[J]. Guangdong Agricultural Sciences, 2015, 42(11): 118-123. DOI:10.3969/j.issn.1004-874X.2015.11.022 |
| [10] |
谢珍玉, 周永灿, 冯永勤. 对虾弧菌病的研究进展——回顾对虾弧菌病的病原种类、致病机制与条件、症状与组织病变及对虾的防御机制[J]. 海南大学学报(自然科学版), 2007, 25(1): 88-95. XIE Z Y, ZHOU Y C, FENG Y Q. The research advance in shrimp vibriosis-reviewed on shrimp pathogens of vibriosis, pathogenic mechanisms and pathogenic conditions, symptoms and histopathological changes of the disease shrimp, and the elimination mechanisms of shrimp to vibrio[J]. Natural Science Journal of Hainan University, 2007, 25(1): 88-95. DOI:10.3969/j.issn.1004-1729.2007.01.022 |
| [11] |
JOSHI J, SRISALA J, TRUONG V H, et al. Variation in Vibrio parahaemolyticus isolates from a single Thai shrimp farm experiencing an outbreak of acute hepatopancreatic necrosis disease (AHPND)[J]. Aquaculture, 2014, 428-429: 297-302. DOI:10.1016/j.aquaculture.2014.03.030 |
| [12] |
贺连华, 吴平芳, 陈妙玲, 等. 食品中副溶血弧菌血清学分型与分子分型的研究[J]. 中国热带医学, 2011, 11(11): 1373-1375. HE L H, WU P F, CHEN M L, et al. Analysis of pulsed-field gel electrophoresis molecular subtyping and serotyping of Vibrio parahaemolyticus in food[J]. China Tropical Medicine, 2011, 11(11): 1373-1375. |
| [13] |
方伟. 副溶血性弧菌分型研究进展[J]. 中华疾病控制杂志, 2008, 12(5): 468-472. FANG W. Review on subtyping study of Vibrio parahaemolyticus[J]. Chinese Journal of Disease Control and Prevention, 2008, 12(5): 468-472. |
| [14] |
丁久法, 潘迎捷, 陈洪友, 等. 副溶血性弧菌ERIC-PCR分型及毒力基因检测研究[J]. 食品工业科技, 2010, 31(8): 137-141. DING J F, PAN Y J, CHEN H Y, et al. Genotyping of Vibrio parahaemolyticus by ERIC-PCR and virulence gene detection[J]. Science and Technology of Food Industry, 2010, 31(8): 137-141. |
| [15] |
帅慧群, 张睿, 詹丽. 多血清型副溶血性弧菌引起食物中毒的毒力基因检测和分子分型[J]. 中国卫生检验杂志, 2013, 23(18): 3524-3526, 3529. SHUAI H Q, ZHANG R, ZHAN L. Virulence gene detection and molecular typing of Vibrio parahaemolyticus in a food poisoning[J]. Chinese Journal of Health Laboratory Technology, 2013, 23(18): 3524-3526, 3529. |
| [16] |
MALUPING R P, RAVELO C, LAVILLA-PITOGO C R, et al. Molecular typing of Vibrio parahaemolyticus strains isolated from the Philippines by PCR-based methods[J]. Journal of Applied Microbiology, 2005, 99(2): 383-391. DOI:10.1111/j.1365-2672.2005.02571.x |
| [17] |
贾丹, 史成银, 黄倢, 等. 凡纳滨对虾急性肝胰腺坏死病(AHPND)病原分离鉴定及其致病性分析[J]. 渔业科学进展, 2018, 39(3): 103-111. JIA D, SHI C Y, HUANG J, et al. Identification and pathogenicity analysis of bacterial pathogen associated with acute hepatopancreatic necrosis disease (AHPND) in the pacific shrimp Litopenaeus vannamei[J]. Progress in Fishery Sciences, 2018, 39(3): 103-111. |
| [18] |
LEE C T, CHEN I T, YANG Y T, et al. The opportunistic marine pathogen Vibrio parahaemolyticus becomes virulent by acquiring a plasmid that expresses a deadly toxin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(34): 10798-10803. DOI:10.1073/pnas.1503129112 |
| [19] |
马月姣, 孙晓红, 赵勇, 等. REP-PCR及ERIC-PCR法对分离自海产品副溶血性弧菌分型分析[J]. 食品科学, 2013, 34(10): 263-267. MA Y J, SUN X H, ZHAO Y, et al. REP-PCR and ERIC-PCR analysis for the typing of Vibrio parahaemolyticus isolated from sea products marketed in Shanghai[J]. Food Science, 2013, 34(10): 263-267. DOI:10.7506/spkx1002-6630-201310058 |
| [20] |
HUNTER P R, GASTON M A. Numerical index of the discriminatory ability of typing systems:an application of Simpson's index of diversity[J]. Journal of Clinical Microbiology, 1988, 26(11): 2465-2466. |
| [21] |
NISHIBUCHI M, KAPER J B. Nucleotide sequence of the thermostable direct hemolysin gene of Vibrio parahaemolyticus[J]. Journal of Bacteriology, 1985, 162(2): 558-564. |
| [22] |
杨振泉, 焦新安. 副溶血弧菌毒力因子及其致病机理研究进展[J]. 中国人兽共患病学报, 2008, 24(11): 1070-1073. YANG Z Q, JIAO X A. Advances research on virulence factors and pathogenic mechanism of Vibrio parahaemolyticus[J]. Chinese Journal of Zoonoses, 2008, 24(11): 1070-1073. DOI:10.3969/j.issn.1002-2694.2008.11.021 |
| [23] |
LIU W W, ZHENG W L, XIE J, et al. Identification of genes related to carcinogenesis of oral leukoplakia by oligo cancer microarray analysis[J]. Oncology Reports, 2011, 26(1): 265-274. |
| [24] |
KONDOH N, OHKURA S, ARAI M, et al. Gene expression signatures that can discriminate oral leukoplakia subtypes and squamous cell carcinoma[J]. Oral Oncology, 2007, 43(5): 455-462. DOI:10.1016/j.oraloncology.2006.04.012 |
| [25] |
LIU L Y, XIAO J Z, ZHANG M M, et al. A Vibrio owensii strain as the causative agent of AHPND in cultured shrimp, Litopenaeus vannamei[J]. Journal of Invertebrate Pathology, 2018, 153: 156-164. DOI:10.1016/j.jip.2018.02.005 |
| [26] |
金莉莉, 王秋雨, 侯潇. ERIC-PCR技术在李斯特氏菌种、菌株鉴定中的应用[J]. 遗传, 2003, 25(2): 195-197. JIN L L, WANG Q Y, HOU X. ERIC-PCR on identification of Listeria species and strain[J]. Hereditas (Beijing), 2003, 25(2): 195-197. DOI:10.3321/j.issn:0253-9772.2003.02.018 |
| [27] |
NAIR G B, RAMAMURTHY T, BHATTACHARYA S K, et al. Global dissemination of Vibrio parahaemolyticus serotype O3:K6 and its serovariants[J]. Clinical Microbiology Reviews, 2007, 20(1): 39-48. DOI:10.1128/CMR.00025-06 |
 2019,
Vol. 28
2019,
Vol. 28

