2. 上海海洋大学 水产与生命学院, 上海 201306
凡纳滨对虾自1988年引进我国后迅速实现了大规模养殖,现今占我国虾类产量首位, 占80%以上[1-3]。在人工养殖条件下,雌虾卵巢发育较为缓慢,不易成熟,因此通常通过摘除眼柄的方式促进卵巢发育[4-5]。摘除眼柄可以减少性腺抑制激素(GIH)的分泌,促进卵黄蛋白原的表达,加速卵巢发育和卵子形成[6-7],但是摘除眼柄以后会对凡纳滨对虾的其他生理活动造成一定的影响。生产实践中需要选育具有优良繁殖性状的新品种以避免继续采用这种促熟方法,优良繁殖性状的种虾选育中急需解决的关键之一是与繁殖性状相关联的分子遗传标记的筛选,本研究选用SNP分子标记方法。
卵黄蛋白原是卵黄蛋白的前体物质,具有储能、运输、免疫、发育、繁殖等功能[8-10]。卵黄蛋白原在对虾中主要由肝胰腺和卵巢共同合成,在卵巢发育过程中大量表达[11-13]并在卵母细胞形成过程中不断累积直到卵子从卵巢释放。由于卵黄蛋白原在凡纳滨对虾的卵巢发育过程中占据重要地位,所以实验选择卵黄蛋白原作为目标基因,来筛选与繁殖性状相关联的SNP位点。
1 材料与方法 1.1 实验材料本实验选取养殖在塞班岛的凡纳滨对虾不同群体的雌虾进行繁殖性状测试,实验时间为2014年10月28日到2015年1月5日。实验开始前对所有待实验的凡纳滨对虾雌虾分别测量体长、体质量,剪除单侧眼柄并戴眼标,随后强化营养以促进其卵巢发育,剪下的眼柄作为DNA分子标记筛选的材料。实验期间每日观察虾的卵巢发育情况,将性腺成熟即将产卵的雌虾分别取出并放入单独产卵桶等待产卵,次日将产出的卵收集放入一定体积的海水中,搅拌均匀,取10 mL直接计数卵子总量,重复3次,取平均值,最后计算出产卵总量。
根据产卵情况挑选了关岛群体(GD2)中30只实验期间不产卵的雌虾和产卵1~5次的70只雌虾,从SyAqua群体(OS)中选择15只不产卵雌虾和15只产卵量各异的雌虾,从V101群体选择10只不产卵雌虾和10只产卵量各异的雌虾,3个养殖群体中合计选择了150只具有代表性的雌虾用于筛选与繁殖性状相关联的SNP位点。其中GD2群体是实验室基地采购自关岛本地的一批亲虾,OS群体是在引自SyAqua公司的亲虾,V101是在塞班岛长期传代养殖的亲虾。
1.2 凡纳滨对虾总DNA提取和引物设计采用天根生物科技有限公司的海洋生物组织DNA提取试剂盒(离心柱型200次)对虾的眼柄进行总DNA提取,用含有核酸染料的1%琼脂糖凝胶电泳检测(本实验使用剪除掉的眼柄进行实验,因为实验雌虾需要进行繁殖下一代)。
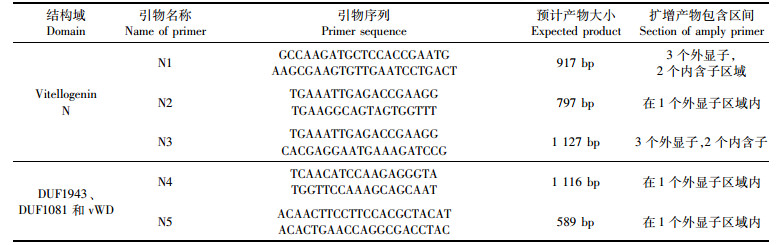
凡纳滨对虾VG基因(AY321153.2)mRNA序列全长7 763 bp,DNA全长10 419 bp,VG基因经蛋白分析后发现含有4个结构域分别是Vitellogenin N(参与脂质的转运)、DUF1943、DUF1081和vWD(von Willebrand factor type D domain,血液凝集因子Ⅶ结合到VWF的必需)。将NCBI数据库里的中国对虾VG-mRNA序列(DQ345690.1),斑节对虾VG-mRNA序列(DQ288843.1),凡纳滨对虾VG-mRNA序列(AY321153.2),刀额新对虾VG-mRNA序列(AF548364.1),刀额新对虾VG-DNA序列(AF548363.1)进行Cluster比对,找出外显子和内含子之间的分界点,在编码4大结构域的DNA序列中筛选SNP位点。在外显子保守区分别设计引物扩增主要的内含子区域及部分外显子区域,具体引物及扩增产物信息见表 1。
|
表 1 凡纳滨对虾VG基因筛选SNP位点所用的引物 Tab.1 Primers used in SNP screening of VG gene of Litopenaeus vannamei |
PCR反应体系:DNA模板2 μL,上下游引物各1 μL,2 ×Taq PCR Master Mix试剂25 μL,加ddH20。所得PCR产物先经电泳检验为单一产物者直接送至上海生工生物公司进行一代Sanger双向测序。其余PCR产物先使用天根生物科技有限公司的克隆试剂盒进行克隆,然后送到生工生物公司进行测序。
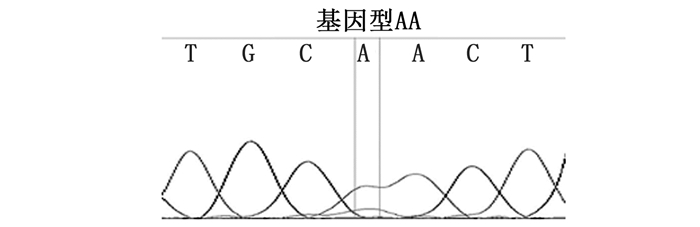
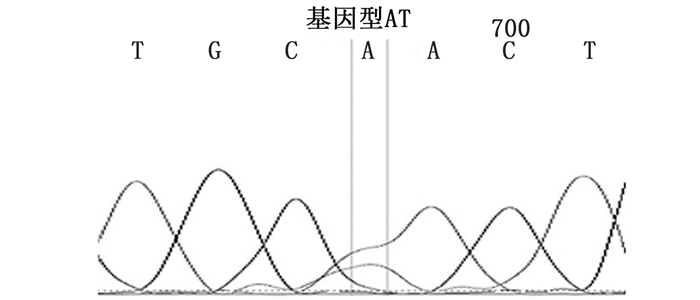
1.4 数据分析(1) 利用BioEdit软件对凡纳滨对虾VG基因测序图谱进行校正,将所测序列进行群体内比对,找到SNP位点。(2)利用DNAMAN软件分析所有SNP位点导致的氨基酸变异。(3)每个SNP位点在读取基因型时,位点出现双峰时,读取方式是测序低峰低于高峰一半的点算作纯合基因型(图 1),测序低峰高于高峰一半的点算作杂合基因型(图 2)。(4)将群体内所有样品的基因型进行比对,当携带某一基因型的个体数量低于3的时候,该位点不计入候选位点;在群体间进行每个位点的基因型比对,当在某一群体的某一位点呈现单一基因型时,该位点不计入候选位点。(5)用PopGene软件分析候选位点的多态性。(6)在GD2群体中,从每个数量型表现性状中挑选40只符合正态分布的凡纳滨对虾样品,采用SPSS 22软件单因素方差分析方法计算基因型和表现型之间的差异。
|
图 1 纯合基因型AA
Fig. 1 Homozygous genetype AA
|
|
图 2 杂合基因型AT
Fig. 2 Heterozygous genetype AT
|
对所提取的凡纳滨对虾总DNA进行电泳分析,所得结果如图 3所示,图中可见清晰明亮的电泳条带,基本无拖尾现象,说明凡纳滨对虾个体总DNA完整无降解,可以用作PCR实验。

|
图 3 凡纳滨对虾总DNA电泳结果
Fig. 3 Electrophoresis result of total DNA of Litopenaeus vannamei
|
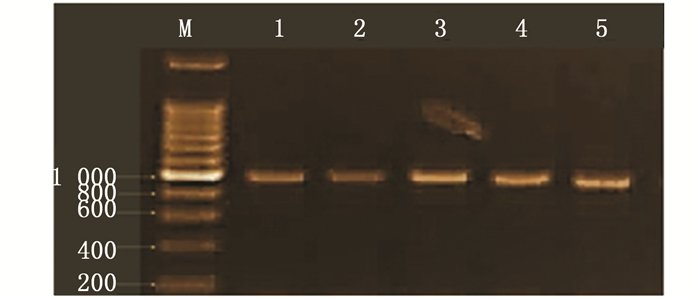
用所设计的VG基因的5对引物对凡纳滨对虾总DNA进行PCR扩增,均获得扩增产物。其中一对引物的扩增产物电泳结果如图 4所示,由图可知,PCR产物经电泳检测结果显示一对引物的PCR产物均为一条特异明亮的条带,与预期预计产物大小相符。将扩增的PCR产物进行测序,在NCBI上Blast,Blast结果显示扩增片段是凡纳滨对虾VG基因的一部分,序列相似度为99%。
|
图 4 凡纳滨对虾VG基因N3引物扩增产物电泳结果
Fig. 4 Amplification products of VG based on N3 primer gene of Litopenaeus vannamei
|
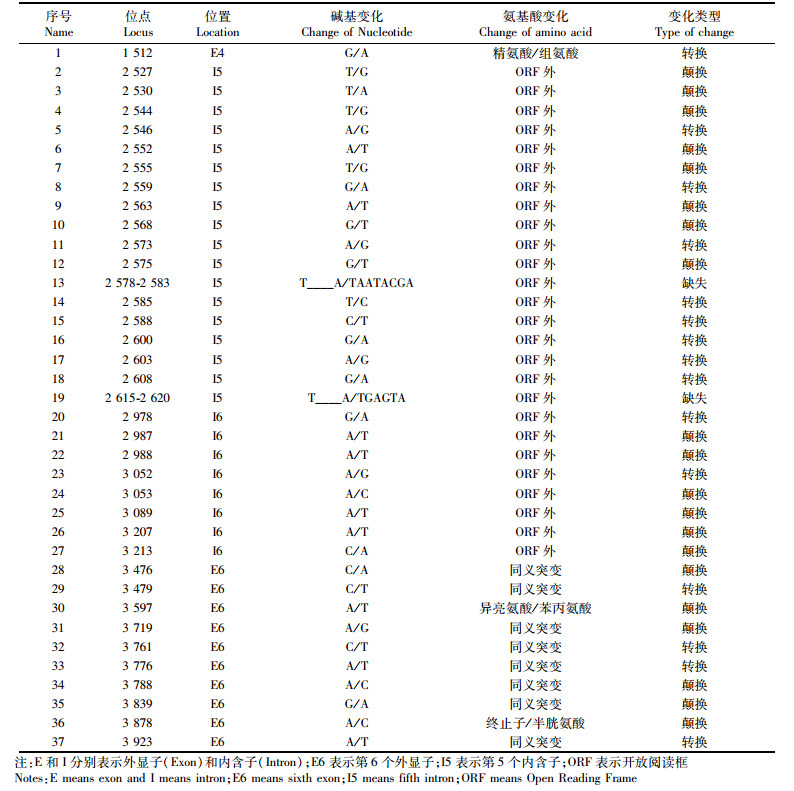
用凡纳滨对虾VG基因5对引物对150个个体进行扩增后得到的750个DNA产物中共检测到37个SNP位点,其中11个位点在外显子区域,其余26个位点在内含子区域(表 2)。11个外显子区域的突变包括8个同义突变和3个错义突变,3个错义突变分别是位点1 512精氨酸和组氨酸之间变异,位点3 597异亮氨酸和苯丙氨酸之间变异,以及位点3 878的终止子和半胱氨酸之间变异。在所得到的37个SNP位点中进一步去除个体数低的基因型以及群体表现单一的基因型,最后得到4个候选SNP位点,分别是两个错义突变位点3 597和位点3 878,两个同义突变位点3 839和3 923,4个位点都位于结构域DUF1 943上。
|
表 2 实验中所获得的凡纳滨对虾VG基因的SNP位点 Tab.2 The SNP locus in VG gene of Litopenaeus vannamei |
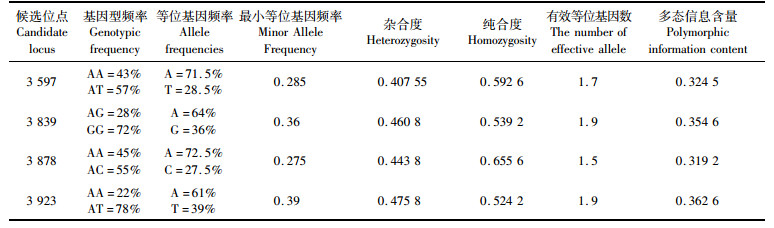
对筛选出的4个VG基因候选SNP位点进行多态性分析,具体结果见表 3。这4个候选位点的杂合度在0.407 55~0.475 80之间,有效等位基因数在1.5~1.9之间,多态信息含量在0.319 2~0.362 6之间。根据BOTSTEIN[14]的理论,4个位点在凡纳滨对虾群体中的遗传多样性属于中等水平。
|
表 3 凡纳滨对虾VG基因候选SNP位点的多态性分析 Tab.3 The polymorphism analysis of potential SNP loci of VG gene in Litopenaeus vannamei |
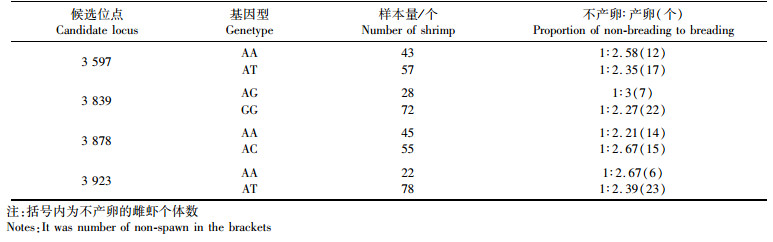
将4个位点在GD2群体中,逐一分析其产卵特性和基因型(表 4),4个候选SNP位点上各自两种基因型所对应的凡纳滨对虾个体在产卵特性上没有明显差异,即每个基因型的个体中既有产卵个体,也有不产卵个体,且产卵个体与不产卵个体比例差异不明显。如位点3 597的AA基因型不产卵和产卵个体比例是1: 2.58,AT基因型不产卵和产卵比例是1: 2.35,该位点在产卵和不产卵这两个性状上未能表现出关联性。其他3个位点的结果与之类似,均未能在是否产卵的性状上表现出关联性。
|
表 4 凡纳滨对虾SNP位点产卵和不产卵个体的样本比例 Tab.4 The proportion of spawn and non-spawn in Litopenaeus vannamei SNP locus |
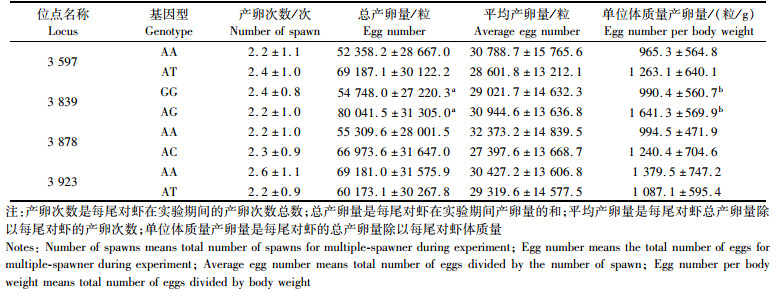
对凡纳滨对虾的SNP位点基因型和产卵数量型表现性状(产卵次数、总产卵量、平均产卵量和单位体质量产卵量)进行单因素方差分析,具体结果如表 5所示。SPSS软件分析可知位点3 839的GG和AG基因型与总产卵量和单位体质量产卵量表现出相关性,其他3个位点的基因型与产卵次数、总产卵量、平均产卵量和单位体质量产卵量等数量性状间没有呈现相关性。对数据分析发现,位点3 839处在总产卵量和单位体质量产卵量这2个性状上,基因型GG和基因型AG存在显著差异(P<0.05);位点3 597处基因型AA和基因型AT在4个性状上不存在显著差异(P>0.05),其他2个位点与之类似。
|
表 5 凡纳滨对虾VG基因SNP位点与繁殖性状的关联性分析 Tab.5 The analysis between spawn character and SNP locus of Litopenaeus vannamei VG gene |
卵黄蛋白是大部分卵生动物胚胎发育所必须的营养物质,在卵巢成熟过程中大量表达。根据本实验结果可知,37个SNP位点包括11个外显子突变和26个内含子突变。11个外显子区域的突变中存在3个错义突变,分别位于Vitellogenin N结构域(1 512位点)和结构域DUF1943(其余两个错义突变)。根据3个位点氨基酸的改变,位点1 512处精氨酸和组氨酸之间的变化,精氨酸和组氨酸都是带正电荷的极性氨基酸,所以不会对卵黄蛋白的功能产生一定的影响;位点3 597处异亮氨酸和苯丙氨酸的变化,两种氨基酸都是非极性氨基酸,对卵黄蛋白原蛋白的三维构象不会产生大的影响,因而对蛋白质的功能方面不会有大的改变。位点3 878处是终止子和半胱氨酸的变化,本来应该翻译结束,此时又有新的氨基酸继续连接,半胱氨酸还能形成二硫键,对卵黄蛋白原的三维结构会产生重大影响,可能导致蛋白质功能的完全改变。产生这种变化的虾在卵黄蛋白原的合成方面会与其他虾出现差异,能否直接影响产卵特性还需进一步实验分析。实验中同时发现结构域vWD和结构域DUF1081完全保守,没有突变位点。而DUF1943上存在多个突变位点,其中就包括3 839处变异。
3.2 对虾产卵特性的遗传标记分析本实验筛选4个候选位点产卵与不产卵之间的差异,没有发现与产卵相关的位点。出现以上结果,推测可能与研究对象的数量和筛选的位点数量有关,遗传背景的干扰和分离群体的大小也是影响QTL定位精度的两个重要因素。在选择凡纳滨对虾样本时我们发现一种现象,在整个繁殖实验期间,部分凡纳滨对虾一直不产卵,实验中也没有找到与是否产卵直接相关联的分子标记。对此存在有两方面可能:首先在分子遗传标记的筛选方面实验中只是选用了VG基因的部分序列,数量有限;其次在整个繁殖实验60天的时间没有产卵的雌虾,有可能是还没有到它们开始产卵的时间。这些未来可能产卵的雌虾在实验中只能当做不产卵虾对待,由此寻找产卵与否的分子遗传标记存在不确定性。
产卵量作为重要的数量性状,在对虾良种选育中具有重要作用,选择产卵量多的个体进行繁育,可以提高育苗效率,获得较大收益。现阶段,利用SNP标记筛选与繁殖性状相关的位点,大多集中在畜禽[15-17]方面, 水生动物则集中在中华绒螯蟹[18]、牙鲆[19],在凡纳滨对虾中尚未见相关研究。
对产卵数量性状分析发现,位点3 839与总产卵量和单位体质量产卵量显著相关,说明体质量和总产卵量之间可能存在一定的相关关系,这与袁瑞鹏等的结果一致[20]。进一步比较发现位点3 839处基因型AG总产卵量显著高于GG基因型,推测等位基因A对产卵量具有促进作用,但是在实验群体中没有发现AA基因型,所以需要加大实验的样本数量做进一步的验证。产卵量是数量遗传性状,由多基因调控,实验中发现的与繁殖性状(总产卵量、单位体重产卵量)相关联的VG基因的1个SNP位点,应该属于这些众多调控因子其中之一,需要进行更多的与繁殖相关的基因筛选。
| [1] |
师尚丽, 叶满, 朱春华, 等. 摘除眼柄与注射眼柄粗提物对凡纳滨对虾性腺发育相关基因表达的影响[J]. 上海海洋大学学报, 2016, 25(3): 344-349. SHI S L, YE M, ZHU C H, et al. Effects of the eyestalk hormones and eyestalk ablation on the gene expreesions related gonadal development of Litopenaeus vannamei[J]. Journal of Shanghai Ocean University, 2016, 25(3): 344-349. |
| [2] |
潘玉洲, 丁茂昌, 李爽. 凡纳滨对虾人工繁育技术研究[J]. 齐鲁渔业, 2010, 27(6): 17-20. PAN Y Z, DING M C, LI S. Study on artificial breeding technology of Litopenaeus vannamei[J]. Shandong Fisheries, 2010, 27(6): 17-20. |
| [3] |
杨琼. 南美白对虾高效生态养殖技术[J]. 中国水产, 2005(11): 87-89. YANG Q. Efficient ecological breeding technology of white shrimp in South America[J]. China Fisheries, 2005(11): 87-89. DOI:10.3969/j.issn.1002-6681.2005.11.028 |
| [4] |
颜婕.凡纳滨对虾卵巢发育差异表达基因的克隆与mRNA相对表达量分析[D].上海: 上海海洋大学, 2016. YAN J. Molecular cloning and mRNA expression of differential expressed genes during ovarian development of white prawn (Litopenaeus vannamei)[D]. Shanghai: Shanghai Ocean University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10264-1016912400.htm |
| [5] |
叶满.凡纳滨对虾性腺抑制激素定位及激素对性腺发育基因表达的影响[D].湛江: 广东海洋大学, 2014. YE M. Localization of gonad-inhibiting hormone in the eyestalk and effects of hormones on gonad development related genes expression in Litopenaeus vannamei[D]. Zhanjiang: Guangdong Ocean University, 2014. http://cdmd.cnki.com.cn/article/cdmd-10566-1014361325.htm |
| [6] |
朱春华, 赵光凤, 李广丽. 眼柄粗提物对凡纳滨对虾性分化的影响[J]. 广东海洋大学学报, 2011, 31(1): 23-27. ZHU C H, ZHAO G F, LI G L. Effects of eyestalk extracts on the sex differentiation on Litopenaeus vannamei[J]. Journal of Guangdong Ocean University, 2011, 31(1): 23-27. DOI:10.3969/j.issn.1673-9159.2011.01.005 |
| [7] |
邓思平, 叶满, 朱春华, 等. 切除眼柄对凡纳滨对虾亲虾卵巢、肝胰腺、血清生化组成的影响[J]. 广东海洋大学学报, 2016, 36(3): 29-34. DENG S P, YE M, ZHU C H, et al. Influence of eyestalk ablation on biochemical composition of ovary, hepatopancreas and serum in Pacific White Shrimp, Litopenaeus vannamei broodstock[J]. Journal of Guangdong Ocean University, 2016, 36(3): 29-34. DOI:10.3969/j.issn.1673-9159.2016.03.005 |
| [8] |
张士璀, 孙旭彤, 李红岩. 卵黄蛋白原研究及其进展[J]. 海洋科学, 2002, 26(7): 32-35. SUN S C, SUN X T, LI H Y. Review on vitelloginin[J]. Marine Sciences, 2002, 26(7): 32-35. DOI:10.3969/j.issn.1000-3096.2002.07.011 |
| [9] |
孙亚宁.文昌鱼一种新卵黄蛋白原的纯化、鉴定及其免疫防御功能的研究[D].青岛: 中国海洋大学, 2004. SUN Y N. Purification and characterization of a new form of vitellogenin from amphioxus (Branchiostoma Belcheri Tsingtaunese) and its immune function study[D]. Qingdao: Ocean University of China, 2004. http://cdmd.cnki.com.cn/Article/CDMD-10423-2004131800.htm |
| [10] |
白鸿坤.青虾卵黄蛋白原和卵黄蛋白原受体基因的克隆及功能研究[D].南京: 南京农业大学, 2015. BAI H K. Gene clone and functional analysis of vitellogenien and vitellogenin receptor in Macrobrachium nipponense[D]. Nanjing: Nanjing Agricultural University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10307-1017045481.htm |
| [11] |
李媛媛.凡纳滨对虾(Litopenaeus vannamei)和罗氏沼虾(Macrobrachium rosenbergii)卵黄蛋白原mRNA合成部位的初步研究[D].上海: 上海海洋大学, 2012. LI Y Y. Study on site of vitellogenin synthesis in the shrimp Litopenaeus vannamei and Macrobrachium rosenbergii[D]. Shanghai: Shanghai Ocean University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10264-1013000228.htm |
| [12] |
张成锋, 刘红, 高祥刚, 等. 中国对虾卵黄蛋白原合成部位的研究[J]. 海洋水产研究, 2006, 27(6): 7-13. ZHANG C F, LIU H, GAO X G, et al. Studies on site of vitellogenin synthesis in the shrimp Fenneropenaeus chinensis[J]. Marine Fisheries Research, 2006, 27(6): 7-13. |
| [13] |
TIU S H K, HUI J H L, MAK A S C, et al. Equal contribution of hepatopancreas and ovary to the production of vitellogenin (PmVg1) transcripts in the tiger shrimp, Penaeus monodon[J]. Aquaculture, 2006, 254(1/4): 666-674. |
| [14] |
BOTSTEIN D, WHITE R L, SKOLNICK M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314-331. |
| [15] |
石磊, 岳文斌. SNP的研究进展及其在家畜育种中的应用[J]. 畜禽业, 2007(3): 2-4. SHI L, YUE W B. Advance in Single Nucleotide Polymorphism and its application in livestock[J]. Livestock and Poultry Industry, 2007(3): 2-4. DOI:10.3969/j.issn.1008-0414.2007.03.001 |
| [16] |
王洋.香猪8个SNP的群体分型及其与繁殖性状的关联研究[D].贵阳: 贵州大学, 2016. WANG Y. Eight SNPs genotyping and their association analysis with the reproduction traits of Xiang pigs[D]. Guiyang: Guizhou University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10657-1016742617.htm |
| [17] |
肖礼华.鸡CRBPs基因家族克隆、表达及其与繁殖性状的关联分析[D].成都: 四川农业大学, 2011. XIAO L H. Molecule clone, expression and the association analysis in reproduction traits of the chicken CRBPs[D]. Chengdu: Sichuan Agricultural University, 2011. http://cdmd.cnki.com.cn/article/cdmd-10626-1011222404.htm |
| [18] |
唐刘秀.中华绒螯蟹育种群体遗传特征分析及性早熟相关SNP标记筛选[D].南京: 南京师范大学, 2014. TANG L X. Analysis on the genetic characteristics of breeding stocks and identification of SNPs associated with precocity of Eriocheir sinensis[D]. Nanjing: Nanjing Normal University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10319-1015529031.htm |
| [19] |
马瑞芹.牙鲆三种细胞色素P450酶基因SNP与繁殖内分泌相关性分析[D].青岛: 中国海洋大学, 2012. MA R Q. Study on associations of SNP of three cytochrome P450 enzymes gene and the reproductive endocrine of Japanese flounder (Paralichthys olivaceus)[D]. Qingdao: Ocean University of China, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10423-1012504994.htm |
| [20] |
袁瑞鹏, 刘建勇, 张嘉晨, 等. 凡纳滨对虾不同家系间繁殖性状的比较[J]. 海洋学报, 2016, 38(6): 89-97. YUAN R P, LIU J Y, ZHANG J C, et al. A comparative study of reproductive traits among different Litopenaeus vannamei families[J]. Haiyang Xuebao, 2016, 38(6): 89-97. DOI:10.3969/j.issn.0253-4193.2016.06.010 |
2. College ofFisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
 2018,
Vol. 27
2018,
Vol. 27


