2. 上海出入境检验检疫局, 上海 200135
2017年9月8日,随着芬兰正式向国际海事组织(IMO),递交了《关于船舶压载水及其沉积物管理和控制的国际公约》(以下简称压载水国际公约)[1]加入文书,公约缔约国船舶吨位总数达到35.1441%,缔约国总数达到52个,满足了公约生效的条件,公约将在2017年9月8日正式生效。该公约由IMO于2004年制定,规定了压载水的置换与性能标准,性能标准里规定了霍乱弧菌、大肠菌群和粪肠球菌的标准,其他致病病原体并未规定。但是据以往的检测数据,大量的致病细菌存在于压载水中,山东从入港的船舶压载水中检出了多达50种细菌,多为致病或条件致病菌,除了霍乱弧菌、副溶血弧菌等大量弧菌,还有大肠埃希氏菌、阪岐杆菌等食源致病菌[2]。
对大连入境的船舶压载水和沉积物48份样本进行检测,发现15种细菌,包括1例霍乱弧菌[3]。对宁波港来自美洲、印度洋和新加坡等海域的压载水中细菌群落多样性研究,也发现了葡萄球菌属、弧菌属和军团菌属等细菌的存在[4]。国外对压载水中携带致病菌的研究也是由来已久,针对到达切萨皮克海湾的压载水细菌总数进行研究发现,平均每毫升的压载水中,有多达8.3×105个细菌[5];针对到达墨西哥湾的五艘船舶的非饮用水进行了研究,检出了致病性霍乱弧菌,而其中有四艘船舶可能是因为途经了有霍乱疫情的拉丁美洲国家[6],这些致病菌以及病原体能直接进入人体,危害人体的健康。因此, 针对处理前后的压载水, 构建病原微生物检测体系, 已成为世界各海洋国家亟待解决的问题。
在这些病原微生物中,包括霍乱弧菌、副溶血弧菌、大肠杆菌O157、阪崎杆菌等食源性致病菌首当其冲。据世界卫生组织(WHO)报道,全球每年因食源性微生物污染引起的腹泻病例达数亿起,死亡的0~15岁儿童约170万人[7]。2010年中国卫生部发布的全国食物中毒事件调查结果显示,微生物性食物中毒事件的报告起数和中毒人数最多, 分别占总数的36.82 %和62.10%。在我国发生的食物中毒事件中,常见的重要致病菌有沙门氏菌、副溶血性弧菌、蜡样芽胞杆菌、金黄色葡萄球菌、肉毒梭菌、单核细胞增生性李斯特菌、空肠弯曲杆菌及肠出血性大肠埃希菌等[8-10]。压载中携带有大量的致病菌,很可能会导致口岸公共卫生事件的发生,本研究针对压载水中可能存在的,口岸重点关注的食源性致病菌,采用高通量基因芯片进行分析和研究,建立压载水中常见致病菌的高通量快速检测方法。
目前采用的传统食品微生物检测方法包括增菌、分离、鉴定等程序,一般需要5~7 d才能完成,检验过程繁琐、费时费力,很难适应公共卫生事件应急处理快速反应的需要。以高通量、灵敏、特异、适用范围广和低成本为特点的快速检测方法受到越来越多的关注,而在这些方法中又以DNA检测为基础的各种分子生物学方法应用尤为广泛。而DNA芯片技术是一种基于核酸杂交的检测方法,是在固相载体(比如玻璃或硅片)上固定靶特异性的寡核苷酸探针,然后与标记的样品(一般指样品的DNA)杂交,通过对杂交信号的检测分析,即可获得样品的遗传信息,用于检测一种或多种致病菌。文中的高通量检测芯片通过在一张芯片上针对常见的食源性致病菌设计多重PCR引物,扩增致病菌的目标基因,并在同一张芯片上完成杂交反应,快速、准确、同步地检测包括大肠埃希氏菌、空肠弯曲菌、副溶血性弧菌、蜡样芽孢杆菌等在内共l2种致病菌。
1 材料与方法 1.1 菌株及压载水来源沙门氏菌、单增李斯特氏菌、蜡样芽孢杆菌、痢疾杆菌、产气荚膜梭菌、副溶血性弧菌、空肠弯曲杆菌、大肠弯曲杆菌和阪崎肠杆菌、肠出血性大肠杆菌O157:H7菌株标准菌株购自美国ATCC。霍乱弧菌DNA来自军事医学科学院。金黄色葡萄球菌为本实验室分离得到的阳性菌株。压载水取自上海口岸入境船舶采集的压载水,已经通过PCR的方法检测为阴性,用来作为模拟样本验证。
1.2 DNA提取方法使用细菌基因组DNA提取试剂盒(离心柱型,Qiagen公司)提取细菌DNA。取1.5 mL增菌液,12 000 r/min离心2 min,弃去上清液,重悬沉淀,按照试剂盒说明书中的提取步骤提取DNA。
1.3 靶基因的扩增 1.3.1 引物和探针由上海交大与威特公司联合开发的高通量病原体检测芯片,主要是利用细菌的16S rDNA或者23S rDNA序列设计通用引物,在此引物扩增片段内设计每种细菌的特异探针,对于在16S rDNA或者23S rDNA引物扩增片段内无法满足探针特异性的菌株,则利用特异基因设计引物和探针。
1.3.2 反应平台为防止引物间的互相作用,分两个反应室进行PCR反应,两个反应室都可以联通到杂交室,PCR后的扩增液可以通过注射压力方式使其流入杂交室进行杂交反应。
PCR反应体系为:2X Mater mix 6.25 μL, PrimerA/B 1 μL,PCR Grade H2O 0.75 μL, PCR Control 2 μL, Sample 2.5 μL。
反应条件相同:94 ℃预变性5 min;94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1 min,8个循环,每个循环退火温度降低1 ℃。8个循环完成后94 ℃变性30 S,56 ℃退火30 S,72 ℃延伸1 min,30个循环;72 ℃,8 min。
1.4 杂交反应分别注入PCR反应室14.5 μL杂交液,使杂交液和扩增液都进入到杂交舱内,反应30 min。杂交后的芯片用washing buffer 3 000 r/min 2 min冲洗,直接用威特荧光扫描仪检测。
1.5 检测体系的灵敏度与特异性DNA水平检测:将参比菌株的过夜培养物用生理盐水制备成菌悬液,通过麦氏比浊法建立标准曲线图 1,判断菌的浓度。将离心后的菌体抽提DNA,并用Thermo公司的Nano-drop 2 000 C测定DNA浓度。根据DNA浓度将各个菌的DNA稀释到大约10、100和1 000 copy/μL。取各个梯度DNA作为模板加入到芯片中进行检测。
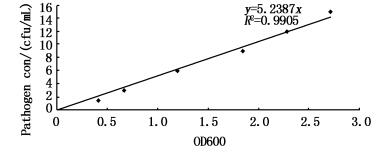
|
图 1 菌浓度测定标准曲线,标准品适用的是麦氏比浊液的BaSO4溶液
Fig. 1 Standard curve for the determination of bacterial concentration. The standard material is the BaSO4 solution.
|
混合菌DNA水平检测:将多种菌的DNA混合到一起,稀释到10、100和1 000 copy/μL的浓度,作为模板进行检测。
菌裂解液直接检测:将参比菌株的过夜培养物用生理盐水制备成菌悬液,通过麦氏比浊法建立标准曲线图 1,测定菌的OD,计算菌的浓度(cfu/mL),根据测定的菌浓度,将菌液进行梯度稀释,并100 ℃裂解10 min,取2.5 μL进行PCR扩增杂交。
1.6 样品添加灵敏度的测定取经荧光PCR检测的12种致病菌阴性的压载水样品3份,每份5 mL, 每份分别同时接种1、10、100 CFU/mL的5种菌悬液各1 mL,同时以不接种病原菌的压载水做阴性对照,加入90 mL优化培养基中,30 ℃培养12 h[11]。取2 mL增菌液离心后加入生理盐水,100 ℃热裂解10 min,进行高通量芯片测定。
2 结果和讨论 2.1 致病菌DNA提取采用Qiagen公司的DNA提取试剂盒提取过夜培养的细菌DNA,结果如表 1所示。由表 1可见样品抽提的DNA,260/280多在2.0左右,说明其中含有较多RNA,加入RNA酶进行处理后备用。
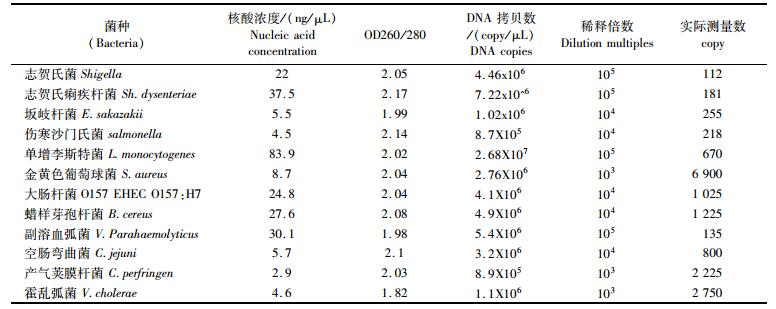
|
表 1 DNA抽提结果以及灵敏度测定 Tab.1 the result of DNA extraction and the detection limit |
设计的微阵列芯片含有112条探针,包括10条PCR阳性对照探针和10条杂交阳性对照探针,8条阴性对照探针,以及84条病源菌专一性探针。
用从美国ATCC购买的参比菌株和实验室自分离菌株共12株菌进行了杂交实验。其中6种单独菌株杂交结果见图 2。

|
图 2 6种食源性致病菌高通量芯片检测灵敏性
Fig. 2 6kinds of pathogens detection by chip for the detection limit
|
图 2显示,用不同的菌株杂交,在芯片的相应位置会出现明显的阳性信号。同时,阳性对照位置出现明显的杂交信号,阴性对照处不出现杂交信号。这表明芯片的特异性良好,不会出现引物的假阳性现象。
我们对多种病源菌同时在同一张芯片上进行特异性检测,其互相之间的干扰程度见图 3所示。可以同时检测到十二种致病菌,这表明该芯片的特异性良好,在所检测的菌株之间无交叉反应,完全可以用于同时检测多种食源性致病菌。

|
图 3 12种致病菌的DNA杂交检测
Fig. 3 Detection of 12 kinds of pathogens
|
对于2.1中检测的DNA模板,我们测定其DNA浓度,并根据DNA分子的大小,估算出具体能检测的copy数如表 1所示,可以看到检测能力最好的为志贺氏菌,能检测到5.5×10-4 ng,即112 copy的DNA;对金黄色葡萄球菌的检测能力最弱,约为2.2×10-2 ng, 约7 000 copy的DNA。
按照我们提取DNA的经验,抽提的效率约10%,预测通过该芯片直接进行原位菌液扩增杂交为10倍的DNA数目,为验证这个假设,我们选取了5种菌,沙门氏菌、金黄色葡萄球菌、单增李斯特菌、副溶血弧菌、志贺氏菌的增菌液稀释后,测定了其灵敏度, 如图 4a.在104~105水平上可以测到5种菌。

|
图 4 增菌裂解液的芯片测定
Fig. 4 Detection of Lysis solution by the chip
a:五种菌(沙门氏菌、金黄色葡萄球菌、单增李斯特菌、副溶血弧菌、志贺氏菌)在104~105 copies时的检测;b:A中5种菌添加到压载水中增菌后的检测结果;c,负对照 a :Five kinds of bacteria detection in the concentration of 104~105 copies; b:Detection of bacteria added to ballast water; c, Negative control |
应用高通量芯片方法对添加到压载水中的菌进行检测, 结果见图 4b.从图中可以看出, 高通量芯片检测认为添加的5种混合菌的敏感性可以达到1 cfu。
3 讨论高通量DNA芯片将DNA杂交检测的扩增过程和杂交检测过程浓缩到一张高通量芯片上进行检测,大大缩减了检测的时间和复杂性,检测设备简单易于携带。检测系统可以定量检测低达110 copy DNA的致病菌,大大提高了当前的检测能力。
当前研究多需要抽提DNA后进行检测,只要菌量足够,该芯片系统可以直接检测细菌的裂解液,其检测的灵敏性也可以达到104~105水平,这就意味着增菌的时间可以大大缩短到几个小时,而从样品到增菌以及检测总时间完全可以在8 h内完成,有益于快速准确地检测食源性致病菌。
当前对于PCR检测的灵敏度研究,主要通过模拟样品添加菌或者检测样品直接增菌后抽提DNA进行DNA水平的检测完成,而对于灵敏度的测定多是添加菌的量来评价,如本研究2.3中的结果,赵新等研究的4种食源致病菌的多重PCR检测,以及KAWASAKI等在肉中检测沙门氏菌、单增李斯特菌和大肠杆菌O157的多重PCR检测[12-13]。本研究既测定了芯片添加菌灵敏性,又直接检测了芯片的检测下限,对于DNA水平的分子,可以在102~103的水平上进行直接检测,但是因为本方法测定下限时采用的是DNA浓度直接除以该菌总DNA分子量的方法,未考虑游离核酸等,所以实际检测的下限可能会更小,灵敏度可能更高一些。
目前该方法与其它PCR方法一样,不能区分待检样品中的阳性样本含有的是死菌还是活菌,因此只能当作一个初步的筛选,检测结果还需要进一步用其他生理生化手段来确认。在本实验中,尚未开展大量对在真实压载水样本的检测,将进一步通过对口岸采集的实际样品进行检测,然后将芯片检测结果与传统检测结果进行比较来评价。
| [1] |
IMO. International convention fo r the control a nd management of ships ballast water and sediments[EB/OL]. [2004-02-13]. http://www.imo.org.
|
| [2] |
贾俊涛, 赵丽青, 李伟才, 等. 入境船舶压载水中细菌调查的多维尺度分析[J]. 中国国境卫生检疫杂志, 2009(1): 31-34. JIA J T, ZHAO L Q, LI W C, et al. Multidimensional scaling analysis of bacteria in ballast water from entry ships[J]. Chinese Frontier Health Quarantine, 2009(1): 31-34. |
| [3] |
李云峰, 吴刚, 薛芳, 宋锋林, 蔡鹏. 16S rDNA序列分析法在国际船舶压载舱沉积物病原微生物检测中的应用[J]. 中国国境卫生检疫杂志, 2009, (6): 475-477. LI Y F, WU G, XUE F, et al. Application of 16S rDNA Sequence Analysis Method in Pathogens Detection from Ballast Sediments of International Vessels[J]. Chinese Frontier Health Quarantine, 2009, (6): 475-477. |
| [4] |
李春丽, 刘兵, 周君, 等. 宁波港压载水的细菌多样性研究[J]. 生态科学, 2012(06): 636-644. LI C L, LIU B, ZHOU J, et al. Study on genetic diversity of bacteria in ballast water from Ningbo port[J]. Ecological Science, 2012(06): 636-644. DOI:10.3969/j.issn.1008-8873.2012.06.007 |
| [5] |
RUIZ G M, RAWLINGS T K, DOBBS F C, et al. Global spread of microorganisms by ships[J]. Nature, 2000, 408: 49-50. DOI:10.1038/35040695 |
| [6] |
MCCARTHY S A, KHAMBATY F M. International dissemination of epidemic Vibrio cholera by cargo shipballast and other nonpotable waters[J]. Appl Environ Microbiol, 1994, 60(7): 2597-601. |
| [7] |
张红波. 我国食品安全现状分析及其对策[J]. 中国安全科学学报, 2004, 14(1): 15-17. ZHANG H B. Analysis on Current Situation of Food Safety in China and Its Countermeasures[J]. China Safety Science Journal, 2004, 14(1): 15-17. |
| [8] |
张芳, 马国柱, 潘立, 等. 陕西省2002-2006年食源性致病菌污染状况[J]. 中国公共卫生, 2008, 24(2): 222-224. ZHANG F, MA G Z, PAN L, et al. Surveillance and analysis for food-borne pathogens in retailed foods in Shaanxi province, 2002-2006[J]. Chin J Public Health, 2008, 24(2): 222-224. DOI:10.11847/zgggws2008-24-02-49 |
| [9] |
畅修军, 刘桂华, 孔祥云, 等. 2002-2007年吉林省食品中食源性致病菌监测结果分析[J]. 中国国境卫生检疫杂志, 2008, 18(7): 1400-1402. CHANG X J, LIU G H, KONG X Y, et al. Monitoring results of foodborne pathogens in food in Jilin Province in 2002-2007[J]. Chinese Frontier Health Quarantine, 2008, 18(7): 1400-1402. |
| [10] |
龚霄, 陈盼. 革兰氏阳性食源性致病菌[J]. 肉类研究, 2008(5): 53-60. GONG X, CHEN P. G+Food-borne Pathogens Bacteria[J]. MEAT RESEARCH, 2008(5): 53-60. |
| [11] |
赵新, 王永, 兰青阔, 等. 四种食源性致病菌多重PCR检测技术的研究及应用[J]. 天津农业科学, 2009, 15(6): 6-8. ZHAO X, WANG Y, LAN Q K, et al. Research and application of multiplex PCR for four foodborne pathogens[J]. Tianjin Agricultural Sciences, 2009, 15(6): 6-8. |
| [12] |
郝玉莲, 孙皆宜, 李艾, 等. 正交优化多重PCR反应体系检测3种食源性致病菌的研究[J]. 安徽农业科学, 2010, 38(2): 602-605. HAO Y L, SUN J Y, LI A, et al. Application of Orthogonal Design to Optimize Multiplex PCR Reaction System for Detection of Three Food-borne Bacteria Pathogens[J]. Journal of Anhui Agri. Sci., 2010, 38(2): 602-605. |
| [13] |
KAWASAKI S, HORIKOSHI N, OKADA Y, et al. Multiplex PCR for simultaneous detection of Salmonella spp., Listeria monocytogenes, and Escherichia coli O157:H7 in meat samples[J]. Journal of Food Protect, 2005, 68(3): 551-556. DOI:10.4315/0362-028X-68.3.551 |
2. Shanghai entry-exit inspection and quarantine bureau, Shanghai 200135, China
 2018,
Vol. 27
2018,
Vol. 27


