2. 上海海洋大学 农业部种质资源与利用重点开放实验室, 上海 201306
纤维素酶(cellulase)是一类专门催化纤维素链中β-1, 4-糖苷键水解,使纤维素转化成葡萄糖、纤维二糖、纤维低聚糖的酶,可被广泛应用于食品、饲料、纺织、环保、医药、生物质能源等领域[1-3]。自然界中许多细菌、真菌、昆虫、动物都能产生纤维素酶[4],尤其是草食性的动物与昆虫,它们的消化道里面存在协助消化的纤维素酶产生菌[5]。研究表明:多数纤维素酶的来源是微生物,尤其是细菌和真菌[6]。
团头鲂(Megalobrama amblycephala)和草鱼(Ctenopharyngodon idellus)为典型的草食性鱼类,两者均能以水草为生,在我国淡水养殖中占据重要的地位,并对维持淡水养殖系统的生态平衡具有显著的作用[7-8]。目前,水产养殖中面临的最大问题是饲料的利用率不高,废物的排放高等问题[9],然而纤维素酶可以改变饲料的品质,补充动物的消化酶,促进营养物质的吸收,消除抗营养因子,某些纤维素酶产生菌的单细胞蛋白可以作为饲料来源,改善动物的饲料品质[10]。已有的全基因组测序结果表明草鱼和团头鲂的基因组内并无编码纤维素酶的基因存在[11-12],这点也佐证了草食性鱼类分解利用纤维素所需的纤维素酶是由其自身消化道内微生物分泌产生[13],然而草食性鱼类肠道纤维素酶产生菌的组成及活力等关键问题仍尚待揭晓。本研究以黑麦草投喂辅以饲料养殖的团头鲂和草鱼为研究对象,分离和筛选到了几株主要的纤维素酶产生菌,对其进行菌种鉴定及发酵趋势的初步研究,旨在为草食性鱼肠道纤维素酶的应用提供科学依据,为草食性鱼类膳食性饲料的配比提供基础性研究。
1 材料与方法 1.1 样品采集实验鱼为二龄草鱼(~1 kg)和团头鲂(~0.8 kg)各3尾,取自农业部团头鲂遗传育种中心。养殖池塘面积约1 hm2,实验鱼投喂复合饲料和种植黑麦草,比例约为1:2。
1.2 培养基[5, 14]LB培养基(g/L):胰蛋白胨10;酵母提取物5;NaCl 10(pH自然)。
PDA培养基(g/L):葡萄糖20;马铃薯粉6;琼脂20(pH 5.6±0.2)。
羧甲基纤维素培养基(g/L):CMC(羧甲基纤维素) 10;KH2PO4 4;Na2HPO4 4;MgSO4·7H2O 0.2;CaCl2 0.001;FeSO4·7H2O 0.001;蛋白胨2;琼脂粉15。
滤纸培养液(g/L):NaNO3 2.5;KH2PO4 2;MgSO4 0.2;CaCl2·6H2O 0.1;NaCl 0.2;1×6 cm滤纸条。
种子培养液(g/L):葡萄糖2;KH2PO4 4;Na2HPO4 4;MgSO4·7H2O 0.2;CaCl2 0.001;FeSO4·7H2O 0.004;蛋白胨2(pH 7)。
发酵培养液(g/L):CMC 10;KH2PO4 4;Na2HPO4 4;MgSO4·7H2O 0.2;CaCl2 0.001;FeSO4·7H2O 0.004;蛋白胨2。
1.3 实验方法 1.3.1 样品的制备解剖团头鲂和草鱼,取肠道的前后两端结扎,剪下结扎好的整个肠道,放进灭菌烧杯内。在净化工作台下解剖肠道,收集肠道内容物,并刮取肠道黏膜,黏膜和内容物混匀后作为样品,置于4 ℃冰箱备用[4-5]。
1.3.2 纤维素酶产生菌的初筛称取1 g样品,用0.9%的生理盐水稀释到合适的浓度备用。
1) 直接初筛方案
移取20 μL稀释液直接涂布于羧甲基纤维素选择培养基上,28 ℃培养数天;另外,移取100 μL稀释液于10 mL滤纸培养液中,到滤纸溃烂时,稀释涂布于羧甲基纤维素培养基上筛选,28 ℃培养数天。
2) 富集分离纯化再筛选方案
移取20 μL稀释液分别涂布于LB或PDA平板上,28 ℃培养48 h后,挑选大而饱满的菌落,接种到羧甲基纤维素培养基上筛选,28 ℃培养数天。
1.3.3 纤维素酶产生菌的复筛1) 透明圈法
在初筛平板上挑选生长良好的菌落平板划线分离得单菌落。纯化的菌株,点种在直径为6 cm的羧甲基纤维素培养基平板上,先后采用革兰氏碘液染色法和刚果红染色法染色[15-16],观察透明圈直径和菌落大小情况。
2) 摇瓶发酵法
选取透明圈大且菌落饱满的菌株,接入100 mL种子培养液中,28 ℃、150 r/min培养36 h。将种子液以3%接种量接入100 mL发酵培养液中,28 ℃、150 r/min培养48 h得到发酵液,测定纤维素酶活。
1.3.4 纤维素酶的酶活测定1) 粗酶液的制备
发酵液经过12 000 r/min、4 ℃离心15 min,取上清液作为胞外酶粗酶液;发酵液抽滤后所得菌体中加入15 mL、0.05 mol/L pH 4.8柠檬酸缓冲液,超声波细胞破碎(600 W)30 min后,12 000 r/min、4 ℃离心15 min,离心所得溶液即为胞内酶粗酶液。
2) 纤维素酶活
1 mL酶反应体系为:含1%羧甲基纤维素的0.05 mol/L pH 4.8柠檬酸缓冲液0.5 mL,粗酶液0.5 mL。混合液在50 ℃水浴反应30 min后,立即加入DNS溶液并煮沸5 min以停止反应,冷却后测定OD540[17]。上述反应以煮沸的粗酶液作为对照,每组3个平行重复实验。一个酶活力单位(U)定义为每分钟释放1 μg葡萄糖所需要的酶量[14, 17]。
1.3.5 菌株鉴定1) 16S rDNA/ITS序列分析
用细菌基因组提取试剂盒[天根生化科技(北京)有限公司]和真菌基因组提取试剂盒[生工生物工程(上海)股份有限公司]提取出菌体总DNA。细菌16S rDNA序列引物为27F:5′-AGAGTTTGATCMTGGCTCAG-3′,1492R:5′-TACGGYTACCTTGTTACGACTT-3′[18]。真菌ITS序列引物为ITSF:5′-TCCGTAGGTGAACCTGCGG-3′,ITSR:5′-TCCTCCGCTTATTGATATGC-3′[19]。PCR反应体系为25 μL:模板DNA 1 μL,上下游引物各1 μL,MIX 10 μL, ddH2O 12 μL。PCR反应过程:95 ℃预变性3 min,95 ℃变性1 min,52 ℃复性1 min,72 ℃延伸2 min,30个循环,72 ℃延伸10 min[20]。PCR扩增产物送往生工生物工程(上海)股份有限公司测序。测序结果与GenBank数据库中的序列进行BLAST检索比对,选取相似性高的微生物序列,利用Cluster W软件进行多重序列比对,在MEGA 7.0软件中,根据Kimura 2-parameter model遗传距离模型,使用邻接法(neighbour-joining,NJ)构建系统发育树[21]。
2) 菌落形态学特征
为了充分地观察菌落的形态学特征,将菌株点种在LB/PDA培养基上,培养数天,不断地观察菌株的培养特征,细菌进行革兰氏染色,普通光学显微镜观察,真菌取生长良好的菌苔制片,进行扫描电子显微镜(日本日立,S-3400N)15 kV高真空模式观察。参照《伯杰氏系统细菌学手册》[22]、《中国真菌志》[23]分析菌株。
2 结果 2.1 纤维素酶产生菌的初筛利用以羧甲基纤维素为碳源的选择性培养基,一共从团头鲂和草鱼肠道初筛到108株单菌落菌株,其中直接以羧甲基纤维素培养基进行筛选的方案得到20株,滤纸溃烂后再筛选得到21株,PDA富集分离后再筛选得到23株,LB富集分离后再筛选得到44株。分离纯化的典型单菌落形态如图 1-a, b所示。
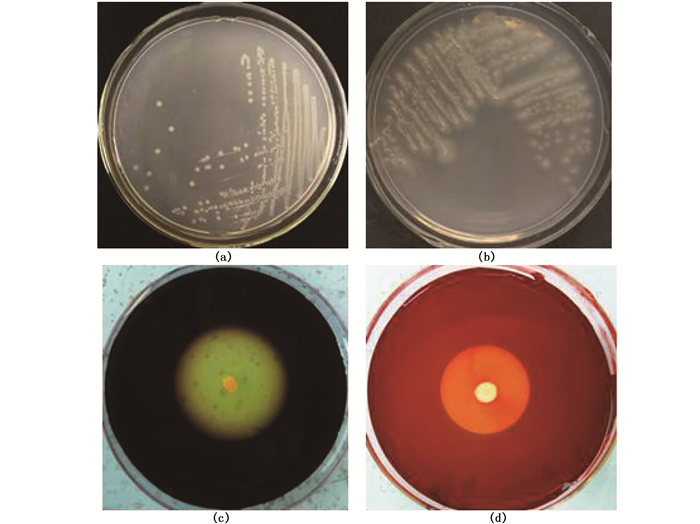
|
图 1 纤维素酶产生菌株的筛选
Fig. 1 Screening of cellulose-producing strains
(a)分离的单菌落;(b)分离的单菌落;(c)革兰氏碘液染色;(d)刚果红染色 (a) Streaked single colony; (b) Streaked single colony; (c) Gram′s iodine staining; (d) Congo red staining |
对初步分离得到的108株单菌落菌株进行革兰氏碘液染色,35株呈现较大的透明圈(>0.8 cm,如图 1-c所示),其水解圈直径D及菌落直径d如表 1所示。另外,对这35株菌株的单菌落进行刚果红染色,发现22株菌株分别出现了明显的透明圈(如图 1-d所示),其水解圈直径D、菌落直径d以及水解圈与菌落直径的比值D/d,如表 1所示。
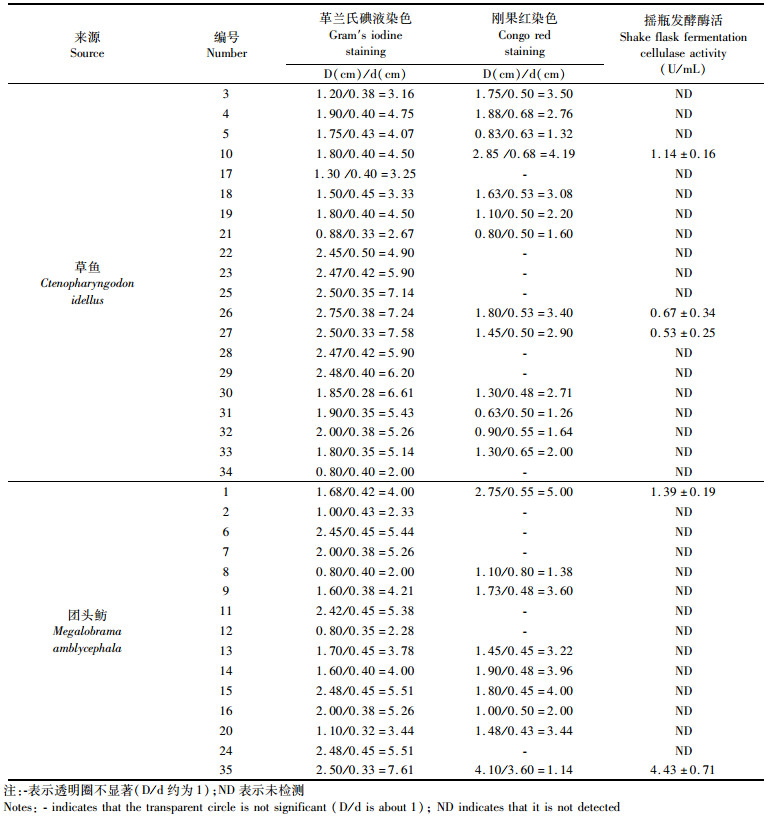
|
表 1 菌株的透明圈与纤维素酶活性 Tab.1 Transparent circle of strains and their cellulase activity |
选取革兰氏碘液法和刚果红染色法中透明圈均较显著的5株菌株(MA1、CI10、CI26、CI27、MA35),进行摇瓶发酵复筛,结果显示各菌株都具有一定的纤维素分解能力,其中MA1、CI10、MA35的发酵液具有较显著的纤维素酶活力,分别为1.39 U/mL、1.14 U/mL、4.43 U/mL,MA35的纤维素酶活最大(表 1)。因此,为了进一步地研究,选取菌株MA1、CI10、MA35作为待鉴定菌株。
2.3 纤维素酶产生菌的鉴定 2.3.1 菌株MA35的鉴定以MA35基因组DNA为模板,扩增菌株ITS序列,序列测定后进行BLAST搜索比对,结果显示MA35与Aspergillus niveus SRRC 333相似度达到99%,选取相似度高的菌株序列,利用Clustal X对菌株序列进行多重序列比对分析,通过MEGA 7.0中的邻接法构建出MA35的系统发育树(图 2),系统发育分析表明MA35属于Aspergillus niveus。

|
图 2 基于ITS基因序列对MA35构建的系统发育树
Fig. 2 Phylogenetic tree of MA35 based on ITS sequence analysis
|
对菌株MA35的培养特征持续性观察,菌株表面呈白色,具有辐射状沟纹和同心环纹,不透明,圆形,边缘整齐,无渗出液,凸起,无光泽,不光滑,不湿润,质地致密絮状,菌落反面淡黄色;随着菌落的老去,菌落会逐渐变黄(图 3-a, c)。扫描电子显微镜观察显示,菌株有透明的有隔菌丝,胞壁平行、光滑,分生孢子头大小不一;孢梗茎壁光滑;顶囊呈半球形(图 3-b, d)。MA35的形态学特征与《中国真菌志》中雪白曲霉的描述一致。分子生物学鉴定和形态学特征观察的结果表明,MA35属于真菌界、半知菌亚门、半知菌纲、壳霉目、杯霉科、曲霉属、雪白曲霉种。
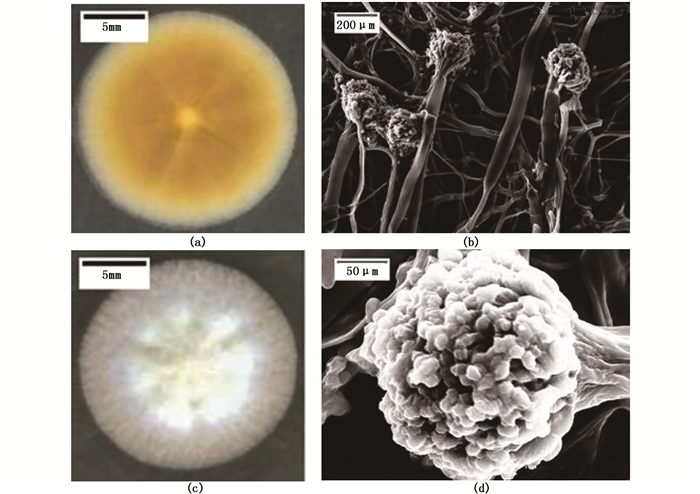
|
图 3 菌株MA35的形态学特性
Fig. 3 Morphological properties of strain MA35
(a)菌落表面;(b)菌丝;(c)菌落反面;(d)顶囊孢子 (a) Colony surface; (b) Mycelium; (c) Colony reverse; (d) Sporangiospore |
分别提取MA1、CI10菌株的基因组DNA,利用PCR扩增出相应的16S rDNA序列,进行测定分析,序列测定结果显示两者序列一致,再与GenBank数据库的序列进行BLAST搜索比对,结果显示MA1、CI10与Aeromonas veronii VM25相似度达到99%,MA1、CI10的系统发育树如图 4所示,系统发育分析表明MA1、CI10属于Aeromonas veronii。根据系统发育分析结果,将MA1、CI10统一命名为MC。

|
图 4 基于16S rDNA基因序列对MA1、CI10构建的系统发育树
Fig. 4 Phylogenetic tree of MA1 and CI10 based on 16S rDNA sequence analysis
|
菌株MC表面白色,不透明,圆形,边缘整齐,中央微凸起,无光泽,光滑,湿润,奶油状(图 5-a)。MC为革兰氏阴性短杆菌(图 5-b)。参照《伯杰名细菌鉴定手册》,鉴定结果表明,MC属于细菌界、变形菌门、γ-变形菌纲、弧菌目、弧菌科、气单胞菌属、维氏气单胞菌种。

|
图 5 菌株MC的形态学特性
Fig. 5 Morphological properties of strain MC
(a)菌落表面;(b)革兰氏染色 (a) Colony surface; (b) Gram's staining |
以羧甲基纤维素为唯一碳源,摇瓶培养细菌MC和真菌MA35(100 mL),在发酵24、48、72 h时分别测定菌株的发酵液(胞外)和菌体(胞内)纤维素酶活力,结果如图 6所示。
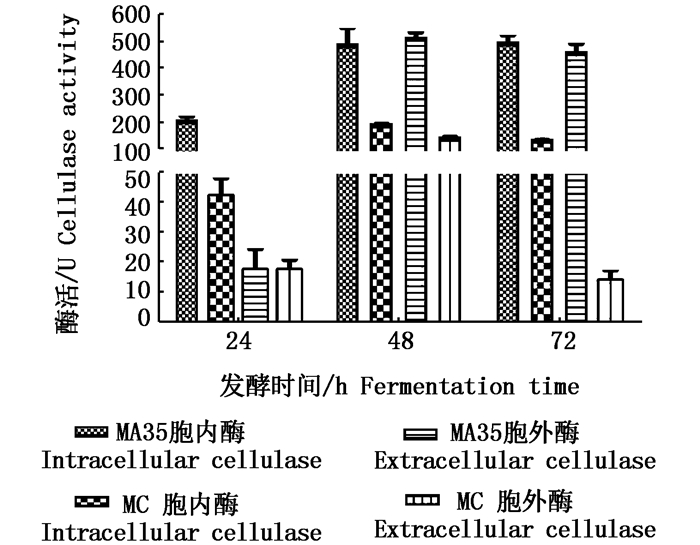
|
图 6 纤维素酶产生菌MC和MA35的发酵趋势
Fig. 6 Fermentation trend of cellulase-producing strains MC and MA35
|
由图 6可知,细菌MC和真菌MA35均能产生胞内和胞外纤维素酶,在不同的发酵时期(24 h、48 h、72 h),胞内纤维素酶的活性均高于胞外纤维素酶的活性,而且在发酵过程中,MA35比MC具有更高的胞外和胞内纤维素酶活。
3 讨论本研究以羧甲基纤维素为唯一碳源的选择性培养基,通过定性初筛及定量复筛的方案,从团头鲂和草鱼肠道获得了2株具有较高纤维素酶活性的菌株。经过分子鉴定及形态学观察的分析表明,来源于团头鲂的1株真菌MA35为雪白曲霉;来源于团头鲂的MA1和来源于草鱼的CI10实为同一种细菌MC,是维氏气单胞菌。进一步的摇瓶发酵趋势研究表明,雪白曲霉MA35,较维氏气单胞菌MC,在发酵过程具有更高的胞外和胞内纤维素酶活力。本实验采用了直接筛选和富集分离纯化再筛选的方案,为了避免菌群数量不够或者菌株调整期差异过大带来的影响。对于初筛的菌株进了革兰氏碘液染色和刚果红染色的透明圈法筛选,避免了两种筛选机制差异带来的少筛或者误筛的情况[24]。关于草鱼肠道纤维素酶产生菌的研究已有报道,如2006年SAHA从草鱼肠道筛选到了Bacillus circulans和Bacillus megaterium[5],2011年JIANG等分离出了Aeromonas hydrophila和Aeromonas veroni,并指出Aeromonas是草鱼肠道的主要菌群[25],2014年水生生物研究所的王微微从草鱼肠道分离出Aeromonas veronii、Aeromonas jandaei、Plesiomonas shigelloides、Enterobacter cloacae、Enterobacter aerogenes、Aeromonas hydrophila[4]。本实验也从草鱼肠道分离出了Aeromonas veronii,与JIANG等的研究结果一致。关于团头鲂肠道纤维素酶产生菌的研究,本研究是首创,并且分离出了Aspergillus niveus和Aeromonas veronii。团头鲂和草鱼肠道都出现了维氏气单胞菌,进一步验证了,维氏气单胞菌是草食性鱼肠道的主要纤维素酶产生菌。对两种菌株进行了发酵趋势的研究,发现两种菌株在48 h左右时均达到最大纤维素酶活,说明两种菌株都具有水解纤维素的能力,只是雪白曲霉的纤维素酶活更高。伴随着水产动物病害发生及抗生素滥用的问题,研究水产动物肠道内存在何种菌,是益生菌还是有害菌,具有何种消化酶能力,是否能减少抗生素使用、提高水产养殖水平的科学研究,越来越受到关注。因为益生菌作为饲料添加剂,能够控制疾病、提高免疫力、提供营养、促进消化能力和控制水质,在水产养殖业中广泛使用[26]。本研究从草鱼和团头鲂肠道筛选出来的雪白曲霉和维氏气单胞菌,都是纤维素酶产生菌,然而,有研究表明雪白曲霉和维氏气单胞菌都是致病菌[27-28],那么,对于依赖肠道纤维素酶产生菌消化纤维素的草食性鱼,这两种菌是益生菌还是有害菌,如何利用纤维素酶或者纤维素酶产生菌合理地养殖草食性鱼的问题,就成了一个值得研究的课题。本研究从团头鲂和草鱼肠道筛选到的纤维素酶产生菌,为后续研究草食性鱼的合理性养殖与膳食性饲料配比,提供了研究素材和科学基础。
| [1] |
SINGHANIA R R, SUKUMARAN R K, PATEL A K, et al. Advancement and comparative profiles in the production technologies using solid-state and submerged fermentation for microbial cellulases[J]. Enzyme and Microbial Technology, 2010, 46(7): 541-549. DOI:10.1016/j.enzmictec.2010.03.010 |
| [2] |
LIN H L, LI W G, GUO C H, et al. Advances in the study of directed evolution for cellulases[J]. Frontiers of Environmental Science & Engineering, 2011, 5(4): 519-525. |
| [3] |
MAKI M, LEUNG K T, QIN W S. The prospects of cellulase-producing bacteria for the bioconversion of lignocellulosic biomass[J]. International Journal of Biological Sciences, 2009, 5(5): 500-516. |
| [4] |
王微微, 吴山功, 邹红, 等. 草鱼肠道纤维素降解细菌的分离与鉴定[J]. 水生生物学报, 2014, 38(2): 291-297. WANG W W, WU S G, ZOU H, et al. Characterization of cellulose-decomposing bacteria in the intestine of grass carp, Ctenopharyngodon idella (Val.)[J]. Acta Hydrobiologica Sinica, 2014, 38(2): 291-297. DOI:10.7541/2014.42 |
| [5] |
SAHA S, ROY R N, SEN S K, et al. Characterization of cellulase-producing bacteria from the digestive tract of tilapia, Oreochromis mossambica (Peters) and grass carp, Ctenopharyngodon idella (Valenciennes)[J]. Aquaculture Research, 2006, 37(4): 380-388. DOI:10.1111/are.2006.37.issue-4 |
| [6] |
张传富, 顾文杰, 彭科峰, 等. 微生物纤维素酶的研究现状[J]. 生物信息学, 2007, 5(1): 34-36. ZHANG C F, GU W J, PENG K F, et al. Present situation of research on microbial cellulase[J]. China Journal of Bioinformatics, 2007, 5(1): 34-36. |
| [7] |
刘寒. 基于基因组学的团头鲂植食性机制研究[D]. 武汉: 华中农业大学, 2016. LIU H. Based on the genomics of Megalobrama amblycephala reveals the adaption to herbivorous diet[D]. Wuhan: Huazhong Agricultural University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10504-1016151564.htm |
| [8] |
吴兴兵, 许璞, 张伟明, 等. 7种重要经济鱼类的ISSR扩增结果分析[J]. 水生生物学报, 2008, 32(3): 445-448. WU X B, XU P, ZHANG W M, et al. Analysis of inter simple sequence repeat (ISSR) amplification results of seven fishes[J]. Acta Hydrobiologica Sinica, 2008, 32(3): 445-448. |
| [9] |
解绶启, 张文兵, 韩冬, 等. 水产养殖动物营养与饲料工程发展战略研究[J]. 中国工程科学, 2016, 18(3): 29-36. XIE S Q, ZHANG W B, HAN D, et al. Study on the developmental strategies for the engineering of aqua nutrition and feed[J]. Engineering Sciences, 2016, 18(3): 29-36. |
| [10] |
刘大森, 鞠涛, 杨方, 等. 纤维素酶在反刍动物饲料中的应用研究进展[J]. 东北农业大学学报, 2011, 42(6): 7-11. LIU D S, JU T, YANG F, et al. Applied research progress of cellulase in ruminant animal feed[J]. Journal of Northeast Agricultural University, 2011, 42(6): 7-11. |
| [11] |
LIU H, CHEN C H, GAO Z X, et al. The draft genome of blunt snout bream (Megalobrama amblycephala) reveals the development of intermuscular bone and adaptation to herbivorous diet[J]. Gigascience, 2017, 6(7): 1-13. |
| [12] |
WANG Y P, LU Y, ZHANG Y, et al. The draft genome of the grass carp (Ctenopharyngodon idellus) provides insights into its evolution and vegetarian adaptation[J]. Nature Genetics, 2015, 47(6): 625-632. DOI:10.1038/ng.3280 |
| [13] |
LI H, WU S G, WIRTH S, et al. Diversity and activity of cellulolytic bacteria, isolated from the gut contents of grass carp (Ctenopharyngodon idellus) (Valenciennes) fed on Sudan grass (Sorghum sudanense) or artificial feedstuffs[J]. Aquaculture Research, 2014, 47(1): 153-164. |
| [14] |
GUPTA P, SAMANT K, SAHU A. Isolation of cellulose-degrading bacteria and determination of their cellulolytic potential[J]. International Journal of Microbiology, 2012, 2012: 578925. |
| [15] |
KASANA R C, SALWAN R, DHAR H, et al. A rapid and easy method for the detection of microbial cellulases on agar plates using Gram's iodine[J]. Current Microbiology, 2008, 57(5): 503-507. DOI:10.1007/s00284-008-9276-8 |
| [16] |
TEATHER R M, WOOD P J. Use of Congo red-polysaccharide interactions in enumeration and characterization of cellulolytic bacteria from the bovine rumen[J]. Applied and Environmental Microbiology, 1982, 43(4): 777-780. |
| [17] |
GHOSE T K. Measurement of cellulase activities[J]. Pure and Applied Chemistry, 1987, 59(2): 257-268. DOI:10.1351/pac198759020257 |
| [18] |
FRANK J A, REICH C I, SHARMA S, et al. Critical evaluation of two primers commonly used for amplification of bacterial 16S rRNA genes[J]. Applied and Environmental Microbiology, 2008, 74(8): 2461-2470. DOI:10.1128/AEM.02272-07 |
| [19] |
WHITE T J, BRUNS T D, LEE S B, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//PCR-Protocols: A Guide to Methods and Applications. New York: Academic Press, Inc., 1994, 38: 315-322.
|
| [20] |
CHENG L D, JIANG X Y, TIAN Y M, et al. The goldfish hAT-family transposon Tgf2 is capable of autonomous excision in zebrafish embryos[J]. Gene, 2014, 536(1): 74-78. DOI:10.1016/j.gene.2013.11.084 |
| [21] |
SAITOU N, NEI M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [22] |
布坎南, 吉本斯. 伯杰细菌鉴定手册[M]. 8版. 北京: 科学出版社, 1984: 482-486. BUCHANAN R E, GIBBONS N E. Bergey's manual of determinative bacteriology[M]. 8th ed. Beijing: Science Press, 1984: 482-486. |
| [23] |
齐祖同, 孔华忠, 孙曾美. 中国真菌志.第五卷, 曲霉属及其相关有性型[M]. 北京: 科学出版社, 1997: 68-69. QI Z T, KONG H Z, SUN Z M. Flora fungorum sinicorum, volume 05, Aspergillus to teleomorphi cognati[M]. Beijing: Science Press, 1997: 68-69. |
| [24] |
MEDDEB-MOUELHI F, MOISAN J K, Beauregard M. A comparison of plate assay methods for detecting extracellular cellulase and xylanase activity[J]. Enzyme and Microbial Technology, 2014, 66: 16-19. DOI:10.1016/j.enzmictec.2014.07.004 |
| [25] |
JIANG Y, XIE C X, YANG G M. Cellulase-producing bacteria of Aeromonas are dominant and indigenous in the gut of Ctenopharyngodon idellus (Valenciennes)[J]. Aquaculture Research, 2011, 42(4): 499-505. DOI:10.1111/are.2011.42.issue-4 |
| [26] |
窦春萌, 左志晗, 刘逸尘, 等. 凡纳滨对虾肠道内产消化酶益生菌的分离与筛选[J]. 水产学报, 2016, 40(4): 537-546. DOU C M, ZUO Z H, LIU Y C, et al. Isolation and screeing of digestive enzyme producing probiotics from intestine of Litopenaeus vannamei[J]. Journal of Fisheries of China, 2016, 40(4): 537-546. |
| [27] |
秦启贤, 秦立模, 章强强. 临床真菌学[M]. 上海: 复旦大学出版社, 2001: 365. QIN Q X, QIN L M, ZHANG Q Q. Clinical mycology[M]. Shanghai: Fudan University Press, 2001: 365. |
| [28] |
马志宏, 杨慧, 李铁梁, 等. 西伯利亚鲟(Acipenser baerii)致病性维氏气单胞菌的分离鉴定[J]. 微生物学报, 2009, 49(10): 1289-1294. MA Z H, YANG H, LI T L, et al. Isolation and identification of pathogenic Aeromonas veronii isolated from infected Siberian sturgeon (Acipenser baerii)[J]. Acta Microbiologica Sinica, 2009, 49(10): 1289-1294. DOI:10.3321/j.issn:0001-6209.2009.10.017 |
2. Key Laboratory of Aquatic Genetic Resources and Utilization Ministry of Agriculture, Shanghai Ocean University, Shanghai 201306, China
 2018,
Vol. 27
2018,
Vol. 27


