2. 上海海洋大学 农业部种质资源与利用重点开放实验室, 上海 201306
转座子(transposon)是一类能够在基因组中移动的遗传因子,其过程称为转座(transposition)。随着多个基因组测序计划的进行,原核和真核生物界陆续发现众多转座子,转座子因此被认为是自然界变异和进化的重要推动力之一[1-2]。转座子可介导基因组插入突变、基因重排、基因修饰及基因治疗[3-6],因此有望开发成为重要的遗传学、生物学工具[7-9]。
DNA转座子在宿主基因组通过“剪切-粘贴”的机制变更位置[10-11]。自主的DNA转座子能编码活性转座酶[12-13]以介导转座过程,但由于受到来自宿主的钝化作用,迄今自然界生物体内仅发现极少数的天然活性转座子,如Tol2、PiggyBac、Tgf2等[14-16]。hAT超家族的Tgf2转座子首先在我国的金鱼(Carassius auratus)中发现,它存在于草金、鹤顶红、硫金等多个品系中,能编码完整、具有活性的转座酶,并在斑马鱼及团头鲂中介导转座[17],是具有潜力的鱼类遗传学工具之一。前期研究显示:大肠杆菌Rosetta(DE3) 中以低温(22 ℃)、低吸光度(OD600≈0.3) 条件下诱导,可表达可溶、活性的Tgf2转座酶[18]。鉴于大肠杆菌BL21菌株表达外源蛋白有更高的效率和稳定性,本研究根据大肠杆菌的密码子偏好性,优化并合成了金鱼Tgf2转座酶编码区基因,与pET-28a(+)载体重组,采用大肠杆菌BL21(DE3) 作为宿主菌株,以期获得Tgf2转座酶的高效表达,为Tgf2转座酶的发酵生产及应用创造先决条件。
1 材料与方法 1.1 质粒、菌种和试剂pET-28a(+)为本实验室保存;BL21(DE3)、质粒小提试剂盒、DNA分子量marker以及T4 DNA连接酶购自天根生化科技(北京)有限公司;蛋白质分子量marker购自上海生工生物技术有限公司;其他试剂均为国产分析纯。
1.2 Tgf2转座酶的密码子优化参考编码Tgf2转座酶的cDNA序列(JN886591),根据大肠杆菌密码子的偏好性对其密码子进行优化,同时在5′端和3′端分别添加BamH Ⅰ和Xho Ⅰ酶切位点,由南京金斯瑞生物科技有限公司合成,优化后的Tgf2转座酶基因命名为Tgf2TP Plus。
1.3 重组表达载体pET-28a(+)-Tgf2TP Plus的构建pET-28a(+)采用BamH Ⅰ和Xho Ⅰ双酶切,经37 ℃孵育3 h,琼脂糖凝胶电泳检测后回收DNA。回收的酶切产物通过琼脂糖凝胶电泳检测。Tgf2TP Plus和pET-28a(+)回收片段通过T4 DNA连接酶,在16 ℃反应4 h,构建重组载体pET-28a(+)-Tgf2TP Plus。将连接产物转化到大肠杆菌感受态细胞DH5α中,37 ℃培养过夜。通过菌落PCR、琼脂糖凝胶电泳检测重组表达载体。
1.4 阳性克隆菌的筛选和鉴定转化成功的DH5α细胞在LB培养基中37 ℃摇床培养约12 h,离心得到菌体后,通过质粒小提试剂盒提取重组表达载体pET-28a(+)-Tgf2TP Plus,将重组载体转化到BL21(DE3) 感受态细胞中,LB液体培养基培养1 h后涂布于含有卡那霉素抗性的固体LB培养基上,37 ℃过夜培养。
选择长势优良的单菌落,利用引物Tgf2TP Plus -F(5′-TGTTAGCAGCCGGATCTCA-3′)和Tgf2TP Plus -R(5′-ACTGGTGGACAGCAAATGG-3′)进行PCR扩增。PCR反应体系(20 μL):上下游引物(10 μmol/L)各1 μL;DNA模板1 μL;10×PCR Buffer 2 μL;dNTPs 2 μL;Taq DNA聚合酶(2.5 U/μL)0.5 μL;加无菌水补足20 μL。PCR反应条件:94 ℃预变性5 min、94 ℃变性30 s、55 ℃退火30 s、72 ℃延伸1 min、30个循环,72 ℃延迟30 s。PCR产物用琼脂糖凝胶电泳检测后,挑选阳性克隆菌落送上海生工生物工程有限公司测序。
1.5 重组蛋白的诱导表达将测序确证的重组单菌落BL21(DE3) 在含有卡那霉素抗性的LB培养液中、37 ℃摇床培养(220 r/min)至OD600≈0.5,加入终浓度为1 mmol/L的异丙基-β-D-硫代半乳糖苷(IPTG),在37 ℃、150 r/min振摇培养。诱导结束后,收集菌液于离心管中,于5 000 r/min离心10 min,收集菌体,重悬于磷酸盐缓冲液中(pH 7.4),用于SDS-PAGE分析(浓缩胶5%,分离胶12%),利用BandScan软件对重组蛋白进行定量分析。
1.6 重组蛋白的纯化及鉴定IPTG诱导宿主菌表达4 h、12 000 r/min离心10 min后收集菌体,磷酸盐缓冲液重悬,在冰上进行超声波破碎30 min (功率100 W,超声破碎时间3 s,间隔6 s)。细菌破碎液以4 ℃、12 000 r/min离心20 min,收集上清。上清液通过0.22 μm滤膜过滤,采用HisTrapTM HP(GE Healthcare)亲和层析,流速为1 mL/min,平衡缓冲液为20 mmol/L磷酸盐、0.5 mol/L NaCl、20 mmol/L咪唑,结合缓冲液为20 mmol/L磷酸盐、0.5 mol/L NaCl、40 mmol/L咪唑,洗脱缓冲液为20 mmol/L磷酸盐、0.5 mol/L NaCl、500 mmol/L咪唑,根据紫外吸收值(OD280)收集目的蛋白组分。
收集的洗脱液通过SDS-PAGE分析,从SDS-PAGE凝胶上切割合适的条带,送至北京华大蛋白质研究中心有限公司进行MALDI-(TOF)/TOF串联质谱鉴定。
1.7 Tgf2转座酶的体外DNA结合活性50 bp核苷酸探针L50(5′-CAGAGGTGTAAAAGTACTTAAGTAATTTTACTTGATTACTGTACTTAAGT-3′), 其序列取自Tgf2转座子左臂反向重复末端。将合成的正义链和反义链均以50 μmol/L浓度混合,加入DNA寡核苷酸退火缓冲液(碧云天生物技术有限公司,中国南通),混匀后放入PCR仪进行退火反应。退火步骤:95 ℃变性2 min;每90 s下降1 ℃、降至25 ℃(约90 min)。探针溶液于-20 ℃保存。
采用Superdex200葡聚糖层析柱,利用分子排阻色谱法,0.5 mol/L NaCl磷酸盐缓冲液洗脱,流速为0.5 mL/min。分别以重组蛋白和探针为空白对照,用两种不同浓度的探针与转座酶混合(Tgf2转座酶与探针L50的摩尔浓度比分别为1: 0.67和1: 2),室温孵育20 min后上样,根据紫外吸收值(OD280, OD260)的变化,判断转座酶是否具有体外DNA结合活性[19-21]。
2 结果与分析 2.1 Tgf2转座酶的密码子优化不同细胞内可用tRNAs量的差异,导致同一密码子在不同生物体的利用率不同,即密码子的偏好性,它是采用原核生物表达真核基因时重要的影响因素之一。Tgf2转座子基因来源于金鱼(C. auratus),其编码的转座酶(Tgf2TP)不但使用了琥珀密码子(UAG)作为终止密码子,还含有多个大肠杆菌难以识别的稀有密码子,如AGG,AGA,AUA,CUA,CGA,CGG,CCC和UCG(图 1)。经稀有密码子分析软件(GenScript Rare Codon Analysis Tool)分析表明:Tgf2TP的密码子适应指数(Codon adaptation index,CAI)为0.61,GC含量为45.62%,密码子使用频率分布系数(Codon frequency distribution,CFD)为13%。依据大肠杆菌密码子的偏好性、优化得到的Tgf2TP Plus对原有的终止密码子和绝大多数稀有密码子进行了同义替换(图 1)。从而,使Tgf2TP Plus的CAI指数为0.86,达到理想值区间(0.8~1.0);GC含量为53.11%,符合理想值区间(30%~70%),避免了低频率密码子的使用;终止密码子替换为UGA。

|
图 1 Tgf2TP和密码子优化后Tgf2TP Plus的序列比对
Fig. 1 Alignment of Tgf2TP and Tgf2TP Plus derived from codon optimization
图中阴影标识不同的稀有密码子及其优化,终止密码子以黑框标识 In the figure, the different codons and their optimization are identified by shadows and the termination codon is identified by a black box |
利用基因全合成方法获得Tgf2TP Plus,借助添加的5′端BamH Ⅰ和3′端Xho Ⅰ酶切位点将Tgf2TP Plus连接到pET-28a(+)上,构建重组表达载体pET-28a(+)-Tgf2TP Plus。将重组载体转化到BL21(DE3) 感受态细胞后,利用卡那霉素抗性平板筛选转化子。挑选长势优良的单菌落用于PCR扩增,琼脂糖凝胶电泳结果(图 2)显示:约1 700 bp处呈现明显的亮条带。阳性单菌落的测序结果表明:Tgf2TP Plus的序列正确、并且正确插入pET-28a(+)载体,未出现阅读框的移码或改变,Tgf2转座酶将融合表达His-Tag组氨酸标签(图 1),利于后期的蛋白质纯化。
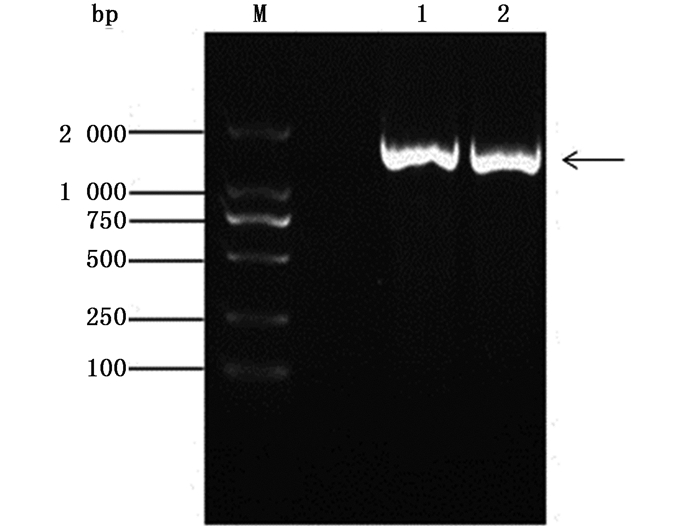
|
图 2 重组质粒pET28a-Tgf2TP Plus的PCR检测
Fig. 2 PCR detection of recombinant plasmid pET28a-Tgf2TP Plus
M. DNA标准分子量marker; 1-2. PCR检测产物 M. DNA marker; 1-2. Product of PCR detection |
将测序正确的重组单菌落BL21(DE3)/pET-28a(+)-Tgf2TP Plus在LB抗性培养液中,37 ℃培养至OD600≈0.5加入IPTG(1.0 mmol/L),诱导表达4 h后,收集菌液,离心重悬后,采用SDS-PAGE分析。结果显示(图 3):与诱导前相比较,诱导后的菌体蛋白在约70 ku处表达量明显增加,即重组蛋白得以表达。经BandScan软件分析,表达的重组蛋白占菌体总蛋白约8.8%。前期研究的Tgf2转座酶表达条件为:低温(22 ℃)培养、低吸光度诱导(OD600≈0.3) [18]。此条件下,大肠杆菌Rosetta(DE3) 表达重组蛋白量占菌体总蛋白的约4.2%。
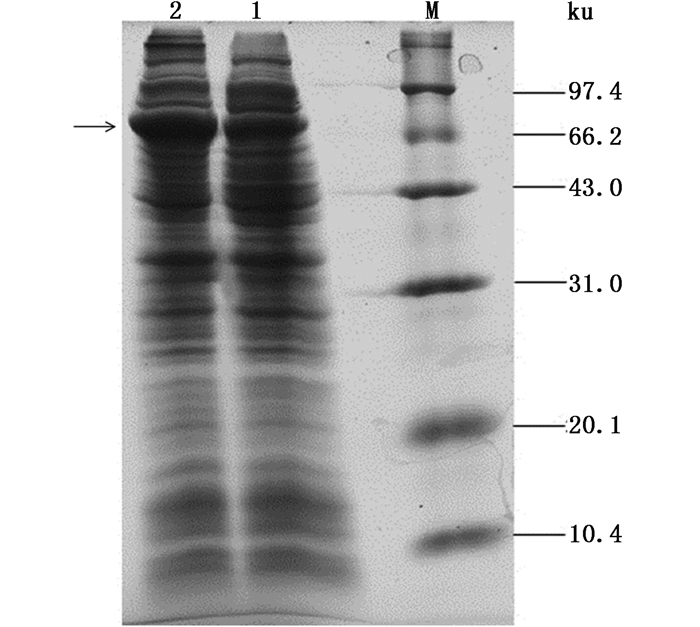
|
图 3 重组蛋白的SDS-PAGE分析
Fig. 3 SDS-PAGE analysis of the recombinant protein
M.低分子量蛋白质marker; 1.未诱导、含有重组质粒的BL21(DE3) 总蛋白; 2.经诱导、含有重组质粒的BL21(DE3) 总蛋白 M. Protein molecular weight marker; 1. Total proteins of bacteria BL21(DE3) with recombinant plasmid without induction; 2. Total proteins of bacteria BL21(DE3) with recombinant plasmid under induced condition |
经过密码子的优化,金鱼Tgf2转座酶在BL21(DE3) 宿主菌中得以表达,表达量从之前的约4.2%提高到了约8.8%,即表达量提高了一倍。培养温度提高到37 ℃,可以缩短发酵周期;对数生长中期诱导能降低发酵过程中染菌的风险。这些为后续发酵生产金鱼Tgf2转座酶提供了保障。
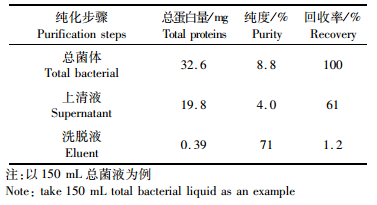
2.4 重组蛋白的纯化收集诱导后培养的菌体、采用超声波破碎后离心得到上清液,通过HisTrapTM HP亲和柱纯化,收集的洗脱液进行SDS-PAGE分析,结果如图 4所示,上清液经亲和层析可得到纯度较高的重组蛋白。通过BandScan分析,从总菌体出发,较纯重组蛋白(>70%)的回收率约为0.28%(表 1)。
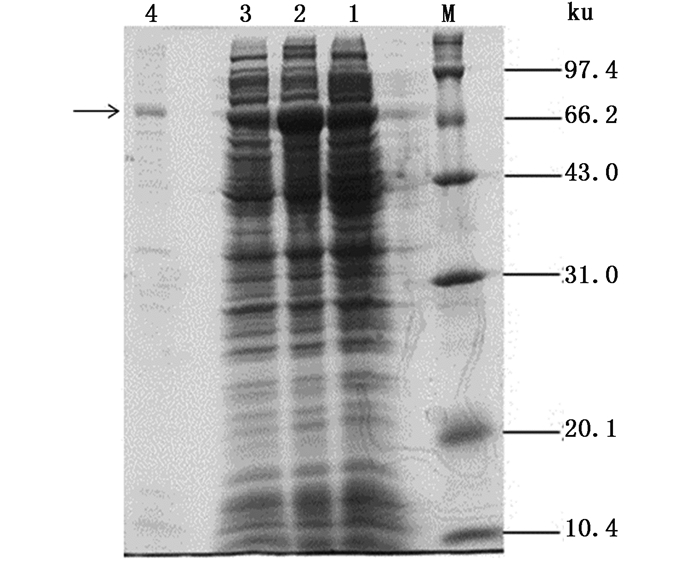
|
图 4 重组蛋白纯化过程的SDS-PAGE分析
Fig. 4 SDS-PAGE analysis of the recombinant protein purification
M.低分子量蛋白质marker; 1.未诱导、含有重组质粒的BL21(DE3) 总蛋白; 2.经诱导、含有重组质粒的BL21(DE3) 总蛋白; 3.经诱导、含有重组质粒的细胞经过超声破壁后的上清液; 4.经过亲和纯化后的重组蛋白 M. Protein molecular weight marker; 1. Total proteins with recombinant plasmid without induction; 2. Total proteins with recombinant plasmid with induction; 3. Supernatant portion of induced bacteria with recombinant plasmid after ultrasonictraetment; 4. Recombinant proteins by affinity chromatography purification |
|
表 1 重组蛋白的纯化 Tab.1 Purification of the recombinant proteins |
从SDS-PAGE胶上切割纯化后的重组蛋白,进行质谱分析,结果如图 5所示:二级质谱检测到的肽片段序列(图 5a中阴影)与GenBank中的Tgf2转座酶相比对,相同片段覆盖率达到64.6%(图 5未显示)。其中3处寡肽段(图 5a中黑框)的MS/MS质谱图如图 5b,c,d所示。质谱结果表明,表达的重组蛋白为金鱼Tgf2转座酶。
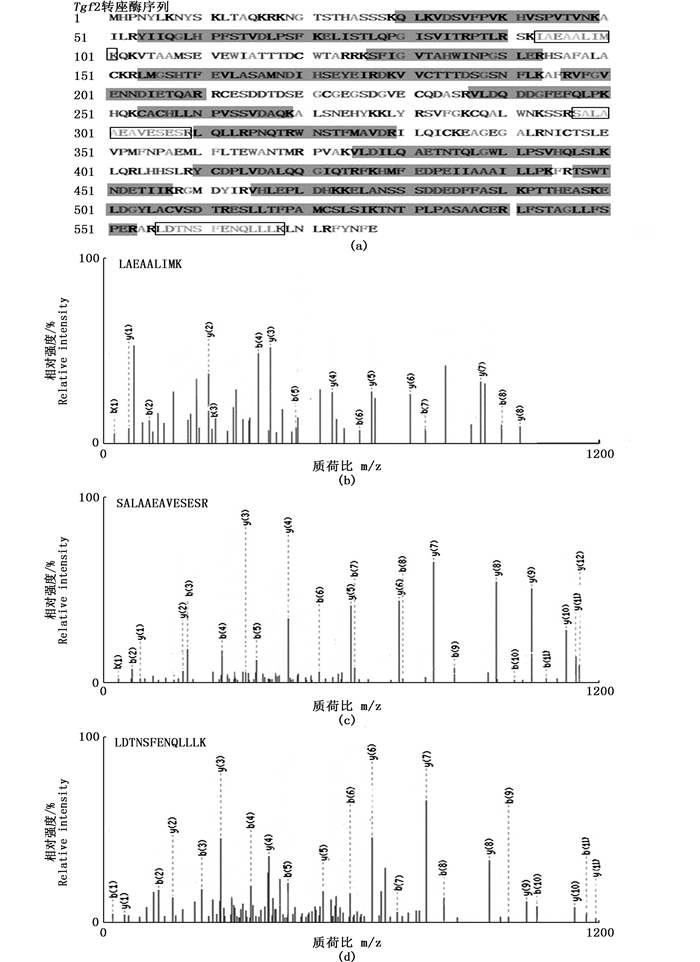
|
图 5 Tgf2转座酶的质谱鉴定
Fig. 5 Identification of Tgf2 transposase by MALDI-TOF/TOF tandem mass spectrometry
(a)推测重组Tgf2转座酶的氨基酸序列; (b-d)串联质谱测序的部分肽段 (a) Putative amino acid sequence of recombinant Tgf2 transposase; (b-d) MS/MS spectra of some peptide fragments |
活性转座酶能识别转座子的左右臂序列、并与之结合,以启动后续的DNA切割、靶位点的整合等转座过程[22-23]。本实验采用的DNA探针L50包含Tgf2左臂反向重复末端,其分子筛层析洗脱曲线呈现核酸洗脱峰(图 6b-Ⅰ, b-Ⅱ),出峰时间约为28 min且OD260 > OD280;单独孵育的重组Tgf2转座酶呈现蛋白洗脱峰(图 6a),出峰时间约为18 min且OD260与OD280相当。
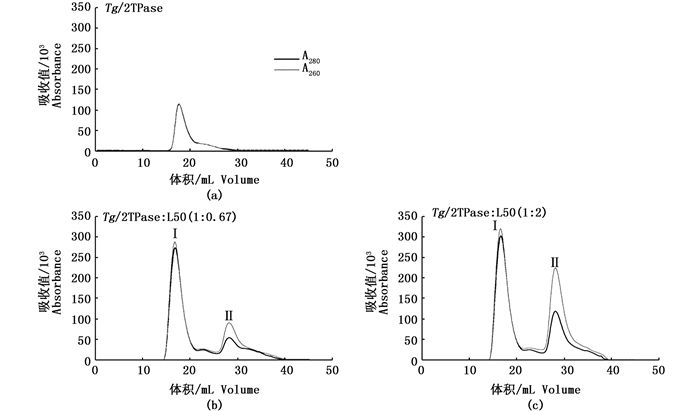
|
图 6 Tgf2转座酶的体外DNA结合活性
Fig. 6 DNA binding activity of Tgf2TP
(a) Tgf2转座酶的洗脱图谱; (b, c) Tgf2转座酶和DNA探针(摩尔比分别为1: 0.67和1: 2) 的洗脱图谱 (a) Elution profile of Tgf2TP alone; (b, c) Elution profiles of incubation mixtures of Tgf2TP and L50 of different proportion(b.1: 0.67, c.1: 2) |
当Tgf2转座酶与探针L50分别以不同摩尔浓度比(1: 0.67和1: 2) 混合孵育,随后的洗脱曲线(图 6b-Ⅰ,c-Ⅰ)都显示:约为18 min时出现的蛋白洗脱峰增高显著,此结果与Hermes转座酶的研究结果一致[20]。hAT家族转座酶以多聚体存在,多聚体通常由若干个二聚体组成空腔结构,以容纳DNA[20],故蛋白与核酸形成复合物的层析行为可能更符合蛋白质特征。
3 讨论本研究通过优化金鱼Tgf2转座酶编码区密码子,构建重组载体pET-28a(+)-Tgf2TP Plus,转化至大肠杆菌BL21(DE3) 细胞中;大肠杆菌培养温度为37 ℃、培养至OD600≈0.5时诱导,菌体中含有8.8%的重组蛋白,其中上清液含有4.0%重组蛋白。通过亲和层析从上清液中纯化得到较高纯度(>70%)的重组蛋白,采用MALDI-TOF/TOF串联质谱对重组蛋白进行鉴定,确定表达的重组蛋白即为金鱼Tgf2转座酶。利用分子筛层析确定了重组金鱼Tgf2转座酶能识别、结合含Tgf2转座子特异性亚末端重复序列的DNA探针,即具有体外DNA结合活性。
转座酶的活性对转座子的功能起到至关重要的作用,如转座子Sleeping beauty和Frog Prince转座活性的唤醒均有赖于其编码转座酶分子水平的重建和关键结构域的改造[24-25]。转座酶能够识别DNA序列,催化切割及整合,使转座得以进行,故重组转座酶的开发是对转座子应用的有益补充。和mRNA转染相比,应用转座酶转染具有的潜在优势为:(1) 避免可能的转座延迟,以提高转座效率;(2) 适于异源系统。在众多的转座子中,报道显示重组Ac、Hermes、Tol2、Tgf2转座酶具有生物活性[18, 26-27],本研究实践了通过优化密码子改善重组Tgf2转座酶表达的方法,并提高了目的蛋白的产量,为后续发酵生产Tgf2转座酶提供保障,也为Tgf2转座酶作为工具酶的应用提供支撑。
| [1] | HUANG C R L, BURNS K H, BOEKE J D. Active transposition in genomes[J]. Annual Review of Genetics, 2012, 46(1): 651–675. DOI:10.1146/annurev-genet-110711-155616 |
| [2] | FESCHOTTE C, PRITHAM E J. DNA transposons and the evolution of eukaryotic genomes[J]. Annual Review of Genetics, 2007, 41(1): 331–368. DOI:10.1146/annurev.genet.40.110405.090448 |
| [3] | KAWAKAMI K. Tol2:a versatile gene transfer vector in vertebrates[J]. Genome Biology, 2007, 8(s1): S7. |
| [4] | NAGAYOSHI S, HAYASHI E, ABE G, et al. Insertional mutagenesis by the Tol2 transposon-mediated enhancer trap approach generated mutations in two developmental genes:tcf7 and synembryn-like[J]. Development, 2008, 135(1): 159–169. |
| [5] | HE X Z, LI J, LONG Y, et al. Gene transfer and mutagenesis mediated by Sleeping Beauty transposon in Nile tilapia (Oreochromis niloticus)[J]. Transgenic Research, 2013, 22(5): 913–924. DOI:10.1007/s11248-013-9693-8 |
| [6] | FURUSHIMA K, JANG C W, CHEN D W, et al. Insertional mutagenesis by a hybrid PiggyBac and Sleeping Beauty transposon in the rat[J]. Genetics, 2012, 192(4): 1235–1248. DOI:10.1534/genetics.112.140855 |
| [7] | GEURTS A M, WILBER A, CARLSON C M, et al. Conditional gene expression in the mouse using a Sleeping Beauty gene-trap transposon[J]. BMC Biotechnology, 2006, 6(1): 1. DOI:10.1186/1472-6750-6-1 |
| [8] | FROSCHAUER A, SPROTT D, GERWIEN F, et al. Effective generation of transgenic reporter and gene trap lines of the medaka(Oryzias latipes)using the Ac/Ds transposon system[J]. Transgenic Research, 2012, 21(1): 149–162. DOI:10.1007/s11248-011-9514-x |
| [9] | CLARK K J, GEURTS A M, BELL J B, et al. Transposon vectors for gene-trap insertional mutagenesis in vertebrates[J]. Genesis, 2004, 39(4): 225–233. DOI:10.1002/(ISSN)1526-968X |
| [10] | BENJAK A, FORNECK A, CASACUBERTA J M. Genome-wide analysis of the "cut-and-paste" transposons of grapevine[J]. PLoS One, 2008, 3(9): e3107. DOI:10.1371/journal.pone.0003107 |
| [11] | ZHANG J B, PETERSON T. Transposition of reversed Ac element ends generates chromosome rearrangements in maize[J]. Genetics, 2004, 167(4): 1929–1937. DOI:10.1534/genetics.103.026229 |
| [12] | CHENG L D, JIANG X Y, TIAN Y M, et al. The goldfish hAT-family transposon Tgf2 is capable of autonomous excision in zebrafish embryos[J]. Gene, 2014, 536(1): 74–78. DOI:10.1016/j.gene.2013.11.084 |
| [13] | CLARK K J, CARLSON D F, LEAVER M J, et al. Passport, a native Tc1 transposon from flatfish, is functionally active in vertebrate cells[J]. Nucleic Acids Research, 2009, 37(4): 1239–1247. DOI:10.1093/nar/gkn1025 |
| [14] | KOGA A, SUZUKI M, INAGAKI H, et al. Transposable element in fish[J]. Nature, 1996, 383(6595): 30. |
| [15] | FRASER M J, SMITH G E, SUMMERS M D. Acquisition of host cell DNA sequences by baculoviruses:relationship between host DNA insertions and FP mutants of Autographa californicaand Galleria mellonella nuclear polyhedrosis viruses[J]. Journal of Virology, 1983, 47(2): 287–300. |
| [16] |
邹曙明, 杜雪地, 袁剑, 等. 金鱼hAT家族转座子Tgf2的克隆及其结构[J].
遗传, 2010, 32(12): 1263–1268.
ZOU S M, DU X D, YUAN J, et al. Cloning of goldfish hAT transposon Tgf2 and its structure[J]. Hereditas, 2010, 32(12): 1263–1268. |
| [17] | JIANG X Y, DU X D, TIAN Y M, et al. Goldfish transposase Tgf2 presumably from recent horizontal transfer is active[J]. The FASEB Journal, 2012, 26(7): 2743–2752. DOI:10.1096/fj.11-199273 |
| [18] | XU H L, SHEN X D, HOU F, et al. Prokaryotic expression and purification of soluble goldfish Tgf2 transposase with transposition activity[J]. Molecular Biotechnology, 2015, 57(1): 94–100. DOI:10.1007/s12033-014-9805-6 |
| [19] | SHIBANO T, TAKEDA M, SUETAKE I, et al. Recombinant Tol2 transposase with activity in Xenopus embryos[J]. FEBS Letters, 2007, 581(22): 4333–4336. DOI:10.1016/j.febslet.2007.08.004 |
| [20] | HICKMAN A B, EWIS H E, LI X H, et al. Structural basis of hAT transposon end recognition by Hermes, an octameric DNA transposase from Musca domestica[J]. Cell, 2014, 158(2): 353–367. DOI:10.1016/j.cell.2014.05.037 |
| [21] | JIANG X Y, HOU F, SHEN X D, et al. The N-terminal zinc finger domain of Tgf2 transposase contributes to DNA binding and to transposition activity[J]. Scientific Reports, 2016, 6: 27101. DOI:10.1038/srep27101 |
| [22] | RICE P A, BAKER T A. Comparative architecture of transposase and integrase complexes[J]. Nature Structural Biology, 2001, 8(4): 302–307. DOI:10.1038/86166 |
| [23] | ESSERS L, ADOLPHS R H, KUNZE R. A highly conserved domain of the maize activator transposase is involved in dimerization[J]. The Plant Cell, 2000, 12(2): 211–223. DOI:10.1105/tpc.12.2.211 |
| [24] | IVICS Z, HACKETT P B, PLASTERK R H, et al. Molecular reconstruction of Sleeping Beauty, a Tc1-like transposon from fish, and its transposition in human cells[J]. Cell, 1997, 91(4): 501–510. DOI:10.1016/S0092-8674(00)80436-5 |
| [25] | GALLARDO-GÁLVEZ J B, MÉNDEZ T, BÉJAR J, et al. Endogenous transposases affect differently Sleeping Beauty and Frog Prince transposons in fish cells[J]. Marine Biotechnology, 2011, 13(4): 695–705. DOI:10.1007/s10126-010-9331-x |
| [26] | GRABUNDZIJA I, IRGANG M, MÁTÉS L, et al. Comparative analysis of transposable element vector systems in human cells[J]. Molecular Therapy, 2010, 18(6): 1200–1209. DOI:10.1038/mt.2010.47 |
| [27] | NI J, WANGENSTEEN K J, NELSEN D, et al. Active recombinant Tol2 transposase for gene transfer and gene discovery applications[J]. Mobile DNA, 2016, 7: 6. DOI:10.1186/s13100-016-0062-z |
2. Key Laboratory of Aquatic Genetic Resources and Utilization, Ministry of Agriculture, Shanghai 201306, China
 2017,
Vol. 26
2017,
Vol. 26


