小黄黝鱼(Micropercops swinhonis)隶属于鲈形目(Perciformes)虾虎鱼亚目(Gobioidei)沙塘鳢科(Eleotridae)小黄黝鱼属(Micropercops),是一种小型底栖淡水鱼类,为中国特有种,分布于长江水系及其以北各大河流中,直至黑龙江。动物志[1]记载小黄黝鱼广泛分布于中国各大水系,但经过野外采样调查和询问当地鱼类爱好者发现闽江、珠江水系均未发现小黄黝鱼,只发现萨氏华黝鱼(Sineleotris saccharae),这两种鱼形态和遗传都有很大差别,动物志可能根据以前资料[2],把侧扁黄黝鱼(Hypseleotris compressocephalus)误当作小黄黝鱼,侧扁黄黝鱼是萨氏华黝鱼的同物异名[1]。目前还没有关于小黄黝鱼种群遗传的分子方面研究,其种群结构以及遗传多样性情况不明,比如小黄黝鱼分为几个地理种群、有效种群的大小及种群遗传多样性情况等有待研究。另外根据伍汉霖教授口述,他与易伯鲁教授曾经探讨中国西南地区小黄黝鱼与其它地区种群存在的形态差异,可能有隐秘种存在,但也未有文章报道(伍汉霖,个人通讯)。
线粒体DNA具有进化速率快、严格按照母系遗传等特点[3-4],本文使用线粒体Cyt b和D-loop基因位点分析不同水系小黄黝鱼的种群结构和遗传分化等方面问题。探讨小黄黝鱼种群可以分为几支,各支之间差异情况以及探讨小黄黝鱼遗传多样性及种群历史动态。
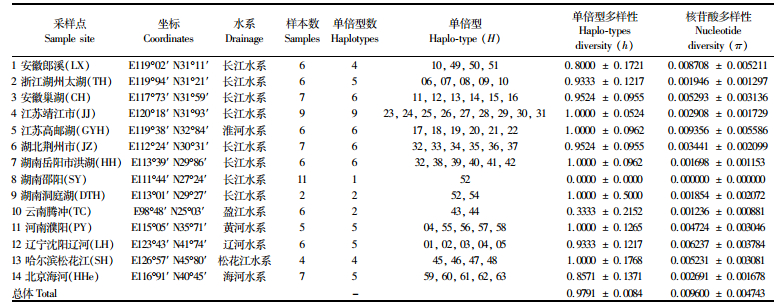
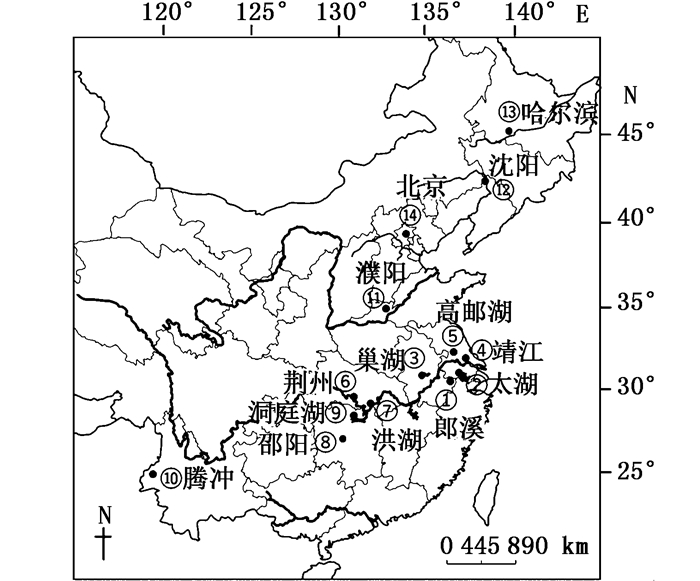
1 材料与方法 1.1 样本采集小黄黝鱼样本采自14个地区共88尾(表 1,图 1),分别采自松花江(SHJ)-哈尔滨、辽河(LH)-沈阳、海河(HHe)-北京、黄河下游-濮阳(PY)、高邮湖(GYH)、长江水系[邵阳资水(SY)、洞庭湖(DTH)、洪湖(HH)、荆州(JZ)、靖江市(JJ)、巢湖(CH)、太湖(TH)、郎溪(LX)]、云南腾冲(TC)。本文每个采样点使用样品数量较少,但是如洞庭湖、洪湖、荆州的采样点相距较近,都属于洞庭湖周边水系,因此同一水系的样本数量充足。样本经鉴定后,取新鲜肌肉保存于95%的乙醇中。标本现存于上海海洋大学标本室。
|
表 1 小黄黝鱼的样本信息及各种群遗传多样性 Tab.1 Sampling information and genetic diversity of M. swinhonis |
|
图 1 小黄黝鱼样本采集地点
Fig. 1 Sampling sites of M. swinhonis
|
用上海生工生物工程公司DNA抽提试剂盒提取DNA。线粒体Cyt b和D-loop基因扩增引物为自己设计CytbL:CTAAAACACCGCAACCCACA,CytbR:CAGTGGTGGGAGTTAAAATCTCC;DLoopL:CAAGTAACMTGCAATAGTAGCTCAGCG,DLoopR:TTAAA GTCAGGACCAAGCTTTTGTGC。25 μL的PCR反应体系包括PCR Master Mix 12.5 μL(含MgCl2、dNTPMix和Taq酶)、上下游引物各1.0 μL (55 ng/μL)、DNA样品1.0 μL,用ddH2O补足25 μL。反应程序:94 ℃预变性4 min,扩增35个循环(94 ℃变性30 s,60 ℃退火45 s,72 ℃延伸1 min), 最后72 ℃延伸8 min,4 ℃保存。PCR产物用1%琼脂糖电泳检测样品扩增情况,送至上海生工生物工程公司纯化和测序。
1.3 数据处理用MEGA 5.0[5]对校对后的序列进行比对剪切;用DnaSP 5.10[6]软件计算群体单倍型多样性(h)核苷酸多态性(π);用Arlequin ver 3.0[7]软件包进行分子方差分析(AMOVA)(重复10 000次抽样检测显著性程度)和种群间遗传分化指数(Fst)的计算;用Arlequin 3.0[7]里的模块Tajima’s D[8]和Fu’s FS[9]中性检验及核苷酸不配对分布(Mismatch distribution)推断种群的历史动态。通过Network 4.6.1.0[10]构建单倍型网络图。利用jModeltest 0.1[11-12]以BIC(Bayesian Information Criterion)为标准确定最佳模型为HKY+G,采用贝叶斯[13]方法建立BI树,运算1 000万代,每1 000次取样一次,去掉运算开始25%的不可信区域,直到链的收敛分离频率平均标准方差小于0.01停止运算,用FigTree 1.4.0[14]查看并编辑贝叶斯树。利用SAMOVA[15]分析做10 000次交换运算,SAMOVA分析共重复100次以上,清除不同起始条件对结果的影响[16]。
2 结果 2.1 遗传多样性对所测得的线粒体Cyt b (1 320 bp)和 D-loop (839 bp)序列进行拼接和人工校对,拼接后长度为2 159 bp,检测到133个变异位点,占6.16%,共发现63个单倍型。整个群体单倍型多样性h =0.979 1,核苷酸多样性π=0.009 6(表 1)。样本碱基含量为T(32.24%)、C(24.43%)、A(27.91%)、G(15.42%)具有明显的反G偏向性。结果显示小黄黝鱼具有较高的单倍型多样性(除云南为引种和邵阳只有一个单倍型),都大于0.800 0±0.172 1;具有较低的核苷酸多样性, 都小于0.01。
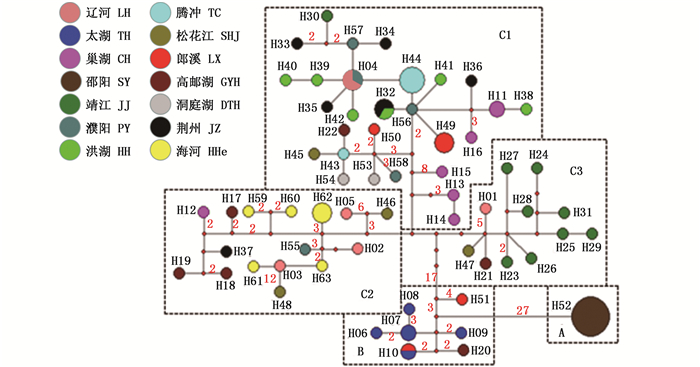
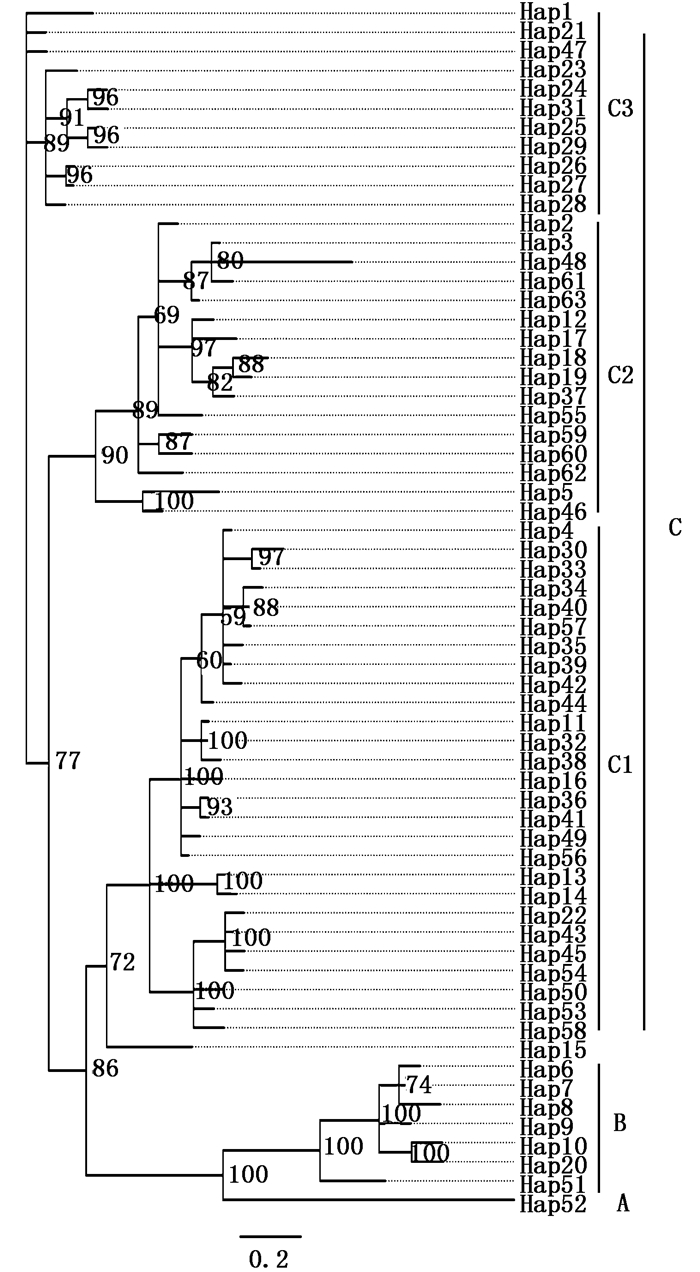
2.2 单倍型网络图和谱系分析88条小黄黝鱼Cyt b和D-loop 串联序列共检测到63个单倍型(表 1),Hap04、Hap10、Hap32存在共享情况:Hap04为辽河与黄河(濮阳)样本所共享;Hap10为太湖和郎溪所共享;Hap32为洪湖和长江(荆州)所共享。构建的群体单倍型网络图(图 2)显示,研究小黄黝鱼群体大致可分为A、B、C三个谱系:A谱系为邵阳群体,所有样本共享单倍型H52(11个样本);B谱系包括H51(长江水系郎溪)和太湖所有样本;C谱系包括除太湖、长江水系资水(邵阳群体)和H51外所有群体,C谱系又可分为C1、C2、C3三个亚谱系:C1主要以长江水系样本为主包括长江水系(洞庭湖、荆州、洪湖、靖江、巢湖、郎溪)和松花江(哈尔滨)、辽河水系(沈阳)、黄河水系(濮阳)、高邮湖、云南腾冲这些地区的少数样本;C2主要包括北方水系(松花江-哈尔滨、辽河-沈阳、海河-北京、黄河-濮阳、高邮湖)样本和少数长江水系(巢湖、荆州)样本;C3主要包括长江水系(靖江样本)和松花江(哈尔滨)、辽河(沈阳)、高邮湖个别样本。贝叶斯(BI)系统发育树(图 3)与单倍型网络图基本一致(图 2),也可分为A、B、C三支。
|
图 2 小黄黝鱼种群的单倍型网络图
Fig. 2 Haplotype network of M. swinhonis
|
|
图 3 小黄黝鱼63个单倍型的贝叶斯无根树
Fig. 3 Bayesian tree of the 63 haplotype of M. swinhonis
|
以地理结构为依据分类的AMOVA分析将所有地理种群分为邵阳资水、太湖、其他3组,结果表明大部分变异来自种群间(79%),种群内只有(21.3%,表 2)。遗传分化指数(FST)是评估种群遗传结构变异的一个重要指数,暗示种群间有明显遗传差异(P ≤ 0.01)。
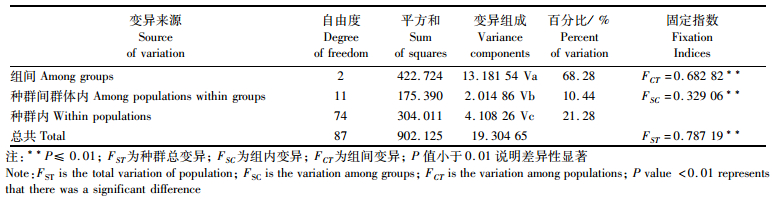
|
表 2 小黄黝鱼种群的AMOVA分析 Tab.2 Analysis of molecular variance (AMOVA) among the populations of M. swinhonis |
SAMOVA结果显示当分组K值等于3,FCT=0.524 87的时候是最佳种群分组结果(图 4)。当K=3时,分为邵阳、太湖和其他3个地理空间种群。遗传分化指数体现了各种群间的分化差异情况(表 3),从表中可以看出种群A、B与C1、C2、C3三亚支的的遗传分化程度较高(FST均大于0.659 30);其中A与C3种群的分化程度最大(FST= 0.896 00),A与C1、C2也存在显著分化(FST =0.884 10、0.843 68);C种群内的各亚支间分化程度略小(FST均小于0.591 17)。
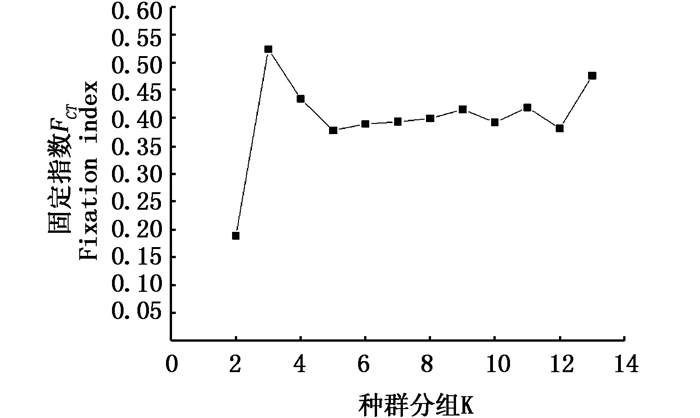
|
图 4 小黄黝鱼种群结构的SAMOVA结果
Fig. 4 SAMOVA of M. swinhonis
当K=3时,FCT最大 When K=3, FCT has maximum value |
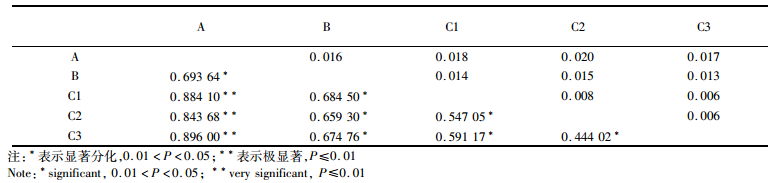
|
表 3 小黄黝鱼种群的遗传分化指数FST(对角线下方)和遗传距离P-distance(对角线上方) Tab.3 Fixation index (FST) (under diagonal) and P-distance (above diagonal) of M. swinhonis |
对14个种群的错配分布(Mismatch distribution)分析(图 5)表明:只有太湖(TH)、靖江(JJ)、荆州(JZ)、洪湖(HH)具有较明显的单峰;而郎溪(LX)、巢湖(CH)、高邮湖(GYH)、濮阳(PY)、海河(HHe)、辽河(LH)、松花江(SHJ)都具有超过2个峰的多峰错配分布,邵阳(SY)、腾冲(TC)、洞庭湖(DTH)分别由于仅有一个单倍型、2个单倍型样本太少,而无法进行错配分析。相对于Tajima’s D,Fu’s FS检测更适合种群的研究[9],结合Tajima’s D和Fu’s FS检测,只有靖江(JJ,Fu’s FS: -4.119,P=0.009)、洪湖(HH,Fu’s FS=:-2.814,P0.016) 群体为负值且P≤0.05,暗示这两个种群历史上发生过种群扩张。太湖和荆州群体虽然Fu’s FS也为负值(Fu’s FS=-0.658 09、-0.377 08),但P值不显著(P=0.266 00、0.331 00)。而其他种群均具有多峰且Fu’s FS多为正值。
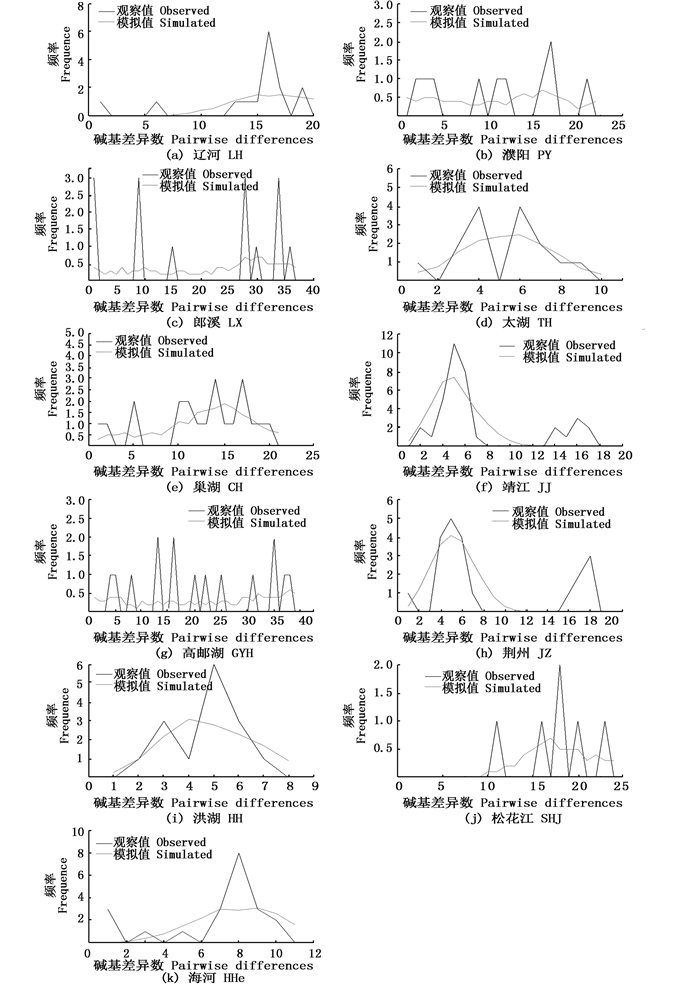
|
图 5 小黄黝鱼单倍型核苷酸错配分布分析
Fig. 5 Mismatch distribution analysis of M. swinhonis sampled from different localities
|
A谱系的种群比较独特,采集样本30尾,测序11尾只有一个单倍型,同时和其他地区遗传差距较远独立成为一个种群。贝叶斯树(BI)与网络图结果基本一致。推测该群体历史上可能发生过严重的瓶颈效应。B谱系主要包括太湖种群,其中H10、H51(郎溪)、H20(高邮湖)可能也是由于引种造成的,也可能是自然扩散而来。
3 讨论单倍型网络图(图 2)结果显示C谱系可分为3个亚谱系C1、C2、C3,这3个亚谱系间的差异较小,其中C1谱系包括长江水系样本(包括巢湖、郎溪、洪湖、荆州的个体)及云南腾冲的样本(H43、H44)。有文献记载云南种群为引种[17-18],结合本文分析我们推断云南种群可能是由长江水系安徽巢湖或者湖南洞庭湖引入,具体的引入源则需要做进一步调查与分析。C1谱系内包括黄河水系濮阳样本(H04、H56、H57、H58)、辽河水系样本(H04)、高邮湖的一个单倍型(H22) 和靖江的一个单倍型(H30),这些地区的单倍型(除H22和H30) 和云南样本在网络图中有着十分相似的情况,推测可能是由于经济物种引种而带入这些地区。因为这些地区原来就存在小黄黝鱼,且目前为止少有分子方面的研究,鱼类资源调查较难发现这种情况,也可能是分子标记不够,导致结果不够准确。错配分布分析结果同时显示郎溪(LX)、巢湖(CH)、高邮湖(GYH)、濮阳(PY)、海河(HHe)、辽河(LH)、松花江(SHJ)都具有超过2个峰的多峰错配分布,说明这些种群内样本具有不同的来源。其中靖江(H30) 可能是一个古老单倍型,而其他靖江样本都聚在C3谱系内。高邮湖的样本(H22) 可能是由长江下游扩散过去,因为前者在扬州市与长江连通。
C2亚谱系种群样本以长江以北水系为主,包括松花江、辽河、海河、黄河和高邮的个体。其中有两个与高邮湖相连的单倍型H12(巢湖)、H37(荆州)可能是由高邮湖扩散而来,也可能是由于引种造成的,因为在湖北荆州到高邮湖中间这些地区没有检测到连续的遗传信号,也可能是由于样本采集不够,信息不足,所以暂不能确定。
对C3亚谱系分析,C3谱系主要包括靖江群体,此种群既有样本聚于C1(H30),也有属于松花江(H47)、辽河(H01) 和高邮湖(H21) 种群聚在该亚谱系,且靖江市属于长江水系靠近与高邮湖连接处,暗示靖江群体可能是一个连接北方种群和长江水系种群的枢纽。
3.2 小黄黝鱼种群结构及历史动态分析AMOVA分析结果显示79%的变异来自种群间,只有28.3%的变异来自种群内,可以确切地表明小黄黝鱼存在系统地理学结构。SAMOVA结果进一步确定存在地理结构,很明显当K=3时,有最大的FCT值(0.524 87), 分为邵阳、太湖和其他地区3组。
结合固定指数Fst和遗传距离P-distance(表 3),A与B的遗传距离为0.016;A与C的遗传距离为0.018;B与C的遗传距离为0.014;C支内部(C1、C2、C3) 的遗传距离最大值为0.008,遗传变异很小。说明A、B、C三支之间已经出现明显的种群分化,其中A支分布于西南地区,与其它群体遗传差异最大,可能是一个古老的种群经过严重的瓶颈效应形成的独特种群;B支太湖种群也表现出特异性单独成为一个种群,原因暂不清楚;C内的3支遗传分化不明显,又同时包含从松花江到长江所有水系所有样本,此种情况文献中也较为少见,其他鱼类种群研究往往可明显根据水系划分不同地理种群。比如PERDICES [19]对宽鳍
小黄黝鱼种群明显分为3个大的种群:邵阳(SY)种群、太湖(TH)种群和其他地区组成的复合体种群。小黄黝鱼具有较高的单倍型多样性和较低的核苷酸多样性,结合错配分析暗示此物种历史上发生过种群扩张。另外发现小黄黝鱼引种现状可能较严重,破坏了小黄黝鱼的自然地理种群分布,建议在经济鱼类引种的同时,加强鱼类自然资源的保护。
| [1] |
中国科学院中国动物志编辑委员会. 中国动物志-五:硬骨鱼纲鲈形目虾虎鱼亚目[M]. 北京: 科学出版社, 2008.
Chinese Academy of Sciences, China Fauna of Editorial Board. Fauna Sinica:Ostichthyes Perciformes Gobioidei[M]. Beijing: Science Press, 2008. |
| [2] |
中国水产科学研究院珠江水产研究所. 广东淡水鱼类志[M]. 广州: 广东科技出版社, 1991.
Pearl River Fishers Research Institute. The Freshwater Fishes of Guangdong Province[M]. Guangdong: Guangdong Science and Technology Press, 1991. |
| [3] | BROWN J R, BECKENBACH A T, SMITH M J. Mitochondrial DNA length variation and heteroplasmy in populations of white sturgeon (Acipenser transmontanus)[J]. Genetics, 1992, 132(1): 221–228. |
| [4] |
肖武汉, 张亚平. 鱼类线粒体DNA的遗传与进化[J].
水生生物学报, 2000, 24(4): 384–391.
XIAO W H, ZHANG Y P. Genetics and evolution of mitochondrial DNA in fish[J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384–391. |
| [5] | TAMURA K, PETERSON D, PETERSON N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731–2739. DOI:10.1093/molbev/msr121 |
| [6] | LIBRADO P, ROZAS J. Dnasp v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451–1452. DOI:10.1093/bioinformatics/btp187 |
| [7] | EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0):an integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2005, 1: 47–50. |
| [8] | TAJIMA F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism[J]. Genetics, 1989, 123(3): 585–595. |
| [9] | FU Y X. Statistical tests of neutrality of mutations against population growth, hitchhiking and background selection[J]. Genetics, 1997, 147(2): 915–925. |
| [10] | BANDELT H J, FORSTER P, RÖHL A. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution, 1999, 16(1): 37–48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [11] | POSADA D. jModelTest:Phylogenetic model averaging[J]. Molecular Biology and Evolution, 2008, 25(7): 1253–1256. DOI:10.1093/molbev/msn083 |
| [12] | GUINDON S, GASCUEL O. A simple, fast, and accurate Algorithm to estimate large phylogenies by maximum likelihood[J]. Systematic Biology, 2003, 52(5): 696–704. DOI:10.1080/10635150390235520 |
| [13] | HUELSENBECK J P, RONQUIST F. MRBAYES:Bayesian inference of phylogenetic trees[J]. Bioinformatics, 2001, 17(8): 754–755. DOI:10.1093/bioinformatics/17.8.754 |
| [14] | DRUMMOND A J, RAMBAUT A J. BEAST:Bayesian evolutionary analysis by sampling trees[J]. BMC Evolutionary Biology, 2007, 7(1): 214. DOI:10.1186/1471-2148-7-214 |
| [15] | DUPANLOUP I, SCHNEIDER S, EXCOFFIER L. A simulated annealing approach to define the genetic structure of populations[J]. Molecular Ecology, 2002, 11(12): 2571–2581. DOI:10.1046/j.1365-294X.2002.01650.x |
| [16] | KUHNER M K. LAMARC 2.0:maximum likelihood and Bayesian estimation of population parameters[J]. Bioinformatics, 2006, 22(6): 768–770. DOI:10.1093/bioinformatics/btk051 |
| [17] |
丁慧萍, 谭剑晖, 林少卿, 等. 拉萨市茶巴朗湿地的外来鱼类[J].
水生态学杂志, 2014, 35(2): 49–55.
DING H P, TAN J H, LIN S Q, et al. Exotic fishes in Chabalang wetland of Lhasa[J]. Journal of Hydroecology, 2014, 35(2): 49–55. |
| [18] |
唐文家, 何德奎. 青海省外来鱼类调查(2001-2014年)[J].
湖泊科学, 2015, 27(3): 502–510.
DOI:10.18307/2015.0318 TANG J W, HE D K. Investigation on alien fishes in Qinghai Province, China (2001-2014)[J]. Journal of Lake Science, 2015, 27(3): 502–510. DOI:10.18307/2015.0318 |
| [19] | PERDICES A, COELHO M M. Comparative phylogeography of Zacco platypus and Opsariichthys bidens (Teleostei, Cyprinidae) in China based on cytochrome b sequences[J]. Journal of Zoological Systematics and Evolutionary Research, 2006, 44(4): 330–338. DOI:10.1111/j.1439-0469.2006.00368.x |
| [20] |
夏曦中, 张训蒲, 刘焕章. 似( XIA X Z, ZHANG X P, LIU H Z. Molecule biogeography analysis of Pseudogobio vaillanti[J]. Progress in Natural Science, 2005, 15(9): 1064–1069. |
| [21] |
库喜英, 周传江, 何舜平. 中国黄颡鱼的线粒体DNA多样性及其分子系统学[J].
生物多样性, 2010, 18(3): 262–274.
KU X Y, ZHOU C J, HE S P. Validity of Pseudobagrus sinensis and mitochondrial DNA diversity of Pseudobagrus fulvidraco populations in China[J]. Biodiversity Science, 2010, 18(3): 262–274. |
| [22] | ZHAO L, ZHANG J, LIU Z J, et al. Complex population genetic and demographic history of the Salangid, Neosalanx taihuensis, based on cytochrome b sequences[J]. BMC Evolutionary Biology, 2008, 8(1): 201. DOI:10.1186/1471-2148-8-201 |
 2017,
Vol. 26
2017,
Vol. 26


