脂鳍位于背鳍和尾鳍之间,往往缺少鳍条等骨骼支撑[1-2],广泛分布在水珍鱼目(Argentiniformes)、仙女鱼目(Aulopiformes)、脂鲤目(Characiformes)、灯笼鱼目(Myctophiformes)、胡瓜鱼目(Osmeriforms)、鲑鲈目(Percopsiformes)、鲑形目(Salmoniformes)、鲇形目(Siluriformes)和巨口鱼目(Stomiiforms)等9个目的75科的六千多种鱼类[3]。鲑形目鲑科的脂鳍,主要由表皮组织、结缔组织和角质组成[4],鲇形目鲿科的脂鳍由表皮层、胶原纤维层和脂肪层组成[5]。有些脂鲤目和鲇形目的脂鳍中存在鳍条状结构[6],尼罗双背鳍鲿(Mochokus niloticus)脂鳍中的鳍条和其他奇鳍的鳍条非常相似[7]。有些种类的脂鳍中有鳍条,也有相应的支持结构,如一些灯笼鱼目和胡瓜鱼目鱼类的脂鳍基底存在小软骨[1]。此外,一些巨口鱼目种类在腹部也有脂鳍[8]。
脂鳍发育有两种模式:一种是鲑形目类型,脂鳍和奇鳍一样,同时在鳍褶尚未完全退化的基础上发育而成; 另一种是脂鲤目类型,脂鳍是在鳍褶完全退化后,再在已形成的背鳍与尾鳍之间发育为脂鳍。鲑形目类型脂鳍发育和背鳍的前期发育类似[8]。背鳍发育被认为分为3个阶段:第一阶段是鳍褶从鱼类头部后到肛门沿中轴线形成一层膜;第二阶段是在背鳍发生的位置出现背鳍原基,随着发育,鳍褶消失;第三阶段是背鳍原基继续分化至出现了支鳍骨和真皮骨骼,背鳍发育完成[9]。如衰白鲑(Coregonus maraena)脂鳍的发育,有类似背鳍发育的第一和第二阶段,但缺少第三阶段[8],脂鳍发育和背鳍前期的发育机制是否相似,目前尚不清楚。脂鲤目类型的脂鳍发育模式,是在鳍褶完全退化后再发育而成的,相比鲑形目类型脂鳍,其似乎与背鳍发育模式相差较大。
为了解脂鲤目鱼类脂鳍发育和背鳍发育的关系,本文观察了脂鲤目的细锯脂鲤(Pristella maxillaris)的脂鳍和背鳍发育,并利用高通量测序技术比较了细锯脂鲤的背鳍原基、背鳍鳍条分化和脂鳍的转录组,为理解脂鳍与背鳍发生的异同提供了分子信息。
1 材料和方法 1.1 材料来源细锯脂鲤饲养在上海海洋大学水产与生命学院实验室。筛选成熟度好的亲鱼进行人工繁殖,受精后24 h仔鱼出膜,在28 ℃恒温光照箱饲养,出膜后第3天开始投喂轮虫,到8 DAH (Day After Hatch,DAH)开始投喂卤虫无节幼体。
1.2 骨骼染色所取样本用4%的多聚甲醛固定过夜,再转移到95%乙醇中保存,用作骨骼染色。对28、30、32和35 DAH的细锯脂鲤骨骼进行了染色。详细骨骼染色方法参考文献[10]~[12]。先用95%乙醇和去离子无菌水3:1、1:1、1:3漂洗样本各30 min,以去除甲醛; 用TBST (50 mmol /L Tris,pH 7.4,150 mmol /L NaCl,体积分数为0.1%的Triton-100)温和震荡漂洗2次,每次30 min,用去离子水洗30 min,以去除残余的TBST溶液; 漂洗完后直接转移至新鲜配置的阿利新蓝溶液[10 mg阿利新蓝,70 mL无水乙醇,30 mL冰乙酸(pH≤1.0)过滤,4 ℃]中,进行软骨染色,染色20 min。染色后转移至1% KOH溶液,漂洗后转移至胰蛋白酶消化液[100 mL体系: 65 mL去离子水,35 mL饱和四硼酸钠盐(Na2B4O7·10H2O)上清液,1 g胰蛋白酶]消化直至头部肌肉组织透明; 将透明的标本转移至新配置的1% KOH溶液中,滴加茜素红染色液进行染色,后以1% KOH溶液洗去组织中残余的染色液; 经含15%、75%、50%、30%、90%和100%的甘油梯度脱水,最后保存在含少许麝香草酚的甘油中。取样样本和骨骼染色后的样本用SMZ1500 (Nikon)解剖显微镜拍照。
1.3 总RNA提取及mRNA分离分别取28、30和35 DAH细锯脂鲤各50尾,MS-222麻醉之后在解剖镜下剪取背鳍原基、背鳍和脂鳍组织,分别抽取总RNA。总RNA提取按照Invitrogen公司的Trizol Reagent说明书进行。用Dynabeads® Oligo (dT)25(Life,美国)进行mRNA纯化。
1.4 cDNA文库构建和转录组测序cDNA文库构建方法主要参考魏利斌等[13]。取100 ng纯化的mRNA,用RNA Library Prep Kit for Illumina (NEB,美国)构建文库。首先加入破碎缓冲液将mRNA随机打断成片段,以这些RNA片段为模板,先合成第1条cDNA链,再合成第2条cDNA链。末端修复cDNA后加poly (A)并连接测序接头,得到300~500 bp范围的ligated cDNA,最后进行PCR扩增得到可测序的文库。取10 μg的文库在Illumina HiSeqTM 2500中进行双向测序。
1.5 序列分析和注释利用Illumina平台将测序所得的图像数据转化为相应的核苷酸序列数据,对所产生的原始序列文件进行质量评估和可信度分析,并去除测序过程中低质量的序列和不确定的序列(Q < 20)。Clean data通过Trinity软件(版本2.0.6,参数min_kmer_cov 2其他默认参数)完成组装。通过Trinity将测序数据组装成转录本,取用每个transcript cluster中最长的转录本作为Unigene,作为后续分析的参考序列。将预测得到的蛋白编码序列与NR、KEGG、GENES、KOG等数据库进行blastp比对,获得最佳注释(E value < 1e-5)。
2 结果 2.1 细锯脂鲤的背鳍和脂鳍的发育细锯脂鲤的背鳍在鳍褶完全退化之后开始发育。28 DAH的细锯脂鲤在后来背鳍形成的位置出现了一个背鳍原基,即在背鳍发育出来的位置长出一个小小的透明凸起(图版-A2),通过阿利新蓝染色发现,尾鳍已经发育出少许鳞质鳍条,支鳍骨形成,臀鳍原基也发育出来,支鳍骨形成。到30 DAH仔鱼背鳍发育成形,可以观察到支鳍骨和鳞质鳍条都已经形成。同时尾鳍和臀鳍的支鳍骨和鳞质鳍条也发育完善。

|
图版 细锯脂鲤背鳍和脂鳍发育及转录组差异的分析 Plate1 Dorsal fin and adipose fin development of P. maxillaris and their comparative transcriptome analysis A1-A4.不同孵化天数细锯脂鲤的背鳍和脂鳍的形态。箭头显示背鳍和脂鳍。B1-B4.不同孵化天数的细锯脂鲤整体骨骼染色。绿色为阿利新蓝染色的软骨,红色为茜素红染色的硬骨。所有标尺都是500 μm。C.细锯脂鲤脂鳍、背鳍原基和背鳍转录组差异分析 A1-A4. Dorsal fin and adipose fin in P. Maxillaris at different days after hatch (DAH), arrows show dorsal fin or adipose fin. B1-B4. Skeletal staining of P. Maxillarisat different days after hatching. Green color shows cartilage, red color shows bone. All bars are 500μm. C. Comparative transcriptome analysis of adipose fin, dorsal fin bud and dorsal fin in P. Maxillaris |
细锯脂鲤的脂鳍也是在鳍褶完全退化之后开始发育。32 DAH的仔鱼,在背鳍与尾鳍之间出现小小的脂鳍,一层透明薄膜, 类似于背鳍原基(图版-A4),此时,背鳍也已发育完成。与背鳍不同的是,脂鳍的下方并没有支鳍骨形成。
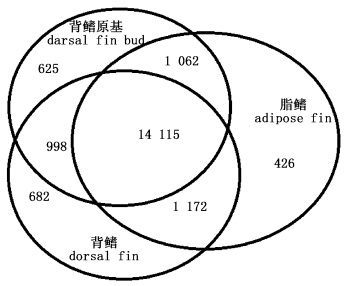
2.2 背鳍原基、背鳍、脂鳍三者转录本基因的异同在细锯脂鲤中,脂鳍组织有16 775个基因表达,背鳍原基组织有16 800个基因表达,背鳍组织基因表达数相对高一些,共有16 967个基因表达(图 1)。背鳍原基、背鳍、脂鳍这3个组织共同表达基因有14 115条,分别占背鳍原基转录组的84.01%、背鳍的83.19%、脂鳍的84.14%。背鳍原基和背鳍共有15 113个基因表达,分别占背鳍原基转录组的89.95%,背鳍转录组的89.07%;脂鳍和背鳍原基共有15 117个基因表达,分别占脂鳍转录组的90.12%,占背鳍原基转录组的89.98%;脂鳍和背鳍共有15 287个基因表达,分别占脂鳍转录组的91.13%,占背鳍转录组的90.1%。
|
图 1 背鳍原基、背鳍、脂鳍的基因异同分析
Fig. 1 Comparison of the gene numbers of dorsal fin bud, dorsal fin and adipose fin
圈内交叉的部分是它们两两或3个共有的基因数量 Circle cross section is both of the two or three common unigene number |
脂鳍单独拥有的基因有426条占2.24%,背鳍原基单独拥有的基因有625条占3.28%,背鳍单独拥有的基因有682条占3.30%。
2.3 背鳍原基、背鳍、脂鳍三者转录本基因的表达水平差异背鳍原基、背鳍、脂鳍三者转录本基因的表达水平差异见图版。图版中纵轴树对这些基因作了一个聚类,同一类的基因可能在一条信号通路上;横轴是3个转录样本,脂鳍和背鳍的相似度小于背鳍和背鳍原基,和背鳍相对背鳍原基要近一些。图版中每一行代表一个基因,显示红色表示基因在该组织表达量高,显示绿色表示基因在该组织表达量低。从图版中能够直观地看出,背鳍基因表达水平总体表现较高。相比脂鳍,背鳍原基和背鳍之间的基因表达模式更为相似。通过统计和比较3个转录本信息,脂鳍和背鳍原基相比,上调基因有186个,下调基因有239个;脂鳍与背鳍相比,上调基因有134个,下调基因有336个;背鳍原基与背鳍相比,上调基因有86个,下调基因有148个。
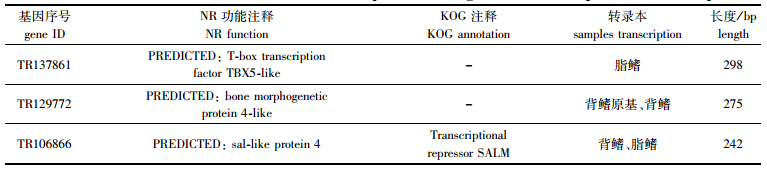
2.4 已知与鱼类鳍发育相关基因在3个样本中的差别已知与鳍发育相关的基因,有105个基因在背鳍原基、背鳍和脂鳍组织都有表达,而少数与鳍发育相关的基因,在此3个组织转录组表达不一样。如bmp4只在背鳍有表达,在背鳍原基和脂鳍里不表达;成对附肢发育中关键的一个基因tbx5在脂鳍中有表达,而在背鳍和背鳍原基中没有表达;而Sall4基因在脂鳍和背鳍的转录本中有,而在背鳍原基处没有(表 1)。
|
表 1 已知鱼类鳍发育相关基因在3个样本转录组的差异分布 Tab.1 Difference distribution of known fish fin development related genes in three samples of the transcriptomes |
细锯脂鲤的脂鳍发育模式属于典型的脂鲤目类型,出膜后由鳍褶从头部到肛门,随后鳍褶退化之后再发育出脂鳍。脂鳍在鳍褶完全退化后再发育出来,也间接提示我们脂鳍不应该是痕迹器官,虽然其目前的功能尚不清楚[2, 14-15]。虹鳟鱼脂鳍基部存在肌肉结构,以及脂鳍在鱼类进化过程中多次进化的现象,也提示脂鳍不应该是痕迹器官[3, 16]。细锯脂鲤背鳍条硬骨化的时间持续较短,从32 DAH到35 DAH,鳍条和支鳍骨骨化已经完成。与背鳍形成相比,细锯脂鲤脂鳍没有鳍条形成,也没有相应的支鳍骨。比较有趣的是,在28 DAH背鳍原基出现的时候,鳍褶已经完全退化,这在其他鱼类中是不多见的[6]。
3.2 脂鳍和背鳍转录组比较分析3个样本的转录组分析表明,脂鳍和背鳍原基,或脂鳍和背鳍都有近似90%相同表达的基因,表明脂鳍发育的主要调控机制应该与背鳍发育非常相似。比较有趣的是,根据共有基因的相互比较,脂鳍和背鳍共有基因数,不但多于脂鳍和背鳍原基的共有基因数,而且多于背鳍原基和背鳍的共有基因数,表明虽然从发育过程上,脂鳍与背鳍原基发育非常相似,但可能在组成脂鳍的性质上,虽然没有鳍条,但具备了形成鳍条的成分,只是不能形成鳍条的形态。例如,鲑形目鲑科的脂鳍,其中已有角质成分存在[4]。背鳍原基、背鳍、脂鳍三者转录本基因的表达水平差异表明,相比脂鳍,背鳍原基与背鳍之间的基因表达模式要更为相似, 和背鳍原基与背鳍之间相比,脂鳍与背鳍原基或背鳍之间的上调和下调的基因数变化更大,表明在基因表达水平上也较多体现了脂鳍与背鳍发育的差异。
tbx5基因可以诱导腹鳍以及前附肢的发育,在附肢发育中有很重要的作用[17-18]。tbx5只有在细锯脂鲤脂鳍中表达,而没有在背鳍和背鳍原基中表达,提示我们tbx5基因可能与脂鳍的出现密切相关。此外,Sal基因与鳍形成发育相关,可以诱导胸鳍的发育[19],而Sall4基因只在细锯脂鲤脂鳍和背鳍的转录本中有,而在背鳍原基处没有,提示我们其可能与角质成分形成有关。bmp4基因只在背鳍有表达,在原基和脂鳍里是不表达的。但在鳍条分化阶段,bmp4不管是胸鳍还是背鳍,都发挥着重要的作用[20-21],提示我们,脂鳍没有鳍条和支鳍骨可能与bmp4没有表达有关。
总之,转录组比较分析发现,无论在基因相似性还是基因表达水平方面,都说明了脂鳍与背鳍发育调控方面存在高度相关性,个别基因在脂鳍和背鳍上的差异表达,也为下一步的功能验证提供了候选基因,为更好地理解脂鳍发育的调控打下了基础。
| [1] | MATSUOKA M, IWAI T.Adipose fin cartilage found in some teleostean fishes[J]. Japanese Journal of Ichthyology, 1983, 30(1): 37–46. |
| [2] | STEWART T A, HALE M E.First description of a musculoskeletal linkage in an adipose fin:innovations for active control in a primitively passive appendage[J]. Proceeding of the Royal Society B, 2013, 280(1750): 20122159. |
| [3] | STEWART T A, SMITH W L, COATES M I.The origins of adipose fins:an analysis of homoplasy and the serial homology of vertebrate appendages[J]. Proceeding of the Royal Society B, 2014, 281(1781): 20133120. DOI:10.1098/rspb.2013.3120 |
| [4] | WEISEL G F.The Salmonoid adipose fin[J]. Copeia, 1968: 626–627. |
| [5] | 钟明超, 罗泉苼, 叶华.大鳍鳠臀鳍和脂鳍的比较组织学研究[J]. 西南师范大学学报(自然科学版), 1996, 21(2): 168–171. ZHONG M C, LUO Q S, YE H.Comparative and histological studies on the anal and adipose fins in freshwater catfish Mystus macropterus (Bleeker)[J]. Journal of Southwest China Normal University (Natural Science), 1996, 21(2): 168–171. |
| [6] | NORMAN J R. A history of fishes[M]. New York: A A Wyn, 1949: 463. |
| [7] | VIGLIOTTA T R.A phylogenetic study of the African catfish family Mochokidae (Osteichthyes, Ostariophysi, Siluriformes), with a key to genera[J]. Proceedings of the Academy of Natural Sciences of Philadelphia, 2008, 157(1): 73–136. DOI:10.1635/0097-3157(2008)157[73:APSOTA]2.0.CO;2 |
| [8] | BENDER A, MORITZ T.Developmental residue and developmental novelty-different modes of adipose-fin formation during ontogeny[J]. Zoosystematics and Evolution, 2013, 89(2): 209–214. DOI:10.1002/zoos.v89.2 |
| [9] | MABEE P M, CROTWELL P L, BIRD N C, et al.Evolution of median fin modules in the axial skeleton of fishes[J]. Journal of Experimental Zoology, 2002, 294(2): 77–90. DOI:10.1002/(ISSN)1097-010X |
| [10] | 柯中和, 张炜, 蒋燕, 等.鲢肌间小骨发育的形态学观察[J]. 动物学杂志, 2008, 43(6): 88–96. KE Z H, ZHANG W, JIANG Y, et al.Developmental morphology of the intermuscular bone in Hypophthalmichthys molitrix[J]. Chinese Journal of Zoology, 2008, 43(6): 88–96. |
| [11] | 姚文杰, 龚小玲, 吕耀平, 等.日本鳗鲡肌间小骨的骨化过程[J]. 上海海洋大学学报, 2014, 23(6): 810–813. YAO W J, GONG X L, LV Y P, et al.The ossificational process of the intermuscular bones in Anguilla japonica[J]. Journal of Shanghai Ocean University, 2014, 23(6): 810–813. |
| [12] | 吕耀平, 陈洁, 鲍宝龙, 等.唇䱻肌间小骨的骨化过程[J]. 上海海洋大学学报, 2012, 21(4): 549–553. LV Y P, CHEN J P, BAO B L, et al.The ossificational process of the intermuscular bones in Hemibarbus labeo[J]. Journal of Shanghai Ocean University, 2012, 21(4): 549–553. |
| [13] | 魏利斌, 苗红梅, 张海洋.芝麻发育转录组分析[J]. 中国农业科学, 2012, 45(7): 1246–1256. WEI L B, MIAO H M, ZHANG H Y.Transcriptomic analysis of sesame development[J]. Scientia Agricultura Sinica, 2012, 45(7): 1246–1256. |
| [14] | 宋洪建, 刘伟, 王继隆, 等.大麻哈鱼卵黄囊期仔鱼异速生长及其生态学意义[J]. 水生生物学报, 2013, 37(2): 329–335. SONG H J, LIU W, WANG J L, et al.Allometric growth during yolk-sac larvae of chum salmon (Oncorhynchus Keta Walbaum) and consequent ecological significance[J]. Acta Hydrobiologica Sinica, 2013, 37(2): 329–335. |
| [15] | REIMCHEN T E, TEMPLE N F.Hydrodynamic and phylogenetic aspects of the adipose fin in fishes[J]. Canadian Journal of Zoology, 2004, 82(6): 910–916. DOI:10.1139/z04-069 |
| [16] | MCMAHON A P, INGHAM P W, TABIN C J.Develop-mental roles and clinical significance of hedgehog signaling[J]. Current Topics in Developmental Biology, 2003, 53: 1–14. DOI:10.1016/S0070-2153(03)53002-2 |
| [17] | AHN D G, KOURAKIS M J, ROHDE L A, et al.T-box gene tbx5 is essential for formation of the pectoral limb bud[J]. Nature, 2002, 417(6890): 754–758. DOI:10.1038/nature00814 |
| [18] | RALLIS C, BRUNEAU B G, DEL BUONO J, et al.Tbx5 is required for forelimb bud formation and continued outgrowth[J]. Development, 2003, 130(12): 2741–2751. DOI:10.1242/dev.00473 |
| [19] | MARÍ-BEFFA M, MURCIANO C.Dermoskeleton morpho-genesis in zebrafish fins[J]. Developmental Dynamics, 2010, 239(11): 2779–2794. DOI:10.1002/dvdy.v239:11 |
| [20] | BASTIDA M F, SHETH R, ROS M A.A BMP-Shh negative-feedback loop restricts Shh expression during limb development[J]. Development, 2009, 136(22): 3779–3789. DOI:10.1242/dev.036418 |
| [21] | SIMTH A, AVARON F, GUAY D, et al.Inhibition of BMP signaling during zebrafish fin regeneration disrupts fin growth and scleroblast differentiation and function[J]. Developmental Biology, 2006, 299(2): 438–454. DOI:10.1016/j.ydbio.2006.08.016 |
 2016,
Vol. 25
2016,
Vol. 25


