2. 河南师范大学 水产学院, 河南 新乡 453007
固醇调节元件结合蛋白(Sterol regulatory element binding protein,SREBP)是脊椎动物调节脂代谢的一类膜结合核转录因子,目前在哺乳动物中已发现3个成员:SREBP-1a、 SREBP-1c和SREBP-2,其中SREBP-1a和 SREBP-1c 由同一个基因SREBP-1的不同启动子转录生成[1]。SREBP-1主要激活与脂肪酸生成有关的基因,如脂肪酸合成酶基因(Fatty acid synthase,FASN),硬脂酰辅酶A脱氢酶基因(Stearoyl-CoA desaturase ,SCD),以及乙酰辅酶A羧化酶基因1(Acetyl-CoA carboxylase 1,ACC1)等,而SREBP-2主要作用于胆固醇调节基因,如3-羟基-3-甲基戊二酸单酰辅酶A还原酶基因(HMG-CoA reducase,HMGCR)、低密度脂蛋白受体基因(Low-density lipoproteinreceptor ,LDLR)等[2]。miRNA-33是参与维持机体脂质稳态的一种小分子核糖核酸,在人类发现有miRNA-33a和miRNA-33b两个亚型,其编码序列分别定位于SREBP-2和SREBP-1基因的内含子上;而在小鼠中只发现一种miRNA-33,定位于SREBP-2内含子上[3-6]。miR-33不仅与其宿主基因共转录,还一起参与维持机体胆固醇和脂质稳态[7]。miR-33的主要靶基因为ATP结合盒转运子 A1 /G1基因(ATP-binding cassette transporter A1 /G1,Abca1/Abcg1)。通过与靶基因的3'-UTR 区相应序列结合,下调其表达,进而降低细胞胆固醇的外流、高密度脂蛋白(High-density lipoprotein,HDL)的合成及胆固醇的逆转运[3];此外,miR-33还能靶向肉毒碱棕榈酰基转移酶(CPT1A)、肉碱 O辛基转移酶(CROT) 和羟烷基辅酶A脱氢酶B(HADHB)等参与脂肪酸 β 氧化的相关基因,从而减少脂肪酸的降解[6]。最近研究发现,miR-33还可靶向SREBP-1[8]。miR-33存在缺陷时,SREBP-1水平升高,脂肪酸合成增加并积聚于肝和脂肪组织。miR-33-/-小鼠出现肥胖和肝脂肪病变的主要机制是由于SREBP-1的过表达。
目前关于SREBP和miR-33的研究结果虽然大多来自哺乳动物,但有证据表明,二者在脊椎动物中高度保守[9-10]。对斑马鱼、大西洋鲑和金头鲷的研究显示,SREBP-1和鱼类的糖脂代谢有关,可调节脂肪酸合成或糖代谢相关基因的表达[9, 11],在此过程中,miRNAs也可能作为上游因子,通过调控SREBP-1的表达而成为SREBP调节网络中的重要成员。我们在之前的研究中已克隆出草鱼(Ctenopharyngodon idella)SREBP-1 基因全长cDNA[12],其中3'-UTR长度为1 282 bp。本研究利用microRNA(miRNA)预测软件预测出该基因3'-URT序列中含有两个与脂代谢相关的miRNA—miR-33和miR-16的靶序列。为进一步研究SREBP-1和miR-33在草鱼脂代谢中的作用及miR-33对 SREBP-1表达的调控,我们克隆了SREBP-1 基因 3'-UTR区含有miR-33靶序列的碱基片断,构建了含miR-33结合位点的报告基因载体,并在草鱼肝细胞中进行荧光素酶活性检测,明确miR-33在草鱼SREBP-1表达中的调控作用。
1 材料与方法 1.1 主要试剂和仪器草鱼肝细胞(L8824),购自中国典型培养物保藏中心(China center for type culture collection,CCTCC),呈贴壁生长;质粒pmirGLO Dual-Luciferase miRNA Target Expression Vector、双荧光素酶检测试剂盒Dual-Luciferase Reporter Assay System购自Promega(美国);gc-miR-33 mimic由上海吉玛制药技术有限公司合成;感受态细胞DH5α由北京鼎国生物科技有限公司提供。其他试剂有T4 DNA 连接酶(Thermo美国)、2×Es Taq Master Mix(CWBIO,北京)、RNAiso Plus、PrimeScript RT Kit (Perfect Real Time)(TaKaRa,大连)、限制性内切酶Sac I和Xba I(NEB,北京)、无支原体优质胎牛血清(四季青,杭州)、青链霉素(Sigma,美国)、DMEM 高糖培养基(Gibico,上海)和lipofectamine 3000(Invitrogen,上海);主要仪器为全波长多功能酶标仪(Varioskan Flash,Thermo)。
1.2 靶向SREBP-1 基因3'-UTR 的miRNA 预测利用microRNA(miRNA)预测软件(http://regrna.mbc.nctu.edu.tw/html/prediction.html),预测可能与SREBP-1基因 3'-UTR 相互作用的miRNA。按照生物信息学预测规则,寻找符合要求的miRNA。一般要符合两个原则,一是miRNA 5'的第2~8位的核苷酸序列(种子序列)要完全与SREBP-1 基因的3'UTR互补;二是考虑在不同物种中其保守性高低和结合的自由能大小。
1.3 构建pmirGLO-SREBP-1-3'-UTR双荧光素酶报告载体根据本课题组已克隆的SREBP-1基因3'-UTR区的碱基序列(KJ162572)和预测出的miR-33所在的靶序列,采用Premier 5.0软件设计PCR引物,序列为:SREBP-3'UTR-F1: 5'CCCGGGTCTAGATTCTTCCCAACAGTTCATCAC3';SREBP-3'UTR-R1: 5'CCCGGGGAGCTCATCCCAGACGATAACAGGTAA3'(加下划线分别为XbaⅠ、SacⅠ酶切位点序列,之前序列为保护碱基,引物的扩增产物长度应为379 bp,位于SREBP-1基因3'-UTR的第148~527 bp处)。提取草鱼肝脏总RNA,RT-PCR法获取SREBP-1的3'UTR片段。反应体系为:2×Taq Master Mix 10 μL,Forward primer(10 μmol/L) 1 μL,Reverse primer(10 μmol/L) 1 μL,cDNA 2 μL,无菌水补足20 μL。反应条件为:95 ℃预变性5 min;94 ℃ 30 s,59.4 ℃ 30 s,72 ℃ 1 min共35个循环;72 ℃延伸10 min;4℃ 保存。1%琼脂糖凝胶电泳分析PCR 反应产物,并对产物进行纯化。用SacⅠ和XbaⅠ对纯化的PCR产物和载体进行双酶切,然后T4酶连接(连接时PCR产物与荧光素酶报告载体的摩尔数之比为3∶1,连接体系共10 μL)。将连接产物导入 DH5α感受态细胞进行阳性克隆验证,双酶切及电泳检测正确后,送公司测序鉴定。鉴定正确的重组载体命名为pmirGLO-SREBP-1-3'-UTR。
1.4 瞬时转染草鱼肝细胞L8824将构建的pmirGLO-SREBP-1-3'-UTR载体感染草鱼肝细胞L8824。采用24孔板进行细胞培养,细胞接种密度为5×104。在含10%胎牛血清的DMEM培养液、28 ℃和5 % CO2条件下,培养至细胞密度达70%~85%时开始转染。转染细胞分为两组:只转染pmirGLO-SREBP-1-3'-UTR报告载体的为对照组,共转染pmirGLO-SREBP-1-3'-UTR报告载体和miR-33 mimic为处理组。每组细胞设6个平行。转染方法:吸去原培养液,每孔加入300 μL新鲜无血清培养液;然后每孔加入200 μL转染试剂[对照组转染试剂:4 μL报告载体(500 ng/μL)+1.5 μL Lipofectamine 3000+1 μL P3000 + 200 μL无血清培养液;处理组转染试剂:4 μL报告载体 + 1.5 μL Lipofectamine3000 + 1 μL P3000 + 2 μL miR-33 mimics(20 μmol/L)+ 200 μL 无血清培养液];培养箱中培养48 h后检测荧光素酶活性。本实验重复3次。
1.5 荧光素酶活性测定采用全波长多功能酶标仪,按照Promega 公司提供的双荧光素酶检测试剂盒操作说明,分别检测对照组和处理组的荧光素酶活性。
1.6 统计学处理所有数据用SPSS 17.0统计软件进行t检验。分析结果以平均数±标准差(Mean ± SD)表示。当P<0.05时 表示差异具有统计学意义。
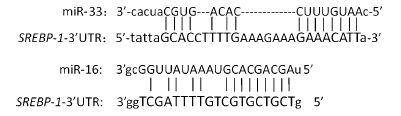
2 结果与分析 2.1 靶向SREBP-1基因 3'-UTR 的miRNA预测结果预测结果显示:SREBP-1的3'UTR区存在两个与脂代谢相关的miRNAs靶序列,即miR-33和miR-16。miR-33的靶序列位于SREBP-1 基因3'-UTR的第433~463 bp处,miR-16位于第650~671 bp处。两个miRNA均从第二位碱基开始与靶序列互补配对(图 1)。
|
图 1 预测的miRNA与SREBP-1基因 3'UTR靶序列的互补情况
Fig. 1 Base complementation between the predicted miRNAs and target sequences in SREBP-1 -3'UTR
|
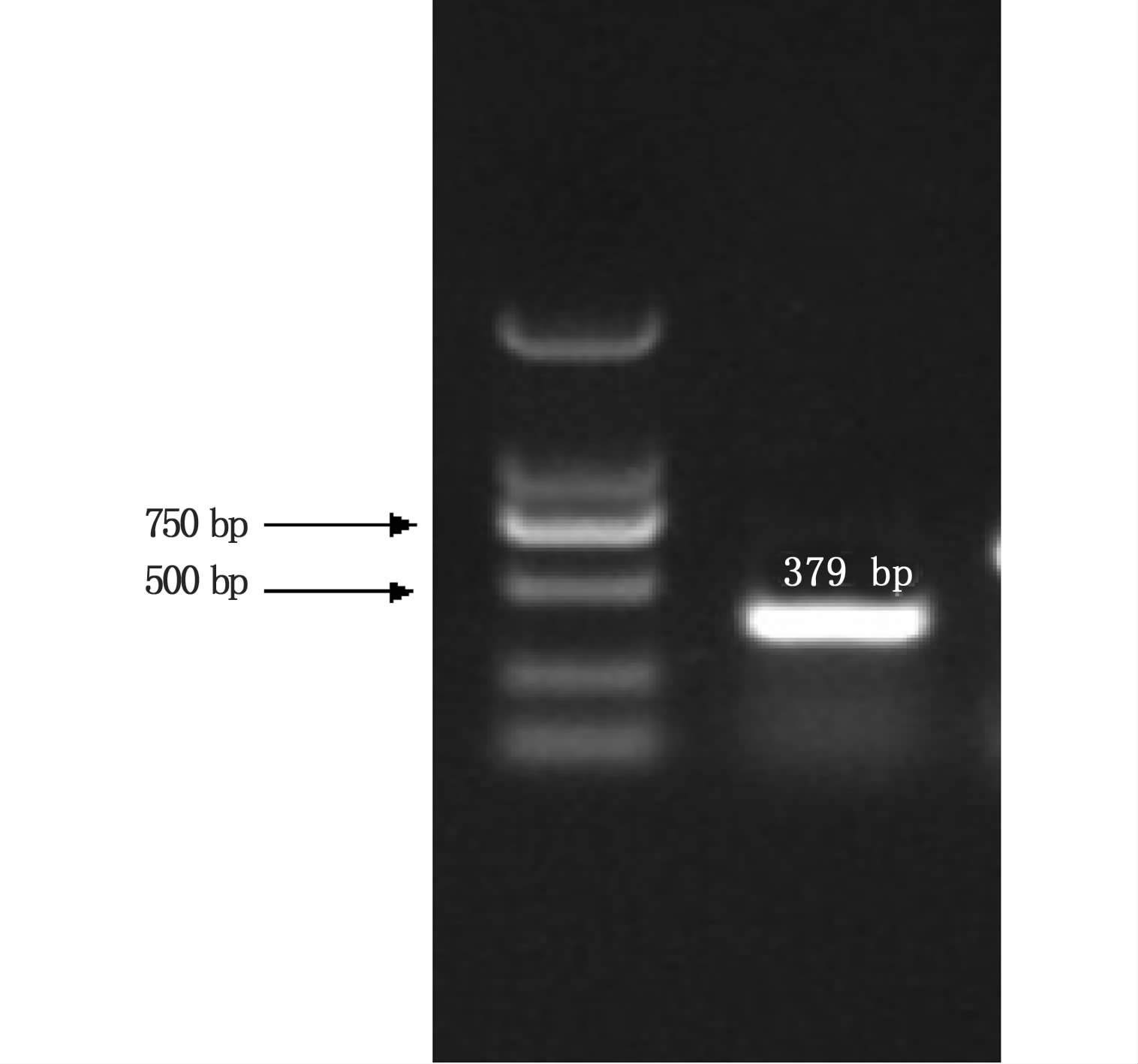
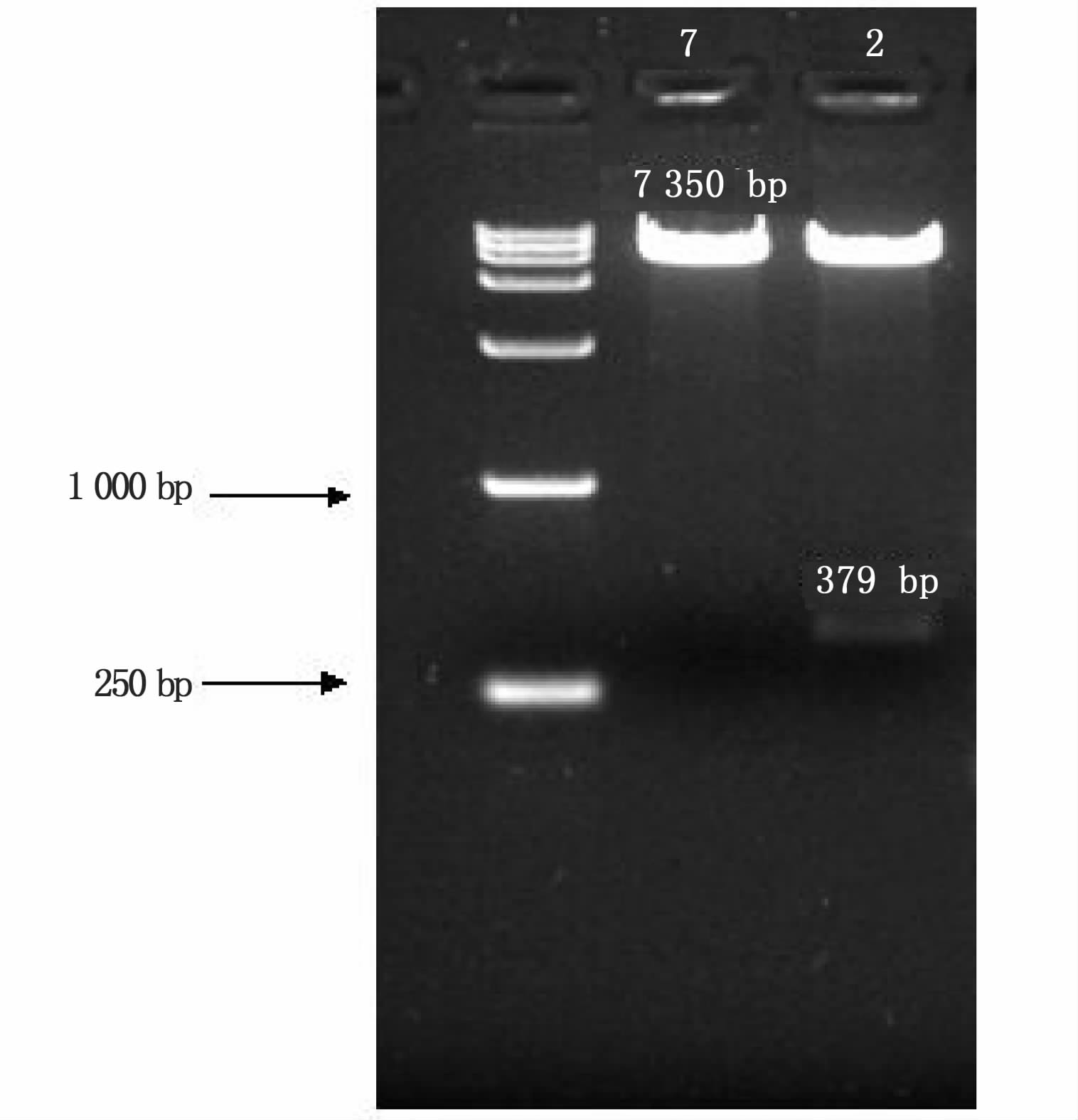
根据预测的miR-33在SREBP-1 3'-UTR的靶序列及其所在位置设计引物,PCR产物经琼脂糖凝胶电泳显示为一条长度约380 bp的条带,与实际长度(379 bp)相符(图 2),测序结果证实该片断基因序列完全正确。将扩增片断插入线性化的pmirGLO载体,再用双酶切鉴定,结果显示酶切后得到的片段与预期片段大小相符(图 3)。双向测序结果显示,重组载体中的SREBP-1-3'-UTR序列与PCR产物完全一致,也与GenBank数据库上的吻合,表明该序列已成功转入pmirGLO载体中。
|
图 2 SREBP-1 基因3'UTR扩增产物电泳图
Fig. 2 PCR products of SREBP-1-3'UTR by 1% agrose gel
|
|
图 3 pmirGLO重组载体双酶切结果
Fig. 3 The double digestion results of pmirGLO recombination plasmid
|
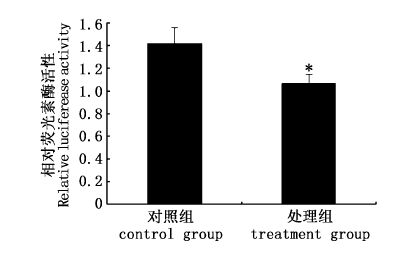
对照组(只转染pmirGLO-SREBP-1-3'-UTR载体)和处理组(共转染pmirGLO-SREBP-1-3'-UTR载体和miR-33 mimic)细胞均检测到荧光素酶活性,但对照组酶活性(1.417±0.141)显著高于处理组(1.064±0.081)(P<0.05,图 4)。miR-33mimics显著下调了pmirGLO-SREBP-1-3'-UTR荧光素酶报告载体的报告荧光,说明SREBP-1应是miR-33的靶基因。
|
图 4 转染细胞的荧光素酶活性
Fig. 4 Relative luciferase activity of transfected cells
|
miRNA和SREBP均是脂代谢调节网络中的重要成员。SREBP是哺乳动物肝脏脂肪生成基因的主要调节者[1-2],不仅在脂类代谢中发挥重要作用,还参与对葡萄糖的代谢调节[13]。miRNA几乎参与了动物脂代谢的所有调节层次,包括脂肪细胞分化、脂肪组织能量代谢、肝脏脂类代谢和脂类代谢调节相关激素的分泌[14-15]。目前已确定包括miR-33、miR-122、miR-370、miR-378、miR-302a 和 miR-106b 在内的多个miRNA参与调控脂类代谢[16],其中miR-33定位在SREBP基因的内含子上,与SREBP共表达,二者共同参与调控机体胆固醇及脂类稳态[7]。miR-33a不仅靶向调控Abca1、Abcg1、CPT1A、CROT等与细胞胆固醇稳态和脂肪酸氧化相关的基因[3, 6],也是SPEBP-1表达的调节因子[8],通过与SREBP-1 3'-UTR结合下调其表达。虽然已证实SREBP-1与miR-33在脊椎动物中高度保守[9-10],但在鱼类中关于二者的研究并不多。我们在之前的研究中已克隆出草鱼SREBP-1 全长cDNA[12]。为验证草鱼SREBP-1是否如哺乳动物一样是miR-33的靶基因,本研究先通过软件预测靶向SREBP-1基因的miRNA,发现miR-33和miR-16在SREBP-1的3'UTR区均存在可以互补配对的靶序列。为避免假阳性需通过实验的方法验证预测结果是否准确[17]。一般采用双荧光素酶检测和免疫印迹进行验证[18-19],本研究选择双荧光素酶基因报告法,通过克隆含miR-33结合位点的SREBP-1的3'UTR序列,成功构建了SREBP-1-3'UTR双荧光素酶基因报告载体,并通过miR-33mimics和报告基因载体共转染草鱼肝细胞L8824,判断miR-33对细胞荧光素酶表达的影响。结果表明,miR-33mimics明显下调了SREBP-1-3'-UTR荧光素酶报告载体的报告荧光,说明SREBP-1是miR-33直接调控的靶基因,miR-33通过与SREBP-1 mRNA 3'UTR结合,对其进行转录后水平调控。本研究为进一步揭示miR-33与SREBP-1在草鱼脂代谢调控网络中的作用奠定了理论和材料基础。
| [1] | HORTON J D, GOLDSTEIN J L, BROWN M S.Brown. SREBPs: activators of the complete program of cholesterol and fatty acid synthesis in the liver[J]. The Journal of Clinical Investigation, 2002, 109(9): 1125–1131. DOI:10.1172/JCI0215593 |
| [2] | ONO K, HORIE T, NISHINO T, et al.MicroRNA-33a/b in lipid metabolism-novel “Thrifty” models[J]. Circulation Journal, 2015, 79(2): 278–284. DOI:10.1253/circj.CJ-14-1252 |
| [3] | HORIE T, ONO K, HORIGUCHI M, et al.MicroRNA-33 encoded by an intron of sterol regulatory element-binding protein 2(Srebp2) regulates HDL in vivo[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(40): 17321–17326. DOI:10.1073/pnas.1008499107 |
| [4] | MARQUART T J, ALLEN R M, ORY D S, et al.miR-33 links SREBP-2 induction to repression of sterol transporters[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(27): 12228–12232. DOI:10.1073/pnas.1005191107 |
| [5] | RAYNER K J, SUáREZ Y, DáVALOS A, et al.miR-33 contributes to the regulation of cholesterol homeostasis[J]. Science, 2010, 328(5985): 1570–1573. DOI:10.1126/science.1189862 |
| [6] | GERIN I, CLERBAUX L A, HAUMONT O, et al.Expression of miR-33 from an SREBP2 intron inhibits cholesterol export and fatty acid oxidation[J]. The Journal of Biological Chemistry, 2010, 285(44): 33652–33661. DOI:10.1074/jbc.M110.152090 |
| [7] | NAJAFI-SHOUSHTARI S H, KRISTO F, LI Y X, et al.MicroRNA-33 and the SREBP host genes cooperate to control cholesterol homeostasis[J]. Science, 2010, 328(5985): 1566–1569. DOI:10.1126/science.1189123 |
| [8] | HORIE T, NISHINO T, BABA O, et al.MicroRNA-33 regulates sterol regulatory element-binding protein 1 expression in mice[J]. Nature Communications, 2013, 4: 2883. |
| [9] | CARMONA-ANTOÑANZAS G, TOCHER D R, MARTINEZ-RUBIO L, et al.Conservation of lipid metabolic gene transcriptional regulatory networks in fish and mammals[J]. Gene, 2014, 534(1): 1–9. DOI:10.1016/j.gene.2013.10.040 |
| [10] | GHARIPOUR M, SADEGHI M.Pivotal role of microRNA-33 in metabolic syndrome: a systematic review[J]. ARYA Atherosclerosis, 2013, 9(6): 372–376. |
| [11] | EGEA M, METÓN I, CORDÓBA M, et al.Role of Sp1 and SREBP-1a in the insulin-mediated regulation of glucokinase transcription in the liver of gilthead sea bream (Sparus aurata)[J]. General and Comparative Endocrinology, 2008, 155(2): 359–367. DOI:10.1016/j.ygcen.2007.06.018 |
| [12] | 孙君君, 卢荣华, 杨峰, 等.草鱼SREBP-1基因的克隆及糖对其在肝脏中表达的影响[J]. 水产学报, 2014, 38(8): 1057–1067. SUN J J, LU R H, YANG F, et al.Molecular cloning of SREBP-1 gene and effects of carbohydrates on its expression in liver of Ctenopharyngodon idella[J]. Journal of Fisheries of China, 2014, 38(8): 1057–1067. |
| [13] | JEON T I, OSBORNE T F.SREBPs: metabolic integrators in physiology and metabolism[J]. Trends in Endocrinology and Metabolism, 2012, 23(2): 65–72. DOI:10.1016/j.tem.2011.10.004 |
| [14] | TARIQ Z, GREEN C J, HODSON L.Are oxidative stress mechanisms the common denominator in the progression from hepatic steatosis towards non-alcoholic steatohepatitis (NASH)[J]. Liver International, 2014, 34(7): e180–e190. DOI:10.1111/liv.2014.34.issue-7 |
| [15] | HUA X X, SAKAI J, HO Y K, et al.Hairpin orientation of sterol regulatory element-binding protein-2 in cell membranes as determined by protease protection[J]. Journal of Biological Chemistry, 1995, 270(49): 29422–29427. DOI:10.1074/jbc.270.49.29422 |
| [16] | 邵芳, 朱斌, 顾志良.microRNA在脂类代谢中的功能研究进展[J]. 生命科学, 2013, 25(7): 676–684. SHAO F, ZHU B, GU Z L.Function of microRNAs in lipid metabolism[J]. Chinese Bulletin of Life Sciences, 2013, 25(7): 676–684. |
| [17] | ØROMU A, LUNDA H.Experimental identification of microRNA targets[J]. Gene, 2010, 451(1/2): 1–5. |
| [18] | LI S J, LI Z G, GUO F J, et al.miR-223 regulates migration and invasion by targeting Artemin in human esophageal carcinoma[J]. Journal of Biomedical Science, 2011, 18: 24. DOI:10.1186/1423-0127-18-24 |
| [19] | ARAU'JO P R, BURLE-CALDAS G A, SILVA-PEREIRA R A, et al.Development of a dual reporter system to identify regulatory cis-acting elements in untranslated regions of Trypanosoma cruzi mRNAs[J]. Parasitology International, 2011, 60(2): 161–169. DOI:10.1016/j.parint.2011.01.006 |
2. College of Fisheries, Henan Normal University, Xinxiang 453007, Henan, China
 2017,
Vol. 26
2017,
Vol. 26


