2. Research Institute for Aquaculture No. 1, Dinh Bang, Tu Son, Bac Ninh, Vietnam;
3. 淮海工学院 海洋生物产业技术协同创新中心, 江苏 连云港 222005
缢蛏(Sinonovacula constricta)俗称蛏子, 属软体动物门(Mollusca), 瓣鳃纲,(Lamellibr-anchia),竹蛏科(Solenidae),缢蛏属(Sinonovacula)贝类。缢蛏为广温广盐性海产双壳类, 具有重要的经济价值, 是我国四大养殖贝类之一。缢蛏的养殖历史悠久,主要在我国浙江、福建沿海地区有大规模养殖,并且有较成熟的养殖技术[1]。
长期以来,贝类的养殖工作一直以表型为基础,但是其表型受到环境因素的影响很大。根据表型对性状的选择,其效率较低。因此,利用分子辅助育种的方法能更有效改善其养殖进程,这对缢蛏养殖尤为重要。近年来,缢蛏人工养殖过程中出现群体种质退化,且养殖群体成活率低的问题,原因可能是在人工选育过程中,过度近亲交配,从而发生后代活力低下所致。为了防止发生近交过度的现象,掌握繁殖亲本的系谱,是解决这一现象的有效途径。因此群体间的亲子鉴定[2-3]尤为重要。
微卫星由于其突变率高和多态性高,且符合孟德尔遗传定律,为共显性遗传,是一种应用价值较高的分子标记。广泛应用于种质资源鉴定、生物遗传作图[4]和遗传多样性分析等研究[5]。在贝类微卫星开发与研究中,国内外均已有较多报道,如虾夷扇贝(Patinopecten yessoensis)[6-7]、紫贻贝(Mytilus galloprovincialis)[8]、文蛤(Meretrix meretrix)[9]和长牡蛎(Crassostrea gigas)[10]的微卫星研究。利用微卫星标记[11]对养殖个体进行亲子鉴定,对于今后的家系选育具有重要的意义。
1 材料与方法 1.1 缢蛏F1家系的构建及样品采集2013年8月,以福建的缢蛏为父母本,在浙江宁海养殖基地,以一对一交配的方式构建了家系,家系父母本在交配结束后,取整个个体浸泡于100%的乙醇中,并且-20 ℃低温保存。缢蛏受精卵在室内育苗池进行为期40 d的养殖至稚贝期后转入室外池塘入泥生长,并且对每个家系进行区域划分养殖。2014年5月,选取1个发育良好的10月龄成贝家系,随机采集家系的300个子代,用泡沫箱4 ℃低温保存带回实验室。
1.2 DNA提取及检测每个样本取0.5 g外套膜组织剪碎,加入490 μL组织匀浆缓冲液(10 mmol/dm3 Tris-HCL, 50 mmol/dm3 EDTA, 1% SDS; pH=8.0),混匀后加入蛋白酶K 10 μL,55 ℃消化澄清。采用苯酚-氯仿法抽提[12]基因组DNA,超微量蛋白核酸分析仪测定DNA浓度,利用2%琼脂糖凝胶电泳检测完整性。将符合要求的基因组DNA置于-20℃保存备用。
1.3 微卫星富集文库构建及引物设计基因组DNA的酶切和富集文库的构建主要参照罗明等[13]和LI等[14]的方法。用RsaⅠ内切酶进行缢蛏基因组DNA的酶切,用试剂盒回收200~1 000 bp大小的片段用于文库构建。微卫星序列的富集采用生物素标记的探针,具体操作步骤与罗明等[13-14]所述相同。利用Sequencher软件去除载体及接头后,用SSRhunter软件来查找其中微卫星位点,用GeneQuest软件查找并去除冗余序列。用PrimerSelect软件在微卫星序列上进行引物设计。
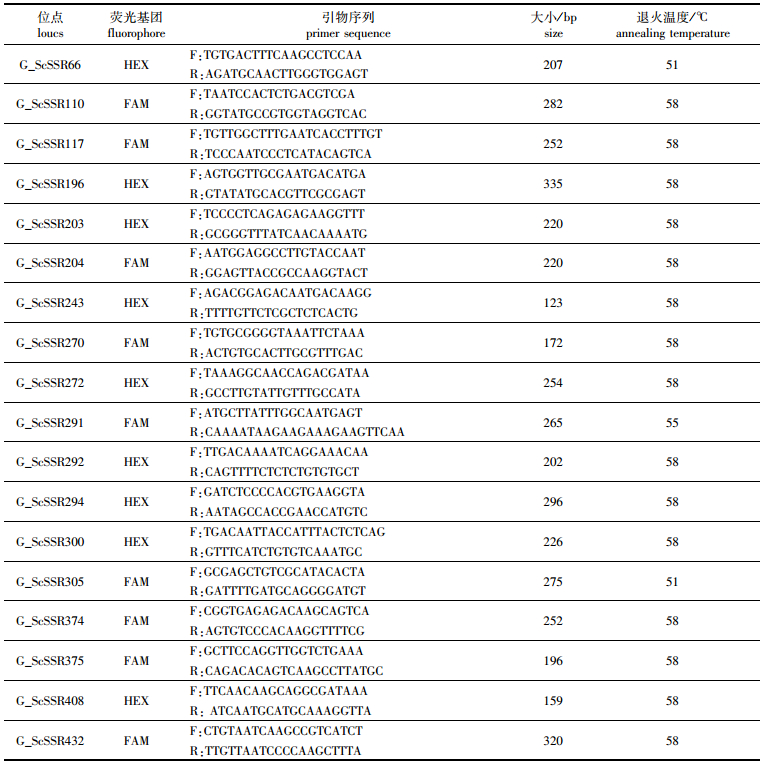
1.4 微卫星标记检验随机选取已筛选的微卫星标记40个,合成5′上游荧光引物(FAM和HEX)[15]。先用家系父母本和10个子代共12个个体的基因组DNA为模板,进行PCR扩增预实验。PCR扩增反应总体积为20 μL,其中DNA模板(20 ng/μL)3 μL,2×Taq PCR MasterMix (上海生工生物工程有限公司)10 μL,正向和反向引物各1 μL,去离子水3 μL。PCR反应体系:94 ℃预变性2 min;94 ℃变性30 s,最佳退火温度根据不同引物分别依次为51 ℃、55 ℃、58 ℃;72 ℃延伸40 s,循环35次;72 ℃延伸10 min;10 ℃保存。在上海迈普生物有限公司进行STR分型检测,用软件Cervus 3.0计算每个微卫星位点的多态信息含量(Polymorphism Information Content, PIC),筛选符合标准的微卫星标记。
1.5 数据分析将筛选出来的微卫星标记,通过PCR扩增检验一个家系中剩余的174个子代在每个位点的基因型, 进行STR分型。用软件GeneMapper 4.0对STR分型结果进行分析,参数标准为ROX500。用Cervus 3.0软件对数据进行分析,计算亲缘关系。
2 结果 2.1 DNA提取将提取出来的缢蛏基因组DNA用超微量核酸蛋白仪检测,结果显示,DNA样品的OD260/OD280均在1.8~2.2之间,基因组DNA纯度较高;DNA浓度约为500~1 000 ng/μL,提取的DNA产量较高;用2%的琼脂糖凝胶电泳检测发现,多数提取的DNA片段主带清晰,说明有较好的完整性。最终将DNA浓度稀释至约20 ng/μL作为DNA模板,4 ℃保存备用
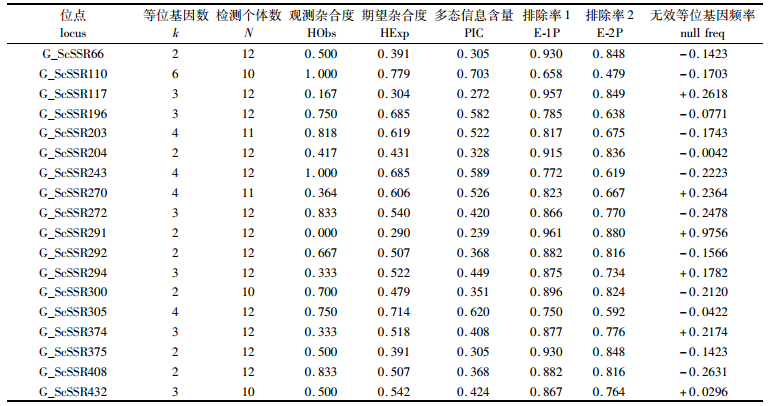
2.2 多态性引物设计及筛选结果用缢蛏父母本及10个F1个体对引物进行初筛,PCR产物扩增结果用2%的琼脂糖凝胶电泳检测发现得到18对稳定扩增、条带清晰的引物。对这18对引物的5′上游荧光引物分别用FAM和HEX[15]两种荧光基团来修饰,用父母本和10个子代个体进行PCR扩增。用GeneMapper 4.0软件对基因组扫描结果进行分析读数,用Cervus 3.0进行遗传多样性分析(表 1)。18对微卫星引物在12个个体中等位基因为2~6,平均每对等位基因为3.00,位点G_ScSSR110等位基因最多,为6个;观测杂合度在0~1.000之间,平均值为0.581 3;期望杂合度为0.290~0.779,平均值为0.528 4;多态信息含量为0.239~0.703,平均值为0.432 2(表 2)。且当两个亲本基因型未知时,单个亲本排除率(E-1P)为0.658~0.930,平均值为0.840 9;当一个亲本基因型已知时,另一个亲本排除率(E-2P)为0.479~0.848,平均值为0.729 1;一个微卫星位点的各种亲子排除率越高,也就证明了该位点越适合于亲子鉴定。
|
表 1 缢蛏18个微卫星位点的序列及引物特征 Tab.1 18 microsatellites repeat motifs and relevant primer characteristics of Sinonovacula constricta |
|
表 2 缢蛏18个微卫星位点的遗传参数 Tab.2 Genetic parameters of 18 microsatellite loci in Sinonovacula constricta |
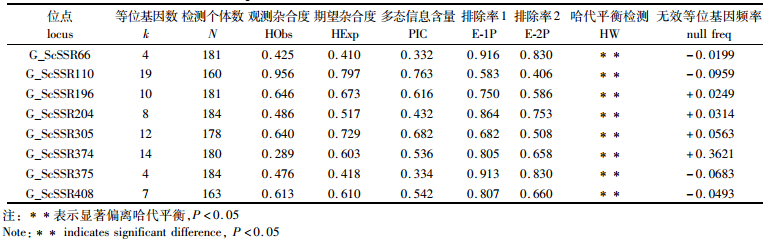
选取8对排除率较高的微卫星引物对剩余174个个体进行PCR扩增,PCR产物进行STR分型,得到数据用GeneMapper 4.0软件进行读数,用Cervus 3.0软件对数据进行分析,计算亲缘关系(表 3)。结果显示,在已知父母本的情况下,在184个F1中,共检测出154个个体为同一家系。其观测杂合度为0.289~0.956,平均值为0.566 4;期望杂合度为0.410~0.797,平均值为0.594 7;多态信息含量为0.332~0.763,平均值为0.529 7。通过Cervus统计得到8个微卫星位点的亲子标记排除率,当两个亲本基因型未知时,单个亲本排除率(E-1P)为0.583~0.916,平均值为0.790 0;当一个亲本基因型已知时,另一个亲本的排除率(E-2P)为0.406~0.830,平均值为0.653 9。同时可知在子代个体数量不断增加的情况下,排除率仍在较高范围。从结果中,我们排除错配在1对引物以上的子代个体。据此,可以确定F1中154个个体为同一家系。
|
表 3 8个微卫星位点在184个个体中的遗传参数 Tab.3 Genetic parameters of 8 microsatellite loci in Sinonovacula constricta |
微卫星分子标记具有多态性丰富、重复性好等优点,是近年来水产养殖中一种较为理想的分子标记手段。微卫星DNA具有孟德尔遗传特性和共显性的特征,可以通过分析基因型特征来确定物种的遗传杂合度,同时可以描述物种的遗传结构。对于共显性特征来说,可以用排除法来进行亲缘关系分析及亲子鉴定;对于孟德尔遗传定律来说,子代分别遗传父本和母本的一个等位基因,因此不符合孟德尔遗传规律的个体便可排除在外[16]。近年来已有许多将微卫星标记应用于水产动物亲子鉴定的报道,如苗贵东等[17]利用8对微卫星引物对人工选育大菱鲆家系进行系谱认证、亲子鉴定和遗传多样性研究;张春雷等[18]通过7对微卫星引物,对哲罗鱼亲子鉴定准确率进行了分析;程鹏等[7]选取8个微卫星引物,用于虾夷扇贝4个不同壳色全同胞和半同胞家系的亲权鉴定。用于亲子鉴定的分子标记需要具有共显性且稳定遗传给子代,同时等位基因的分离符合孟德尔遗传定律。微卫星分子标记正符合这些特征。
开发筛选微卫星标记的方法有很多,本实验采用磁珠富集法开发微卫星标记,此方法利用了吸附微卫星核心序列的探针来寻找微卫星片段序列,通过PCR将其富集,能得到较好的结果,并且效率高,筛选结果高。同时用微卫星荧光标记基因组扫描,其PCR产物可以分析微卫星长度,且精确长度为1 bp。其优点是可以区分复制时滑动而引起的次斑,次斑对PCR产物长度有些许干扰,是影响数据可靠性的重要原因[13]。荧光引物合成的缺点是成本相对较高。
影响物种遗传多样性的主要特征是杂合度[19],杂合度越高表明该物种遗传多样性高;反之则说明遗传一致性高。本实验18对引物观测杂合度平均值为0.581 4,期望杂合度平均值为0.528 4,表明杂合度与物种的遗传多样性均属于较高水平。其中G_ScSSR374,G_ScSSR270,G_ScSSR291,G_ScSSR294,G_ScSSR374,G_ScSSR432的观测杂合度低于期望杂合度,可能是因为出现无效等位基因所致[20]。部分个体由于在引物结合位点存在碱基突变、插入或缺失而导致该位点等位基因不能被合成引物通过PCR扩增出来,称为无效等位基因。PIC是能较好地体现群体多态性和遗传信息含量的标准[21]。PIC平均值0.432 2,一般认为0.25 < PIC < 0.5为中度多态,PIC>0.5即为高度多态。本实验微卫星位点显著偏离哈代平衡(HWE,P < 0.05),偏离现象的发生说明在缢蛏养殖群体中某种类型基因型个体缺少或者偏少,原因可能是在受精过程中配子间存在选择或者环境对某些致死基因进行了选择[22-24]。其中环境对致死基因的选择对微卫星标记偏离现象的发生影响较大[6, 24-25]。同时有研究发现在双壳类中遗传负荷较大,使得双壳类中出现偏离比例较高的问题[26-27]。这些致死基因存在于幼虫发育阶段,导致幼虫在成为成贝之前大量死亡。LAUNEY等[27]的研究中,用幼虫期大量死亡后存活的长牡蛎成贝作为实验材料,导致了标记发生偏离现象。
在用微卫星引物进行亲子鉴定时,微卫星位点的排除率高低取决于亲本的数量以及位点多态性[28]。本实验选取用于亲子鉴定的引物,均在双亲未知排除率和单亲未知排除率上表现较高的水平。同时个别微卫星位点存在无效等位基因,会降低亲子鉴定的成功率[29]。DNA模板质量低,配子在减数分裂过程中个别微卫星位点发生变异,设计合成的引物序列不具备高保守性而导致退火效率降低[30-31]等因素都会发生出现无效等位基因的情况。本实验的无效等位基因均为纯合,且在一个家系中,分析其原因可能是由于微卫星位点发生变异,造成在进行PCR扩增时出现等位基因缺失。在鉴定子代个体数增加的情况下,两种亲缘排除率均出现下降的情况,可能是因为基因分型的错误而导致子代与亲本发生错配。有研究表明,每个基因座位都存在2%~3%的基因分型错误[32]而发生错配。本实验亲缘排除率在个体增加的情况下出现下降,仍属于正确结果。
| [1] | 周炳元. 缢蛏养殖技术[M]. 北京: 金盾出版社, 2002. ZHOU B Y. The breeding technology of Sinonovacula constricta[M]. Beijing: Jindun Publishing House, 2002. |
| [2] | 何小燕, 白俊杰, 刘小林, 等.微卫星DNA标记在大口黑鲈亲权鉴定中的应用[J]. 西北农林科技大学学报(自然科学版), 2009, 37(8): 55–62. HE X Y, BAI J J, LIU X L, et al.Application of microsatellite makers for parentage determination in Micropterus salmoides[J]. Journal of Northwest University (Natural Science), 2009, 37(8): 55–62. |
| [3] | HERLIN M, DELGHANDI M, WESMAJERVI M, et al.Analysis of the parental contribution to a group of fry from a single day of spawning from a commercial Atlantic cod (Gadus morhua) breeding tank[J]. Aquaculture, 2008, 274(2/3/4): 218–224. |
| [4] | RUAN X H, WANG W J, KONG J, et al.Genetic linkage mapping of turbot (Scophthalmus maximus L.) using microsatellite markers and its application in QTL analysis[J]. Aquaculture, 2010, 308(3/4): 89–100. |
| [5] | 盛岩, 郑蔚虹, 裴克全, 等.微卫星标记在种群生物学研究中的应用[J]. 植物生态学报, 2002, 26(s1): 119–126. SHENG Y, ZHENG W H, PEI K Q, et al.Applications of microsatellites in population biology[J]. Acta Phytoecologica Sinica, 2002, 26(s1): 119–126. |
| [6] | SATO M, KAWAMATA K, ZASLAVSKAYA N, et al.Development of microsatellite markers for Japanese scallop (Mizuhopecten yessoensis) and their application to a population genetic study[J]. Marine Biotechnology, 2005, 7(6): 713–728. DOI:10.1007/s10126-004-0127-8 |
| [7] | 程鹏, 杨爱国, 吴彪, 等.微卫星标记在不同壳色虾夷扇贝家系亲权鉴定的适用性[J]. 水生生物学报, 2011, 35(5): 768–775. CHENG P, YANG A G, WU B, et al.The applicability analysis on microsatellite markers for parentage determination of different shell color lines of Japanese scallop Patinopecten yessoensis[J]. Acta Hydrobiologica Sinica, 2011, 35(5): 768–775. |
| [8] | 齐明君.紫贻贝和虾夷扇贝雌核发育二倍体的遗传学研究[D].青岛:中国海洋大学, 2013. QI M J. Genetic studies on gynogenetic diploids in Mytilus edulis and Patinopecten yessoensis[D]. Qingdao:Ocean University of China, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10423-1013368585.htm |
| [9] | 卢霞.分子标记应用于文蛤遗传评价、图谱构建及QTL筛选的研究[D].北京:中国科学院研究生院, 2012. LU X. Investigation of molecular markers applied in genetic estimation, linkage mapping and QTL identification in the Clam[D]. Beijing:Graduate University of Chinese Academy of Sciences, 2012. http://cdmd.cnki.com.cn/Article/CDMD-80068-1012411009.htm |
| [10] | 李莉.长牡蛎的分子标记筛选和遗传图谱构建[D].北京:中国科学院研究生院, 2003. LI L. Molecular marker development and linkage mapping in the Pacific oyster Crassostrea gigas Thunberg[D]. Beijing:Graduate University of Chinese Academy of Sciences, 2003. http://cdmd.cnki.com.cn/Article/CDMD-80068-2003099115.htm |
| [11] | GOLDSTEIN D B, SCHLOTTERER C. Microsatellites:evolution and applications[M]. Oxford: Oxford University Press, 1999. |
| [12] | SAMBROOK J E, FRITSCH E F M, MANIATIS T E.T Molecular cloning:A laboratory manual[J]. Immunology, 1983, 49(1): 895–909. |
| [13] | 罗明, 白志毅, 李应森, 等.三角帆蚌微卫星位点筛选及多态性分析[J]. 淡水渔业, 2012, 42(1): 80–84. LUO M, BAI Z Y, LI Y S, et al.Development and genetic diversity analysis of microsatellite in Hyriopsis cumingii[J]. Freshwater Fisheries, 2012, 42(1): 80–84. |
| [14] | LI J L, WANG G L, BAI Z Y, et al.Ten polymorphic microsatellites from freshwater pearl mussel, Hyriopsis cumingii[J]. Molecular Ecology Resources, 2007, 7(6): 1357–1359. |
| [15] | KALINOWSKI S T, TAPER M L, MARSHALL T C.Revising how the computer program cervus accommodates genotyping error increases success in paternity assignment[J]. Molecular Ecology, 2007, 16(5): 1099–1106. DOI:10.1111/j.1365-294X.2007.03089.x |
| [16] | WILSON A J, FERGUSON M M.Molecular pedigree analysis in natural populations of fishes:approaches, applications, and practical considerations[J]. Canadian Journal of Fisheries and Aquatic Sciences, 2002, 59(10): 1696–1707. DOI:10.1139/f02-127 |
| [17] | 苗贵东, 杜民, 杨景峰, 等.大菱鲆亲子鉴定的微卫星多重PCR技术建立及应用[J]. 中国海洋大学学报(自然科学版), 2011, 41(1): 97–106. MIAO G D, DU M, YANG J F, et al.Development of two microsatellite multiplex PCR systems for turbot (Scophthalmu smaximus) and its application for family management[J]. Periodical of Ocean University of China (Natural Science), 2011, 41(1): 97–106. |
| [18] | 张春雷, 佟广香, 匡友谊, 等.哲罗鱼微卫星亲子鉴定的应用[J]. 动物学研究, 2010, 31(4): 395–400. ZHANG C L, DONG G X, KUANG Y Y, et al.Applicability of microsatellite DNA markers to the parental identification of Hucho taimen (Pallas)[J]. Zoological Research, 2010, 31(4): 395–400. |
| [19] | 李莉, 孙振兴, 杨树德, 等.用微卫星标记分析皱纹盘鲍群体的遗传变异[J]. 遗传, 2006, 28(12): 1549–1554. LI L, SUN Z X, YANG S D, et al.Analysis of genetic variation of abalone (Haliotis discus hannai) populations with microsatellite markers[J]. Hereditas, 2006, 28(12): 1549–1554. |
| [20] | 陈微, 张全启, 于海洋, 等.牙鲆微卫星标记的筛选及群体多态性分析[J]. 中国水产科学, 2005, 12(6): 682–687. CHEN W, ZHANG Q Q, YU H Y, et al.Isolation and characterization of polymorphic microsatellite loci in olive flounder (Paralichthys olivaceus)[J]. Journal of Fishery Sciences of China, 2005, 12(6): 682–687. |
| [21] | BOTSTEIN D, WHITE R L, SKOLNICK M, et al.Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314–331. |
| [22] | LYTTLE T W.Segregation distorters[J]. Annual Review of Genetics, 1991, 25: 511–581. DOI:10.1146/annurev.ge.25.120191.002455 |
| [23] | HEDGECOCK D, LI G, HUBERT S, et al.Widespread null alleles and poor cross-species amplification of microsatellite DNA loci cloned from the Pacific oyster, Crassostrea gigas[J]. Journal of Shellfish Research, 2004, 23(2): 379–385. |
| [24] | LANDERGOTT U, HOLDEREGGER R, KOZLOWSKI G, et al.Historical bottlenecks decrease genetic diversity in natural populations of Dryopteris cristata[J]. Heredity, 2001, 87(3): 344–355. DOI:10.1046/j.1365-2540.2001.00912.x |
| [25] | LIU B Z, DONG B, TANG B J, et al.Effect of stocking density on growth, settlement and survival of clam larvae, Meretrix meretrix[J]. Aquaculture, 2006, 258(1/2/3/4): 344–349. |
| [26] | FARIS J D, LADDOMADA B, GILL B S.Molecular mapping of segregation distortion loci in Aegilops tauschii[J]. Genetics, 1998, 149(1): 319–327. |
| [27] | LAUNEY S, HEDGECOCK D.High genetic load in the Pacific oyster Crassostrea gigas[J]. Genetics, 2001, 159(1): 255–265. |
| [28] | DONG S R, KONG J, ZHANG T S, et al.Microsatellite markers simulation and application for parentage determination on Fenneropenaeus chinensis[J]. Acta Hydrobiologica Sinica, 2008, 32(1): 96–101. DOI:10.3724/SP.J.1035.2008.00096 |
| [29] | MARSHALL T C, SLATE J, KRUUK L E B, et al.Statistical confidence for likelihood-based paternity inference in natural populations[J]. Molecular Ecology, 1998, 7(5): 639–655. DOI:10.1046/j.1365-294x.1998.00374.x |
| [30] | PEMBERTON J M, SLATE J, BANCROFT D R, et al.Nonamplifying alleles at microsatellite loci:a caution for parentage and population studies[J]. Molecular Ecology, 1995, 4(2): 249–252. DOI:10.1111/j.1365-294X.1995.tb00214.x |
| [31] | JONES A G, STOCKWELL C A, WALKER D, et al.The molecular basis of a microsatellite null allele from the White Sands Pupfish[J]. The Journal of Heredity, 1998, 89(4): 339–342. DOI:10.1093/jhered/89.4.339 |
| [32] | O'REILLY P T, HERBINGER C, WRIGHT J M.Analysis of parentage determination in Atlantic salmon (Salmo salar) using microsatellites[J]. Animal Genetics, 1998, 29(5): 363–370. DOI:10.1046/j.1365-2052.1998.295359.x |
2. Research Institute for Aquaculture No. 1, Dinh Bang, Tu Son, Bac Ninh, Vietnam;
3. Co-Innovation Center of Jiangsu Marine Bio-industry Technology, Huaihai Institute of Technology, Lianyungang 222005, Jiangsu, China
 2016,
Vol. 25
2016,
Vol. 25


