2. 上海海洋大学 水产与生命学院, 上海 201306
抗生素的发现和应用拯救了数十亿人的生命,同时也推动了陆生和水生动物养殖业的发展。随着抗菌药物在人类医学临床尤其是在食品动物养殖中的广泛使用,细菌耐药性问题日趋严重。整合子是介导革兰氏阴性菌抗菌药物耐药产生和迅速传播的重要机制之一[1]。它是细菌的一种天然的克隆和表达系统,可在整合酶的催化下介导耐药基因盒的捕获、整合和切除,造成耐药基因在菌群中迅速传播[2]。目前,已报道的耐药相关的整合子包括五类,其中1型整合子在临床菌株中最为常见。整合子携带的耐药基因盒种类多达130余种,涉及到的抗菌药物包括:β-内酰胺类、氨基糖苷类、磺胺类、酰胺醇类、氨基糖苷类、利福霉素类等[3]。在一些复杂的1类整合子3′端保守区的下游通常存在有一段长为2 154 bp、可编码513个氨基酸的DNA序列,因此被称为Orf513,后来又被命名为1型插入序列共同区(Insertion Sequence Common Region, ISCR1)[4]。ISCR1是一种通过“滚环转座”机制转移其相邻耐药基因的可移动遗传元件;大量的证据表明,ISCR1与多种抗菌药物耐药基因的传播扩散密切相关,如氯霉素类、甲氧苄啶类、喹诺酮类、氨基糖苷类耐药基因以及A类和C类β内酰胺酶基因等[5-6]。4类整合子即超级整合子SXT首次发现于霍乱弧菌,被认为是指导宿主基因进化的主要因素,它可携带耐药基因、毒力基因等上百个基因盒,可使细菌迅速适应环境的变化,是细菌生存和进化的重要遗传元件[7]。目前,上述3种整合子相关元件已在国内外多种来源的弧菌中被大量报道,涉及到耐药基因的种类包括:酰胺醇耐药基因(cat2、floR)、β-内酰胺耐药基因(blaPER、blaVIM-1、blaOXA-10)、氨基糖苷类耐药基因(aadA1、aacA3、aadA2、aadB、strAB)、四环素类耐药基因(tetA)、喹诺酮类耐药基因(qnrVC)、利福霉素类耐药基因(arr)、甲氧苄啶耐药基因(dfrA1、dfr16)、磺胺类耐药基因(sul1、sul2)等[8-12]。本实验室在开展海水养殖源弧菌耐药性研究的过程中,从江苏、山东、辽宁、福建和海南等地区分离的多种弧菌中检测到了Int1、SXT、ISCR1的流行,但未检测到其他类型整合子元件(如1和2类整合子)的存在。然而,到目前为止还没有一种能同时检测Int1、SXT、ISCR1这3种元件的方法。
为了建立一种能同时检测1类整合子Int1、4类超级整合子SXT、1型插入序列共同区ISCR1的方法,本研究根据GenBank中int1、intSXT及ISCR1的基因序列,利用PrimerPlex 2.61设计了3对PCR引物,建立一种多重PCR体系,并通过改变引物浓度、退火温度等对其进行了优化。本研究的成果提供了一种同时检测上述3种整合子相关元件的方法,为整合子相关细菌耐药性研究的开展提供了便利。
1 材料与方法 1.1 实验材料 1.1.1 菌株来源阳性对照菌:溶藻弧菌VA-001含有Int1、SXT和ISCR1,创伤弧菌VV-311含有Int1和ISCR1,溶藻弧菌VA-352含有Int1和SXT,副溶血弧菌VP-046含有SXT、ISCR1,均为本实验室从海水养殖中临床分离的菌株,其耐药元件PCR产物经测序确认。阴性对照菌:副溶血弧菌CICC21617、创伤弧菌CICC10383和溶藻弧菌CICC10484购自中国工业微生物菌种保藏中心。受试弧菌:副溶血弧菌133株、哈维氏弧菌32株、溶藻弧菌36株、创伤弧菌10株、霍乱弧菌7株和鳗弧菌4株,共计222株,分离自上海、江苏、福建和海南地区养殖的发病对虾、舌鳎和青蟹等。
1.1.2 主要实验试剂TCBS琼脂培养基、胰蛋白胨大豆肉汤(TSB)购自青岛海博生物技术有限公司;PCR相关试剂(Premix Ex TaqTM)、克隆载体pMD18Tvector (含连接酶、Buffer)、DNA Maker DL2000和胶回收试剂盒购于宝生物工程(大连)有限公司;Wizard®基因组DNA纯化试剂盒购于普洛麦格(北京)生物技术有限公司。
1.1.3 主要实验仪器恒温培养箱,MIR-254,日本Sanyo;恒温摇床,SHKE5000-8CE,美国Thermo;PCR仪,Mastercycler pro,德国Eppendorf;电泳仪,DYY-7C,北京六一仪器厂;凝胶成像仪,G05-7500,美国BIO-RAD;微量移液器,德国,Eppendorf;超微量紫外可见分光光度计,ND5000,北京百泰克。
1.2 细菌培养和DNA提取在无菌操作环境下,将保存于-80 ℃的甘油菌分区划线于TCBS琼脂平板,28 ℃培养16~24 h;然后,挑取典型弧菌单菌落接种于2 mL的TSB肉汤(盐度25)中,28 ℃摇床中200 r/min振荡培养16~18 h;取1mL菌液置于1.5 mL离心管中12 000 r/min离心2 min收集菌体,参照Wizard®基因组DNA纯化试剂盒的操作说明提取基因组DNA,用作PCR模板。
1.3 引物设计与单引物特异性检测 1.3.1 引物的设计与退火温度的优化根据GenBank中1类整合子整合酶基因int1(Accession No.KU130396.1)、4类超级整合子整合酶基因intSXT(Accession No.GQ495075.1)和1型插入序列共同区转座酶基因ISCR1(Accession No.KT725789.1)的序列,利用多重PCR引物设计软件PrimerPlex 2.61(PREMIER Biosoft)设计1套(3对) PCR引物,交由生工生物工程(上海)股份有限公司合成(表 1)。
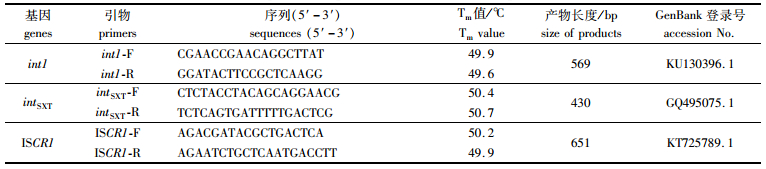
|
表 1 多重PCR扩增所用引物序列 Tab.1 Sequences of primers for multi-PCR amplification |
之后,在各对引物TM值基础之上,分别通过改变PCR反应程序中的退火温度值进行梯度扩增(3次重复试验),以确定各对引物PCR反应的最佳退火温度。PCR反应体系:Premix Ex TaqTM12.5 μL、上下游引物(10 pmol/L)各0.5 μL、DNA模板0.5 μL,超纯水定容至25 μL。梯度PCR扩增程序:95 ℃预变性5 min;95 ℃变性40 s,47~52.5 ℃(即:47.0 ℃、47.5 ℃、48.0 ℃、48.5 ℃、49.0 ℃、49.5 ℃、50.0 ℃、50.5 ℃、51.0 ℃、51.5 ℃、52.0 ℃和52.5 ℃)退火35 s,72 ℃延伸40 s,30个循环;最后72 ℃再延伸10 min,4 ℃保存。
1.3.2 单引物特异性及灵敏度检测以第1.1.1节中的阳性对照菌和阴性对照菌基因组DNA为模板,按照第1.3.1节所述的PCR反应体系及确定的最佳PCR反应程序(退火温度),使用上述合成的3对引物分别进行PCR扩增,PCR产物经TA克隆后测序,并将结果与GenBank上公布的序列进行比对分析,以检测引物的特异性。
将第1.1.1节中的阳性对照菌VA-001基因组DNA (浓度700 ng/μL)依次梯度稀释为:350、160、80、40、20、10、5、2.5、1.2、0.6、0.3、0.15、0.08、0.04、0.02、0.01、0.005、0.002、0.001、0.000 5、0.000 2、0.000 1和0.000 05 ng/μL,用作模板DNA。然后,利用上述PCR反应程序对设计的3对引物分别进行单独PCR扩增,并将PCR产物进行1%的琼脂糖凝胶电泳,以获得所设计各对引物的灵敏度。同时,采用上述方法检测文献中3对单引物(表 2)的灵敏度。
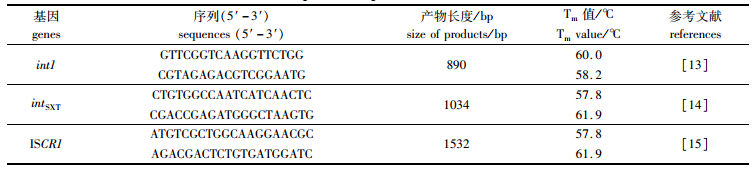
|
表 2 参考文献引物序列 Tab.2 Sequences of primers from references |
以第1.1.1节中的阳性对照菌基因组DNA为模板,按照第1.3.1节中的描述建立25 μL多重PCR体系,其中int1、intSXT和ISCR1的上下游引物(10 pmol/L)体积均为0.5 μL。在第1.3.1节中所获得的各引物最佳退火温度的基础上,选取适合的多重PCR反应退火条件,按照第1.3.1节所述的PCR程序进行目的基因的扩增。扩增产物经1%的琼脂糖凝胶电泳(120 V,40 min)后,利用凝胶成像系统观察结果,依据结果对多重PCR反应体系进行优化。
1.4.2 多重PCR体系引物体积组合的优化结合第1.3.2节中单引物灵敏度检测结果及第1.4.1节中初始多重PCR反应体系的检测结果,将退火温度固定为50 ℃,然后对int1、intSXT和ISCR1 3对目的基因引物(10 pmol/L)的体积组合进行优化。在预设的int1(0.3、0.4和0.5 μL)、intSXT(0.4、0.5和0.6 μL)和ISCR1(0.4、0.5和0.6 μL)体积范围内,进行三因素三水平的全面实验,以确定各引物的最佳体积组合。
1.4.3 退火温度的优化按照第1.4.1节中描述的PCR体系和第1.4.2节中得到的各引物最优体积组合,建立25 μL多重PCR体系,设定一系列退火温度:47.0、47.5、48.0、48.5、49.0、49.5、50.0、50.5、51.0、51.5、52.0和52.5 ℃,以确定多重PCR反应的最佳退火温度。
1.5 多重PCR体系的灵敏度检测以阳性对照菌株溶藻弧菌VA-001的基因组DNA (浓度为700 ng/μL)为模板,按照第1.3.2节中描述的方法对其进行系列的梯度稀释,用作多重PCR扩增的模板,以检测经第1.4节优化后的多重PCR体系的灵敏度(3次重复实验)。
1.6 多重PCR体系的应用利用所建立多重PCR反应体系对临床分离的222株弧菌进行int1、intSXT和ISCR1基因的检测(3次重复试验),以溶藻弧菌VA-001、创伤弧菌VV-311、溶藻弧菌VA-352、溶藻弧菌VA-368、副溶血弧菌VP-046作为阳性对照,副溶血弧菌CICC21617、创伤弧菌CICC10484、溶藻弧菌CICC10484作为阴性对照,以灭菌去离子水作为DNA模板的空白对照。并且,利用文献中报道的int1[13]、intSXT[14]和ISCR1[15]3对单引物对222株分离株进行检测,并将这2种检测方法的结果进行比较分析。
2 结果 2.1 多重PCR引物及最佳退火温度依据GenBank公布的DNA序列,利用PrimerPlex 2.61设计得到3对PCR引物,各引物的具体参数详见表 1。在引物软件给出的TM值基础之上,通过调整退火温度的大小进行梯度PCR扩增的结果表明,退火温度在48.5~51.0 ℃范围时,3对PCR引物均能实现良好的扩增,PCR产物的电泳条带清晰。基于此,本研究取50.0 ℃作为多重PCR反应的初始退火温度。
2.2 单引物特异性及灵敏度检测结果以int1、intSXT和ISCR1基因设计的3对引物能分别特异性地扩增出约550 bp (菌株VA-001、VV-311和VA-352)、450 bp (菌株VA-001、VA-352和VP-046)和650 bp (菌株VA-001、VV-311和VP-046)左右的目的片段。PCR产物测序的结果与Genbank上的序列比较显示,int1的测序结果与Genbank中LN831185.1序列的相似度为99%~100%;intSXT的测序结果与GenBank中DQ180349.1的相似度为97%~100%;ISCR1的测序结果与GenBank中的KT725788.1序列的相似度为100%。此外,以阴性对照溶藻弧菌CICC10484、副溶血弧菌CICC21617和创伤弧菌CICC10383的基因组DNA为模板的PCR扩增没有得到目的条带。
利用阳性对照株VA-001弧菌基因组不同浓度DNA (700~0.000 05 ng/μL)为模板,电泳结果显示:本实验中设计的int1、intSXT和ISCR1基因引物可分别在模板浓度≥0.005 ng/μL、0. 01 ng/μL和0.02 ng/μL的条件下扩增出单一可辨的特异性目的条带,且随着模板浓度的降低,条带亮度也随之依次降低,因此该3对引物的灵敏度分别是0.005 ng/μL、0.01 ng/μL和0.02 ng/μL。文献中int1、intSXT和ISCR1基因引物的灵敏度检测结果分别是0.02 ng/μL、0.02 ng/μL和0.02 ng/μL。
2.3 多重PCR反应体系的建立与优化单引物灵敏度检测结果发现,int1基因引物灵敏度高,且当多重PCR反应体系中3对引物的体积均为0.5 μL (10 pmol/L)时,该引物扩增的目的条带的亮度要明显高于intSXT和ISCR1目的条带的亮度。鉴于此,本研究拟通过减少int1引物体积和/或增加intSXT和ISCR1引物体积的方式来优化该多重PCR体系。故而,将int1(0.3 μL、0.4 μL和0.5 μL)、intSXT(0.4 μL、0.5μL和0.6 μL)和ISCR1(0.4 μL、0.5 μL和0.6 μL)引物体积限定后,进行三因素三水平的全面实验。结果显示,当上述3对基因引物的体积组合为:0.3 μL (int1)、0.6 μL (intSXT)和0.6 μL (ISCR1)时,该反应体系扩增出的3条目的条带清晰,亮度均一,区别度较好。
如表 1所示,利用PrimerPlex 2.61设计6条(3对) PCR单引物的Tm值在49.6~50.7 ℃之间。因此,选择在47.5~52.5 ℃范围内对多重PCR体系的退火温度进行优化。结果显示,在此温度范围内均可特异性地扩增出3个目的基因条带,但扩增效率存在一定差异。当退火温度在47.5~49 ℃时,intSXT基因的电泳条带相对略弱;而退火温度在52 ℃及以上时,int1基因的电泳条带相对较弱。最终发现,多重PCR体系的退火温度为50 ℃时,3对引物扩增效率基本一致,扩增的目的条带清晰,均一性好。
2.4 多重PCR体系的灵敏度分析同时对含有Int1、ISCR1和SXT 3种元件的阳性对照株溶藻弧菌VA-001基因组DNA做梯度稀释(700 ng/μL~0.000 05 ng/μL),以测定多重PCR体系的敏感性。结果如图 1所示,随着多重PCR体系中加入模板DNA浓度的降低,扩增出的条带亮度呈现出逐渐减弱趋势,到0.02 ng/μL以下浓度基本已观察不清条带;0.02 ng/μL及以上浓度条带清晰,扩增效果稳定。
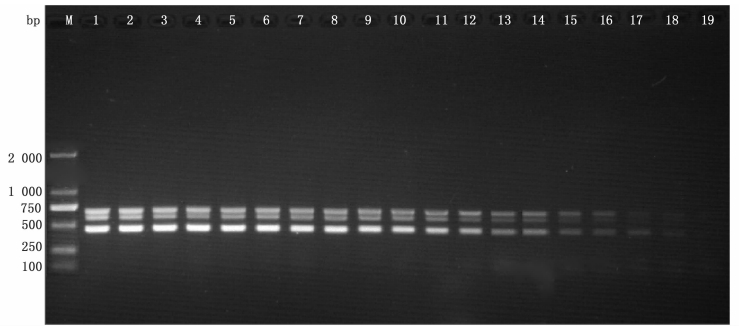
|
图 1 多重PCR反应灵敏度检测
Fig. 1 Sensitivity tests of the multiplex PCR using VA-001 DNA
M. DL2000 DNA Marker; 1-19. VA-001 DNA模板浓度依次为700、350、160、80、40、20、10、5、2.5、1.2、0.6、0.3、0.15、0.08、0.04、0.02、0.01、0.005和0.002 ng/μL M. DL2000 DNA Marker; 1-19. the concentrations of VA-001 DNA temples is 700、350、160、80、40、20、10、5、2.5、1.2、0.6、0.3、0.15、0.08、0.04、0.02、0.01、0.005、0.002 ng/μL, respectively |
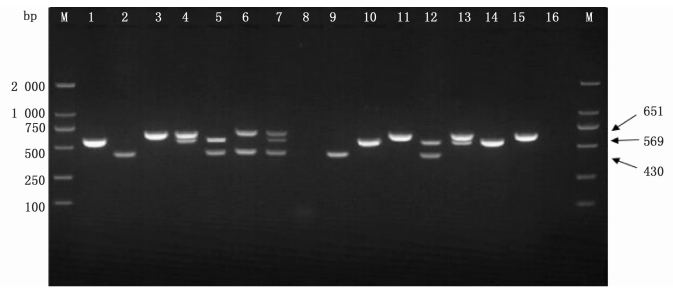
应用所建立的多重PCR体系检测222株临床分离弧菌Int1、SXT和ISCR1的携带情况,3组平行试验检测结果一致(表 3),图 2为部分菌株的多重PCR电泳图。1类整合子Int1阳性弧菌24株,4类超级整合子SXT阳性弧菌37株,1型插入序列共同区ISCR1阳性弧菌25株;其中,同时含有Int1和ISCR1的弧菌有13株,同时含有Int1和SXT的弧菌4株,3种基因同时存在的弧菌仅1株。同时,222株弧菌的多重PCR检测结果与利用文献上公布的int1、intSXT和ISCR1单引物(表 2) PCR扩增得到的结果相一致。
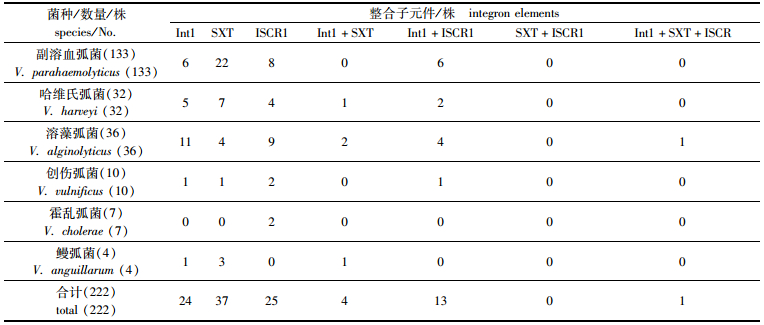
|
表 3 222株临床分离弧菌3种整合子相关元件检出结果 Tab.3 The results of three kinds of integron-related elements test in 222 clinical Vibrio strains |
|
图 2 不同DNA模板的多重PCR扩增电泳图谱
Fig. 2 Electrophoresis profile of multiplex PCR amplication against various DNA templates
M. DL2000 DNA Marker; 1. Int1(569 bp); 2. SXT (430 bp); 3. ISCR1 (651 bp); 4-7.阳性对照(依次为:VV-311, VA-352, VP-046和VA-001); 8.阴性对照(溶藻弧菌CICC10484); 9-15.临床分离弧菌样品(依次为:V-317, V-345, V-307, V-366, V-368, V-022和V-013); 16.空白对照(无菌水) M. DL2000 DNA Marker; 1. Int1(569 bp); 2. SXT (430 bp); 3. ISCR1 (651 bp); 4-7. Positive control (VV-311, VA-352, VP-046 and VA-001, respectively); 8. Negative control (V. alginolyticus CICC10484); 9-15. Clinical Vibrio isolates (V-317, V-345, V-307, V-366, V-368, V-022 and V-013, respectively); 16. Blank control (Sterile water) |
整合子在细菌耐药性尤其是多重耐药性的转移扩散中起到了非常重要的作用,是革兰氏阴性菌耐药趋势逐年升高的主要原因之一[16-17],因此整合子的流行情况可作为评判细菌抗菌药物耐药严重性的指征之一。整合子相关元件也是介导水产养殖源弧菌耐药性传播的一类可移动遗传元件,且以1类整合子、4类超级整合子SXT和1型插入序列共同ISCR1元件为主,而2、3类整合子极少。目前,整合子元件的检测以单对PCR引物扩增为主。为了能够实现养殖源弧菌中三类常见整合子相关元件的同时检测,本研究建立了一种多重PCR检测方法。与单对引物PCR相比,该方法更为省时省力,且能够节约检测成本;与荧光定量PCR方法相比,其具备操作相对简单、成本低和不易出现假阳性等优点。因此,整体上来说,本研究针对3种整合子相关元件建立的多重PCR检测方法,具有操作简便、节省时间和成本等优点,且该方法的检测限能够满足临床检测的要求。
多重PCR技术是通过在同一PCR反应体系中添加多对引物,对多个靶点序列同时进行扩增的一种分子生物学诊断方法,具有操作简单、节约时间和经济高效等优点。引物的特异性是多重PCR方法的关键。本研究根据GenBank中公布的int1、intSXT和ISCR1基因序列,利用多重PCR引物设计软件PrimerPlex 2.61设计了1套(3对) PCR引物。单引物特异性及灵敏度检测实验显示,上述设计的3对引物在DNA模板浓度分别在0.005 ng/μL、0.01 ng/μL和0.02 ng/μL及以上时,均能扩增得到预期大小的目的片段,且阴性和空白对照样品没有条带出现。由此说明,利用这些引物能实现对int1、intSXT和ISCR1基因的特异性扩增检测且敏感性好。基于此,本研究利用这3对引物初步建立了同时检测Int1、SXT和ISCR1元件的多重PCR体系。
影响多重PCR扩增反应的因素很多,包括:Mg2+浓度、Taq酶浓度、dNTP浓度、引物的浓度与退火温度等[18]。考虑到实际情况,多数的PCR扩增实验使用的是Mg2+、Taq酶和4种dNTP的预混液。本研究使用的是Premix Ex TaqTM(TaKaRa, RR902A),故而未对所建立的多重PCR体系中Mg2+浓度、Taq酶浓度和dNTP浓度进行优化。引物的浓度是PCR扩增体系中的关键因素之一,不同的引物在同一PCR反应体系中形成竞争关系;在有限量的Mg2+、Taq酶和dNTP情况下,引物的浓度与其扩增产物电泳条带的亮度成正相关。此外,PCR扩增的效率与引物的碱基序列有关,这也就造成了不同引物扩增效率的差异。本实验结果显示,当多重PCR体系中3对引物体积相等时,int1引物扩增得到的目的电泳条带亮度明显高于其他两对引物得到的目的条带,表明在该体系中int1引物竞争能力强,因此扩增效率相对更高;当int1、intSXT和ISCR1引物体积分别为0.3 μL、0.6 μL和0.6 μL时,多重反应体系中各引物之间的竞争能力相互平衡,故扩增得到的三条目的条带亮度清晰一致。此外,退火温度是影响多重PCR扩增的另外一个重要因素。本研究在优化出PCR反应最优引物体积组合的基础上进一步优化退火温度,当退火温度为50 ℃时扩增得到目的电泳条带清晰,扩增效率最佳。实验中发现,当退火温度低于49 ℃时,intSXT目的条带较弱,有微弱的非特异性条带出现;当退火温度高于52 ℃时,int1的扩增效率降低,条带亮度减弱。灵敏度实验结果显示,本研究建立的多重PCR反应体系最低能在0.02 ng/μL DNA浓度时扩增出目的条带,各单引物的检测灵敏度均略高于多重PCR的检测灵敏度。在实验室检测中,通过水煮法或利用细菌基因组试剂盒提取的基因组DNA模板浓度均可达到100 ng/μL以上。本研究对参考文献中所涉及的3对引物灵敏度进行了测试,并与本文设计的引物的灵敏度进行了比较,结果发现就单一引物之间比较而言,所设计的引物具有更好的灵敏度,可达1~2个数量级的差别;多重PCR反应的灵敏度则与文献中各基因单引物的检测灵敏度相当,对阳性菌株的检出结果完全一致。因此,本研究建立的多重PCR反应体系的灵敏度完全能满足细菌中Int1、SXT和ISCR1元件的检测要求。在实际应用过程中发现,利用所建立的多重PCR体系对222株对虾源分离弧菌3次平行试验检测结果与文献公布的单引物PCR方法检测结果一致,无假阴性或假阳性出现,说明该多重PCR体系重复性好,具有良好的敏感性与特异性。
综上所述,本研究建立了一种可同时检测3种整合子相关元件(Int1、SXT和ISCR1)的多重PCR方法,且扩增得到的目的电泳条带清晰,区分度良好。实际应用结果表明,该方法能够满足在临床分离菌株中实现3种整合子相关元件的快速检测要求。
| [1] | WU K H, WANG F P, SUN J J, et al.Class 1 integron gene cassettes in multidrug-resistant Gram-negative bacteria in southern China[J]. International Journal of Antimicrobial Agents, 2012, 40(3): 264–267. DOI:10.1016/j.ijantimicag.2012.05.017 |
| [2] | HALL R M, COLLIS C M.Mobile gene cassettes and integrons:capture and spread of genes by site-specific recombination[J]. Molecular Microbiology, 1995, 15(4): 593–600. |
| [3] | RECCHIA G D, HALL R M.Gene cassettes:a new class of mobile element[J]. Microbiology, 1996, 141(12): 3015–3027. |
| [4] | TOLEMAN M A, BENNETT P M, WALSH T R.ISCR elements:novel gene-capturing systems of the 21st Century?[J]. Microbiology and Molecular Biology Reviews, 2006, 70(2): 296–316. DOI:10.1128/MMBR.00048-05 |
| [5] | 孙景勇, 倪语星.非O1、非O139型霍乱弧菌中首次发现PER-1超广谱β内酰胺酶[J]. 中国感染与化疗杂志, 2010, 10(5): 338–341. SUN J Y, NI Y X.First identification and genetic analysis of extended-spectrum β-lactamase PER-1 in a strain of non-O1, non-O139 Vibrio cholerae[J]. Chinese Journal of Infection and Chemotherapy, 2010, 10(5): 338–341. |
| [6] | 邓玉婷, 曾振灵, 刘健华, 等.插入序列共同区元件:细菌中新出现的一种基因捕获系统[J]. 微生物学报, 2009, 48(8): 987–993. DENG Y T, ZENG Z L, LIU J H, et al.Insertion sequence common region element:a novel gene-capturing system in bacteria-A review[J]. Acta Microbiologica Sinica, 2009, 48(8): 987–993. |
| [7] | VAISVILA R, MORGAN R D, POSFAI J, et al.Discovery and distribution of super-integrons among pseudomonads[J]. Molecular Microbiology, 2001, 42(3): 587–601. |
| [8] | JAIN M, KUMAR P, GOEL A K, et al.Class 1 integrons and SXT elements conferring multidrug resistance in Vibrio cholerae O1 strains associated with a recent large cholera outbreak in Orissa, Eastern India[J]. International Journal of Antimicrobial Agents, 2008, 32(5): 459–460. DOI:10.1016/j.ijantimicag.2008.05.003 |
| [9] | RAJPARA N, KUTAR B M, SINHA R, et al.Role of integrons, plasmids and SXT elements in multidrug resistance of Vibrio cholerae and Providencia vermicola obtained from a clinical isolate of diarrhea[J]. Frontiers in Microbiology, 2015, 6(57): 1–11. |
| [10] | KUMAR P, THOMAS S.Presence of qnrVC3 gene cassette in SXT and class 1 integrons of Vibrio cholerae[J]. International Journal of Antimicrobial Agents, 2011, 37(3): 280–281. DOI:10.1016/j.ijantimicag.2010.12.006 |
| [11] | IWANAGA M, TOMA C, MIYAZATO T, et al.Antibiotic resistance conferred by a class I integron and SXT constin in Vibrio cholerae O1 strains isolated in Laos[J]. Antimicrobial Agents and Chemotherapy, 2004, 48(7): 2364–2369. DOI:10.1128/AAC.48.7.2364-2369.2004 |
| [12] | 姚小娟.海水养殖源弧菌耐药性检测与整合子分析[D].上海:上海海洋大学, 2014. YAO X J. Drug resistance detection and integrons analysis of Vibrios from mariculture source[D]. Shanghai:Shanghai Ocean University, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10264-1014051802.htm |
| [13] | XU H, DAVIES J, MIAO V.Molecular characterization of class 3 integrons from Delftia spp[J]. Journal of Bacteriology, 2007, 189(17): 389–394. |
| [14] | RODRíGUEZ-BLANCO A, LEMOS M L, OSORIO C R.Integrating conjugative elements as vectors of antibiotic, mercury, and quaternary ammonium compound resistance in marine aquaculture environments[J]. Antimicrobial Agents and Chemotherapy, 2012, 56(5): 2619–2626. DOI:10.1128/AAC.05997-11 |
| [15] | 陈霞, 袁敏, 李桂喜, 等.基因捕获元件int1和ISCR1在临床菌株中的分布及与细菌耐药性的关系研究[J]. 中国人兽共患病学报, 2013, 29(7): 646–652. CHEN X, YUAN M, LI G X, et al.Dissemination of int1 gene and ISCR1 and their relations with multi-drug resistance in clinical isolates[J]. Chinese Journal of Zoonoses, 2013, 29(7): 646–652. |
| [16] | ROWE-MAGNUS D A, GUEROUT A M, MAZEL D.Bacterial resistance evolution by recruitment of super-integron gene cassettes[J]. Molecular Microbiology, 2002, 43(6): 1657–1669. DOI:10.1046/j.1365-2958.2002.02861.x |
| [17] | 冯洁, 石磊, 肖增璜, 等.细菌耐药整合子intl分类的多重PCR方法的建立及应用[J]. 中国抗生素杂志, 2005, 30(10): 599–603, 630. FENG J, SHI L, XIAO Z H, et al.Construction and application of multi-PCR assay to screen antibiotic resistance integrons in bacteria[J]. Chinese Journal of Antibiotics, 2005, 30(10): 599–603, 630. |
| [18] | 潘孝明.基于LM-PCR预扩增法的建立及其在水产品致病菌检测中的应用[D].青岛:中国海洋大学, 2014. PAN X M. Development of a pre-amplificatin method based on LM-PCR and its applications on detection of pathogens in seafood[D]. Qingdao:Ocean University of China, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10423-1014329423.htm |
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
 2016,
Vol. 25
2016,
Vol. 25


