2. 上海海洋大学 水产与生命学院, 上海 201306
缢蛏 (Sinonovacula constricta Lamarck) 俗称蛏子、泥蛏,属软体动物门(Mollusca),双壳纲(Bivalve),竹蛏科(Solenidae),缢蛏属(Sinonovacula),为广温、广盐性埋栖型海产双壳类,广泛分布于西太平洋沿海海域,在食用贝类中占有相当重要的地位[1-2]。由于缢蛏具有较为重要的经济价值,对它的研究和开发利用受到越来越多科研工作者的重视。目前,国内外已利用形态学指标[3]、同工酶 [4-5]、线粒体序列[6-10]、RAPD[11-13]、微卫星标记[10, 14-15]、ISSR[16]等标记对中国沿海不同缢蛏群体的遗传多样性进行了研究。本研究将从形态学结合SRAP(Sequence-based amplified polymorphism,SRAP)标记,较为全面分析乐清湾缢蛏的资源特色,旨在为浙江乐清湾缢蛏资源的管护和开发利用提供依据。
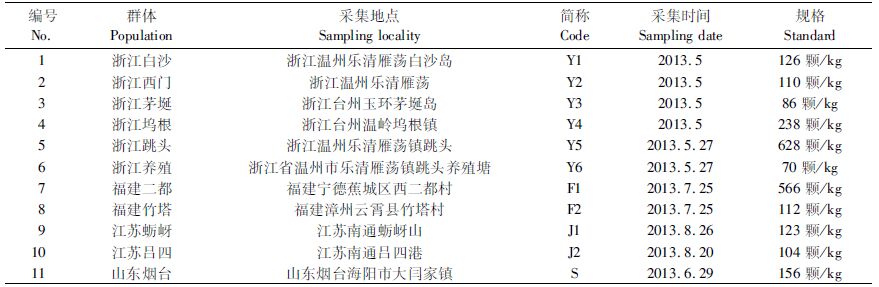
1 材料与方法 1.1 材料采集2013年5月至8月间采集了浙江、福建、江苏及山东等中国沿海4省11个区域缢蛏样本,其中浙江省6个,全部来自乐清湾,除6号(Y6)为养殖群体外,其余均为野生群体。7-11号来自中国沿海其他省份,全部为野生群体,作为对照组,具体采集信息见表 1。
|
表 1 缢蛏采样信息 Tab.1 Sampling information of S.constricta |
形态学性状的测量参考刘达博等[3],采用游标卡尺,精确到0.1 mm,每个个体分别测量壳长(CG)、壳宽(AE)、壳厚(IK)及AB、AC、AD、AF、AG、AH、BC、BD、BE、BF、BG、BH、CD、CE、CF、CH、DE、DF、DG、DH、EF、EG、EH、FG、FH、GH等共29个可量形态学和框架数据(图 1)。11个群体,每个群体测量50个个体,共获得15 950个形态学和框架数据。

|
图 1 缢蛏形态学分析测量位点
Fig. 1 Landmark points for morphological measurement of S. constricta
A.壳顶点; B.背前端; C.体前端; D.腹前端; E.腹缘底端; F.腹后端; G.体后端; H.背后端; I.左壳外侧顶; K.右壳外侧顶 A. Beak (Umbo) ; B. Front-end of dorsal; C. Anterior of body; D. Front-end of dorsal; E. Bottom of ventral; F. Back-end of ventral; G. Posterior of body; H. Back-end of dorsal; I. Left outboard of umbo; K. Right out board of umbo |
SRAP 是基于PCR的随机扩增标记技术,具有多态性高、共显性、操作简便等特点,目前已成为遗传多样性、目的基因标定、遗传图谱构建等众多研究领域的重要技术手段之一[17-19]。
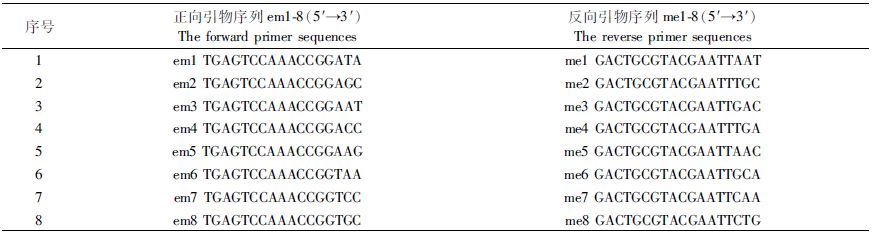
本研究取SRAP正、反向引物各8条(em1~em8、me1~me8),正、反引物两两组合,共构成64对引物。先以每个群体2个个体进行引物筛选,PCR反应体系为20 μL (10 μL 2×PCR Mix、6.5 μL H2O、1.5 μL DNA、正反向引物各1 μL);反应程序为94 ℃预变性5 min; 94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸1 min,5个循环;94 ℃ 变性1 min,50 ℃退火1 min,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;1.5%琼脂糖电泳预检,具有条带的用8%非变性聚丙烯胶电泳检测。选取6对PCR扩增条带清晰、条带多的引物组合分别对11个群体,每个群体20个个体,共220个样本进行分析。SRAP的聚丙烯胶电泳条带按照软件包TFPGA[20]格式记分,在同一基因座位上,1表示有条带,0表示没有条带。
|
表 2 SRAP引物序列 Tab.2 The primer sequences of SRAP |
采用SPSS 19.0软件对形态学数据进行了聚类分析、主成分分析和判别分析;SRAP数据以Popgene32进行分析[20],根据NEI[21]的无偏差杂合率计算公式估算各个基因座位的平均遗传杂合度。
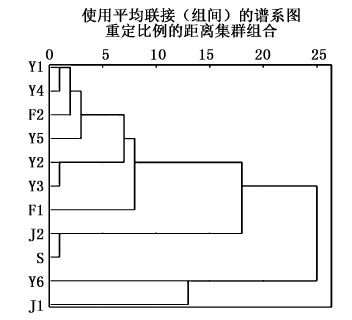
2 结果 2.1 形态学分析 2.1.1 聚类分析为消除缢蛏规格大小对参数值的影响,先将每个个体所有参数分别除以壳长(CG)予以校正,得到29个比例性状,再求出各样本每个参数校正值的平均值,用校正平均值进行聚类分析。聚类分析采用层次聚类分析中的Q型聚类,聚类结果见图 2。
|
图 2 11个缢蛏群体的聚类图
Fig. 2 Cluster dendrogram of eleven populations of S. constricta
|
聚类结果显示我国沿海缢蛏11个群体共聚为2大支:浙江乐清湾养殖群体(Y6)和江苏南通群体(J1)聚为一支,其余9个群体聚为另一大支;在9个群体这一支上,浙江乐清湾(5个)和福建2个、共7个野生群体聚为其中的一支,这表明浙江省乐清湾缢蛏野生群体在形态上具有很高的相似性,且与福建野生群体的相似性也比较高。
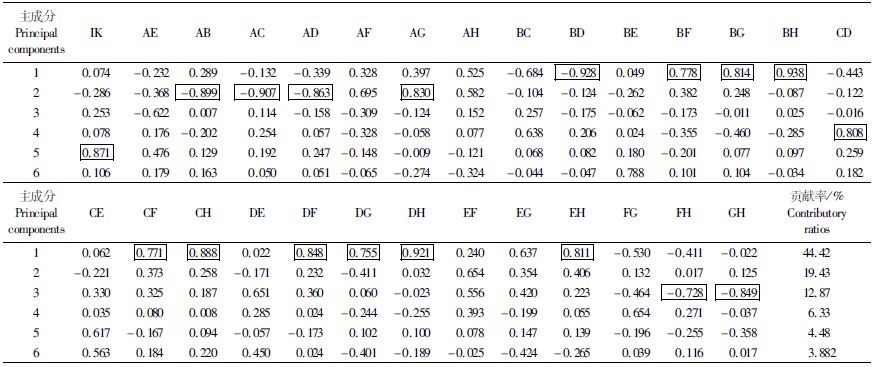
2.1.2 主成分分析主成分分析为将多个指标作为小数指标的统计方法,所有数据都是校正比例值,从29个比例性状中通过分析得出前6个互不关联的主成分。前5个主成分分析结果见表 3。
|
表 3 11个缢蛏群体29个性状前6个主成分的贡献率及负荷值 Tab.3 Contributory ratios and eigenvector loading of six principal components for twenty-nine characters of eleven populations of S. constricta |
从表 3中可以得出,总方差的贡献率分别为: 主成分1为44.42%,主成分2为19.43%,前5的主成分累积贡献率为87.53%。主成分负荷绝对值超过0.9的分别为壳顶点到体前端(BD)、背前端到腹前端(BH)、背前端到背后端(AC)3项负荷值比较大,是重要的特征参数。
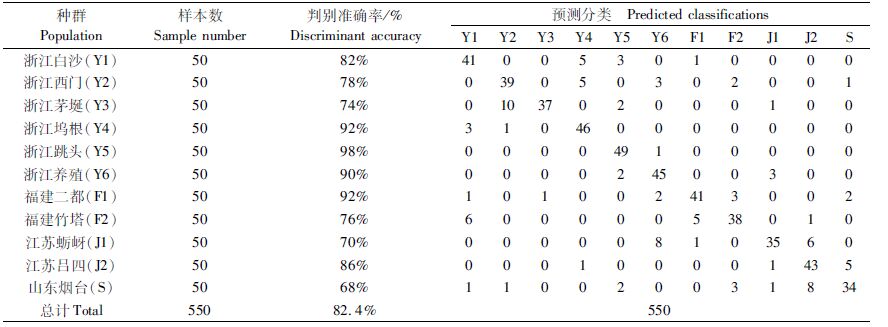
2.1.3 判别分析对11个群体29个校正比例性状进行判别分析(表 4)。其F检验结果表明,判别效果较好,说明不同群体之间形态差异显著。其中浙江跳头(Y5)的判别准确率最高,达98%;山东烟台(S)群体判别准确率最低,仅68%,有16%的个体被判入到江苏吕四(J2)群体中;浙江乐清湾野生群体之间易相互出现错判,11个群体平均判别准确率为82.4%。
|
表 4 11个缢蛏群体形态学判别分析 Tab.4 The discriminant analysis of morphology of eleven populations of S. constricta |
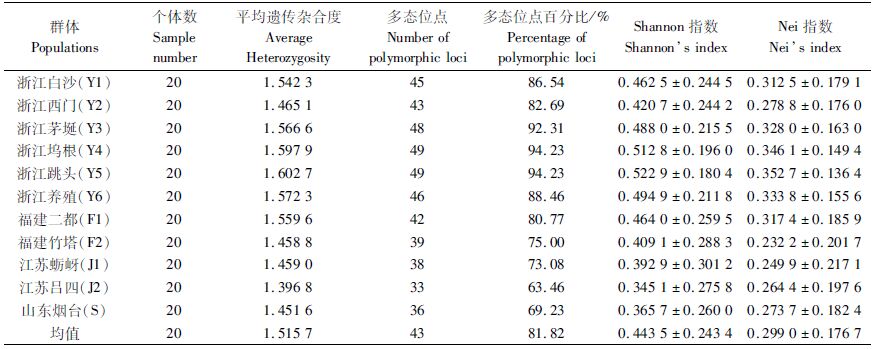
根据48对引物组合在11个群体、每个群体2个样本的预分析结果,选取其中em2/me6、em3/me5、em3/me6、em3/me2、em1/me4、em4/me3共6对扩增条带多、条带清晰的引物组合在11个群体、共220个个体中进行扩增,共产生52条条带,平均每对引物产生8.67条条带,各群体的遗传多样性分析见表 5。
|
表 5 基于SRAP的11个缢蛏群体的遗传多样性 Tab.5 Result of genetic diversity between 11 populations based on SRAP |
浙江乐清雁湾野生群体Shannon多样性指数在0.420 7~0.522 9之间,均值为0.481 4;浙江养殖(Y6)群体的遗传多样性处在11个采样群体的中间状态。浙江以外的区域,Shannon多样性指数在0.464 0~0.345 1之间,均值为0.402 8。浙江乐清湾缢蛏野生群体的Shannon多样性指数比浙江以外区域的群体高16.3%。
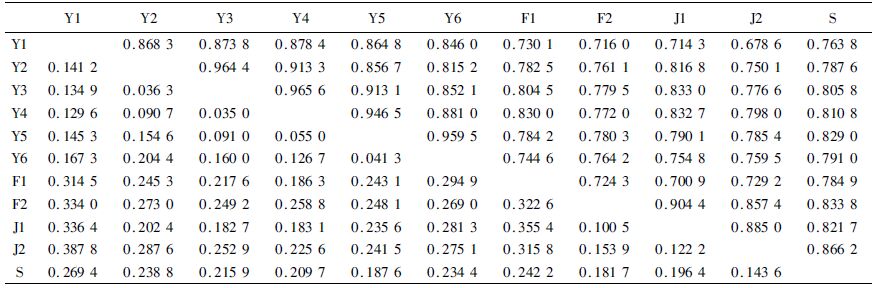
进一步分析各群体间的遗传分化程度(表 6)。11个群体之间的基因流Nm=1.258 7,遗传分化系数Gst=0.284 3。遗传距离在0.035 0~0.388 7之间;乐清湾6个群体之间的遗传距离较小,在0.035 0~0.204 4之间,除雁荡西门岛(Y2)与雁荡跳头养殖塘(Y6)外,其余均小于0.2。而乐清湾群体与其他沿海群体之间的遗传距离相对较大,在0.186 3~0.387 8之间,只有3组群体之间遗传距离小于0.2。福建、江苏和山东群体之间的遗传距离在0.100 5~0.355 4之间。
|
表 6 11个缢蛏群体之间遗传距离(对角线以下)及遗传相似度(对角线以上) Tab.6 Genetic distances (below diagonal) and genetic similarity (above diagonal) between 11 populations |
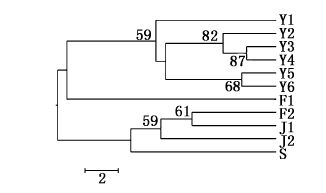
以遗传距离构建11个群体的NJ系统关系树(图 3)发现,浙江乐清湾群体与福建宁德蕉城区西二都村(F1)共7个聚成一支,而浙江群体聚为更为紧密的一小支;福建漳州云霄县竹塔村(F2)与江苏南通蛎岈山(J1)、江苏南通吕四港(J2)及山东烟台海阳市大闫家镇(S)聚为一支。
|
图 3 基于SRAP缢蛏11个群体间的NJ分子系统树
Fig. 3 NJ tree among 11 populations of S. constricta based on SARP
|
通过形态学可量及框架数据分析的结果显示11个群体的缢蛏聚为两支,它们之间的聚类关系与地理位置有一定关系,浙江与福建野生群体之间具有更高的形态相似性,江苏与山东烟台野生群体之间形态相似度也更高些,这与刘达博等[3]的研究结果基本一致。但浙江野生群体和福建群体之间的聚类关系与地理距离不存在相关性,这可能是由于养殖环境的相似性导致的,这种情况在其他水生生物中也同样存在[22-24]。
SRAP对中国沿海缢蛏11个群体分析结果存在较高的一致性:(1)浙江乐清湾野生和养殖共6个群体遗传多样性比较高,遗传分化指数大,但是它们却是在同一聚类分支上,显示了它们同祖同宗、相似的遗传背景,以及人为干扰少、遗传多样性高、优良的遗传资源,这与刘达博等[25]、王冬群等[4]、刘博等[15]的研究结果是相似的;(2)江苏、福建、山东群体尽管是在地理位置上相隔较远的野生群体,但遗传多样性却相对较低、遗传分化不显著,这暗示江苏、福建、山东的缢蛏野生群体遗传资源可能受人为干扰大,抑或有相似的遗传背景。
尽管贝类自主迁移能力较差,但在生活史的早期,随着洋流的漂移,还是导致相隔较远的群体间存在基因流,只是基因交流会存在时间上和交流程度上的局限[4]。利用SRAP标记,从全基因组进行扫描发现了乐清湾群体的一些特异性条带。通过扩大群体进行分析以后发现乐清湾群体可以与其他地点的群体能够一定程度上与沿海群体区分开来,证明SRAP技术可以对乐清湾基因组中的特异性进行筛选。群体遗传多样性可以用于评估物种的种质资源。结果表明乐清湾缢蛏野生群体的遗传多样性仍处于较高水平,可以推断目前湾内的种质情况基本良好,但是乐清湾存在异地引苗的现象,因此要加强缢蛏种质资源保护和管理,在苗种引进后,要防止与当地群体混杂[25]。而对于养殖塘群体,长期的养殖过程,异地苗种的引进和繁殖,对当地缢蛏群体的遗传结构可能造成影响,从而打破了原有的遗传结构,虽然遗传相似度等依然较高,但是逐渐形成了适应当地的新种群[10]。
| [1] | 薛兴华, 岑大华.缢蛏人工养殖技术[J]. 黑龙江水产, 2007: 17–18. XUE X H, CEN D H.The artificial cultivation technology of Sinonovacula constricta[J]. Heilongjiang Aquaculture, 2007: 17–18. |
| [2] | 朱亚珠, 翁佩芳.蛏子软罐头加工工艺的研究[J]. 浙江海洋学院学报(自然科学版), 2002, 21(2): 119–122. ZHU Y Z, WENG P F.Studies on processing technology of soft-can Razor Clam[J]. Journal of Zhejiang Ocean University (Natural Science), 2002, 21(2): 119–122. |
| [3] | 刘达博, 牛东红, 姜志勇, 等.缢蛏7群体的形态差异与判别分析[J]. 海洋渔业, 2009, 31(4): 363–368. LIU D B, NIU D H, JIANG Z Y, et al.Morphological variations and discriminant analysis of seven populations of Sinonovacula constricta[J]. Marine Fisheries, 2009, 31(4): 363–368. |
| [4] | 王冬群, 李太武, 苏秀榕.三种蛏不同组织同工酶的比较分析[J]. 台湾海峡, 2004, 23(2): 131–137. WANG D Q, LI T W, SU X R.Comparison and analysis of isozymes in defferent tissues of three species in Solenidae[J]. Journal of Oceanography in Taiwan Strait, 2004, 23(2): 131–137. |
| [5] | 王冬群, 李太武, 苏秀榕.象山缢蛏养殖群体和野生群体遗传多样性的比较[J]. 中国水产科学, 2005, 12(2): 138–143. WANG D Q, LI T W, SU X R.Comparison of genetic diversity between hatchery stock and wild population of Sinonovacula constricta Lamarck in Xiangshan Bay[J]. Journal of Fishery Sciences of China, 2005, 12(2): 138–143. |
| [6] | 姜志勇, 牛东红, 陈慧, 等.福建缢蛏野生群体与养殖群体的ITS-1和ITS-2分析[J]. 海洋渔业, 2007, 29(4): 314–318. JIANG Z Y, NIU D H, CHEN H, et al.The genetic analysis of ITS-1 and ITS-2 between wild and cultured populations of Sinonovacula constricta in Fujian[J]. Marine Fisheries, 2007, 29(4): 314–318. |
| [7] | 牛东红, 李家乐, 汪桂玲, 等.缢蛏六群体16s rRNA基因片段序列的差异分析[J]. 上海水产大学学报, 2007, 16(1): 1–6. NIU D H, LI J L, WANG G L, et al.The genetic diversity of mitochondrial 16S rRNA gene fragment in six populations of Sinonovacula constricta[J]. Journal of Shanghai Fisheries University, 2007, 16(1): 1–6. |
| [8] | 牛东红, 李家乐, 沈和定, 等.缢蛏六群体线粒体DNA-COI基因序列变异及群体遗传结构分析[J]. 海洋学报, 2008, 30(3): 109–116. NIU D H, LI J L, SHEN H D, et al.Sequence variability of mitochondrial DNA-COI gene fragment and population genetic structure of six Sinonovacula constricta populations[J]. Acta Oceanologica Sinica, 2008, 30(3): 109–116. |
| [9] | 牛东红, 陈慧, 王树亮, 等.我国沿海缢蛏群体遗传结构的mtDNA-COI分析[J]. 动物学杂志, 2010, 45(2): 11–18. NIU D H, CHEN H, WANG S L, et al.Population genetic structure of Sinonovacula constricta along the coast of China[J]. Chinese Journal of Zoology, 2010, 45(2): 11–18. |
| [10] | 牛东红, 冯冰冰, 刘达博, 等.浙闽沿海缢蛏群体遗传结构的微卫星和线粒体COI序列分析[J]. 水产学报, 2011, 35(12): 1805–1813. NIU D H, FENG B B, LIU D B, et al.Microsatellites and mitochondrial COI analysis of population genetic structure of Sinonovacula constricta along the coast of Zhejiang and Fujian Provinces[J]. Journal of Fisheries of China, 2011, 35(12): 1805–1813. |
| [11] | 李成华, 李太武, 苏秀榕, 等.宁波长街缢蛏遗传结构的RAPD分析[J]. 海洋科学, 2004, 28(10): 48–51. LI C H, LI T W, SU X R, et al.Genetic structure analysis of the natural and the cultural stocks of Sinonovacula constricta in Changjie, Ningbo[J]. Marine Sciences, 2004, 28(10): 48–51. |
| [12] | 李成华, 李太武, 宋林生, 等.4个缢蛏群体遗传结构的RAPD分析[J]. 水产科学, 2004, 23(12): 26–28. LI C H, LI T W, SONG L S, et al.Genetic variations among four populations of Sinonovacula constricta by using random amplified polymorphic DNA[J]. Fisheries Science, 2004, 23(12): 26–28. |
| [13] | 于颖, 孟祥盈, 王秀利, 等.缢蛏遗传多样性的RAPD分析[J]. 生物技术通报, 2007: 138–140. YU Y, MENG X Y, WANG X L, et al.Genetic diversity analysis of razor clam (Sinonovaeula constricta) by RAPD[J]. Biotechnology Bulletin, 2007: 138–140. |
| [14] | 刘博, 邵艳卿, 滕爽爽, 等.乐清湾养殖缢蛏群体遗传结构的微卫星标记[J]. 中国农学通报, 2012, 28(2): 69–73. LIU B, SHAO Y Q, TENG S S, et al.Genetic variation of cultured population structure in Sinonovacula constricta using microsatellites[J]. Chinese Agricultural Science Bulletin, 2012, 28(2): 69–73. |
| [15] | 刘博, 邵艳卿, 王侃, 等.4个缢蛏群体遗传多样性和系统发生关系的微卫星分析[J]. 海洋科学, 2013, 37(8): 96–102. LIU B, SHAO Y Q, WANG K, et al.Microsatellite analysis of genetic diversity and phylogenetic relationship of four different geographical populations of Sinonovacula constricta[J]. Marine Sciences, 2013, 37(8): 96–102. |
| [16] | 牛东红, 李家乐, 冯冰冰, 等.缢蛏6个群体遗传结构的ISSR分析[J]. 应用与环境生物学报, 2009, 15(3): 332–336. NIU D H, LI J L, FENG B B, et al.ISSR Analysis on genetic structure of six Sinonovacula constricta populations[J]. Chinese Journal of Applied & Environmental Biology, 2009, 15(3): 332–336. |
| [17] | LI G, QUIROS C F.Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2/3): 455–461. |
| [18] | 徐操, 赵宝华.SRAP分子标记的研究进展及其应用[J]. 生命科学仪器, 2009, 7(4): 24–27. XU C, ZHAO B H.The development and application of SRAP molecular markers[J]. Life Science Instruments, 2009, 7(4): 24–27. |
| [19] | 周剑虹, 郑金娟, 杨受保, 等.SRAP分子标记及其在水生动物中的应用[J]. 湖北农业科学, 2013, 52(17): 4038–4040. ZHOU J H, ZHENG J J, YANG S B, et al.Application of SRAP molecular marker in aquatic animal research[J]. Hubei Agricultural Sciences, 2013, 52(17): 4038–4040. |
| [20] | MILLER M P. Tools for population genetic analysis (TFPGA), version 1.3. A windows program for the analysis of allozyme and molecular population Genetic Data[M]. Flagstaff, AZ: Department of Biological Sciences, Northern Arizona University, 1997. |
| [21] | NEI M.Estimation of average heterozygosity and genetic distance from a small number of individuals[J]. Genetics, 1978, 89(3): 583–590. |
| [22] | 曹侦, 冯广朋, 庄平, 等.长江中华绒螯蟹洄游群体与养殖群体形态差异分析[J]. 淡水渔业, 2013, 43(6): 3–7. CAO Z, FENG G P, ZHUANG P, et al.Morphological difference analysis between migratory population and cultured population of Eriocheir sinensis in the Yangtze river[J]. Freshwater Fisheries, 2013, 43(6): 3–7. |
| [23] | 许凡, 王婷婷, 陈太丰, 等.日本沼虾野生群体与养殖群体杂交、回交后代的形态特征和生产性能[J]. 水产科学, 2011, 30(4): 215–220. XU F, WANG T T, CHEN T F, et al.The morphological characters and growth performance of hybrid and backcross progenies from crosses between wild and cultured prawn Macrobrachium nipponensis[J]. Fisheries Science, 2011, 30(4): 215–220. |
| [24] | 刘汉生, 易祖盛, 梁健宏, 等.唐鱼野生种群和养殖群体的形态差异分析[J]. 暨南大学学报(自然科学版), 2008, 29(3): 295–299. LIU H S, YI Z S, LIANG J H, et al.Morphological variations between the wild population and hatchery stock of Tanichtys albonubes[J]. Journal of Jinan University (Natural Science), 2008, 29(3): 295–299. |
| [25] | 刘达博, 牛东红, 冯冰冰, 等.乐清湾和三沙湾缢蛏群体遗传多样性的微卫星分析[J]. 上海海洋大学学报, 2011, 20(3): 350–357. LIU D B, NIU D H, FENG B B, et al.Microsatellite analysis on genetic diversity of Sinonovacula constricta stocks in Yueqing Bay and Sansha Bay[J]. Journal of Shanghai Ocean University, 2011, 20(3): 350–357. |
2. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306, China
 2017,
Vol. 26
2017,
Vol. 26

