2. 红河学院 生命科学与技术学院, 云南 蒙自 661199
巨魾(Bagarius yarrelli)在中国分布于云南省境内的澜沧江、怒江、元江水系[1],其肉呈黄色,故地方名叫“黄鱼”,隶属于硬骨鱼纲(Osterichthyes)、鲶形目(Siluriformes)、鮡科(Sisordae)、魾属(Bagarius)。巨魾是云南特有的大型底栖食肉鱼类,其体质量可超过50 kg[2]。目前对巨魾的研究主要集中在巨魾的外部形态特征[3]、人工繁殖[2]和人工驯养[4]等方面,2012年糯扎渡水电站有效地解决了池塘驯养和人工繁殖技术问题,但是鱼苗成活时间较短,通过对研究方法不断改进,到目前为止人工繁殖技术已经得到提升,并成功记录了巨魾的胚胎发育过程[5]。
线粒体DNA是目前分子生物学研究的一个重要领域,而鱼类mtDNA是鱼类分子系统学研究和群体遗传分析的重要标记[6]。近年来越来越多的研究者将线粒体基因作为分子标记应用到鱼类的分类及系统发育[7-8]、物种鉴别[9]和系统进化关系[10]等研究领域中。动物的细胞色素C氧化酶由13个亚基组成,其中3大亚基(COⅠ、COⅡ、COⅢ)由线粒体基因(mitochondrial DNA,mtDNA)编码合成,具有酶催化活性[11],是线粒体内膜上呼吸链的重要组成部分,具有较高的保守性,是研究远缘物种系统分类和演化的有效基因[12]。目前,学者利用COⅠ基因、COⅡ基因、COⅢ基因对鱼类的遗传变异与系统发育进行了研究。如:齐兴柱等[13]利用COⅡ基因分析了5种中国南海裸胸鳝属鱼类的系统进化关系;李京等[14]通过部分COⅡ基因分析了28个沟胫天牛亚种的系统发育状况;董徐辉等[15]运用细胞色素b和细胞色素氧化酶Ⅱ基因分析了日本鬼鲉和28种鲉形目线粒体系统发育关系;张望等[16]通过细胞色素氧化酶Ⅰ对鳅科鱼类89尾个体的序列特征进行了分析;崔文涛等[17]通过细胞色素氧化酶Ⅱ基因序列对中国近海12种蛸亚科动物的分子系统进化关系进行了分析;乔德亮等[18]通过细胞色素氧化酶Ⅱ对细鳞斜颌鲴3个群体线粒体的遗传与变异进行了研究。由于人类活动对河干的干扰,以及对野生巨魾的滥捕滥杀,使得种群数量和产卵场破坏较为严重,巨魾数量急剧减少。为了保护巨魾的种质资源,并对巨魾的遗传变异进行研究,本实验通过设计特异性引物对巨魾线粒体COⅠ、COⅡ和COⅢ的基因克隆,进行序列多态性分析并研究巨魾的进化关系,为下一步的研究提供分子依据。
1 材料与方法 1.1 实验材料本研究的12条巨魾采自云南省河口县,剪取肌肉组织放于1.5 mL EP管中贴上对应标签,加入无水乙醇,置于4 ℃保存备用。
1.2 实验方法高纯度的巨魾基因组DNA的提取采用杜民等[19]的酚-氯仿-异戊醇方法。在100 mL 1%的琼脂糖凝胶中加入6 μL溴化乙锭(EB)来检测提取的DNA样品纯度。利用设计的3对特异性引物(表 1)对提取的DNA进行PCR扩增,1%的琼脂糖凝胶检测扩增片段,用凝胶成像系统观察并进行拍照保存。筛选出目的条带利用1.5%的琼脂糖凝胶电泳,在暗箱式紫外分光仪下切取目的片段,用琼脂糖凝胶DNA回收试剂盒(天根生化科技北京有限公司)回收DNA,于1%琼脂糖凝胶电泳检验后与PMD18-T载体(北京博迈德生物技术有限公司)进行连接,利用TOP10感受态细胞进行转化并克隆,挑选阳性克隆利用M13通用引物进行菌液PCR,扩增产物在1%的琼脂糖凝胶上进行电泳,使用凝胶成像系统进行检测并拍照保存,筛选出具有阳性克隆的菌株,送南京金斯瑞生物科技有限公司测序。

|
表 1 引物名称及序列 Tab.1 Primer name and sequence |
利用DNAMAN 5.0对克隆得到的序列进行手动剪切并与GenBank中巨魾序列进行配对剪切得到目的基因的序列全长,对序列进行同源性分析;利用MEGA 5.0软件进行碱基组成分析、遗传距离分析并利用最小进化法(minimum evolution,ME)构建系统发育树。
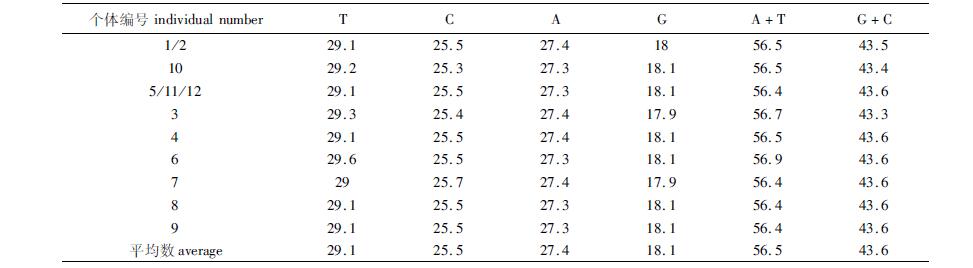
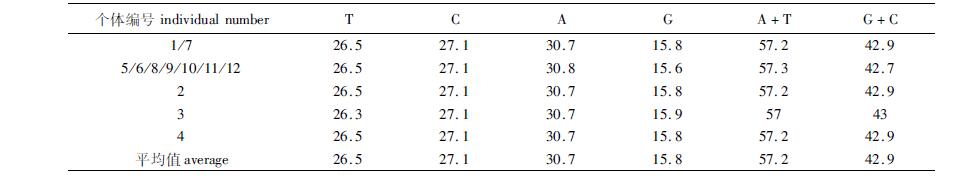
2 结果与分析 2.1 序列分析采用DNAMAN 5.0和MEGA 5.0软件对获得的巨魾序列进行拼接并分析,表明巨魾COⅠ基因序列全长1 551 bp,起始密码子为ATG,终止密码子为TAA,12个个体共出现了9个单倍型(1号和2号为一个单倍型,5号、10号和12号为一个单倍型),13个突变位点;COⅡ基因序列全长691 bp,12个个体共出现了5个单倍型(5号、6号、8号、9号和10号为一个单倍型),5个突变点,起始密码子为ATG,终止密码子为TGA;COⅢ基因序列全长784 bp,12个个体出现了7个单倍型(1号、5号、6号、8号、9号和10号为一个单倍型),8个突变位点,起始密码子为ATG,终止密码子为TGA。COⅠ碱基组成T含量最高(29.1%),G含量最低(18.1%),A+T(56.5%)明显高于G+C(43.5%);COⅡ碱基组成A含量最高(30.7%),G含量最低(15.8%),A+T(57.2%)明显高于G+C(42.9%);COⅢ碱基组成T含量最高(27.6%),G含量最低(17.4%),A+T(55.0%)明显高于G+C(45.0%)。具体碱基含量见表 2~4。
|
表 2 巨魾线粒体COⅠ碱基组成 Tab.2 The base composition of B. yarrelli mitochondrial COⅠ gene |
|
表 3 巨魾线粒体COⅡ碱基组成 Tab.3 The base composition of B. yarrelli mitochondrial COⅡgene |
|
表 4 巨魾线粒体COⅢ碱基组成 Tab.4 The base composition of B. yarrelli mitochondrial COⅢ gene |
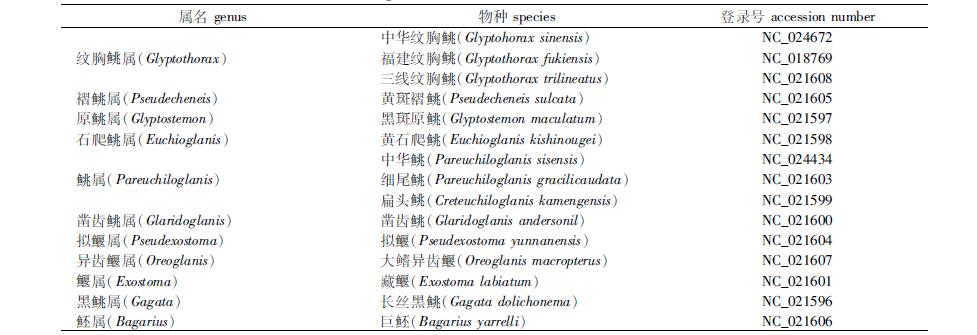
对12个个体的遗传距离进行分析表明:COⅠ的10号个体与3号、4号和8号个体(0.003 9),COⅡ的2号个体与1号、2号和5号个体(0.004 4),COⅢ的2号个体与3号个体(0.003 9)遗传距离最大。利用MAGE 5.0软件中的最小进化法(minimum evolution,ME)将巨魾与中华纹胸鮡(Glyptohorax sinensis)、中华鮡(Pareuchiloglanis sisensis)、福建纹胸鮡(Glyptothorax fukiensis)、三线纹胸鮡(Glyptothorax trilineatus)、大鳍异齿鰋(Oreoglanis macropterus)、黄斑褶鮡(Pseudecheneis sulcata)、拟鰋(Pseudexostoma yunnanensis)、细尾鮡(Pareuchiloglanis gracilicaudata)、藏鰋(Exostoma labiatum)、凿齿鮡(Glaridoglanis andersonil)、扁头鮡(Creteuchiloglanis kamengensis)、黄石爬鮡(Euchioglanis kishinougei)、黑斑原鮡(Glyptostemon maculatum)、长丝黑鮡(Gagata dolichonema)等14种鮡科鱼碱基序列构建系统发育树,结果显示COⅠ、COⅡ和COⅢ都与中华纹胸鮡、福建纹胸鮡、三线纹胸鮡和长丝黑鮡在同一个分支,表明巨魾与纹胸鮡属和黑鮡属的亲缘关系较近。14种鮡科鱼类基因片段的登录号见表 5,具体系统发育树见图 1~3。
|
表 5 15种鮡科种类及登录号 Tab.5 15 species and accession numbers of Sisoridae |
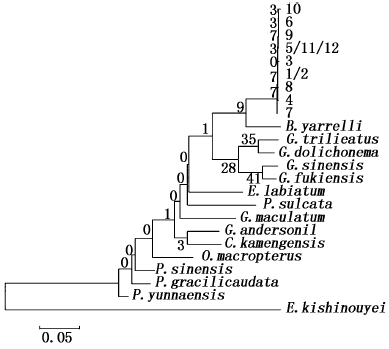
|
图 1 利用ME法构建15种鮡科鱼线粒体COⅠ基因分子系统树
Fig. 1 Molecular phylogenetic tree constructed by ME method of mitochondrial COⅠ gene from 15 species of Sisoridae
|
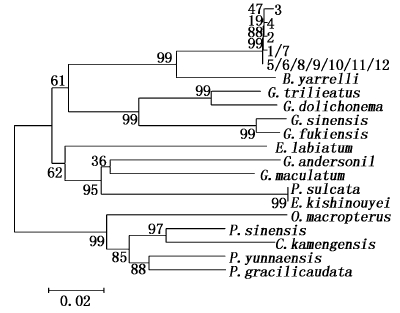
|
图 2 利用ME法构建15种鮡科鱼线粒体COⅡ分子基因系统树
Fig. 2 Molecular phylogenetic tree constructed by ME method of mitochondrial COⅡ gene from 15 species of Sisoridae
|
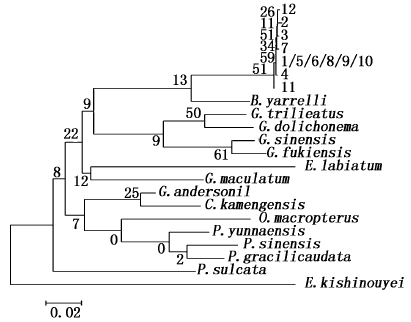
|
图 3 利用ME法构建15种鮡科鱼线粒体COⅢ分子系统树
Fig. 3 Molecular phylogenetic tree constructed by ME method of mitochondrial COⅢgene from 15 species of Sisoridae
|
巨魾在中国主要分布于云南省的怒江、澜沧江以及元江水系。红河州河口县位于元江下游,元江是该河流流经云南省玉溪市元江地段的名称,流经红河州地段就称之为红河[20]。本实验研究的巨魾来源于地势比较平坦的河口县地段,该流域较适合巨魾的生存。通过对巨魾的多态性研究可以探讨在该流域的巨魾遗传变异程度。
多态性指两个无关个体的碱基平均相差3%但不改变氨基酸排列[6]。核苷酸多态性是衡量一个群体遗传多样性大小的重要指标[18]。目前,基因的多态性研究主要应用于人类疾病方面,到目前为止全球已有1 831种疾病可以用基因多态性技术进行诊断[21],但是,运用基因的多态性对巨魾鱼类遗传和变异方面进行研究的资料不多。本研究对巨魾的COⅠ、COⅡ、COⅢ基因进行克隆,并对其多态性进行分析,建立与鮡科的系统进化关系来对河口巨魾进行深入研究,以确立其种群关系。
本研究通过克隆得到巨魾12个个体COⅠ、COⅡ、COⅢ基因序列,并利用MEGA 5.0分析表明COⅠ基因序列全长1 551 bp,起始密码子为AUG,终止密码子为TAA;出现了5个T与C的转换,1个A与C的转换,5个G与A的转换,1个G与T的转换,1个A与T的颠换;一个12个位点的插入,插入序列为CAACACTCCATG,共14个变异位点。COⅡ基因序列全长691 bp,起始密码子为ATG,终止密码子为TGA;出现了4个A与G的转换,1个T与C的转换,共5个变异位点。COⅢ基因序列全长784 bp,起始密码子为ATG,终止密码子为TGA,出现了5个A与G的转换,2个T与C的转换,1个缺失。符合DNA进化转换比颠换发生频率高的特点[22]。多态性分析在序列上都具有较高的同源性,COⅠ基因遗传距离在0.000 6~0.003 9之间,COⅡ基因遗传距离在0.001 5~0.004 4之间,COⅢ基因遗传距离在0.001 3~0.003 9之间,都非常接近表现出了遗传的高度稳定。可初步断定来自同一流域的不同个体COⅠ、COⅡ、COⅢ具有较高的遗传稳定性,可能由于生活在同一河流的原因,这些基因的突变率较低,序列较为保守。通过碱基成分分析,虽然3个基因的碱基含量有一定的差别,但是都同时表现为A+T含量明显高于G+C含量,符合脊椎动物线粒体DNA碱基组成的特点。系统发育研究表明,COⅠ、COⅡ、COⅢ都与中华纹胸鮡、福建纹胸鮡、三线纹胸鮡和长丝黑鮡在同一个分支,此结果与报道的巨魾属于鮡科的外缘种类[20]相符合,同时与报道的黑鮡属包含鮡属和纹胸鮡属的分类相一致[23]。通过系统进化树分析还可以看出河口巨魾与GenBank中巨魾(云南省澜沧江流域)的基因序列有一定的差异,可为下一步的研究提供分子依据。
| [1] | 陈小勇.云南鱼类名录[J]. 动物学研究, 2013, 34(4): 281–343. CHEN X Y.Checklist of fishes of Yunnan[J]. Zoological Research, 2013, 34(4): 281–343. |
| [2] | 刘跃天, 田树魁, 冷云, 等.野生巨魾生物学特性研究[J]. 现代农业科技, 2010: 302–303. LIU Y T, TIAN S K, LENG Y, et al.Study on the biological characteristics of wild Bagarius yarrelli[J]. Modern Agricultural Science and Technology, 2010: 302–303. |
| [3] | 田树魁, 薛晨江, 冷云, 等.巨魾的生物学特性初步研究[J]. 水生态学杂志, 2009, 30(3): 115–117. TIAN S K, XUE C J, LENG Y, et al.Preliminary research on the biological characteristics of Bagarius yarrelli[J]. Journal of Hydroecology, 2009, 30(3): 115–117. |
| [4] | 杨质高. 全球首次人工育成巨魾[N]. 中国渔业报, 2012-09-10. |
| [5] | 薛晨江, 张正雄, 马建颜, 等.巨魾人工繁殖初报与胚胎发育观察[J]. 水生态学杂志, 2012, 33(5): 54–56. XUE C J, ZHANG Z X, MA J Y, et al.Preliminary studies on artificial propagation and embryonic development of Bagarius yarrelli[J]. Journal of Hydroecology, 2012, 33(5): 54–56. |
| [6] | 陈四海, 区又君, 李加儿.鱼类线粒体DNA及其研究进展[J]. 生物技术通报, 2011: 13–20. CHEN S H, OU Y J, LI J E.Mitochondrial DNA and its progresses in fish[J]. Biotechnology Bulletin, 2011: 13–20. |
| [7] | KEITH P, LORD C, LORION J, et al.Phylogeny and biogeography of Sicydiinae (Teleostei:Gobiidae) inferred from mitochon drial and nuclear genes[J]. Marine Biology, 2011, 158(2): 311–326. |
| [8] | LORD C, LORION J, DETTAI A, et al.From endemism to widespread distribution:phylogeography of three amphidromous Sicyopterus species (Teleostei:Gobioidei:Sicydiinae)[J]. Marine Ecology, 2012, 455: 269–285. |
| [9] | JEON H B, CHOI S H, SUK H Y.Exploring the utility of partial cytochrome C oxidase subunit 1 for DNA barcoding of gobies[J]. Animal Systematics, Evolution and Diversity, 2012, 28(4): 269–278. |
| [10] | 李林, 梁宏伟, 李忠, 等.瓦氏黄颡鱼线粒体全基因组序列分析及系统进化[J]. 遗传, 2011, 33(6): 627–635. LI L, LIANG H W, LI Z, et al.Sequence and phylogeny analysis of the complete mitochondrial genome of Pelteobagrus vachelli[J]. Hereditas (Beijing), 2011, 33(6): 627–635. |
| [11] | 晁燕, 祁得林, 申志新, 等.黄河裸裂尻鱼细胞色素C氧化酶Ⅰ, Ⅱ和Ⅲ亚基基因的克隆及序列特征分析[J]. 动物学杂志, 2011, 46(4): 16–24. CHAO Y, QI D L, SHEN Z X, et al.Cloning and sequence analysis of genes encoding mtDNA cytochrome C oxidase subunits Ⅰ, Ⅱ and Ⅲ in Schizopygopsis pylzovi[J]. Chinese Journal of Zoology, 2011, 46(4): 16–24. |
| [12] | 邵爱华. 暗纹东方鲀(Takifugu fasciatus)线粒体DNA的分离纯化及其几个重要基因的克隆与序列分析[D]. 苏州:苏州大学, 2005. SHAO A H. Purification of mitochondrial DNA and cloning and analysis of the sequences of several important genes from Takifugu fasciatus[D]. Suzhou:Soochow University, 2005. |
| [13] | 齐兴柱, 尹绍武, 张本, 等.基于mtDNA-COⅡ基因序列对中国南海裸胸鳝属鱼类分子系统进化关系的研究[J]. 水产科学, 2010, 29(10): 605–609. QI X Z, YIN S W, ZHANG B, et al.Molecular phylogenetic relationships among six species in moray eels Gymnothorax sp in south China Sea by cytochrome oxidase Ⅱ gene sequences[J]. Fisheries Science, 2010, 29(10): 605–609. |
| [14] | 李京, 安世恒, 张鸿飞, 等.沟胫天牛亚科部分种类线粒体COⅡ基因特征及其系统发育研究[J]. 河南农业大学学报, 2013, 47(6): 715–721. LI J, AN S H, ZHANG H F, et al.Study on mithochondrial COⅡ gene characters and its phylogenetic analysis of Lamiinae partial species[J]. Journal of Henan Agricultural University, 2013, 47(6): 715–721. |
| [15] | 董徐辉, 李明云, 陈炯, 等.日本鬼鲉(Inimicus japonicus)线粒体Cyt b和COⅡ基因的克隆及鲉形目系统发育分析[J]. 海洋与湖沼, 2011, 42(1): 47–54. DONG X H, LI M Y, CHEN J, et al.Cloning of Inimicus japonicus mithochondrial Cyt b and COⅡ gene and their phylogenetic analysis in Scorpaeniformes[J]. Oceanologia et Limnologia Sinica, 2011, 42(1): 47–54. |
| [16] | 张望, 陈秀开, 彭勇, 等.鳅科鱼类DNA条形码分析及泥鳅和大鳞副泥鳅的分子鉴定[J]. 淡水渔业, 2015, 45(3): 19–24. ZHANG W, CHEN X K, PENG Y, et al.DNA barcoding analysis of cobitidae fishes and molecular indentification for misgurnus anguillicaudatus and paramisgurnus dabryanus[J]. Freshwater Fisheries, 2015, 45(3): 19–24. |
| [17] | 崔文涛, 刘立芹, 李红梅, 等.基于COⅡ基因序列的中国近海12种蛸亚科动物的分子系统进化研究[J]. 渔业科学进展, 2013, 34(3): 21–28. CUI W T, LIU L Q, LI H M, et al.Phylogenic study of Octopodinae in the China seas based on COⅡc partial sequences[J]. Progress in Fishery Sciences, 2013, 34(3): 21–28. |
| [18] | 乔德亮, 何晓梅, 韦传宝, 等.细鳞斜颌鲴三个群体线粒体COⅡ基因的遗传变异[J]. 淡水渔业, 2011, 41(5): 17–21. QIAO D L, HE X M, WEI C B, et al.Genetic variation of mtDNA COⅡ gene in three populations of Plagiognathops microlepis[J]. Freshwater Fisheries, 2011, 41(5): 17–21. |
| [19] | 杜民, 牛宝珍, 罗彩艳, 等.巨魾野生群体遗传多样性的RAPD分析[J]. 淡水渔业, 2015, 45(1): 15–19. DU M, NIU B Z, LUO C Y, et al.RAPD analysis of genetic diversity in the wild populations of Bagarius yarrelli[J]. Freshwater Fisheries, 2015, 45(1): 15–19. |
| [20] | 周用武, 庞峻峰, 周伟, 等.鮡科褶鮡属鱼类部分线粒体DNA序列分析与分子进化[J]. 西南林学院学报, 2007, 27(3): 45–51. ZHOU Y W, PANG J F, ZHOU W, et al.Taxonomy and molecular evolution of genus Pseudecheneis in family Sisoridae inferred from partial mitochondrial DNA sequences[J]. Journal of Southwest Forestry College, 2007, 27(3): 45–51. |
| [21] | 梁辰.基因多态性检测项目综合评价研究[D].北京:中国科学院大学,2014. LIANG C.Comprehensive evalution of gene polymorphism detection project[D]. Beijing:University of Chinese Academy of Sciences,2014. |
| [22] | 单云晶, 鲁翠云, 李超, 等.基于线粒体COⅠ基因序列的5种鲤养殖品种遗传多样性研究[J]. 中国水产科学, 2013, 20(5): 931–938. SHAN Y J, LU C Y, LI C, et al.Study of DNA barcoding and genetic variation based on the mitochondrial COⅠ gene sequences in five carp varieties[J]. Journal of Fishery Sciences of China, 2013, 20(5): 931–938. |
| [23] | 肖海, 代应贵, 张晓杰, 等.我国鮡科鱼类种质资源研究进展[J]. 广东农业科学, 2010: 180–182. XIAO H, DAI Y G, ZHANG X J, et al.Advances in the study on the genetic resources of Sisoridae in China[J]. Guangdong Agricultural Sciences, 2010: 180–182. |
2. College of Life Science and Technology, Honghe University, Mengzi 661199, Yunnan, China
 2016,
Vol. 25
2016,
Vol. 25


