鳊鲂鱼类,包括鳊属(Parabramis)和鲂属(Megalobrama),它们都属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)、鲌亚科(Culterinae)。鲂属主要包括厚颌鲂(M. pellegrini)、三角鲂(M. terminalis)、团头鲂(M. amblycephala)、广东鲂(M. hoffmanni);而鳊属只有长春鳊(P. pekinensis)。鲂属鱼类由于形态差异不显著,在命名上存在一定分歧[1, 2]。鳊鲂鱼类分布在我国大河、湖泊、水库中,在我国淡水养殖业及渔业捕捞中占有重要比例。由于20世纪六七十年代的杂交研究,鳊鲂鱼类的种质纯度受到一定影响,种质资源衰退严重[3]。在鳊鲂鱼类研究中,利用染色体多态性和DNA含量双重方法,进行鳊鲂鱼类的种属区分和进化地位分析还未见报道。虽然有学者通过形态学方法对鳊鲂鱼类进行种群区分,但宏观鉴定方法易受外界环境影响,具有一定的局限性[4, 5]。
染色体是细胞内遗传物质深度压缩形成的聚合体,易被碱性染料染成深色,所以称为染色体,它是遗传信息(基因)的主要载体。20世纪70年代[6],只有初步的单个物种的染色体核型研究,没有系统开展鳊鲂鱼类染色体核型多态性及进化地位分析。本研究以鲂属(团头鲂、广东鲂、厚颌鲂和三角鲂)和鳊属(长春鳊)为实验材料,进行染色体核型分析,并讨论了鳊鲂鱼类在进化上的地位;同时以鸡血细胞DNA含量为标准(2.30 pg),鳊鲂鱼类外周血细胞为样本,使用流式细胞仪(BD AccuriTM C6)测定了鳊鲂鱼类二倍体细胞的DNA含量。为今后鳊鲂鱼类育种、分类、鉴定和基因组研究提供理论依据。
1 材料与方法 1.1 实验材料实验所用的4种鲂属鱼类为团头鲂“浦江1号”(TTF)、广东鲂(GDF)、厚颌鲂(HHF)、三角鲂(SJF)和1种鳊属鱼类长春鳊(CCB),形态见图1。其中:团头鲂 “浦江1号”取自于上海海洋大学农业部团头鲂遗传育种中心;广东鲂取自广东南海沙头镇大健鱼苗场;厚颌鲂取自重庆市东平水产养殖有限公司;三角鲂取自广东珠江东江支流河源段;长春鳊取自长江江苏靖江段。每个群体取样雌雄各5尾,共50尾,在DNA含量测定和染色体制备的前一周停止投喂。

|
图1
5种鳊鲂鱼类的形态
Fig.1
The morphology of five species of Megalobrama and Parabramis
TTF 团头鲂; GDF 广东鲂; HHF 厚颌鲂; SJF 三角鲂; CCB 长春鳊。 TTF M. amblycephala; GDF M. hoffmanni; HHF M. pellegrini; SJF M.terminalis; CCB P. pekinensis. |
选用鸡血细胞(DNA含量2.30 pg)[7]作为对照标准,进行鳊鲂鱼类DNA含量测定。用1 mL注射器抽0.1 mL肝素钠,在实验鱼尾静脉采血0.2~0.4 mL,立即混匀。随后加入到含有1 mL磷酸缓冲液(PBS)的离心管中,1 000 r/min离心5 min,重复清洗2次,用预冷PBS 70%乙醇4 ℃固定过夜,加PI和RNase处理30 min,用流式细胞仪(BD AccuriTM C6)进行检测。
实验鱼DNA含量依照以下公式计算:

放鱼前1天水族箱加满水(用加热器升温至24 ℃),曝气备用,实验前将鱼暂养3 d,停止投喂。参照林义浩[8]体内注射植物血细胞凝集素(PHA)和秋水仙素方法:胸鳍基部注射PHA(10 μg/g),22 h后注射秋水仙素(4 μg/g),4 h后,立即剪断鱼两侧鳃耙,在水中放血10 min,解剖,取鱼头肾于 0.75%的生理盐水中,用两把镊子充分撕扯,直至淋巴细胞充分释放。
将细胞悬浮液移入离心管中(用300目筛网过滤,1 000 r/min离心5 min,吸去上清。然后加入6 mL 0.5%的KCl低渗液(37 ℃预热,现配),立即混匀,于37 ℃静置低渗40 min;低渗结束后,1 000 r/min离心5 min,吸去上清。加入6 mL冰冻(-20 ℃)固定液[V(甲醇)∶V(冰醋酸)=3∶1],用吸管小心混匀,固定30 min,1 000 r/min离心5 min,吸去上清。再以同样方法各固定、离心1次;最后加入适量的冷冻固定液(1~2 mL),混匀后,进行滴片,37 ℃烘箱放1 h,干燥后即可染色。
2 结果与分析 2.1 染色体数目分析在显微镜油镜下选取5种鳊鲂鱼类各100个染色体中期分裂相,进行染色体计数,其计数结果见表1。其中,团头鲂染色体数目为2n=48的占70%;广东鲂2n=48的占68%;厚颌鲂2n=48的占78%;三角鲂2n=48的占81%;长春鳊2n=48的占72%。所以可以确定这5种鳊鲂鱼类为二倍体,染色体数目均为2n=48。
|
表1 5种鳊鲂鱼类染色体数目分布 Tab. 1 The number of chromosomes of five species of Megalobrama and Parabramis |
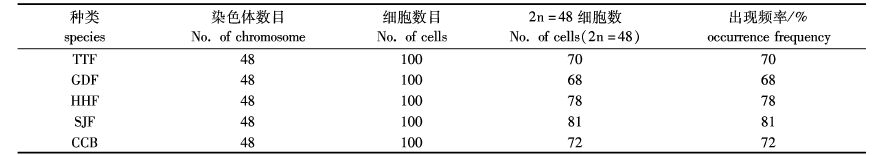
各选6个染色体数目完整、形态清晰的中期分裂相,用Photoshop软件放大,Image J测定染色体长、短臂长度,然后计算染色体的相对长度、臂比,并进行统计学分析。得到鳊鲂鱼类的染色体核型参数(表2)。
|
表2 5种鳊鲂鱼类的核型参数 Tab. 2 Indices of karyotypes of five species of Megalobrama and Parabramis |
根据LEVAN等[9]按照着丝点的位置进行染色体分类和命名的原则,可将每一种鲂属鱼类(团头鲂、广东鲂、厚颌鲂、三角鲂)染色体分为3组,鳊属(长春鳊)染色体分为2组(图2)。其中,团头鲂的染色体核型为2n=48=24m+20sm+4st,NF=92,具有1对特大染色体位于m组;广东鲂的染色体核型为2n=48=26m+18sm+4st,NF=92,在m和sm组均具有一对较大染色体;厚颌鲂的染色体核型为2n=48=26m+18sm+4st,NF=92,在m组有一对特大染色体;三角鲂的染色体核型为2n=48=24m+20sm+4st,NF=92,一对较大染色体在sm组中;长春鳊的染色体核型为2n=48=26m+22sm,NF=96,没有发现st组染色体(表2,图2)。此外,在鳊鲂鱼类染色体中未发现次缢痕及性染色体,也未发现随体。

|
图2
5种鳊鲂鱼类染色体及其核型
Fig.2
Chromosome and karyotype of five species of Megalobrama and Parabramis
TTF 团头鲂; GDF 广东鲂; HHF 厚颌鲂; SJF 三角鲂; CCB 长春鳊。 TTF M. amblycephala; GDF M. hoffmanni; HHF M. pellegrini; SJF M.terminalis; CCB P. pekinensis. |
选公鸡血细胞作为参照,用流式细胞仪对5种鳊鲂鱼类外周血细胞DNA含量进行测定。图3分别表示:鸡(DZ)、团头鲂(TTF)、广东鲂(GDF)、厚颌鲂(HHF)、三角鲂(SJF)、长春鳊(CCB)血细胞的消光值。以公鸡血细胞DNA绝对含量值 2.30 pg为标准,计算出团头鲂血细胞DNA绝对含量为(2.66±0.03) pg;广东鲂为(2.57±0.05) pg;厚颌鲂为(2.81±0.08) pg;三角鲂为(2.60±0.02) pg;长春鳊为(2.34±0.04) pg(表3)。通过表3可以看出:厚颌鲂血细胞DNA含量最高,长春鳊血细胞DNA含量最少。
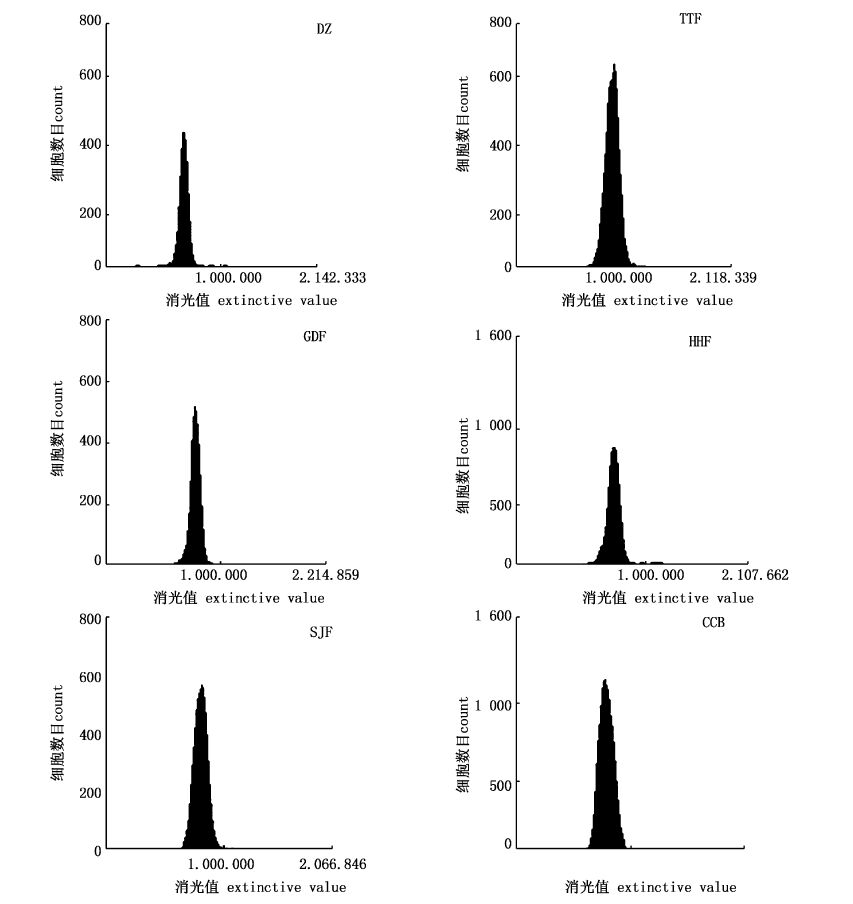
|
图3
5种鳊鲂鱼类血细胞DNA含量
Fig.3
DNA contents of five species of Megalobrama and Parabramis
DZ 鸡;TTF 团头鲂;GDF 广东鲂;HHF 厚颌鲂;SJF 三角鲂;CCB 长春鳊。 DZ Chicken; TTF M. amblycephala; GDF M. hoffmanni; HHF M. pellegrini; SJF M.terminalis; CCB P. pekinensis. |

|
表3 流式细胞仪检测5种鳊鲂鱼类DNA含量 Tab. 3 DNA content of five species of Megalobrama and Parabramis examined by flow cytometry (FCM) |
从表1可以看出,5种鳊鲂鱼的染色体数目(2n=48)出现频率在68%到81%之间,对于非重数染色体细胞,可能是低渗或制片操作中导致少数染色体丢失的结果,每种鱼的个体差异不会造成染色体数目的变化。所以,可以确定5种鳊鲂鱼类的染色体数目都为2n=48。
研究显示:团头鲂的核型公式为2n=24m+20sm+4st,NF=92;三角鲂的染色体核型公式为2n=24m+20sm+4st,NF=92;广东鲂的染色体核型公式为2n=26m+18sm+4st,NF=92;厚颌 鲂的染色体核型公式为2n=26m+18sm+4st,NF=92。表明鲂属鱼类中团头鲂和三角鲂的核型相同,广东鲂与厚颌鲂核型相同。长春鳊核型公式为2n=26m+22sm,NF=96,与鲂属鱼类差别较大,并且缺少2对st型染色体。本文获得团头鲂的核型与昝瑞光和宋峥[6] 的结果一致,与李渝成等[10]和叶星等[11]的结果存在明显差异。厚颌鲂的核型与易少奎等[12]结果存在差异。三角鲂核型与陈淑群[13]的结果存在差异。获得的广东鲂核型与叶星等[11]和朱新平等[14]的结果一致。鳊属长春鳊核型与林义浩[15]的结果相一致。这些结果表明团头鲂、三角鲂和厚颌鲂的染色体核型存在差异,这些不一致固然有研究准确性上的差异,但也不排除3个鲂属物种在不同群体核型上存在差异。卓孝磊和邹记兴[16]曾分析了77种海水鱼类核型,有9种存在种内核型差异。余先觉等[17]总结分析了200多种淡水鱼类核型,有47种也存在种内核型差异。
鱼类核型特征与其演化情况有直接关系,一种鱼类的染色体数目、形态的稳定是相对的,同一属近缘种的核型不一定相似,同种鱼类的染色体数目或形态也会出现差异。鲂属鱼类染色体核型尽管存在一定的多态性,但在演化过程中存在着趋同性,与不同属的长春鳊的核型存在明显差异,核型反映出来的亲缘关系与线粒体基因组、形态学和微卫星分析的结果相似[18, 19, 20]。
3.2 鳊鲂鱼类的DNA含量20世纪六十年代,OHNO和ATKIN等[21]测定了数种鱼类体细胞的DNA含量。七十年代末开始有人用流式细胞仪(flow cytometry)测定鱼类细胞的DNA含量[22, 23]。流式细胞仪测定鱼类细胞DNA含量具有操作方便、快速以及测量结果准确的优点。同一种鱼类的DNA含量是恒定的,具有种的特异性,可作为物种种质遗传的特征性参数[24]。
本研究显示,团头鲂DNA绝对含量为(2.66±0.03)pg,这同范兆廷等[24]所测结果一致,不同于张新辉等[25]的(2.90±0.08) pg。厚颌鲂DNA含量为(2.81±0.08)pg,所测结果和易少奎等[12]相近。广东鲂DNA含量为(2.57±0.05) pg;三角鲂DNA含量为(2.60±0.02) pg;长春鳊DNA含量为(2.34±0.04) pg,这3种鳊鲂鱼类的DNA含量属首次测定。由于团头鲂、广东鲂、厚颌鲂、三角鲂为同属鱼类,亲缘关系较近,DNA含量差异较小。而长春鳊与鲂属鱼类为不同属鱼类,亲缘关系较远,因而DNA含量差异较大。
3.3 染色体核型与鱼类进化的关系鳊鲂鱼类染色体核型的明显差异,可能与鱼类染色体发生了变异有关。染色体变异方式有染色体结构重排如臂间倒位(Pericentric inversion),以及罗伯逊易位(Robertsonian translocation) [26, 27]。罗伯逊易位是通过着丝点融合、解离和衔接融合从而形成整臂易位。若为罗伯逊易位,则是那些具有双臂的中部或亚中部着丝点染色体解离后成为具有单臂的端部着丝点染色体,或者那些具有单臂的端部着丝点染色体,其着丝点融合可成为具有双臂的中部或亚中部着丝点染色体,从而导致染色体数目非整倍性的增加或减少;臂间倒位则可使端部或亚端部着丝点染色体变为中部或亚中部着丝点染色体,只改变染色体形态,不改变染色体数目,但改变染色体总臂数[28]。在鱼类进化过程中,鳊属鱼类的变异核型可能是由具有4st相对原始核型的鲂属鱼类通过臂间倒位形成的。染色体核型变化与生物进化有着极其密切的关系。李树深[29]研究指出:在特定的分类阶元中,臂数少的为较原始类群,臂数多的为特化类群。鲂属鱼类臂数NF=92,鳊属鱼类臂数NF=96,可认为长春鳊为特化类群,鲂属鱼类为较原始类群。
鳊鲂鱼类是我国主要经济鱼类,为保证种质和遗传性状开展对其染色体核型的研究是很有必要的。同时,良好的染色体图像为以后功能基因在染色体上的定位打下了很好基础。
| [1] | 成庆泰,郑葆珊.中国鱼类系统检索[M].北京:科学出版社, 1987:427-431.CHENG Q T, ZHENG B S. Systematic Synopsis of Chinese Fishes[M]. Beijing:Science Press, 1987:427-431. |
| [2] | 罗云林.鲂属鱼类的分类整理[J].水生生物学报, 1990, 14(2):160-165.LUO Y L. A revision of fishes of the cyprinid genus Megalobrama[J]. Acta Hydrobiological Sinica, 1990, 14(2):160-165. |
| [3] | 李思发,朱泽闻,邹曙明,等.鲂属团头鲂、三角鲂及广东鲂种间遗传关系及种内遗传差异[J].动物学报, 2002, 48(3):339-345.LI S F, ZHU Z W, ZOU S M, et al. Interspecific phylogenesis and intraspecific genetic differences of genus Megalobrama:bluntnose black bream (M. amblycephala), guangdong black bream (M. hoffmanni) and black bream (M. terminalis)[J]. Acta Zoologica Sinica, 2002, 48(3):339-345. |
| [4] | 蔡鸣俊,张敏莹,曾青兰,等.鲂属鱼类形态度量学研究[J].水生生物学报, 2001, 25(6):631-635.CAI M J, ZHANG M Y, ZENG Q L, et al. A study on morphometrics of the genus Megalobrama[J]. Acta Hydrobiologica Sinica, 2001, 25(6):631-635. |
| [5] | 宋文,王艺舟,祝东梅,等.鲂属鱼类形态差异分析[J].淡水渔业, 2013, 43(3):21-27.SONG W, WANG Y Z, ZHU D M, et al. Morphological variations among the genus Megalobrama[J]. Freshwater Fisheries, 2013, 43(3):21-27. |
| [6] | 昝瑞光,宋峥.草鱼、团头鲂染色体组型的分析比较[J].遗传学报, 1979, 6(2):205-210.ZAN R G, SONG Z. Analysis and comparison between the karyotypes of ctenopharyngodon idellus and Megalobrama amblycephala[J]. Acta Genetica Sinica, 1979, 6(2):205-210. |
| [7] | TIERSCH T R, CHANDLER R W. Chicken erythrocytes as an internal reference for analysis of DNA content by flow cytometry in grass carp[J]. Transactions of the American Fisheries Society, 1989, 118(6):713-717. |
| [8] | 林义浩.快速获得大量鱼类肾细胞中期分裂相的PHA体内注射法[J].水产学报, 1982, 6(3):201-208.LIN Y H. A PHA injection method in vivo for the rapid obtainment of large numbers of metaphase figures from kidney cells of teleosts[J]. Journal of Fisheries of China, 1982, 6(3):201-208. |
| [9] | LEVAN A, FREDGA K, SANDBERG A A. Nomenclature for centromeric position on Chromosome[J]. Hereditas, 1964, 52(2):201-220. |
| [10] | 李渝成,李康,周暾.中国鲤科鱼类染色体组型的研究:I.鳊亚科10种鱼的染色体组型[J].遗传学报, 1983, 10(3):216-222.LI Y C, LI K, ZHOU D. Studies on the karyotypes of Chinese Cyprinid fishes:Ⅰ.karyotypes of ten species of Abramidinae[J]. Acta Genetica Sinica, 1983, 10(3):216-222. |
| [11] | 叶星,谢刚,祁宝伦,等.广东鲂(♀)×团头鲂(♂)杂交子一代及其双亲染色体组型的分析[J].大连水产学院学报, 2002, 17(2):102-107.YE X, XIE G, QI B L, et al. Comparison of karyotypes of the F1 hybrid (Megalobrama hoffmanni ♀×M. amblycephala ♂)with its Parents[J]. Journal of Dalian Fisheries University, 2002, 17(2):102-107. |
| [12] | 易少奎,张新辉,杨坤,等.厚颌鲂核型分析与DNA含量的测定[J].华中农业大学学报, 2014, 33(1):80-84.YI S K, ZHANG X H, YANG K, et al. Karyotype and cellar DNA content analysis of Megalobrama pellegrini[J]. Journal of Huazhong Agricultural University, 2014, 33(1):80-84. |
| [13] | 陈淑群.青鱼(♀)×三角鲂(♂)不同亚科之间的杂交研究1、青鱼(♀)、三角鲂(♂)及其子一代的比较细胞遗传学研究[J].湖南师范大学学报:自然科学版, 1984, (4):71-80.CHEN S Q. Investigation on the inter-subfamily hybridization (Mylopharyngodon piceus R. (♀)×Megalobrama terminalis R.(♂)Ⅰ. Comparative cytogenetic study on Mylopharyngodon piceus R. (♀), Megalobrama Terminalis R. (♂) and their F1 generation[J]. Journal of Natural Science of Hunan China Normal University, 1984, (4):71-80. |
| [14] | 朱新平,林礼堂,夏仕玲.广东鲂的染色体组型[J].淡水渔业, 1990, (5):12-13.ZHU X P, LIN L T, XIA S L. Karyotype of M. hoffmanni [J]. Freshwater Fisheries, 1990, (5):12-13. |
| [15] | 林义浩.团头鲂×长春鳊两性全育原因的探讨[J].水产学报, 1985, 9(1):63-69.LIN Y H. An approach to the factor causing the fertility of the hybrids of blunted snout bream (Megalobrama amblycephala)♀×Chinese bream (Parabramis pekinensis)♂[J]. Journal of Fisheries of China, 1985, 9(1):63-69. |
| [16] | 卓孝磊,邹记兴.我国海水鱼类核型及染色体显带研究进展[J].热带海洋学报, 2007, 26(5):73-80.ZHUO X L, ZOU J X. Advances in karyotype and chromosome banding studies of marine fish in China[J]. Journal of Tropical Oceanography, 2007, 26(5):73-80. |
| [17] | 余先觉,周暾,李康,等.中国淡水鱼类染色体[M].北京:科学出版社, 1989:23-25.YU X J, ZHOU D, LI K, et al. Chromosomes of Chinese Fresh-water Fishes[M]. Beijing:Science Press, 1989:23-25. |
| [18] | 赖瑞芳,张秀杰,李艳和,等.鲂属鱼类线粒体基因组的比较及其系统发育分析[J].水产学报, 2014, 38(1):1-14.LAI R F, ZHANG X J, LI Y H, et al. Comparison of mitochondrial genomes of the genus Megalobrama and their phylogenetic analysis[J]. Journal of Fisheries of China, 2014, 38(1):1-14. |
| [19] | 陈杰,李福贵,黄创新,等.不同鳊鲂属鱼类群体的形态差异分析[J].上海海洋大学学报, 2014, 23(3):388-394.CHEN J, LI F G, HUANG C X, et al. Morphological variations of genera Parabramis and Megalobrama teleost populations[J]. Journal of Shanghai Ocean University, 2014, 23(3):388-394. |
| [20] | 张倩倩,陈杰,蒋霞云,等.不同鳊鲂鱼类群体微卫星DNA指纹图谱的构建和遗传结构分析[J].水产学报, 2014, 38(1):15-22.ZHANG Q Q, CHEN J, JIANG X Y, et al. Establishment of DNA fingerprinting and analysis on genetic structure of different Parabramis and Megalobrama populations with microsatellite[J]. Journal of Fisheries of China, 2014, 38(1):15-22. |
| [21] | OHNO S, ATKIN N B. Comparative DNA values and chromosome complements of eight species of fishes[J]. Chromosoma, 1966, 18(3):445-466. |
| [22] | KRAEMER P M, DEAVEN L L, CRISSMAN H A, et al. DNA constancy despite variability in chromosome number[J]//DUORAW E J. Advances in cells and molecular biology. New York:Academic Press, 1972, 2:47-108. |
| [23] | TIERCH T R, CHANDLER R W, WACHTEL S S, et al. Reference standards for flow cytometry and application in comparative studies of nuclear DNA content[J]. Cytometry, 1989, 10(6):706-710. |
| [24] | 范兆廷,尹洪滨,宋苏祥.十三种淡水养殖鱼类的DNA含量[J].水产学报, 1995, 19(4):322-326.FAN Z T, YI H B, SONG S X. DNA content from thirteen species of cultured freshwater fishes in China[J]. Journal of Fisheries of China, 1995, 19(4):322-326. |
| [25] | 张新辉,罗伟,高泽霞,等.团头鲂三倍体的诱导及其鉴定[J].水产科学, 2013, 32(9):503-508.ZHANG X H, LUO W, GAO Z X, et al. Induction and identification of triploid in bluntnose black bream Megalobrama amblycephala[J]. Fisheries Science, 2013, 32(9):503-508. |
| [26] | 小岛吉雄.鱼类细胞遗传学[M].林义浩,译.广州:广东科技出版社, 1990:8-33.小岛吉雄. Fish Cell Genetics[M]. LIN Y H, trans. Guangzhou:Guangdong Science and Technology Press, 1990:8-33. |
| [27] | 桂建芳,李渝成,李康,等. (鱼叚)虎鱼亚目三种鱼染色体的比较研究[J].动物学研究, 1984, 5(s3):67-68.GUI J F, LI Y C, LI K, et al. A comparative study of the karyotypes of three species of Gobioid Fishes[J]. Zoological Research, 1984, 5(s3):67-68. |
| [28] | 毛连菊,李雅娟. 5种海水鱼类染色体的组型分析[J].大连水产学院学报, 2002, 17(2):108-113.MAO L J, LI Y J. Karyotype analyses of five species of marine fishes[J]. Journal of Dalian Fisheries University, 2002, 17(2):108-113. |
| [29] | 李树深.鱼类细胞分类学[J].生物科学动态, 1981(2):8-15.LI S S. Fish cytotaxonomy[J]. Biological Sciences Dynamic, 1981(2):8-15. |
 2015, Vol. 24
2015, Vol. 24


