2. 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
3. 农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
西藏开菲尔粒(Tibetan kefir grains,TKGs),俗称“藏灵菇”,因源于西藏而得名。与其他品系的开菲尔粒相似,TKGs外形也呈花椰菜状,白色或者白色偏黄,上面栖息着多种微生物。有时,TKGs的某些个体会以伸展开的片层状形态生长,状如盛开的雪莲花,故也被称之为“西藏雪莲”。
西藏开菲尔粒是一种天然的乳品发酵剂,由其发酵牛奶得到的发酵乳,称为西藏开菲尔或开菲尔发酵乳。开菲尔发酵乳具有一定的保健作用。据研究,开菲尔具有调节免疫系统[1]、改善消化机能[2]、抗炎、杀菌及促进伤口愈合[3, 4, 5]等功效,但其保健机理迄今依然未知。
西藏开菲尔粒中的微生物多样性因地域和分析研究方法不同而差异较大,但基本上主要由细菌与酵母菌两大类微生物群体组成[6, 7, 8, 9, 10, 11]。而有关西藏开菲尔粒中微生物的数量特点及变化关系还不是很清楚。实时荧光定量PCR(real-time quantitative PCR)技术是在PCR 反应体系中加入荧光染料或荧光探针,利用荧光信号的积累,实时监测整个PCR 反应进程,最后通过标准曲线对未知模板进行定量分析,具有操作简便、快速高效、高通量、高敏感性等特点。该技术在分子诊断、分子生物学研究、动植物检疫以及食品安全检测等方面有着广泛的应用[12, 13, 14, 15, 16]。
将实时荧光定量 PCR技术用于分析西藏开菲尔粒中微生物基因的拷贝数与比例,探究西藏开菲尔粒中细菌与酵母菌之间的数量变化规律,有助于认识西藏开菲尔粒生长特点以及细菌与酵母菌间的共生关系。
1 材料与方法 1.1 主要材料西藏开菲尔粒(本实验室保存),1 L光明纯牛奶。
1.2 主要试验仪器设备与试剂 1.2.1 仪器组织研磨器(OSE-Y20,天根)、PCR仪(Eppendorf 6325,德国)、电泳仪、凝胶成像系统(BioRad Molecular Imager® Gel DocTM XR+System,美国)、荧光定量PCR仪(7500 Fast Real-time PCR system,ABI)。
1.2.2 试剂溶菌酶、溶壁酶(天根,RT401、RT410),缓冲液(20 mmol/L Tris,pH 8.0;2 mmol/L Na2-EDTA; 1.2% Triton)、2 × Taq PCR MasterMix (天根,KT201)、 pGM-T载体(天根,VT202)、大肠杆菌TOP 10细胞(天根,CB104)、海洋动物组织基因组提取试剂盒(天根,DP324)、普通产物纯化试剂盒(天根,DP204)、高纯度质粒提取试剂盒QIAprep Spin Miniprep (Qiagen,德国)、Quant-iTTM PicoGreen® dsDNA Reagent and Kits(Invitrogen,美国)、SYBR® Premix Ex TaqTM Ⅱ (2×)(TaKaRa,日本)。
1.2 mol/L山梨醇缓冲液: 用0.1 mol/L磷酸钠缓冲液配制,最终pH定为7.4。
本实验所用的引物均由上海生工生物工程有限公司合成,合成的引物信息如表1所示。
| 表 1 实验中用到的引物 Tab.1 Primers used in this study |
将-20 ℃低温保存的西藏开菲尔粒按10%(w/v)接种量接入光明纯牛奶中,置于恒温培养箱中(温度25 ℃)静置培养24 h。重复活化3次后,重新接种培养24 h。过滤出西藏开菲尔粒,用无菌水将西藏开菲尔粒冲洗干净,再加入相应量的光明纯牛奶中继续培养[10]。对培养1、4、8和12周的西藏开菲尔粒分别取样,进行微生物总基因组提取后用于后续荧光定量PCR实验研究。
1.3.2 西藏开菲尔粒中总微生物基因组DNA的提取样品前处理:(1)随机剪取3个不同部位共40 mg的3个西藏开菲尔粒样品分别放至1.5 mL离心管内研磨2 min,添加600 μL山梨醇缓冲液与5 μL溶壁酶,30 ℃处理90 min。(2)7 500 r/min离心10 min,加入108 μL缓冲液(20 mmol/L Tris,pH 8.0; 2 mmol/L Na2-EDTA; 1.2% Triton)和72 μL溶菌酶,37 ℃反应50 min。(3)之后的步骤完全按照天根海洋动物组织基因组提取试剂盒的操作步骤进行。对培养1、4、8和12周的西藏开菲尔粒进行基因组提取的步骤参照上述步骤进行,每个时间点3个平行样品,随机取每个西藏开菲尔粒上3个不同部位共40 mg进行微生物基因组的提取。
1.3.3 琼脂糖凝胶电泳检测DNA电泳条件:电泳缓冲液1×TAE,电压130 V、30 min。用1%的琼脂糖凝胶检测,入DNA作Marker,通过凝胶成像系统观察最终结果。
1.3.4 荧光定量PCR检测样品(1)细菌和酵母菌标准质粒的制备
首先提取西藏开菲尔粒中微生物的总基因组DNA(天根海洋动物组织基因组提取试剂盒),将其作为模板进行普通PCR扩增,采用的扩增引物为表1中的27F和1 492R[17],扩增片段为细菌16S rRNA genes的全长。PCR反应体系为50 μL,包含有25 μL的2× Taq PCR MasterMix(天根),正反向引物各1 μL(浓度为10 μmol/L),模板大约100 ng以及适量的ddH2O补足反应体系至50 μL。PCR扩增程序:预变性94 ℃、5 min;变性94 ℃、30 s,退火55 ℃、30 s,延伸72 ℃、1 min,35个循环;最后72 ℃、10 min。
得到的PCR扩增产物进行纯化后克隆到pGM-T载体上来获得重组质粒,然后将重组质粒导入到大肠杆菌T0P10感受态细胞,采用蓝白斑筛选、菌落PCR和2%的琼脂糖凝胶电泳筛选出阳性克隆。将阳性克隆送去测序(生工生物工程上海股份有限公司),进行插入片段序列验证,得到的阳性克隆序列信息与NCBI核酸数据库比对确定具体细菌种类。
挑取经测序验证的阳性菌落于LB液体培养基(北京路桥)中进行过夜增菌培养,用高纯度质粒提取试剂盒QIAprep Spin Miniprep (Qiagen,德国)从菌液中提取质粒,该质粒即为细菌16S rRNA基因标准质粒。随后用Quant-iT PicoGreen® dsDNA Reagent and Kits试剂盒绝对定量,并计算出上述细菌标准质粒的拷贝数[20],定量后的标准细菌质粒样品保存于-20 ℃冰箱备用。
酵母菌标准质粒的构建与细菌标准质粒的构建方法类似,只是采用的引物为表1中的NL-1与NL-4,扩增酵母菌的26S rRNA genes的D1/D2区[21]。
(2)荧光定量PCR试验
采用TaKaRa公司的SYBR® Premix Ex TaqTM Ⅱ 试剂盒与ABI 公司的7500 Fast Real-time PCR 系统进行实时荧光定量PCR反应。
首先将提取的西藏开菲尔粒中微生物的总基因组(即1.3.2 中提取的基因组)DNA经过梯度稀释至终浓度在每微升0.1~1.0 ng之间;其次将上述构建的细菌与酵母菌标准质粒均进行10倍的梯度稀释,选择每微升108~104拷贝数浓度梯度的标准质粒用于荧光定量PCR反应并绘制标准曲线。荧光定量PCR反应体系为20 μL,包含有2.0 μL模板、0.4 μmol/L的引物各0.8 μL、10 μL的SYBR® Premix Ex TaqTM Ⅱ(2×) (TaKaRa,日本)、0.4 μL的Rox Reference Dye (50×),ddH2O补足PCR反应体系至 20 μL。细菌荧光定量PCR反应中引物采用表1中的338F、518R;酵母菌荧光定量PCR反应中引物采用表1中的Yeast-F、Yeast-R。PCR的扩增程序为:预变性95 ℃、10 min;变性94 ℃、15 s,退火59 ℃、30 s,延伸72 ℃、30 s,共40个循环。定量PCR反应中每个样品3个平行,重复3次。
定量PCR反应结束后,从软件生成的细菌与酵母菌各自标准曲线中读取并换算出对应样品的细菌与酵母菌基因拷贝数。
1.4 数据处理采用Geneious Pro 6.07对涉及到的基因序列进行分析;应用BLASTN程序(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列相似性鉴定;Origin 8软件对荧光定量PCR数据结果进行统计学分析;SPSS 19.0软件对1、4、8和12周西藏开菲尔粒中细菌与酵母菌含量进行ANOVA 单因素方差分析(P﹤0.05为存在显著性差异),数值以均值±标准差表示。
2 结果与分析 2.1 电泳检测提取的西藏开菲尔粒中微生物的总基因组DNA按照1.3.2 基因组提取方法,将从样品TKGs中提取的微生物总基因组DNA用琼脂糖凝胶电泳检测,结果如图1所示。3个样品主要在23 kbp位置均出现一条亮度强的DNA带,这表明提取的西藏开菲尔粒中微生物总基因组DNA分子大小较为完整,提取过程中物理机械剪切力对DNA分子的破坏较小,且方法的重现性好。
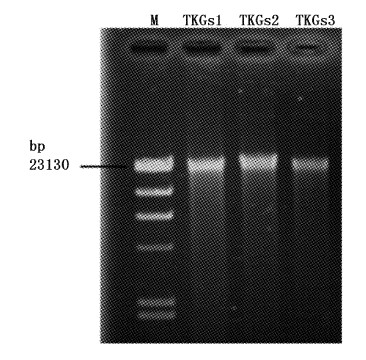
|
图 1
提取的基因组DNA电泳图
Fig. 1
Isolated genomic DNAs
M: marker; TKGs1、TKGs2、TKGs3为3组平行样品基因组。 M: marker; TKGs1,TKGs2,TKGs3: genomic DNAs of samples in 3 parallel. |
对上述西藏开菲尔粒微生物总基因组DNA进行荧光定量PCR反应,分析细菌16S rRNA 基因拷贝数,结果见图2。qPCR反应的扩增效率为103.154%,属于合理的范围;3个平行样品总细菌基因拷贝数扩增曲线几乎完全重叠,平均每微升3.40×107拷贝,这表明以提取的西藏开菲尔粒微生物总基因组DNA为模板,运用qPCR定量分析细菌16S rRNA基因的拷贝数是可行的。
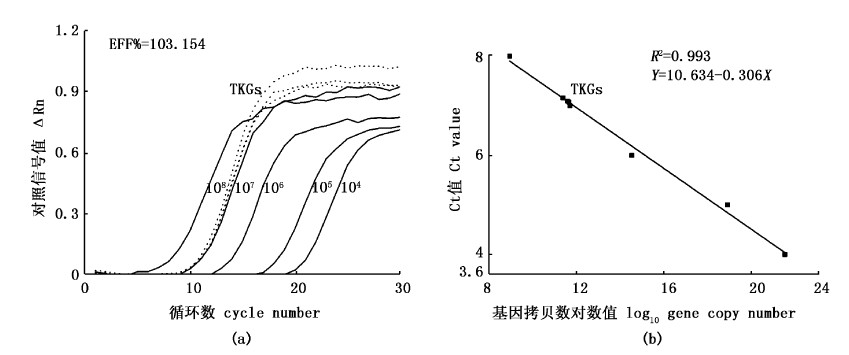
|
图 2
TKGs中细菌基因拷贝数
Fig. 2
Gene copy number of bacteria in TKGs
a为扩增曲线图,虚线代表TKGs样品;b为标准曲线图,TKGs为样品。 a: amplification plot, the dotted curves stand for TKGs samples; b: standard curve, TKGs were samples. |
与2.2类似,同样对西藏开菲尔粒微生物总基因组DNA进行荧光定量PCR反应,分析酵母菌26S rRNA 基因拷贝数,结果如图3所示。qPCR反应扩增效率为93.845%,属于合理的范围;3个平行样品总酵母菌基因拷贝数扩增曲线几乎完全重叠,平均每微升样品酵母菌基因3.71×105拷贝,表明以提取的西藏开菲尔粒微生物总基因组DNA为模板,运用qPCR定量分析酵母菌26S rRNA基因的拷贝数是可行的。
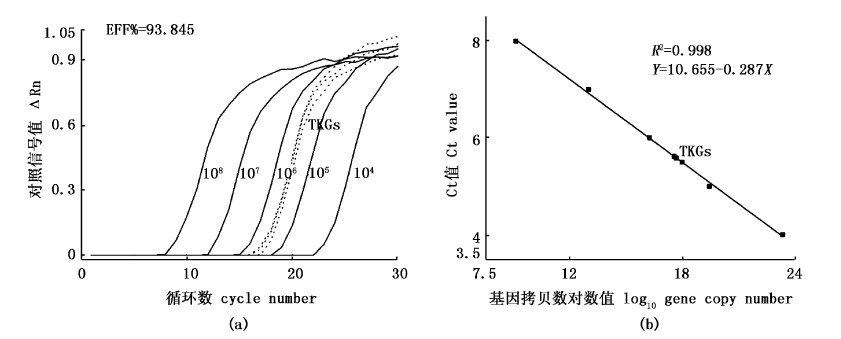
|
图 3
TKGs中酵母菌基因拷贝数
Fig. 3
Gene copy number of yeasts in TKGs
a为扩增曲线图,虚线代表TKGs样品;b为标准曲线图,TKGs为样品。 a: amplification plot, the dotted curves stand for TKGs samples; b: standard curve, TKGs were samples. |
基于2.1-2.3确立的实验方案与结果,进行4个培养时间点(1、4、8和12周)西藏开菲尔粒中微生物总基因组DNA的提取与细菌及酵母菌数量定量实验,测定总细菌与总酵母菌基因拷贝数。图4a所示为4个时间点西藏开菲尔粒中总细菌的数量变化统计图。尽管培养时间不断延长,但细菌的基因拷贝数稳定在大约每微升107 拷贝;培养1周与4周之间,8周与12周之间的细菌基因拷贝数不存在显著性差异(P﹤0.05)。图4b 为4个时间点西藏开菲尔粒中总酵母菌的数量变化统计图,酵母菌的数量稳定在大约每微升105 拷贝。尽管培养时间变化,但4个采样时间点酵母菌的数量相对稳定,无显著性差异(P﹤0.05)。
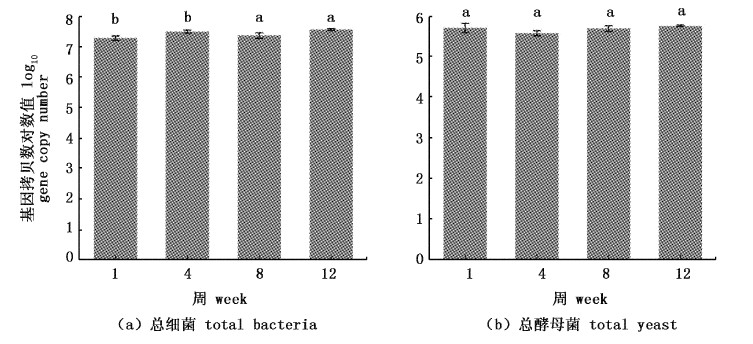
|
图 4
实时荧光定量PCR定量4个时间点(1、4、8和12周)总细菌与总酵母菌的基因拷贝数
Fig. 4
The gene copy numbers of bacteria and yeasts by real-time qPCR at four different time points
a: 总细菌基因拷贝数; b: 总酵母菌基因拷贝数;柱状图上的不同字母a、b表示显著性差异(P﹤0.05),标准偏差为误差线所示。 a: the gene copy number of bacteria; b: the gene copy number of yeasts; Differing letters within each subfigure (a,b) indicate statistically significant differences (P < 0.05). Standard deviations are shown by error bars. |
本文目的在于建立可靠的西藏开菲尔粒微生物总基因组DNA提取方法,并分析西藏开菲尔粒中细菌与酵母菌的数量变化规律。之前的研究表明,不同的培养方法和生长环境会导致开菲尔粒中的微生物组成存在差别[9, 22]。研究发现西藏开菲尔粒在大约相差10个月的培养时间内,不同种的酵母菌数量发生了很大变化,亚罗酵母菌由几乎零含量上升为第二大优势酵母菌,而克鲁为酵母菌的含量却大幅度下降[21]。酵母菌数量的变化是否会导致西藏开菲尔粒中细菌数量发生变化,两者之间是否存在一定的比例关系。要探索这些问题,首先需要建立高质量的西藏开菲尔粒微生物总基因组DNA 的提取方法。本文通过采用优化过的试剂盒实验方案(添加溶菌酶和溶壁酶同时处理样品的步骤)进行西藏开菲尔粒微生物总基因组提取,获得了较高质量的西藏开菲尔粒微生物总基因组DNA,且用这种方法提取的西藏开菲尔粒微生物总基因组DNA为模板进行实时荧光定量PCR分析,可以得到可信的总细菌与总酵母菌的基因拷贝数,进而为之后采用real-time qPCR技术来进行西藏开菲尔粒中微生物数量变化规律的研究奠定一定的基础,为探究西藏开菲尔粒中主要菌群细菌与酵母菌的变化规律,揭秘西藏开菲尔粒中微生物的共生机制提供了一种途径。
值得一提的是,针对实验室备有的两种基因组提取试剂盒给出的基因组DNA提取方案,在未做优化情况下进行西藏开菲尔粒中微生物基因组DNA的提取预实验,结果均不理想。但在样品前处理部分添加溶菌酶和溶壁酶来同时处理样品后,再用两种试剂盒来提取西藏开菲尔粒中总微生物基因组DNA都可以成功,但天根海洋动物组织基因组提取试剂盒较另外一种试剂盒提取到的基因组质量优且含量高。此外,天根试剂盒在价格方面也较另外一种试剂盒便宜很多,相对而言节约了实验成本。因此对提取西藏开菲尔粒中总微生物基因组DNA而言,天根试剂盒可以更好地满足我们的实验需求。
西藏开菲尔粒在培养12周期间,酵母菌总基因拷贝数相对稳定,不存在显著性差异(P﹤0.05),这一结果与LU等[21]对培养10个月前后的西藏开菲尔粒中酵母菌总含量稳定性的研究结果一致。对总细菌数量而言,培养1周与4周的西藏开菲尔粒样品中细菌基因拷贝数之间无显著性差异,8周与12周期间细菌基因拷贝数之间无显著性差异,培养1周到12周期间总细菌基因拷贝数每微升略上升至3.1×107拷贝。这些研究结果表明,西藏开菲尔粒中微生物(细菌与酵母菌)菌群数量相对稳定。有趣的是,尽管总酵母菌数量相对稳定,但其种属组成随着培养时间不同会发生改变[21]。这些特点或许是西藏开菲尔粒在长期的进化中为了保持其结构稳定而对环境选择性压力作出的一种适应性调节机制。
| [1] | VINDEROLA C G, DUARTE J, THANGAVEL D, et al. Immunomodulating capacity of kefir[J]. Journal of Dairy Research, 2005, 72(2): 195-202. |
| [2] | URDANETA E, BARRENETXE J, ARANGUREN P, et al. Intestinal beneficial effects of kefir-supplemented diet in rats[J]. Nutrition Research, 2007, 27(10): 653-658. |
| [3] | DINIZ R O, GARLA L K, SCHNEEDORF J M, et al. Study of anti-inflammatory activity of Tibetan mushroom, a symbiotic culture of bacteria and fungi encapsulated into a polysaccharide matrix[J]. Pharmacological Research, 2003, 47(1): 49-52. |
| [4] | RODRIGUES K L, CAPUTO L R G, CARVALHO J C T, et al. Antimicrobial and healing activity of kefir and kefiran extract[J]. International Journal of Antimicrobial Agents, 2005, 25(5): 404-408. |
| [5] | AHMED Z, WANG Y P, AHMAD A, et al. Kefir and health: a contemporary perspective[J]. Critical Reviews in Food Science and Nutrition, 2013, 53(5): 422-434. |
| [6] | 周剑忠, 董明盛, 江汉湖. PCR-DGGE指纹技术与分离技术结合筛选藏灵菇奶发酵过程的优势菌[J]. 中国农业科学, 2006, 39(8): 1632-1638. ZHOU J Z, DONG M S, JIANG H H. Screen of dominating microbial species isolated from Tibetan kefir using an integrated approach of PCR-DGGE and culture-dependent methods[J]. Scientia Agricultura Sinica, 2006, 39(8): 1632-1638. |
| [7] | 李剑, 周剑忠, 孙宇辉, 等. 藏灵菇中有益微生物的分离与鉴定[J]. 江苏农业科学, 2009, (1): 242-244. LI J, ZHOU J Z, SUN Y H, et al. Isolation and identification of valuable microorganism from Tibetan kefir grain [J]. Jiangsu Agricultural Sciences, 2009, (1): 242-244. |
| [8] | 杨希娟, 樊明涛, 师俊玲, 等. 西藏灵菇发酵乳中优势菌群的分离鉴定[J]. 中国酿造, 2007(6): 52-55. YANG X J, FAN M T, SHI J L, et al. Isolation and identification of preponderant flora in Tibetan fermented linggu milk[J]. China Brewing, 2007(6): 52-55. |
| [9] | GAO J, GU F Y, ABDELLA N H, et al. Investigation on culturable microflora in Tibetan kefir grains from different areas of China[J]. Journal of Food Science, 2012, 77(8): M425-M433. |
| [10] | 卢曼, 陈小兵, 王兴兴, 等. 西藏开菲尔粒中酵母菌的分离与初步鉴定[J]. 上海海洋大学学报, 2015, 24(1): 138-145. LU M, CHEN X B, WANG X X, et al. Isolation and characterization of yeasts in Tibetan kefir grains[J]. Journal of Shanghai Ocean University, 2015, 24(1): 138-145. |
| [11] | GAO J, GU F Y, HE J, et al. Metagenome analysis of bacterial diversity in Tibetan kefir grains[J]. European Food Research and Technology, 2013, 236(3): 549-556. |
| [12] | IMAMOVIC L, BALLESTE E, JOFRE J, et al. Quantification of Shiga toxin-converting bacteriophages in wastewater and in fecal samples by real-time quantitative PCR[J]. Applied and Environmental Microbiology, 2010, 76(17): 5693-5701. |
| [13] | SHANKS O C, KELTY C A, PEED L, et al. Age-related shifts in the density and distribution of genetic marker water quality indicators in cow and calf feces[J]. Applied and Environmental Microbiology, 2014, 80(5): 1588-1594. |
| [14] | GAMBAROTTA G, RONCHI G, FRIARD O, et al. Identification and validation of suitable housekeeping genes for normalizing quantitative real-time PCR assays in injured peripheral nerves[J]. PLoS One, 2014, 9(8): e105601. |
| [15] | LÓPEZ-ENRÍQUEZ L, RODRÍGUEZ-LÁZARO D, HERNÁNDEZ M. Quantitative detection of Clostridium tyrobutyricum in milk by real-time PCR[J]. Applied and Environmental Microbiology, 2007, 73(11): 3747-3751. |
| [16] | ZHAO Y, KONG C C, CUI X L, et al. Detection of infectious laryngotracheitis virus by real-time PCR in naturally and experimentally infected chickens[J]. PLoS One, 2013, 8(6): e67598. |
| [17] | LANE D J. 16S/23S rRNA sequencing[M]. Nucleic Acid Techniques in Bacterial Systematics, 1991: 125-175. |
| [18] | KURTZMAN C P, ROBNETT C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences[J]. Antonie van Leeuwenhoek, 1998, 73(4): 331-371. |
| [19] | PARK E J, CHANG H W, KIM K H, et al. Application of quantitative real-time PCR for enumeration of total bacterial, archaeal, and yeast populations in kimchi[J]. The Journal of Microbiology, 2009, 47(6): 682-685. |
| [20] | 孙国伟, 肖劲洲, 陈小兵, 等. 洋山深水港海域表层海水中古菌多样性特点[J]. 微生物学杂志, 2014, 34(5): 13-20. SUN G W, XIAO J Z, CHEN X B, et al. Archaeal diversity in surface seawater from the Yangshan Deep-Water Port[J]. Journal of Microbiology, 2014, 34(5):13-20. |
| [21] | LU M, WANG X X, SUN G W, et al. Fine structure of Tibetan kefir grains and their yeast distribution, diversity, and shift[J]. PLoS One, 2014, 9(6): e101387. |
| [22] | KÖK-TAS T, SEYDIM A C, ÖZER B, et al. Effects of different fermentation parameters on quality characteristics of kefir[J]. Journal of Dairy Science, 2013, 96(2): 780-789. |
2. Shanghai Engineering Research Center of Aquatic-Product Processing & Preservation, Shanghai 201306, China;
3. Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation, Ministry of Agriculture, Shanghai 201306, China
 2015, Vol. 24
2015, Vol. 24


