鳊鲂鱼类隶属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)、鲌亚科,归属鳊属(Parabramis)和鲂属(Megalobrama)。鳊鲂鱼类分为5个种,即鲂属的三角鲂(M. terminalis)、广东鲂(M. hoffmanni)、厚颌鲂(M. pellegrini)和团头鲂(M. amblycephala),以及鳊属的长春鳊(P. pekinensis)。鳊鲂鱼类是我国广大淡水水域的重要经济鱼类资源,尤其是团头鲂在淡水养殖业中养殖比例较大[1]。
迄今为止,鱼类等生物的遗传育种主要是发现突变体,并通过选育和杂交的方法获得良种。这些水产良种的育成,从本质而言,均是利用自然界已存在的携带优良性状基因的突变体,在此基础上加以选择、固定,从而建立优良品系,因此,优良性状突变体的获得是实现育种目标的关键[2, 3, 4]。
本实验室首次在自然界中发现了5尾红色的鳊鲂鱼类突变体,其外形似三角鲂和团头鲂。为鉴定其到底为何种鳊鲂鱼类,本研究分别从外部形态框架、微卫星标记及线粒体基因[5, 6, 7, 8, 9, 10]聚类关系对其进行了鉴别,鳊鲂红色突变体的发现将提供宝贵的遗传中间材料,有助于相关性状的主控基因的筛选以及遗传育种研究。
1 材料与方法 1.1 实验材料5尾红色鳊鱼(雌鱼3尾,雄鱼2尾,2龄)采自浙江省金华市武义县王宅镇东升村的山塘水库,属于钱塘江水系,是传统的三角鲂栖息地;实验所用的5个鲂属鱼类群体(广东东江三角鲂、浙江钱塘江三角鲂、广东鲂、团头鲂、厚颌鲂)和1个鳊属鱼类群体(长春鳊),保存于上海海洋大学农业部团头鲂遗传育种中心[11],均为一周龄。
1.2 实验方法 1.2.1 形态学参数的测量测量记录红色团头鲂突变体的可数、可量性状。可数性状有侧线鳞数、侧线上鳞数、侧线下鳞数、背鳍条数、胸鳍条数、腹鳍条数和臀鳍条数,共7项。测量的可量性状有全长、体长、体高、头长、吻长、眼径、眼间距、尾柄长、尾柄高和背棘长,并转化为相应比例参数,有全长/体长、体长/体高、体长/头长、头长/吻长、头长/眼径、头长/眼间距、体长/尾柄长、尾柄长/尾柄高、背棘长/体长,共9项。框架结构数据的测定参照图1[12],有D1-2、D1-3、D3-5、D5-7、D7-9、D2-4、D4-6、D6-8、D8-10、D4-1、D4-3、D4-5、D6-3、D6-5、D6-7、D8-5、D8-7、D8-9、D10-7、D10-9,共20项。20项框架结构数据转化为与全长的比例参数以减少鱼体大小引起的统计误差。数据采用两脚规配合直尺测量,精确到0.1 mm。

|
图1
鳊鲂鱼类框架结构示意图
Fig.1
Truss network of distance measures assessed in bream species
10 个定位点之间的距离为20 个框架数据。1. 胸鳍起点; 2. 吻前端; 3. 腹鳍起点; 4. 头背部末端; 5. 臀鳍起点; 6. 背鳍起点; 7. 臀鳍末端; 8. 背鳍末端; 9. 尾鳍腹部起点; 10. 尾鳍背部起点。 20 truss parameter measurements are the distances between the two of 10 landmark points. 1. Origin of operculum fin; 2. Tip of snout; 3. Origin of pelvic fin; 4. Terminus of head back; 5. Origin of anal fin; 6. Origin of dorsal fin; 7. Terminus of caudal fin; 8. Terminus of dorsal fin; 9. Ventral origin of caudal fin; 10. Dorsal origin of caudal fin. |
基于9项传统形态学可量比例参数和20项框架结构比例参数,使用SPSS 13.0软件对数据进行主成分分析,对突变体和这6类鳊鲂属群体间形态学差异进行评估。在进行主成分分析之前,数据已经过旋转以提高正态分布性,鱼体大小的影响也通过使用可量比例参数和框架比例参数得到最大程度的减小[11]。主成分贡献率和累积贡献率的计算参照文献[13, 14]。
1.2.3 基因组DNA的提取参照北京天根生物科技有限公司的海洋动物组织基因组DNA 提取试剂盒(离心柱型) 说明书介绍的方法提取样品基因组DNA。基因组DNA 提取完成后,用0.8%的琼脂糖凝胶电泳和分光光度计检测DNA 质量和浓度,-20 ℃保存备用。
1.2.4 微卫星和线粒体基因扩增引物及PCR扩增微卫星引物:其中TTF2、TTF3和TTF10这3对引物来自TANG等[15]利用5′锚定PCR技术开发的团头鲂微卫星引物,Mam03引物来自LI等[16]利用磁珠富集得到的SSR引物,引物EST37 和EST66来自GAO等[17]经转录组测序分析得到的EST-SSR引物。线粒体COX1和COX2基因的引物以团头鲂线粒体基因(gi|166897644|gb|EU434747.1|)为参照使用Primer 5软件设计。所有引物均由上海生工生物工程股份有限公司合成。微卫星及线粒体基因扩增引物信息见表1。
| 表1 微卫星及线粒体COX1和COX2基因扩增引物 Tab 1. Microsatellite and mitochondrial COX1 and COX2 gene amplification primers |
微卫星反应体系为10 μL,包含5 μL 含染料的2 × Taq PCR Master Mix (Taq DNA Polymerase: 0.1 U /μL; MgCl2: 4 mmol /L; dNTPs each: 0.4 mmol /L) ,上下游引物各0.5 μL(10 μmol /L),0.5 μL 模板DNA(30~50 ng),3.5 μL ddH2O。PCR反应在Eppendorf Mastercycler ep gradients 型PCR仪上进行,反应程序为94 ℃预变性5 min,94 ℃ 30 s,50~65 ℃(根据表1中各引物的退火温度进行调整) 30 s,72 ℃ 30 s,30个循环,最后72 ℃延伸10 min。
线粒体COX1和COX2基因PCR的反应体系为50 μL,包含25 μL 含染料的2 ×Taq PCR Master Mix (Taq DNA Polymerase: 0.1 U /μL; MgCl2: 4 mmol /L; dNTPs each: 0.4 mmol /L) ,上下游引物各2 μL(10 μmol /L),2 μL 模板DNA(30~50 ng),19 μL ddH2O。反应程序为94 ℃预变性5 min,94 ℃ 30 s,55 ℃ 1 min,72 ℃ 30 s,35个循环,最后72 ℃延伸10 min。线粒体COX1和COX2基因的PCR产物送上海生工生物工程股份有限公司测序。
1.2.5 微卫星PCR产物凝胶电泳检测微卫星PCR产物在8%的非变性聚丙烯酰胺凝胶上进行电泳,胶片大小为195 mm(长)×120 mm(宽)×1 mm(厚)。产物上样量均为1 μL,DNA Marker (pBR322 DNA /MspⅠ)上样量为0.5 μL。电泳条件: 电泳缓冲液为1×TBE,电压200 V,电泳1.5~2 h (具体时间根据PCR产物分子量大小而定)。电泳完成后,进行硝酸银染色,然后显色。最后将胶片平铺于观片灯箱上,用数码相机照相并存入电脑。用Popgene(Version 1.32)软件计算6种鳊鲂鱼及红色鳊鱼间微卫星位点的遗传距离,并使用MEGA 5软件构件UPGMA聚类关系图。
1.2.6 红色突变体与相关鱼类的聚类分析将5尾红色鳊鱼突变体的线粒体COX1和COX2基因序列,与NCBI中搜索得到的相关鱼类线粒体序列进行聚类分析。鱼类相关序列的GenBank登录号分别为:三角鲂(gi|407954722|dbj| AB626850.1|);团头鲂(gi|166897644|gb|EU434747.1|);长春鳊(gi|399157651|gb|J X242531.1|);大眼华鳊(gi|429125639|gb|KC122916.1| Sinibrama macrops);翘嘴红鲌(gi|269996108|gb|GU190362.1| Culter alburnus);蒙古鲌(gi|485474638|gb|KC701385.1| Chanodichthys mongolicus);鲤(gi|391223878|ref| NC_018035.1|Cyprinus carpio);鲫(gi|5835778|ref|NC_ 002079.1| Carassius auratus);斑马鱼(gi|15079186|ref|NC_002333.2| Danio rerio)。聚类关系图使用MEGA 5软件进行分析。
2 结果与分析 2.1 红色鳊鱼突变体与鳊鲂鱼类可数性状比较红色鳊鱼突变体(图2),侧线鳞数、侧线上鳞数、侧线下鳞数、背鳍条数、胸鳍条数、腹鳍条数、臀鳍条数7项可数性状的结果如表2所示。通过与文献[11]所测的东江三角鲂、钱塘江三角鲂、广东鲂、团头鲂、厚颌鲂和长春鳊6群体数据比较,发现红色鳊鱼突变体的侧线鳞数、侧线上鳞数、侧线下鳞数、腹鳍条数差异不显著(P>0.05)。然而,红色鳊鱼突变体胸鳍条数为14,比实验室所测6群体鳊鲂鱼类的16~19少;背鳍和臀鳍棘数均为2,而所测6群体均为3,有明显差异。

|
图2 红色鳊鱼突变体(上)与野生型团头鲂(下) Fig.2 Red bream mutant (upper) and wild-type bream (lower) |
| 表2 红色鳊鱼突变体的可数性状均值和标准差 Tab 2. Mean and standard deviation of meristic characters of red bream mutant |
实验通过对5尾红色鳊鱼突变体11项可量性状的测量,得出其9项传统形态可量比例参数(表3)。
| 表3 红色鳊鱼突变体9项传统形态学可量比例参数 Tab 3. Nine traditional morphological parameters of red bream mutants |
基于这9项传统形态学可量比例参数和所测20项框架结构比例参数,按陈杰等[11]构建的6群体鳊鲂属鱼类的判别函数,与这6群体鳊鲂鱼类[11]做主成分分析,得出主成分散点图(图3)。从图中可看出红色鳊鱼突变体在形态上与团头鲂较近,形态差异较小。

|
图3 红色鳊鱼突变体与6个鳊鲂属鱼类主成分分析 Fig.3 Principal component analysis between red bream mutants and other six breams |
用6对微卫星引物EST37、Mam03、TTF2、TTF10、TTF3和EST66分别对6种鳊鲂鱼(东江三角鲂、长春鳊、厚颌鲂、广东鲂、钱塘江三角鲂、团头鲂)与红色鳊鱼突变体进行微卫星鉴别,其中,EST37、Mam03引物显示5尾红色鳊鱼突变体与团头鲂的微卫星指纹图谱更为接近(图4)。
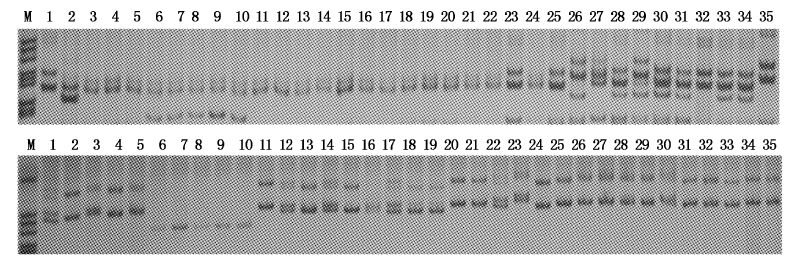
|
图4
红色鳊鱼突变体EST37(上图)和Mam03(下图)微卫星产物电泳图
Fig.4
Microsatellite electrophoresis analysis of red bream mutant by EST37 (upper) and Mam03 (lower) primers
M. PBR 322 DNA /MspⅠ标准分子量; 1~5.东江三角鲂; 6~10.长春鳊; 11~15.厚颌鲂; 16~20.广东鲂; 21~25.钱塘江三角鲂; 26~30.红色突变体; 31~35.团头鲂。 M. PBR 322 DNA /MspⅠladder; 1~5. M. terminalis from Dong River; 6~10. P. pekinensis; 11~15. M. pellegrini; 16~20. M. hoffmanni; 21~25. M. terminalis from Qiantang River; 26~30. red bream mutants; 31~35. M. amblycephala. |
用Popgene(Version 1.32)软件计算的Nei’s遗传距离如表4所示,红色鳊鱼突变体与6种鳊鲂群体间的遗传距离为0.309 4~3.743 9。根据群体间的遗传距离,使用MEGA 5软件构建UPGMA聚类关系图(图5),结果显示红色突变体与团头鲂聚为一类,然后与钱塘江三角鲂聚为一类。红色突变体与团头鲂遗传距离最近,为0.309 4,与长春鳊的遗传距离最远,为3.743 9。
| 表4 红色鳊鱼突变体与6群体鳊鲂鱼类的欧氏遗传距离 Tab 4. Nei’s genetic distance of red bream mutant and the six genera Megalobrama and Parabramis |

|
图5 红色鳊鱼突变体与6类鳊鲂鱼UPGMA聚类图 Fig.5 Dendrogram of relationships among the red bream mutant and the six genera Megalobrama and Parabramis using UPGMA method of clustering |
将红色鳊鱼突变体的线粒体COX1-COX2合并序列与其他鱼类相应序列进行比对,结果显示5尾红色鳊鱼突变体间的核苷酸序列相似度大于99%;它们与团头鲂、三角鲂、长春鳊、大眼华鳊、翘嘴鲌、蒙古鲌、鲫、鲤和斑马鱼的遗传相似度分别为99%、95%、95%、94%、94%、93%、86%、86%和82%,表明红色鳊鱼突变体与团头鲂间存在极近的亲缘关系。NJ聚类分析也显示5尾红色鳊鱼突变个体聚为一类,然后再与团头鲂聚为一类(图6),这些结果说明红色鳊鱼可能属于团头鲂的红色突变体。

|
图6 红色鳊鱼突变体与亲缘相近鱼类线粒体COX1和COX2基因聚类关系 Fig.6 Clustering relationships between red bream mutants and close relatives by mitochondrial COX1 and COX2 genes |
鳊鲂鱼种间的传统形态性状存在一定的差异,可用以突变体分类鉴别[5, 7, 11, 18]。本研究对红色鳊鱼突变体的可量数据进行了主成分分析,发现红色鳊鱼突变体与团头鲂处于同一象限,形态可量性状较为较近。在形态学可数性状中,红色鳊鱼突变体胸鳍条数为16、背鳍棘数和臀鳍棘数均为2,与文献[11, 19]报道的鳊鲂鱼类的背鳍棘数和臀鳍棘数(均为3)存在明显差异。但在其他文献中也曾发现团头鲂的背鳍棘数和臀鳍棘数为2,如在梁子湖团头鲂中就发现存在背鳍棘数为2,臀鳍棘数为3的类型[20],另外,在伊河团头鲂中也发现背鳍棘数为2的类型[21],造成这些团头鲂可数形态性状差异的原因不清楚,在大多数形态学参数上红色鳊鱼突变体与鳊鲂鱼类是一致的。
微卫星分析发现,5尾红色鳊鱼突变体与团头鲂的EST37、Mam03微卫星指纹图谱较为接近,其与三角鲂的遗传距离大于与团头鲂的遗传距离。聚类结果同样显示红色鳊鱼突变体先与团头鲂聚为一类,然后再与钱塘江三角鲂聚为一类。线粒体基因序列显示中间种内遗传差异现已广泛用于鱼类种类鉴定和聚类分析[9, 22, 23]。通过进一步测定红色鳊鱼突变体的线粒体COX1和COX2基因序列,发现5尾红色鳊鱼突变体与团头鲂聚为一类,它们与团头鲂的相似度高达99%,这些结果说明红色鳊鱼突变体应归属团头鲂,而非三角鲂。
鉴于红色鳊鱼突变体的线粒体基因序列与团头鲂高度相似,可排除为与其他鲤科鱼类的杂交种类,应该是体色自发突变的结果。鱼类所表现出的各种体色是由体内色素细胞(黑色素细胞、虹彩细胞、红色素细胞、黄色素细胞和白色素细胞等)所形成[24]。不同鱼类的色素细胞组成不同,叶元土等[25]在一般光学显微镜下观察到养殖团头鲂鳞片色素细胞含黑色素细胞和黄色素细胞,且发现以黑色体色为主的养殖鱼类体色变浅、发白的直接原因来源于黑色素细胞。黑色素细胞可分泌产生黑色和棕色表型的真黑色素(eumelanin)和产生黄色和红色表型的褐黑色素(pheomelanin),且这两种色素产生于共同的遗传信号通路[26],都依赖于酪氨酸酶基因的作用[27]。黑色素主控基因如酪氨酸酶基因的突变可能是团头鲂红色突变体产生的根本原因[28],这有待进一步研究。
| [1] | 李思发.中国淡水主要养殖鱼类的种质研究[M]. 上海: 上海科学技术出版社, 1996.LI S F. Genetic study on main freshwater aquaculture fish species in China [M]. Shanghai: Shanghai Scientific & Technical Press,1996. |
| [2] | YU X. Bulletin of Minstry of Agriculture: aquacultural new breeds[J]. Journal of Modern Fisheries Information, 2011, 8: 30. |
| [3] | FUJI K, KOBAYASHI K, HASEGAWA O, et al. Identification of a single major genetic locus controlling the resistance to lymphocystis disease in Japanese flounder (Paralichthys olivaceus) [J]. Aquaculture, 2006, 254(1/4): 203-210. |
| [4] | FUJI K, HASEGAWA O, HONDA K, et al. Marker-assisted breeding of a lymphocystis disease-resistant Japanese flounder (Paralichthys olivaceus) [J]. Aquaculture, 2007, 272(1/4): 291-295. |
| [5] | 李思发, 朱泽闻, 邹曙明, 等. 鲂属团头鲂、三角鲂及广东鲂种间遗传关系及种内遗传差异[J]. 动物学报, 2002, 48(3): 339-345.LI S F, ZHU Z W, ZOU S M, et al. Interspecific phylogensis and intraspecific genetic differences of genus megalobrama: bluntnose black bream(M.amblycephala),guangong blackream(M. hoffmanni) and black bream(M. terminalis) [J]. Acta Zoologica Sinica, 2002, 48(3): 339-345. |
| [6] | 宋文, 王艺舟, 祝东梅, 等. 三个地理种群鲂的形态差异与判别分析[J]. 水产学杂志, 2013, 26(4): 1-7.SONG W, WANG Y Z, ZHU D M, et al. Morphological variation and discriminant analysis of three geographical populations of freshwater bream Megalobrama skolkovii [J]. Chinese Journal of Fisheries, 2013, 26(4): 1-7. |
| [7] | 李绍戊, 常玉梅, 梁利群, 等. 团头鲂微卫星标记的快速制备[J]. 中国水产科学, 2006, 13(2): 187-192.LI S W, CHANG Y M, LIANG L Q, et al. Rapid isolation of microsatellites from genome of Bluntnose black bream (Megalobrama amblycephala) [J]. Journal of Fishery Sciences of China, 2006, 13(2): 187-192. |
| [8] | 易少奎, 高泽霞, 罗伟, 等. 团头鲂EST-SSR在厚颌鲂中的跨种扩增及杂交F1的鉴定[J]. 水产学报, 2013, 37(7): 970-977.YI S K, GAO Z X, LUO W, et al. Cross-species amplification of Megalobrama amblycephala EST-SSR in M.pellegrini and identification of their reciprocal hybrids [J]. Journal of Fisheries of China, 2013, 37(7): 970-977. |
| [9] | 张倩倩, 陈杰, 蒋霞云, 等. 不同鳊鲂鱼类群体微卫星DNA指纹图谱的构建和遗传结构分析[J]. 水产学报, 2014, 38(1): 15-22.ZHANG Q Q, CHEN J, JIANG X Y, et al. Establishment of DNA fingerprinting and analysis on genetic structure of different Parabramis and Megalobrama populations with microsatellite [J]. Journal of Fisheries of China, 2014, 38(1): 15-22. |
| [10] | 赖瑞芳, 张秀杰, 李艳和, 等. 鲂属鱼类线粒体基因组的比较及其系统发育分析[J]. 水产学报, 2014, 38(1): 1-14.LAI R F, ZHANG X J, LI Y H, et al. Comparison of mitochondrial genomes of the genus Megalobrama and their phylogenetic analysis [J]. Journal of Fisheries of China, 2014, 38(1): 1-14. |
| [11] | 陈杰, 李福贵, 黄创新, 等. 不同鳊鲂属鱼类群体的形态差异分析[J]. 上海海洋大学学报, 2014, 23(3):388-394.CHEN J, LI F G, HUANG C X, et al. Morphological variations of genera Parabramis and Megalobrama teleost populations [J]. Journal of Shanghai Ocean University, 2014, 23(3): 388-394. |
| [12] | 李思发, 蔡完其, 周碧云. 团头鲂种群间的形态差异和生化遗传差异[J]. 水产学报, 1991, 15(3): 204-211.LI S F, CAI W Q, ZHOU B Y. Morphological and biochemical genetic variations among populations of blunt snout bream (Megalobrama amblycephala) [J]. Journal of Fisheries of China, 1991, 15(3): 204-211. |
| [13] | DEBUSE V J, ADDISION J T, REYNOLDS J D. Morphometric variability in UK populations of the European lobster [J]. Journal of the Marine Biological Association of the United Kingdom, 2001, 81(2): 469-474. |
| [14] | 张尧庭, 方开泰. 多元统计分析引论[M]. 北京:科学出版社, 1999:393-454.ZHANG Y T, FANG K T. Introduction to multivariate statistical analysis [M]. Beijing:Science Press, 1999: 393-454. |
| [15] | TANG S J, LI S F, CAI W Q. Development of microsatellite markers for blunt snout bream Megalobrama amblycephala using 5'-anchored PCR [J]. Molecular Ecology Resources, 2009, 9(3): 971-974. |
| [16] | LI W T, LIAO X L, YU X M, et al. Isolation and characterization of polymorphic microsatellite loci in Wuchang bream (Megalobrama amblycephala) [J]. Molecular Ecology Notes, 2007, 7(5): 771-773. |
| [17] | GAO Z X, LUO W, LIU H, et al. Transcriptome analysis and SSR/SNP markers information of the blunt snout bream (Megalobrama amblycephala) [J]. PLoS One, 2012, 7(8): e42637. |
| [18] | 曾聪, 阎里清, 高泽霞, 等. 梁子湖、鄱阳湖和淤泥湖团头鲂的形态学比较[J]. 华中农业大学学报, 2012, 31(1): 88-94.ZENG C, YAN L Q, GAO Z X, et al. Comparison of mophology parameters of blunt snout bream (Megalobrama amblycephala Yih) among Liangzi Lake, Poyang Lake and Yuni Lake [J]. Journal of Huazhong Agricultural University, 2012, 31(1): 88-94. |
| [19] | 宋文, 王艺舟, 祝东梅, 等. 鲂属鱼类形态差异分析[J]. 淡水渔业, 2013, 43(3): 21-27.SONG W, WANG Y Z, ZHU D M, et al. Morphological variations among the genus Megalobrama [J]. Freshwater Fisheries, 2013, 43(3): 21-27. |
| [20] | 曹文宣.梁子湖的团头鲂与三角鲂[J]. 水生生物学集刊, 1960(1): 57-77.CAO W X. Megalobrama amblycephala and Megalobrama terminalis in Liangzi Lake [J]. Acta Hydrobiologica Sinica, 1960(1): 57-77. |
| [21] | 屈长义, 冯建新, 张芹, 等. 伊河团头鲂主要形态学性状研究[J]. 河南农业, 2013(1):47-48.QU C Y, FENG J X, ZHANG Q, et al. Studying the main morphological characters of Megalobrama amblycephala in Yihe [J]. Henan Nongye, 2013(1):47-48. |
| [22] | 黄丽英, 丁诗华, 张海琪, 等. 利用Cytb基因序列研究翘嘴红鲌的系统发生[J]. 西南农业大学学报:自然科学版, 2005, 27(6): 909-913.HUANG L Y, DING S H, ZHANG H Q, et al. Phylogenetic relationship of erythrocul terilishaeformis with other four abramidinae species based on cytochrome b gene sequences[J]. Journal of Southwest Agricultural University:Natural Science, 2005, 27(6): 909-913. |
| [23] | 唐首杰, 李思发, 蔡完其. 团头鲂野生、驯养、选育3 类遗传生态群体遗传变异的线粒体DNA分析[J]. 中国水产科学, 2011, 18(3): 483-492.TANG S J, LI S F, CAI W Q. Mitochondrial DNA analysis on genetic variation of wild, domesticated, and genetically selected populations of blunt snout bream (Megalobrama amblycephala) [J]. Journal of Fishery Sciences of China, 2011, 18(3): 483-492. |
| [24] | FUJI R.Cytophysiology of fish chromatophores [J]. International Review of Cytology, 1993, 143: 191,255. |
| [25] | 叶元土, 郭建林, 萧培珍, 等. 养殖武昌鱼体色与鳞片黑色素细胞初步观察[J]. 饲料工业, 2006, 27(22): 25-27.YE Y T, GUO J L, XIAO P Z, et al. Preliminary observation of Wuchang bream body color and flake melanocytes [J]. Feed Industry, 2006, 27(22): 25-27. |
| [26] | HOEKSTRA H E.Genetics, development and evolution of adaptive pigmentation in vertebrates [J]. Heredity, 2006,97(3): 222-234. |
| [27] | LIN J Y, FISHER D E.Melanocyte biology and skin pigmentation [J]. Nature, 2007, 445(7130): 843-850. |
| [28] | 黄永政. 鱼类体色研究进展[J]. 水产学杂志, 2008, 21(1): 89-94.HUANG Y Z. Advances in fish body color [J]. Chinese Journal of Fisheries, 2008, 21(1): 89-94. |
 2015, Vol. 24
2015, Vol. 24


