2. 上海水产品加工及贮藏工程技术研究中心, 上海 201306;
3. 农业部水产品贮藏保鲜质量安全风险评估实验室, 上海 201306
西藏开菲尔粒(Tibetan kefir grains,TKGs),俗称“藏灵菇”,因源于西藏而得名,是目前已知可用于酸奶制作的唯一天然野生菌种资源。鉴于“藏灵菇”的命名易与食用菌混淆,且已证实其属于开菲尔粒的一个品系,因此,本文称其为西藏开菲尔粒。
西藏开菲尔粒主要是由多个直径2~9 mm、厚约200 μm的中空囊状结构集结而成的类似菜花状结构。该囊状结构培养至一定程度后,会再生出新的小囊状结构。西藏开菲尔粒微生物菌相组成复杂,尽管不同来源的开菲尔粒中微生物组成存在差异,但主要是乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)以及酿酒酵母属(Saccharomyces)和克鲁维酵母属(Kluyveromyces),并以乳酸菌为优势菌群。
近年来,西藏开菲尔粒中微生物群落组成及分布备受科研人员的关注,针对细菌多样性的研究较多[1, 2, 3],而对于西藏开菲尔粒中酵母菌的研究报道则相对较少[1, 2]。酵母菌作为西藏开菲尔粒中的第二大优势菌群,对于维持西藏开菲尔粒共生体的稳定性有关键作用,并且它对乳制品发酵和成熟过程中风味的形成也有重要的影响[1, 4]。此外,目前研究表明某些酵母菌还有益生保健作用[5]。因此,西藏开菲尔粒中酵母菌的研究对认识西藏开菲尔粒至关重要。
本研究采用扫描电镜观察西藏开菲尔粒中酵母菌的形态特征,并通过分离培养、26S rRNA基因测序与序列分析、荧光原位杂交(FISH)技术和方法,对西藏开菲尔粒中酵母菌进行了分离培养和初步鉴定,以期为研究西藏开菲尔粒中各菌系之间的共生机理奠定基础。
1 材料与方法 1.1 实验材料西藏开菲尔粒由本实验室保存。主要试剂包括冰冻切片包埋剂(莱卡)、DAPI(Vector Laboratories,Cat. H-1200)、硝酸纤维素滤膜(0.22 μm孔径,密理博,Cat. GTTP02500)、盖玻片(0.17 mm,25×25 mm)、载玻片(25×75 mm,于玻片盒中存放和保藏切片样品);无菌超纯水、福尔马林(37%~40%甲醛溶液,生工生物工程,Cat. F1912-500 mL)、无水乙醇(生工生物工程,Cat. ET0737)、Zeiss “Immersol” 518F无荧光镜油(卡尔蔡司,Cat. 444960-0000-000);包埋剂、封片剂(或者无色指甲油);酵母基因组提取试剂盒(天根,Cat. DP307)、2 ×Taq PCR MasterMix (天根,Cat. KT201-02)、PCR产物回收试剂盒(天根,Cat. DP201)。
YPD培养基:用于酵母菌的分离纯化。葡萄糖20 g,胰蛋白胨20 g,酵母粉10 g,琼脂20 g,去离子水1 000 mL,pH 6.6,121 ℃灭菌15 min。
1.2 实验器材实验器材包括:荧光显微镜(卡尔蔡司,Scope.A1)、临界点干燥器(日立,HCP-2),扫描电镜(日立,HCP-2)、PCR仪(艾本德,Mastercycler nexus GSX1)、冷冻切片机(莱卡,CM1950)、杂交仪(UVP,HL-2000)、微量分析玻璃换膜过滤器(密理博)、真空泵(密理博,220V/50Hz)、移液枪(艾本德,量程:1 mL,100 μL,20 μL,10 μL)、组织研磨器(天根,OSE-Y20)。
1.3 方法 1.3.1 样品前处理及准备工作将-20 ℃低温保存的西藏开菲尔粒按10%(w/v)接种量接入灭菌光明纯牛奶中,置于恒温培养箱中25 ℃静置培养24 h,重复活化3次后,重新接种培养24 h,过滤,用无菌水将西藏开菲尔粒冲洗至干净,再加入相应量的纯牛奶培养。
1.3.2 扫描电镜取10个西藏开菲尔粒,用无菌水清洗干净,之后将其分别置于2.5%的戊二醛中过夜固定(4 ℃)。固定好的样品用0.1 mol/L PBS清洗3次(15 min/次),之后依次过梯度乙醇30%、50%、70%、90%和100%,各15 min。然后4 ℃下用1%锇酸(OsO4)溶液固定1 h,重复上述PBS清洗操作后依次通过70%、80%、90%和100%的梯度乙醇(10 min/次),其中无水乙醇冲洗3次,其余分别1次。然后样品依次过乙酸异戊酯2次(15 min/次),最后用临界点干燥器干燥样品,干燥好的样品用双面胶固定在样品托上,并用喷金仪对样品进行喷金镀膜后即可用扫描电镜观察。
1.3.3 西藏开菲尔粒中酵母菌的分离培养取0.1 g无菌水洗净的西藏开菲尔粒,用组织研磨器研碎,加入1 000 μL无菌生理盐水,混匀,而后梯度稀释(10 μL研磨液+90 μL无菌生理盐水),分别获得10-1、10-2、10-3和10-4梯度稀释液。将每个梯度的研磨液分别涂布在YPD酵母菌选择培养基上,并在25 ℃下培养24~48 h。挑选白色、圆形或椭圆形典型菌落制成涂片简单染色镜检。观察到椭圆形、长形或卵形菌株后,将其在YPD培养基上划线纯化。将纯化的菌株先通过菌落形态进行初筛,选择代表菌株(16个菌落)用已灭菌的枪头挑取适量菌落至YPD液体培养基中进行过夜增菌培养,然后依据酵母菌基因组提取试剂盒进行酵母菌总基因组DNA的提取。
1.3.4 PCR扩增酵母26S rRNA基因以酵母菌基因组DNA为模板,NL-1 (5′-GCATATCAATAAGCGGAGGAAAAG-3′)和NL-4 (5′-GGTCCGTGTTTCAAGACGG-3′)[6]为引物,对酵母菌26S rRNA基因的D1/D2区进行扩增。50 μL的PCR反应体系包含25 μL的2×Taq PCR MasterMix,10 μmol/L引物各1 μL,1 μL(100 ng左右)模板基因组DNA。PCR扩增程序为:95 ℃变性5 min,然后进入35个循环:94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min;最后72 ℃下延伸10 min。根据PCR产物回收试剂盒,对PCR扩增产物进行纯化,纯化产物送去测序(上海生工生物工程股份有限公司)。
1.3.5 序列分析得到的26S rRNA基因的D1/D2区序列先进行嵌合体检测(http://decipher.cee.wisc. edu/FindChimeras.html),然后将序列上传到NCBI数据库进行BLAST比对,得到最相近序列的种属信息。依据相似度进行排序分类。相似度大于97%而小于99%,将其归于属的水平上;若相似度大于等于99%,则根据其最相似序列归在种的水平上,同时记录下每条序列的详细信息,并做统计分析。此外,下载BLAST比对结果中最相似的序列,运用BioEdit将其与测得的序列整理在一个FASTA文件中,而后使用MEGA 6 (http://www.megasoftware.net/) 进行ClustalW序列比对,之后采用Neighbor-Joining的建树方法构建系统发育树,并且进行100次重复的bootstrap分析来评估系统发育树的可信度。
1.3.6 荧光原位杂交法针对酿酒酵母属(5′-ACGAGATTCCCCTACCCA-3′),克鲁维酵母属(5′-CCAACCGCTAAAACTGAT-3′和5′-CAGAGCCACATTCCCGAG-3′)(未正式发表数据)和亚罗酵母属(5′-CACTCATTTCCTTCCC-3′)[7]的探针分别用FITC和CY3荧光染料标记。挑选经测序鉴定后的菌种为代表菌株(序列相似性达100%的菌株),用灭菌的枪头挑取适量菌落于YPD液体培养基中25 ℃培养16 h,而后梯度稀释(1 mL菌液+ 9 mL 0.1 mol/L PBS),获得10-1、10-2和10-3梯度稀释液。分别吸取980 μL各个梯度菌液加入20 μL经0.22 μm滤膜过滤的甲醛溶液,使甲醛的终浓度达到2% (V/V)。在4 ℃冰箱过夜固定后,取1 mL过滤至0.22 μm膜上。将载有菌体的滤膜依次通过70%、80%、90%和100%系列乙醇进行脱水。分别吸取27 μL杂交液(20%甲酰胺)和3 μL相应探针(浓度10 μmol/L)于适当大小的封口膜上混匀,将载有相应酵母菌的滤膜置于上述封口膜上,放入50 mL用锡箔纸避光的离心管,管内放有用杂交液(不含探针)润湿的吸水纸,于杂交炉内46 ℃杂交3 h。杂交完成后立即将滤膜转移至48 ℃洗涤液中,48 ℃下洗涤30 min。膜杂交中杂交液及洗涤液的配制参考之前的研究[8]。洗涤过后的滤膜再次通过去离子水、70%、80%、90%和100%梯度乙醇进行洗脱。最后用DAPI进行复染5 min,之后即可选取最适稀释度的菌液滤膜用荧光显微镜分别在DAPI、FITC、CY3光道下观察。
2 结果与分析 2.1 扫描电镜观察西藏开菲尔粒中的酵母菌应用扫描电镜观察西藏开菲尔粒中的酵母菌,结果如图版Ⅰ所示:酵母菌大多呈球形(2.0~5.0 μm)或椭圆形(1.0~3.0 μm× 2.0~4.0μm),主要位于西藏开菲尔粒的内外表面及其邻近区域,包埋或镶嵌在细菌群体中,但其数量远不及细菌。
2.2 西藏开菲尔粒中酵母菌的分离鉴定对分离培养后获得的菌株,首先根据其菌落形态特点进行初筛,可分为3类,如图版Ⅱ所示。第一类菌落(图版Ⅱ-1)呈圆形、表面光滑、低凸起、边缘整齐、乳白色;第二类菌落(图版Ⅱ-2)呈圆形、较大、暗乳白色、高突起,表面略粗糙;第三类菌落(图版Ⅱ-3)圆形、较大、乳白色、低突起,表面粗糙且有褶皱。

|
图版Ⅰ
西藏开菲尔粒扫描电镜观察
PlateⅠ
Scanning electron micrographs of TKGs
1、2.西藏开菲尔粒内表面; 3、4.西藏开菲尔粒外表面; 箭头指示酵母菌。 1,2. The inner surface of TKGs; 3,4. The outer surface of TKGs; The arrows indicate the representative yeasts in TKGs. |

|
图版Ⅱ 西藏开菲尔粒中分离培养的酵母菌菌落特点 PlateⅡ Colony features of isolated yeasts from TKGs |

|
图版Ⅲ
FISH分析3种酵母菌
PlateⅢ
FISH analysis of three cultured representative strains of Kluyveromyces,Saccharomyces and Yarrowia
1、3、5. DAPI染色; 2.酿酒酵母属(Saccharomyces)特异探针杂交; 4.克鲁维酵母属(Kluyveromyces)特异探针杂交; 6.亚罗酵母属(Yarrowia)特异探针杂交; 其他是相应重叠的图片。 1,3,5. DAPI staining; 2.Saccharomyces hybridized with the probe Sacch; 4.Kluyveromyces hybridized with the probes Kluyv 1 and 2; 6.Yarrowia hybridized with the probe Ylip. Others represent merged photographs. |
随机从第一类菌落中选出5个,第二类中选出8个,第三类中选出3个,共16株酵母菌进行26S rRNA基因测序,序列相似性比对分析如表 1。所有序列与GenBank中最相近序列的相似度为97%~100%,根据方法中提到的分类鉴定标准将其归为3个属:酿酒酵母属(Saccharomyces)、克鲁维酵母属(Kluyveromyces)、亚罗酵母属(Yarrowia)。与菌落分类结果一致,它们所代表的酵母菌属分别对应着上述的第一、二、三类菌落(表 1)。在种的水平上,16株中有14株可分别归为酿酒酵母菌(Saccharomyces cerevisiae)、马克斯克鲁维酵母菌(Kluyveromyces marxianus)和解脂亚罗酵母菌(Yarrowia lipolytica)。这表明,西藏开菲尔粒中可分离培养的酵母菌多样性相对较低。
| 表 1 26S rRNA序列比对分析 Tab.1 Sequence analysis of 26S rRNA genes |
系统发育分析26S rRNA基因序列结果如图 1所示。16株酵母菌归为3个属和4个种,这与上述序列相似性分析结果一致,进一步证实了酵母菌分类地位的可靠性。
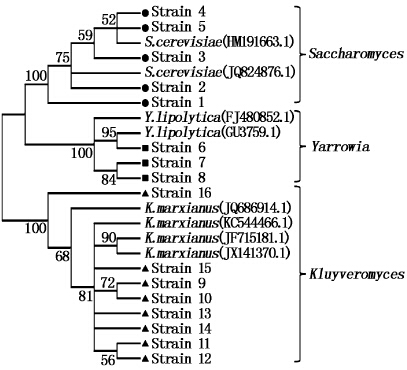
|
图 1
26S rRNA基因序列的系统发育分析
Fig. 1
Phylogenetic affiliations of yeast 26S rRNA genes
本研究中的序列用圆形、黑色方块以及三角形标记;大于50的Bootstrap值显示在系统发育树的各节点处;酿酒酵母属(Saccharomyces)、亚罗酵母属(Yarrowia)和克鲁维酵母属(Kluyveromyces)用括号标出;菌株5,8,15分别用于FISH研究。 The sequences obtained in the present study are marked with circle,square and triangle,respectively. Bootstrap values of >50% are shown at nodes. The } indicates the three dominant yeast genera of Saccharomyces,Yarrowia and Kluyveromyces. The strains 5,8 and 15 were applied in FISH analysis. |
从分离鉴定为酿酒酵母菌(S. cerevisiae)、马克斯克鲁维酵母菌(K. marxianus)和解脂亚罗酵母菌(Y. lipolytica)的14株酵母菌中各选取1株作为代表菌株开展后续研究(图 1)。为了排除潜在的菌株间的交叉污染,分别对这3株酵母菌进行了特异性荧光探针的原位杂交,结果如图版Ⅲ所示。3种酵母菌细胞只与对应的特异探针有100%的杂交信号,说明菌种细胞是同质的,不存在污染,同时也证明了它们确实分属于酿酒酵母属(Saccharomyces)、克鲁维酵母属(Kluyveromyces)和亚罗酵母属(Yarrowia)。
3 讨论本文采用分离培养的方法,从西藏开菲尔粒中分离出16株酵母菌并对其进行了初步鉴定。26S rRNA基因序列分析及系统发育分析结果显示,其中8株属于克鲁维酵母属(Kluyveromyces)、5株为酿酒酵母属(Saccharomyces)、3株归为亚罗酵母属(Yarrowia)。DIOSMA等人从阿根廷开菲尔粒中分离鉴定出酿酒酵母菌(Saccharomyces cerevisiae)、单孢酵母菌(Saccharomyces unisporus)、Issatchenkia occidentalis以及马克斯克鲁维酵母菌(Kluyveromyces marxianus)[9];台湾开菲尔粒中鉴定出马克斯克鲁维酵母菌(K. marxianus)、单孢酵母菌(S. unisporus)和发酵毕赤氏酵母(Pichia fermentans)[10]。这些研究结果说明,克鲁维酵母属(Kluyveromyces)和酿酒酵母属(Saccharomyces)酵母菌作为优势菌普遍存在于不同来源的开菲尔粒品系中[11]。不过,值得注意的是,它们中可被分离培养的酵母菌均不多。
实验表明,亚罗酵母属(Yarrowia)尚未在其他西藏开菲尔粒或开菲尔粒中发现。相比之下,在其他西藏开菲尔粒及其他开菲尔粒中很常见的Kazachstania并未从我们的样品中分离出来[12, 13]。与之类似的是,WITTHUHN等应用ID32C方法(API系统,MontalieuVercieu)鉴定分离自南非开菲尔粒中酵母菌时,发现了一种Zygosaccharomyces spp.,这个属的酵母菌在其他传统的开菲尔粒中从未被发现过[14]。这一现象与之前许多学者对不同地区开菲尔粒中微生物分离培养研究结果相一致,即不同来源、不同培养条件下的开菲尔粒,其菌群多样性是存在差异的[15, 16]。因此,亚罗酵母属(Yarrowia)只在本研究的西藏开菲尔粒中分离到是可以理解的,可能与本研究中用于培养开菲尔粒的牛奶品质有关。此外,本文对西藏开菲尔粒中酵母菌的鉴定是基于测序及序列分析,因此序列相似性低于99%的菌株只能将其归于属的水平,通过后续的生理生化试验则有望将其鉴定到种的水平上。
| [1] | GAO J, GU F, ABDELLA N H, et al. Investigation on culturable microflora in Tibetan kefir grains from different areas of China[J].Journal of food science,2012,77:425-433. |
| [2] | ZHOU J Z, LIU X L, JIANG H H, et al. Analysis of the microflora in Tibetan kefir grains using denaturing gradient gel electrophoresis[J].Food Microbiology,2009,26:770-775. |
| [3] | GAO J, GU F, HE J, et al. Metagenome analysis of bacterial diversity in Tibetan kefir grains[J].European Food Research Technology, 2013, 236:549-556. |
| [4] | FARNWORTH E R. Kefir-a complex probiotic[J].Food Science and Technology Bulletin: Functional Foods,2006,2:1-17. |
| [5] | FLEET G, BALIA R. The public health and probiotic significance of yeasts in foods and beverages[J].Yeasts in Food and Beverages, 2006:381-397. |
| [6] | KURTZMAN C P, ROBNETT C J. Identification and phylogeny of ascomycetous yeasts from analysis of nuclear large subunit (26S) ribosomal DNA partial sequences[J].Antonie Van Leeuwenhoek,1998,73(4):331-371. |
| [7] | PARK E J, CHANG H W, KIM K H, et al. Application of quantitative real-time PCR for enumeration of total bacterial, archaeal, and yeast populations in kimchi[J].Journal of Microbiology,2009,47:682-685. |
| [8] | FOSTER H A, STURMEY R G, STOKES P J, et al. Fluorescence in situ hybridization on early porcine embryos[J].Methods in Molecular Biology,2009,659:427-436. |
| [9] | DIOSMA G, ROMANIN D E, et al. Yeasts from kefir grains: isolation, identification, and probiotic characterization[J].World Journal of Microbiology and Biotechnology,2014,30(1):43-53. |
| [10] | LIN C W, CHEN H L, LIU J R. Identification and characterisation of lactic acid bacteria and yeasts isolated from kefir grains in Taiwan[J].Australian Journal of Dairy Technology,1999,54:14-18. |
| [11] | MARSH A J, O'SULLIVAN O, HILL C, et al. Sequencing-based analysis of the bacterial and fungal composition of kefir grains and milks from multiple sources[J].PloS one,2013,8:69371. |
| [12] | SIMOVA E, BESHKOVA D, ANGELOV A, et al. Lactic acid bacteria and yeasts in kefir grains and kefir made from them[J].Journal of Industrial Microbiology and Biotechnology,2002,28:1-6. |
| [13] | WANG S Y, CHEN H C, LIU J R, et al. Identification of yeasts and evaluation of their distribution in Taiwanese kefir and viili starters[J].Journal of dairy science,2008,91:3798-3805. |
| [14] | WITTHUHN R C, SCHOEMAN T, BRITZ T J. Isolation and characterization of the microbial population of different South African kefir grains[J]. International Journal of Dairy Technology, 2004,57(1):33-37. |
| [15] | GARROTE G L, ABRAHAM A G, de ANTONI G L. Chemical and microbiological characterisation of kefir grains[J].Journal of Dairy Research,2001,68(4):639-652. |
| [16] | MARSHALL V M, COLE W M, BROOKER B E. Observations on the structure of kefir grains and the distribution of the microflora[J]. Journal of Applied Microbiology,1984,57(3):491-497. |
2. Shanghai Engineering Research Center of Aquatic Product Processing & Preservation, Shanghai 201306, China;
3. Laboratory of Quality & Safety Risk Assessment for Aquatic Production on Storage and Preservation (Shanghai), Ministry of Agriculture, Shanghai 201306, China
 2015, Vol. 24
2015, Vol. 24


