扩展功能
文章信息
- 杨苗, 孙洁, 沈富军, 凌珊珊, 吴虹林, 张亮, 侯蓉, 黄炎, 岳碧松, 张修月
- YANG Miao, SUN Jie, SHEN Fujun, LING Shanshan, WU Honglin, ZHANG Liang, HOU Rong, HUANG Yan, YUE Bisong, ZHANG Xiuyue
- 幼年早期和成年大熊猫胰腺组织miRNA初步研究
- Preliminary Study on miRNAs in Pancreatic Tissue Between Early Childhood Giant Pandas and Adults
- 四川动物, 2022, 41(2): 121-131
- Sichuan Journal of Zoology, 2022, 41(2): 121-131
- 10.11984/j.issn.1000-7083.20210169
-
文章历史
- 收稿日期: 2021-05-12
- 接受日期: 2021-05-20
2. 成都大熊猫繁育研究基地, 四川省濒危动物保护生物学重点实验室, 成都 610081;
3. 中国大熊猫保护研究中心, 大熊猫国家公园珍稀动物保护生物学国家林业和草原局重点实验室, 四川都江堰 623006
2. Key Laboratory of Endangered Animal Conservation Biology of Sichuan Province, Chengdu Research Base of Giant Panda, Chengdu 610081, China;
3. Key Laboratory of National Forestry and Grassland Administration on Conservation Biology of Rare Animals in the Giant Panda National Park, China Conservation and Research Center for Giant Panda, Dujiangyan, Sichuan Province 623006, China
大熊猫Ailuropoda melanoleuca属于食肉目Carnivora,是我国珍稀濒危野生动物,幼年早期主要以母乳为食,成年却专性以竹子为食。大熊猫母乳营养丰富,含有大量的蛋白质、碳水化合物和脂肪(Nakamura et al., 2003),而竹子属于低营养高纤维食物。不仅如此,大熊猫消化道短且无盲肠,食物通过时间短,依然保留着肉食性消化系统结构和遗传系统特征(邹兴淮等,1998)。因此,大熊猫生长过程中,营养利用与食物代谢及其变化可能与一般的草食动物和肉食动物均不同,具有其独特性。研究大熊猫生长过程中的营养利用及调控特征对其保护具有重要意义。
胰腺是动物重要的消化代谢器官,作为第二大消化腺,兼具内分泌和外分泌的双重功能。胰腺分泌胰液经胰导管输送至十二指肠的过程即为外分泌,胰液中含有的碱性碳酸氢盐及各种消化酶可中和胃酸,消化糖类、蛋白质及脂肪。胰腺的内分泌主要依靠内部的胰岛细胞产生胰岛素、胰高血糖素及生长激素释放抑制激素和胃泌素等(Leung,2010;Lorberbaum et al., 2020)。
转录组水平研究表明,大熊猫在幼年早期的胆固醇代谢、蛋白质消化吸收相关基因高表达,以满足其对营养的高需求,有利于出生后的快速生长。碳水化合物代谢、能量产生、氨基酸和蛋白质代谢等相关基因表达上调,表明成年大熊猫具有高的代谢水平(Ma et al., 2020)。但其具体代谢调控的分子机制有待进一步研究。microRNA(miRNA)作为一种非编码的短序列RNA,已被证实能够参与多种形式的生命活动,如疾病、免疫、细胞凋亡、代谢、生殖和发育等(Ambros,2004;Wienholds & Plasterk,2005)。胰腺组织相关miRNA的研究表明,一些miRNA可能参与胰腺的发生,包括miR-124(Baroukh et al., 2007),miR-15a、miR-15b、miR-16、miR-195(Joglekar et al., 2007),miR-376、miR-375(Kloosterman et al., 2007;Joglekar et al., 2009)和miR-7(Correa-Medina et al., 2009;Joglekar et al., 2009)等,这些miRNA的异常表达会影响胰腺的正常发育和生理功能(Dumortier & Obberghen,2012)。此外,miRNA可以调控营养物质的代谢过程,包括脂类代谢(Esau et al., 2006)、氨基酸代谢(Dang,2009;Sengupta et al., 2020)、糖代谢(Eichner et al., 2010;Kefas et al., 2010)等。因此,通过对大熊猫不同年龄阶段胰腺组织中差异miRNA的分析,可以了解在大熊猫从幼年到成年的食性转变过程中,miRNA对营养代谢的调控作用。
本研究探讨幼年早期与成年大熊猫胰腺组织中miRNA表达谱的差异,以了解miRNA对幼年早期和成年大熊猫营养代谢的调控差异。
1 实验材料和方法 1.1 样本采集大熊猫胰腺组织样本由成都大熊猫繁育研究基地和中国大熊猫保护研究中心提供。从4只死亡个体中采集到了4份胰腺样本,其中,样本YR和LT为1日龄和6日龄,死亡原因为意外窒息;样本DN和CC为成年个体,为野外严重受伤,抢救无效死亡。所有样本的胰腺组织均未见病理改变。采样工作由专业兽医进行,项目研究经四川大学生命科学学院伦理委员会批准(批准号:20190506001)。
1.2 Small RNA提取和测序对胰腺组织匀浆后,使用Trizol试剂按照说明提取RNA。使用Agilent 2100 Bioanalyzer检测样本的完整性和质量,RNA完整值(RIN)≥7.5,说明样本具有较好完整性,可用于后续small RNA建库测序。使用Small RNA Sample Pre Kit构建文库,将提取的总RNA过滤后得到15~40个核苷酸的small RNA。用T4连接酶将small RNA的3'和5'端加上接头,PCR反转录扩增得到small RNA文库。扩增产物纯化检测合格后,由北京诺禾致源科技股份有限公司测序,测序平台为Illumina HiSeqTM 2000。
1.3 Small RNA数据处理测序得到的原始数据需经质控后才可用于后续分析,因此对原始数据进行以下处理:首先,删除质量值≤5的reads占总读段>50%的低质量序列,去除N(无法确定碱基信息)比例>10%的序列。其次,删除5'接头污染的序列和没有3'接头和插入片段的序列。修剪3'接头序列后,去除有poly A、poly T、poly C和poly G的序列。最后,去除过长或过短的序列,保留18~30 nt的序列用于后续分析。为避免非miRNA序列对结果的影响,使用BLASTN对非miRNA进行注释。为确保每个small RNA仅分配1个注释,使用以下规则:rRNA>tRNA>snRNA>snoRNA>repeat sequences。删除非miRNA后,使用miRDeep2将剩余序列比对到大熊猫参考基因组,鉴定保守miRNA及预测新miRNA。
1.4 保守miRNA差异表达分析使用miRDeep2中的quantifier.pl脚本计算每个样本miRNA的表达量,得到原始表达矩阵,导入R软件的DEseq2程序包进行差异表达分析。DESeq2的estimateSizeFactors函数可对数据进行标准化处理,以消除测序文库的影响。使用标准化计数计算每个miRNA的表达量并采用Benjamini-Hochberg修正方法对P值进行调整。以|log2(fold change)|≥1且Padj<0.05作为差异表达miRNA(differentially expressed miRNA,DE miRNA)筛选的阈值。
1.5 预测靶基因使用miRTarBase数据库预测miRNA的靶基因,该数据库中的miRNA靶基因已通过实验验证(Huang et al., 2020),可提高分析结果的准确性。
1.6 miRNA与mRNA的相互作用关系为探究miRNA与mRNA之间的靶向调控关系,对已有mRNA样本的表达量进行差异表达分析。mRNA数据来自Ma等(2020)的研究,获得了该论文中与本研究一致的YR、LT与DN、CC 4只个体胰腺组织的原始表达量数据进行后续分析。将YR、LT与DN、CC的表达量数据同样采用DEseq2程序包进行差异表达分析,并认为符合|log2(fold change)|≥1且Padj<0.05筛选条件的基因是显著差异表达基因(differentially expressed gene,DEG)。miRNA主要通过使其靶基因沉默表达来起调控作用(Huntzinger & Izaurralde,2011),因此,本研究主要讨论与其靶向基因表达有负相关的DE miRNA。
1.7 miRNA靶基因的富集分析为了解miRNA的功能,分别对鉴定出的DE miRNA的靶向DEG进行富集分析。使用g: Profiler将其映射到基因本体论(GO)(http://www.geneontology.org/)和京都基因与基因组百科全书(KEGG)(http://www.genome.jp/kegg/)数据库,其中,Padj<0.05的条目和通路被认为是显著富集的。
2 结果 2.1 Small RNA序列特征测序获得4只大熊猫胰腺组织的small RNA数据,质控保留长度18~30 nt的序列后,共获得46 357 895条small RNA序列(图 1)。
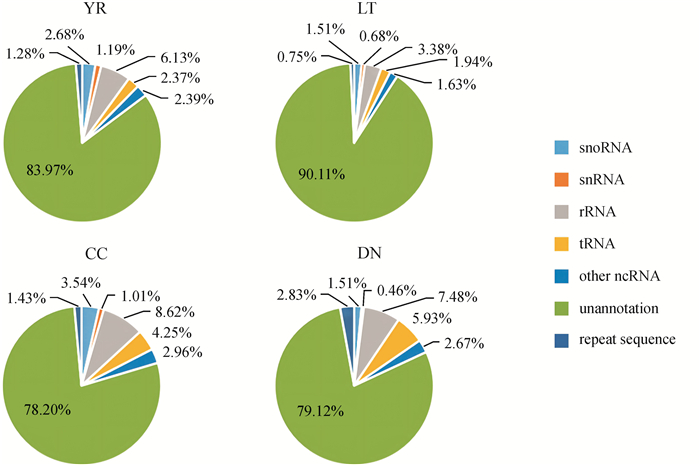
|
| 图 1 幼年早期(YR、LT)和成年大熊猫(CC、DN)胰腺组织中small RNA分类注释及其所占比例统计 Fig. 1 Small RNA categories and their proportions in pancreatic tissues of early childhood (YR, LT) and adult giant pandas (CC, DN) |
| |
将miRNA与miRBase数据库进行比对,共获得202个保守miRNA和8个新miRNA。其中,保守miRNA在幼年早期大熊猫胰腺组织中较为丰富,在成年大熊猫胰腺组织中较少。202个保守miRNA中,有36个在幼年早期大熊猫胰腺中特有表达,成年大熊猫胰腺组织未见表达(图 2)。
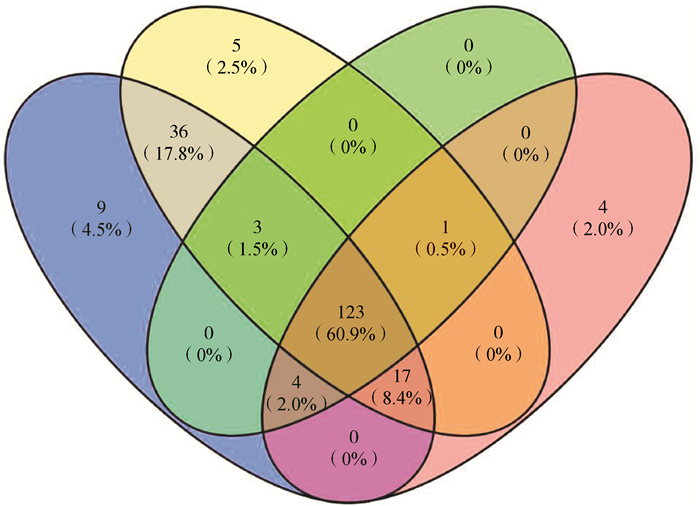
|
| 图 2 幼年早期(YR、LT)和成年大熊猫(CC、DN)胰腺组织中保守miRNA数目维恩图 Fig. 2 Venn diagram of the number of conserved miRNAs in pancreatic tissues of early childhood (YR, LT) and adult giant pandas (CC, DN) 蓝色为YR,黄色为LT,绿色为DN,红色为CC;重叠区域为共有miRNA YR. blue, LT. yellow, DN. green, CC. red; the overlapping area represents the number of shared miRNAs |
| |
miRNA的差异表达分析显示,幼年早期与成年大熊猫胰腺组织中miRNA表达存在较大差异。共获得77个DE miRNA,其中,在成年大熊猫胰腺组织中,39个上调,38个下调(图 3)。
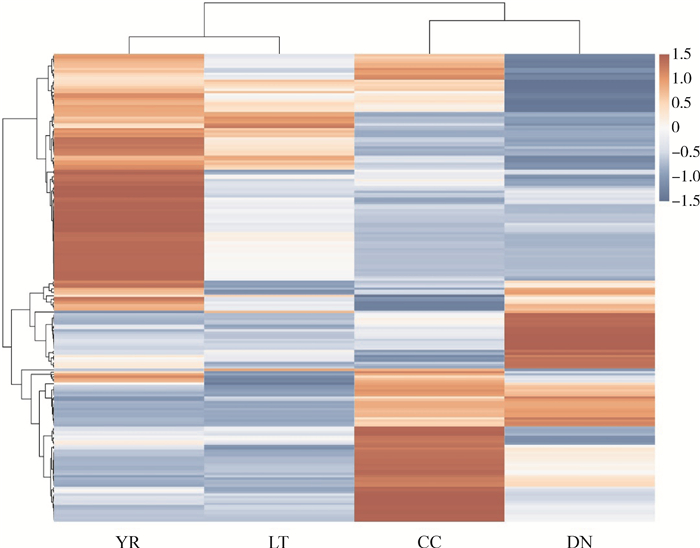
|
| 图 3 DE miRNA的聚类热图 Fig. 3 Clustering heat map of DE miRNAs 红色为表达量上调,蓝色为表达量下调; 下同 Red indicates up-regulated miRNAs, and blue indicates down-regulated miRNAs; the same below |
| |
通过miRTarBase数据库的预测,幼年早期个体中特有的36个miRNA共预测到3 488个有实验验证的靶基因。DE miRNA中,成年大熊猫胰腺组织中39个上调的miRNA预测到5 836个有实验验证的靶基因,38个下调的预测到4 193个有实验验证的靶基因。
2.5 miRNA靶基因功能富集分析幼年早期个体中特有的36个miRNA的靶基因GO富集分析显示,幼年早期个体特有的miRNA主要参与了物质代谢及合成等生物过程的调控,如,细胞代谢过程的调控(GO: 0031323)、生物合成过程的调控(GO: 0009889)和大分子代谢过程的调控(GO: 0060255)等。
DE miRNA的靶基因富集分析显示,上调DE miRNA靶基因GO富集结果多与细胞、大分子、含氮化合物等代谢的正调控有关,KEGG富集结果多与信号通路和疾病通路等有关。下调DE miRNA靶基因GO富集结果与上调DE miRNA功能相反,主要参与细胞、大分子及含氮化合物等代谢的负调控,KEGG富集结果也多与信号通路和疾病通路相关。
2.6 miRNA与mRNA的相互作用关系的构建mRNA差异表达分析显示,成年大熊猫胰腺组织中获得661个上调DEG和691个下调DEG。将39个上调DE miRNA对应的靶基因与691个下调DEG取交集处理,得到240个由上调DE miRNA调控的下调DEG(图 4)。将38个下调DE miRNA对应的靶基因与661个上调DEG取交集处理,得到138个由下调DE miRNA调控的上调DEG(图 5)。
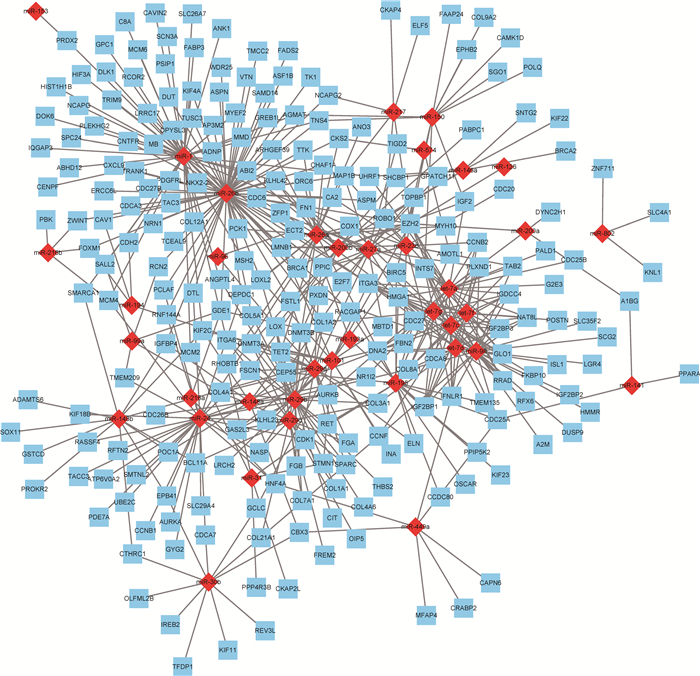
|
| 图 4 上调DE miRNA调控的下调DEG Fig. 4 The down-regulated DEGs regulated by up-regulated DE miRNAs 菱形代表DE miRNA,正方形代表DEG; 下同 Diamonds represent DE miRNAs, squares represent DEGs; the same below |
| |
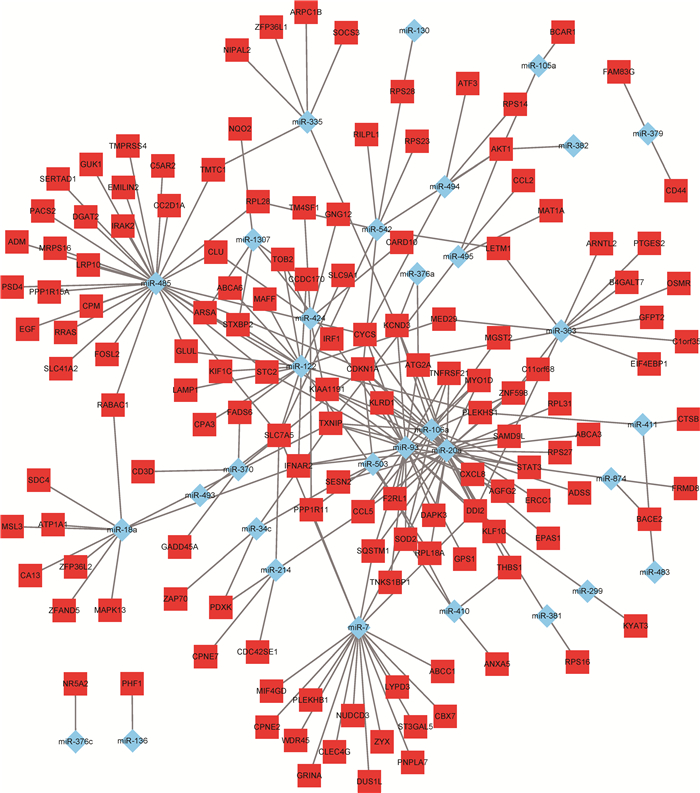
|
| 图 5 下调DE miRNA调控的上调DEG Fig. 5 The up-regulated DEGs regulated by down-regulated DE miRNA |
| |
为探究交集基因的生物学功能,对由DE miRNA调控的DEG进行了富集分析。KEGG富集结果显示,成年组中上调DE miRNA调控的下调DEG主要富集到蛋白质消化吸收等通路(图 6:A)。成年组中下调DE miRNA调控的上调DEG主要富集到疾病、信号通路及氨基酸代谢等相关通路(图 6:B)。
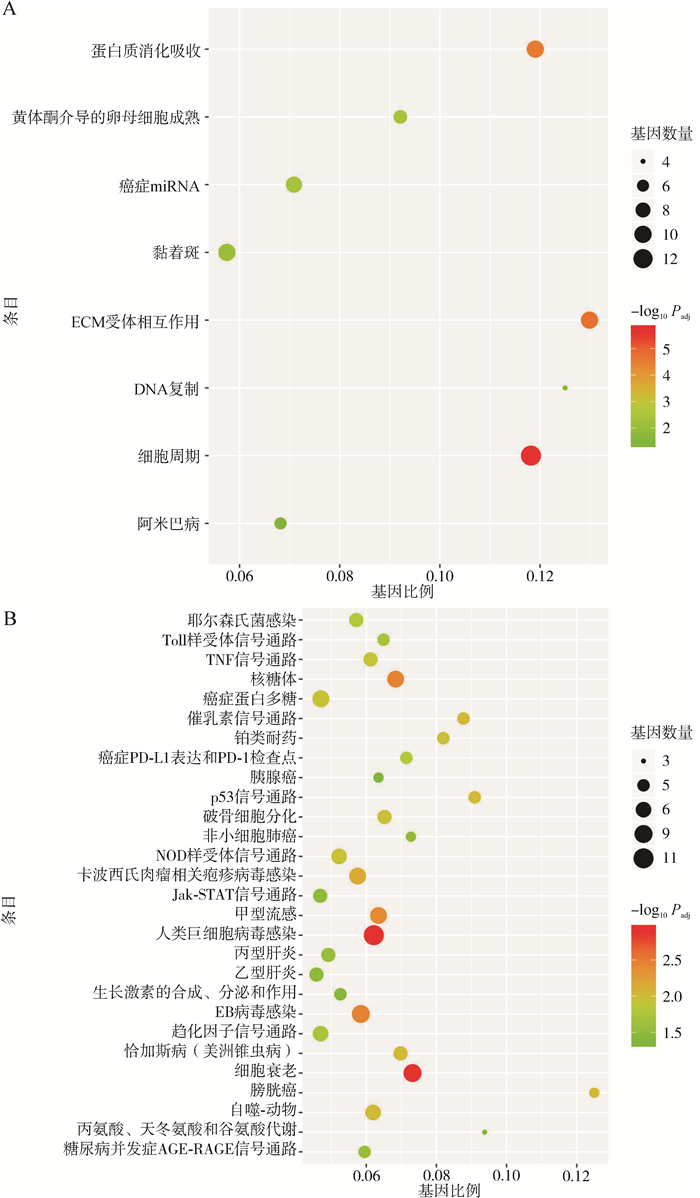
|
| 图 6 DE miRNA调控的DEG的KEGG富集结果 Fig. 6 KEGG enrichment of DEGs regulated by DE miRNAs A.上调DE miRNA调控的下调DEG, B. 下调DE miRNA调控的上调DEG A. down-regulated DEGs regulated by up-regulated DE miRNAs, B. up-regulated DEGs regulated by down-regulated DE miRNAs |
| |
胰腺是重要的消化代谢器官,为探究miRNA对不同年龄大熊猫消化代谢的调控作用,对参与糖代谢、脂质代谢以及蛋白质代谢等生物过程的相关基因及其调控miRNA进行分析。其中,NQO2编码电子传递链主要蛋白,在成年组中显著上调,是miR-1307的靶基因之一。ANGPTL4与胆固醇代谢有关,在成年组中显著下调,是miR-1的靶基因之一。蛋白质消化代谢相关基因中,COL12A1在成年组中显著下调,是miR-1的靶基因之一,这些miRNA与它们的靶基因表达均呈反向负调控趋势。
3 讨论大熊猫的营养代谢一直是遗传学家和进化生物学家最感兴趣的问题。胰腺是动物重要的消化器官,在消化和营养代谢中起着至关重要的作用。分析幼年早期和成年大熊猫胰腺组织中miRNA的差异表达,有助于了解不同年龄阶段miRNA对大熊猫食物代谢的调控作用及其变化规律。
本文鉴别到幼年早期大熊猫胰腺组织中特有表达的36个保守miRNA,主要参与合成及代谢的调控,包括含氮化合物、大分子化合物、核酸代谢过程的调控,暗示幼年早期大熊猫大分子合成较弱;而在成年大熊猫体内,这些miRNA的沉默表达可能促进了蛋白质、脂质等生物大分子的合成过程。对DE miRNA的靶基因进行功能分析,发现成年组中上调DE miRNA的靶基因对大分子和含氮化合物代谢过程的调控是正调控作用,而成年组下调DE miRNA的靶基因对这些物质合成的生物学过程主要是负调控。这与mRNA分析结果相吻合,即成年大熊猫胰腺组织中参与这些大分子合成的相关基因表达上调(Ma et al., 2020)。
生物体在正常营养代谢过程中,会产生活性氧物质(Poyton et al., 2009)。幼年早期大熊猫胰腺组织中上调DE miRNA调控的下调DEG富集到了活性氧代谢过程的正调控通路上,说明相较于成年大熊猫,幼年大熊猫活性氧代谢的能力较弱。研究表明,当需求能量增加但摄入能量较少时,会激活体内脂肪的分解代谢,而造成细胞的活性氧代谢产物大量增加(张帆等,2020)。这一方面可能由于成年大熊猫代谢水平高(Ma et al., 2020),产生的活性氧物质多,另一方面可能由于幼年早期大熊猫从母体乳汁中摄入的能量较多,因此体内活性氧化物质较少,而成年大熊猫以竹子为食,竹子中所含能量物质少,体内脂肪代谢途径可能被激活从而产生了较多的活性氧物质,提高活性氧物质的代谢反应以维持机体内正常的活性氧化物水平。本研究结果表明,miRNA参与了成年大熊猫活性氧代谢的调控作用。
幼年早期组中下调DE miRNA调控的上调DEG富集到了蛋白质消化吸收的通路上,而成年组中下调DE miRNA调控的上调DEG富集到了氨基酸代谢的相关通路上。mRNA分析结果也显示,成年大熊猫胰腺组织内上调DEG富集到了与氨基酸和蛋白质代谢有关的通路上,而在幼体大熊猫胰腺组织中的上调DEG富集到了与蛋白质消化吸收有关的通路上(Ma et al., 2020),miRNA结果与mRNA分析结果一致。蛋白质是幼体生长发育期间所必需的重要营养物质(Wiedmeier et al., 2011),蛋白质利用率下降会导致哺乳期幼崽生长受限(Aguinaga et al., 2011)。负调控蛋白质消化吸收相关基因的miRNA在幼年早期大熊猫胰腺组织中低表达,而在成年后高表达,可能由于乳汁中蛋白质含量高,幼年大熊猫对乳汁中蛋白质吸收充分,以保证出生后的快速生长。成年大熊猫调控氨基酸和蛋白质代谢相关基因的miRNA下调,可能是因为成年大熊猫代谢水平的全面提高(Ma et al., 2020)。
针对一些DE miRNA的功能分析发现,它们可以调节许多重要的代谢过程,将这些代谢过程分为脂质代谢、能量代谢以及蛋白质代谢。其中,miR-27a、miR-122和miR-370与脂质代谢有关。miR-27a可以促进脂肪分解,释放出更多的甘油及游离脂肪酸(Wang et al., 2011),它在成年大熊猫中的表达上调表明了成年大熊猫胰腺组织中的脂质代谢较幼年早期个体有提高。miR-122可以参与脂类代谢,Esau等(2006)的研究表明,抑制miR-122的表达对小鼠Mus musculus血浆的胆固醇和脂肪酸代谢均有促进作用。成年大熊猫体内miR-122的表达相较于幼年早期下降,也说明成年大熊猫体内的脂肪酸代谢较幼年早期旺盛。miR-370也在成年大熊猫体内表达下调。miR-370对miR-122的表达具有正向调控作用;此外,miR-370还与miR-122的调控作用类似,可通过调控基因的表达来调控脂质代谢过程(Iliopoulos et al., 2010)。这些miRNA的差异表达说明,成年大熊猫体内的脂质代谢水平高,与mRNA的研究结果一致。Li等(2011)研究显示,包括miR-363、miR-382和miR-494等27个miRNA被预测为靶向参与氧化磷酸化的基因,且蛋白质组学实验显示miRNA与其靶基因呈反向表达。miR-494、miR-382和miR-363在成年大熊猫中的显著下调可能促进其能量代谢。此外,ATP合酶是氧化磷酸化过程中一个重要因子,而miR-127可以调控ATP合酶的亚基——ATP5B,miR-127靶向β-mRNA的3'UTR区域,并抑制ATP5B翻译(Willers et al., 2012)。在成年组中,miR-127的表达下调保证了ATP合酶的生成,促进了能量代谢。这一结果也与mRNA研究一致,即在成年大熊猫体内的能量代谢较幼年早期旺盛(Ma et al., 2020)。目前,有关miRNA对动物体内氨基酸代谢过程中的调控研究较少,在差异表达的miRNA中仅发现miR-1对氨基酸代谢有调控作用。有研究表明,仅摄取10 g必需氨基酸后3 h内,miR-1的表达会出现显著上调,从而加速氨基酸代谢与后期蛋白质的合成(Mcgregor et al., 2014),miR-1在成年大熊猫胰腺组织中的表达上调可能与其相关代谢水平的提高有关。
综上,miRNA在大熊猫生长发育、营养代谢的动态变化中发挥了重要的调控作用,miRNA的差异表达模式与幼年早期大熊猫对高营养的需求和成年大熊猫胰腺组织中高水平的脂质代谢、氨基酸代谢和能量代谢相一致,这些结果与mRNA的分析一致。本研究丰富了大熊猫消化代谢的基础研究,为进一步了解大熊猫食性转变与营养利用提供了重要的参考。然而,由于研究对象及组织的特殊性,目前样本量较小,因此后续可增加样本量继续开展深入研究,进一步证实本研究的结果。
张帆, 呙于明, 熊本海. 2020. 围产期奶牛能量负平衡营养调控研究进展[J]. 动物营养学报, 32(7): 2966-2974. |
邹兴淮, 曾鲁军, 孙中武, 等. 1998. 大熊猫疾病死亡因素分析及其保护对策[J]. 东北林业大学学报, 26(1): 53-56. |
Aguinaga MA, Gómez-Carballar F, Nieto R, et al. 2011. Utilization of milk amino acids by the suckling Iberian piglet[J]. Journal of Animal Physiology and Animal Nutrition, 95(6): 771-780. DOI:10.1111/j.1439-0396.2010.01108.x |
Ambros V. 2004. The functions of animal microRNAs[J]. Nature, 431(7006): 350-355. DOI:10.1038/nature02871 |
Baroukh N, Ravier MA, Loder MK, et al. 2007. MicroRNA-124a regulates Foxa2 expression and intracellular signaling in pancreatic beta-cell lines[J]. Journal of Biological Chemistry, 282(27): 19575-19588. DOI:10.1074/jbc.M611841200 |
Correa-Medina M, Bravo-Egana V, Rosero S, et al. 2009. MicroRNA miR-7 is preferentially expressed in endocrine cells of the developing and adult human pancreas[J]. Gene Expression Patterns, 9(4): 193-199. DOI:10.1016/j.gep.2008.12.003 |
Dang CV. 2009. MYC, microRNAs and glutamine addiction in cancers[J]. Cell Cycle, 8(20): 3243-3245. DOI:10.4161/cc.8.20.9522 |
Dumortier O, Obberghen EV. 2012. MicroRNAs in pancreas development[J]. Diabetes, Obesity and Metabolism, 14(s3): 22-28. |
Eichner LJ, Perry MC, Dufour CR, et al. 2010. miR-378* mediates metabolic shift in breast cancer cells via the PGC-1β/ERRγ transcriptional pathway[J]. Cell Metabolism, 12(4): 352-361. DOI:10.1016/j.cmet.2010.09.002 |
Esau C, Davis S, Murray SF, et al. 2006. miR-122 regulation of lipid metabolism revealed by in vivo antisense targeting[J]. Cell Metabolism, 3(2): 87-98. DOI:10.1016/j.cmet.2006.01.005 |
Huang HY, Lin YC, Li J, et al. 2020. miRTarBase 2020:updates to the experimentally validated microRNA-target interaction database[J]. Nucleic Acids Research, 48(D1): 148-154. |
Huntzinger E, Izaurralde E. 2011. Gene silencing by microRNAs: contributions of translational repression and mRNA decay[J]. Nature Reviews Genetics, 12(2): 99-110. DOI:10.1038/nrg2936 |
Iliopoulos D, Drosatos K, Hiyama Y, et al. 2010. MicroRNA-370 controls the expression of microRNA-122 and Cpt1alpha and affects lipid metabolism[J]. Journal of Lipid Research, 51(6): 1513-1523. DOI:10.1194/jlr.M004812 |
Joglekar MV, Joglekar VM, Hardikar AA. 2009. Expression of islet-specific microRNAs during human pancreatic development[J]. Gene Expression Patterns, 9(2): 109-113. DOI:10.1016/j.gep.2008.10.001 |
Joglekar MV, Parekh VS, Mehta S, et al. 2007. MicroRNA profiling of developing and regenerating pancreas reveal post-transcriptional regulation of neurogenin3[J]. Developmental Biology, 311(2): 603-612. DOI:10.1016/j.ydbio.2007.09.008 |
Kefas B, Comeau L, Erdle N, et al. 2010. Pyruvate kinase M2 is a target of the tumor-suppressive microRNA-326 and regulates the survival of glioma cells[J]. Neuro-Oncology, 12(11): 1102-1112. DOI:10.1093/neuonc/noq080 |
Kloosterman WP, Lagendijk AK, Ketting RF, et al. 2007. Targeted inhibition of miRNA maturation with morpholinos reveals a role for miR-375 in pancreatic islet development[J/OL]. PLoS Biology, 5(8): e203 [2021-01-20]. https://doi.org/10.1371/journal.pbio.0050203.
|
Leung PS. 2010. Overview of the pancreas[J]. Advances in Experimental Medicine & Biology, 690(3): 3-12. |
Li N, Bates DJ, An J, et al. 2011. Up-regulation of key microRNAs, and inverse down-regulation of their predicted oxidative phosphorylation target genes, during aging in mouse brain[J]. Neurobiology of Aging, 32(5): 944-955. DOI:10.1016/j.neurobiolaging.2009.04.020 |
Lorberbaum DS, Docherty FM, Sussel L. 2020. Animal models of pancreas development, developmental disorders, and disease[J]. Advances in Experimental Medicine and Biology, 1236: 65-85. |
Ma J, Shen F, Chen L, et al. 2020. Gene expression profiles during postnatal development of the liver and pancreas in giant pandas[J]. Aging (Albany NY), 12(15): 15705-15729. |
McGregor RA, Poppitt SD, Cameron-Smith D. 2014. Role of microRNAs in the age-related changes in skeletal muscle and diet or exercise interventions to promote healthy aging in humans[J]. Ageing Research Reviews, 17: 25-33. DOI:10.1016/j.arr.2014.05.001 |
Nakamura T, Urashima T, Mizukami T, et al. 2003. Composition and oligosaccharides of a milk sample of the giant panda, Ailuropoda melanoleuca[J]. Comparative Biochemistry and Physiology B: Biochemistry and Molecular Biology, 135(3): 439-448. DOI:10.1016/S1096-4959(03)00093-9 |
Poyton RO, Ball KA, Castello PR. 2009. Mitochondrial generation of free radicals and hypoxic signaling[J]. Trends in Endocrinology & Metabolism, 20(7): 332-340. |
Sengupta D, Cassel T, Teng KY, et al. 2020. Regulation of hepatic glutamine metabolism by miR-122[J]. Molecular Metabolism, 34: 174-186. DOI:10.1016/j.molmet.2020.01.003 |
Wang T, Li M, Guan J, et al. 2011. MicroRNAs miR-27a and miR-143 regulate porcine adipocyte lipid metabolism[J]. International Journal of Molecular Sciences, 12(11): 7950-7959. DOI:10.3390/ijms12117950 |
Wiedmeier JE, Joss-Moore LA, Lane RH, et al. 2011. Early postnatal nutrition and programming of the preterm neonate[J]. Nutrition Reviews, 69(2): 76-82. DOI:10.1111/j.1753-4887.2010.00370.x |
Wienholds E, Plasterk RH. 2005. MicroRNA function in animal development[J]. FEBS Letters, 579(26): 5911-5922. DOI:10.1016/j.febslet.2005.07.070 |
Willers IM, Martínez-Reyes I, Martínez-Diez M, et al. 2012. miR-127-5p targets the 3'UTR of human β-F1-ATPase mRNA and inhibits its translation[J]. BBA Bioenergetics, 1817(5): 838-848. DOI:10.1016/j.bbabio.2012.03.005 |
 2022, Vol. 41
2022, Vol. 41




