扩展功能
文章信息
- 朱英, 李裕冬, 何鸣, 胡兰, 吴虹林
- ZHU Ying, LI Yudong, HE Ming, HU Lan, WU Honglin
- 直接测序法构建圈养大熊猫MHC遗传档案
- MHC Genotyping for Captive Giant Pandas by PCR Sequencing
- 四川动物, 2021, 40(1): 46-52
- Sichuan Journal of Zoology, 2021, 40(1): 46-52
- 10.11984/j.issn.1000-7083.20200145
-
文章历史
- 收稿日期: 2020-04-26
- 接受日期: 2020-08-20
2. 中国大熊猫保护研究中心, 四川都江堰 611800
2. China Conservation and Research Center for the Giant Panda, Dujiangyan, Sichuan Province 611800, China
主要组织相容性复合体(major histocompatibility complex,MHC)是脊椎动物基因组中多态性极高的功能性多基因家族之一,它编码的蛋白分子能够识别、结合、呈递抗原并激发一系列免疫反应(Klein,1986)。MHC基因家族包括呈递胞内病原菌抗原的Ⅰ类MHC基因和呈递胞外病原菌抗原的Ⅱ类MHC基因(Piertney & Oliver,2006)。
由于MHC基因编码的蛋白质分子在脊椎动物的免疫系统中扮演着重要的角色,濒危物种的MHC基因得到了动物保护工作者的关注,学界陆续开展了诸如大熊猫Ailuropoda melanoleuca(Chen et al., 2010, 2013;Zhu et al., 2013a, 2013b)、川金丝猴Rhinopithecus roxellana(Luo & Pan,2013;Zhang et al., 2018)、林麝Moschus berezovskii(Li et al., 2014;Yao et al., 2015)、红腹锦鸡Chrysolophus pictus(Zeng et al., 2016a, 2016b)、扬子鳄Alligator sinensis(Zhai et al., 2017;Han et al., 2019)和朱鹮Nipponia nippon(Lan et al., 2019;Sun et al., 2019)等濒危动物的MHC遗传多样性评估、MHC配偶选择机制的解析、MHC与疾病的关系等研究,推动了MHC基因在濒危动物保护中的应用。
Zhu等(2019, 2020)研究表明,MHC基因与大熊猫的配偶选择以及抗寄生虫感染能力有密切的关系,前者研究表明大熊猫的MHC配偶选择的机制影响了大熊猫的自然交配行为,以及大熊猫的繁殖成功率; 雌性大熊猫偏爱特定MHC基因(Aime-C、Aime-I、DQ)是杂合子的雄性以及倾向于选择与自身MHC(Aime-C、DQ、DR)基因型相异的雄性交配; MHC基因型差异越大的配偶之间交配成功率越高。后者研究表明大熊猫拥有西氏贝蛔虫Baylisascaris schroederi的MHC抗性基因; 与MHC基因纯合的大熊猫相比,MHC杂合的大熊猫不易感染西氏贝蛔虫,且感染程度也远低于MHC基因纯合的个体。这些发现表明MHC基因的遗传参数可以用于指导大熊猫的繁育计划、寄生虫防控、野外放归个体选择等种群管理工作。因此,建立圈养种群的MHC遗传档案,并获得遗传多样性等相关参数对于大熊猫的遗传管理具有重要的应用价值。
中国大熊猫保护研究中心拥有目前世界上最大的大熊猫人工圈养种群(截止2019年底,313只,约占人工圈养种群数量的50%以上),该中心的大熊猫MHC遗传参数将是我国人工圈养种群的遗传管理的重要参考指标。大熊猫MHC基因分型主要采取以聚丙烯酰胺凝胶电泳和银染技术为基础的单链构象多态性的方法(Chen et al., 2010, 2013;Zhu et al., 2013a, 2013b)。该方法已成功应用于大熊猫的10个经典的MHC基因的分型工作中,包括4个Ⅰ类MHC(Aime-C、Aime-F、Aime-I、Aime-L; Zhu et al., 2013a)和6个Ⅱ类MHC基因(Aime-DRA、Aime-DQA1、Aime-DQA2、Aime-DQB1、Aime-DQB2和Aime-DRB3;Chen et al., 2013)。但该方法需掌握聚丙烯酰胺凝胶电泳和银染技术,且需要较高的时间成本(电泳8~12 h,银染1~3 h)和劳动成本(Sunnucks et al., 2000)。故为每一只圈养大熊猫建立全面的MHC遗传档案是费时费力的工作。鉴于MHC遗传档案的重要性,本研究在已有研究的基础上,建立一种不需聚丙烯酰胺凝胶电泳和银染技术且具备较低实验成本的分型方法,并对大熊猫的所有多态MHC基因进行分型,构建大熊猫的MHC遗传档案,以完善我国人工圈养种群的遗传管理体系。
1 方法 1.1 样品采集本研究于2016年8月共收集91只大熊猫的粪便样品,分别来自中国大熊猫保护研究中心雅安碧峰峡基地(n=32)、都江堰基地(n=21)、卧龙神树坪基地(n=38)。为了不干扰大熊猫活动,粪便样品由中国大熊猫保护研究中心饲养员在08: 30大熊猫进入室外活动场地后在室内采集,粪便的排出时长小于12 h。粪便样品在4 ℃的条件下2 h内运输至实验室。
1.2 DNA提取、PCR扩增、基因分型在大熊猫的10个MHC基因中,Aime-F、Aime-DQB2和Aime-DRA位点在圈养种群中均只有1条等位基因(Chen et al., 2010;Zhu et al., 2013a),不具有多态性,本研究排除这3个位点,采用其他7个多态的MHC位点(Aime-C、Aime-I、Aime-L、Aime-DQA1、Aime-DQA2、Aime-DQB1和Aime-DRB3)进行遗传变异分析。粪便DNA提取参考Zhu等(2017)的方法。
现有的单链构象多态性分型方法的开发,在一定程度上归因于高昂的测序费用。随着测序成本的大幅降低,直接测序法时间成本和劳动成本的优势便得以凸显。我们建立了直接测序法进行基因分型的流程(图 1)。对于纯合子个体的基因型,可以直接进行PCR产物测序并结合峰图确认。对于杂合个体的基因型,如果不同基因型的峰图不同,可以通过碱基组合的特征进行区分。如果不同基因型峰图一致,需通过克隆分离等位基因进行基因型的确认。所有PCR产物都进行双向测序,以保证结果的准确性。
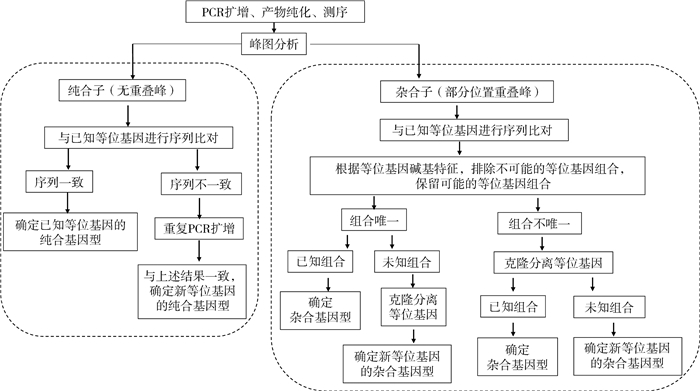
|
| 图 1 直接测序法进行大熊猫MHC基因分型的流程 Fig. 1 Flow chart of MHC genotyping in giant panda by PCR sequencing |
| |
利用已发表的基因特异性分型引物(Chen et al., 2013;Zhu et al., 2013b),对采集的所有熊猫粪便DNA样品进行PCR扩增、PCR产物纯化、直接测序、峰图分析,进行Ⅰ类MHC基因的exon2-exon3(编码α1和α2结构域)以及Ⅱ类MHC基因的exon2的分型。
Ⅰ类MHC基因需要进行exon2-exon3单倍型分型。单链构象多态性法是分别对exon2和exon3进行分型,再通过谱系验证确定exon2和exon3的连锁关系。本研究将exon2的上游引物和exon3的下游引物进行组合后进行PCR扩增、产物直接测序,通过测序峰图碱基特征直接获得exon2-exon3的单倍型信息。
1.3 数据分析所有序列经过手工校对后,利用MEGA X(Kumar et al., 2018)的Clustal W算法进行比对,并计算核苷酸和氨基酸的变异位点数目。大熊猫Ⅰ类MHC基因的抗原结合位点(antigen binding site,ABS)根据人Homo sapiens的ABS进行推断(Zhu et al., 2013a)。使用Genepop 4.0进行等位基因频率计算和等位基因数目的统计(Rousset,2008)。
2 结果除DQA1*0104和DQA1*0506外,大熊猫7个多态的MHC基因各自的等位基因两两组合,都能够通过等位基因碱基组合的特征进行区分。对于DQA1*0104或DQA1*0506杂合型,通过PCR扩增、克隆分离等位基因,确定基因型。所有疑似为DQA1*0104或DQA1*0506这2种组合的最终均确认为DQA1*0104杂合型,试验的大熊猫种群中不含DQA1*0506基因型的个体。
利用直接测序分型方法,从91只大熊猫中分离到45条MHC等位基因(22条Ⅰ类MHC等位基因和23条Ⅱ类MHC等位基因)。与已报道的Ⅰ类和Ⅱ类MHC等位基因进行比对,发现了3条新的等位基因,1条来自Aime-I,命名为Aime-I*08;2条来自Aime-L,命名Aime-L*08和Aime-L*09。对于Aime-I位点,新等位基因Aime-I*08与已报道的Aime-I*01~07的差异核苷酸个数为26~35,差异氨基酸个数为13~16,ABS的差异氨基酸个数为9~12。对于Aime-L位点,新等位基因Aime-L*08与已报道的Aime-L*01~07差异核苷酸个数、差异氨基酸个数、ABS的差异氨基酸个数分别为1~26、1~15、1~11;新等位基因Aime-L*09与已报道的Aime-L*01~07的差异核苷酸个数、差异氨基酸个数、ABS的差异氨基酸个数分别为2~17、1~9、0~7。Aime-L*08与Aime-L*02核苷酸序列相似度为99.8%(1个碱基差异),氨基酸序列相似度为99.5%(1个氨基酸差异且位于ABS上)。Aime-L*09与Aime-L*03、Aime-L*04均具有2个核苷酸差异(相似度99.6%)和1个氨基酸差异(相似度为99.5%)。但Aime-L*09与Aime-L*04具有完全相同的ABS(表 1;图 2)。
| Aime-I*08 | Aime-L*08 | Aime-L*09 | |||||||
| Nucl | AA | ABS | Nucl | AA | ABS | Nucl | AA | ABS | |
| 01 | 30 | 16 | 12 | 14 | 9 | 4 | 6 | 3 | 2 |
| 02 | 34 | 15 | 12 | 1 | 1 | 1 | 9 | 7 | 3 |
| 03 | 33 | 15 | 12 | 10 | 9 | 5 | 2 | 1 | 1 |
| 04 | 26 | 13 | 9 | 12 | 9 | 4 | 2 | 1 | 0 |
| 05 | 29 | 16 | 12 | 15 | 10 | 5 | 5 | 2 | 1 |
| 06 | 35 | 16 | 12 | 26 | 15 | 11 | 17 | 9 | 7 |
| 07 | 30 | 13 | 10 | 5 | 5 | 3 | 5 | 3 | 1 |
| 08 | 0 | 0 | 0 | 0 | 0 | 0 | 10 | 8 | 4 |
| 注:Nucl、AA、ABS分别表示等位基因核苷酸序列、氨基酸序列和抗原结合位点,长度分别为546 bp、182 bp和56 bp Notes:Nucl,AA,ABS represent nucleotide sequences,deduced amino acid sequences and antigen binding sites,respectively,and their length are 546 bp,182 bp and 56 bp,respectively | |||||||||
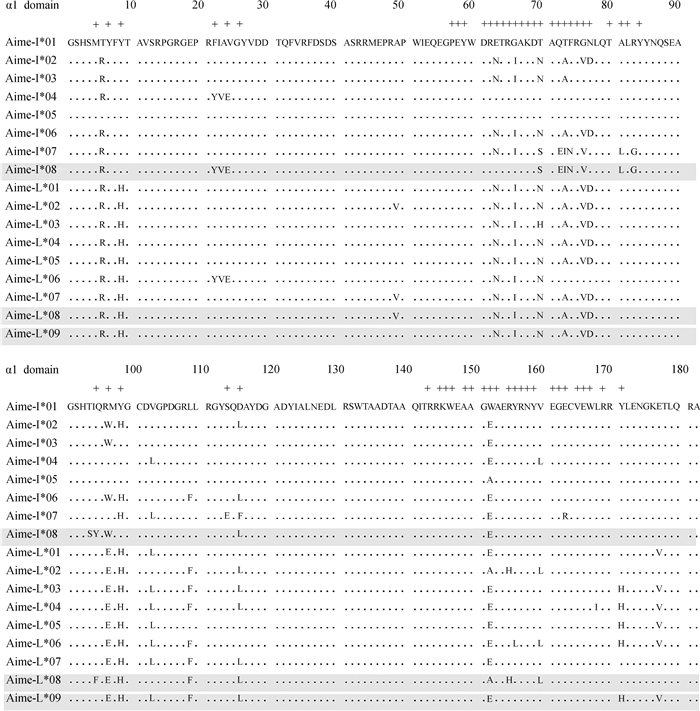
|
| 图 2 大熊猫Aime-I和Aime-L基因α1和α2结构域氨基酸序列比对结果 Fig. 2 Alignment of the amino acid sequences deduced from Aime-I and Aime-L genes 阴影部分表示本研究新分离到的等位基因,“.”表示该氨基酸与第一条序列一致,“+”表示推断的抗原结合位点 The light-grey regions indicate the new alleles isolated in this study, dots indicate identities to the first sequence, plus signs represent putative antigen binding sites |
| |
圈养大熊猫7个多态MHC基因的高频等位基因(频率>0.2)为:Aime-C*03、Aime-I*02、Aime-L*02和Aime-L*03、Aime-DQA1*01和Aime-DQA1*03、Aime-DQA2*01、Aime-DQB1*02和Aime-DQB1*04、Aime-DRB3*01和Aime-DRB3*09(表 2)。
| Aime-C (91) |
Aime-I (85) |
Aime-L (91) |
DQA1 (77) |
DQA2 (78) |
DQB1 (76) |
DRB3 (71) |
|
| 01 | 0.016 5 | 0.111 8 | 0.109 9 | 0.272 7 | 0.807 7 | 0.046 1 | 0.239 4 |
| 02 | 0.159 3 | 0.541 2 | 0.225 3 | 0.084 4 | 0.173 1 | 0.348 7 | — |
| 03 | 0.373 6 | 0.123 5 | 0.395 6 | 0.266 2 | 0.019 2 | 0.131 6 | 0.133 8 |
| 04 | 0.060 4 | 0.082 4 | 0.011 0 | 0.155 8 | — | 0.421 1 | 0.014 1 |
| 05 | 0.186 8 | — | 0.022 0 | 0.188 3 | — | — | 0.077 5 |
| 06 | 0.197 8 | 0.017 6 | 0.060 4 | 0.006 5 | — | 0.013 2 | — |
| 07 | 0.005 5 | 0.082 4 | — | 0.026 0 | — | 0.039 5 | 0.042 3 |
| 08# | — | 0.041 2 | 0.164 8 | — | — | — | 0.197 2 |
| 09# | — | — | 0.011 0 | — | — | — | 0.295 8 |
| 注:括号里面的数字表示该位点分型成功的个体数目; 粗体表示频率在0.2以上的等位基因; #本研究分离到的新等位基因 Notes:Figures in parentless indicate successful genotyped individuals; bolds indicate alleles frequencies larger than 0.2; # new alleles isolated in this study | |||||||
本研究利用直接测序法成功构建了91只大熊猫的7个多态MHC基因遗传档案,并分离得到3条新的Ⅰ类MHC等位基因。现有的大熊猫Ⅰ类MHC基因的单倍型分型采用单链构象多态性的方法,该技术虽然能检测1个碱基的突变,但是当突变的位置靠近引物上下游时,检测的灵敏度不佳。核苷酸序列比对结果显示,分离到的Aime-L*08与已报道的Aime-L*02相差1个碱基,且该差异碱基位于exon3的起始位置(靠近引物上游)。而Aime-L*09与Aime-L*04的2个差异碱基分别位于exon3的起始(靠近引物上游)和结束位置(靠近引物下游)。因此,推测Aime-L*08与Aime-L*09的单链多态性构象与Aime-L*02、04非常相似,导致这2条等位基因没有被单链构象多态性的方法检测出来。新等位基因Aime-I*08只在2只个体中被检测到,低频可能是导致Aime-I*08在其他种群中没有被检测到的原因。
单链构象多态性分型方法的良好分型效果仅限于100~300 bp短片段(单碱基变异检测率达到99%以上),长度大于400 bp的片段,20%左右的变异不能被检出(Sunnucks et al., 2000),不能满足Ⅰ类MHC基因exon2-exon3的单倍型的准确分型(约600 bp)。本研究采用的PCR产物直接测序法可以直接得到Ⅰ类MHC基因exon2-exon3的单倍型信息,不需要谱系进行辅助推断,而且不需要进行耗时耗力的电泳和银染工作。
直接测序法与单链构象多态性法相比,时间成本和劳动成本低,不需要掌握聚丙烯酰胺凝胶电泳及银染的显色技术,可检测出引物附近的碱基突变,可直接用于长片段的分型(Ⅰ类MHC基因的单倍型分型)。但是该方法适用于多数等位基因已被分离且序列已知、杂合子序列峰图易于区分的MHC研究。对于MHC各基因的等位基因已知的物种,如金丝猴(Luo & Pan,2013;Zhang et al., 2018)、林麝(Li et al., 2014;Yao et al., 2015)、红腹锦鸡(Zeng et al., 2016a, 2016b)、扬子鳄(Zhai et al., 2017;Han et al., 2019)和朱鹮(Lan et al., 2019;Sun et al., 2019),如果具备不同杂合子基因型峰图碱基易于区分的特点,也可采用直接测序法进行基因分型。随着测序技术的发展,能够自动化且进行长片段的精确分型技术,如Pacbio单分子实时测序将为MHC基因研究工作带来便利。
致谢: 感谢峨眉山方好濒危动物保育有限公司杨海琼博士、南京林业大学刘宏毅博士提出的宝贵建议。
Chen YY, Zhang YY, Zhang HM, et al. 2010. Natural selection coupled with intragenic recombination shapes diversity patterns in the major histocompatibility complex class Ⅱ genes of the giant panda[J]. Journal of Experimental Zoology Part B:Molecular and Developmental Evolution, 314(3): 208-223. |
Chen YY, Zhu Y, Wan QH, et al. 2013. Patterns of adaptive and neutral diversity identify the Xiaoxiangling Mountains as a refuge for the giant panda[J/OL]. PLoS ONE, 8(7): e70229[2020-01-20]. https://doi.org/10.1371/journal.pone.0070229.
|
Han QH, Sun RN, Yang HQ, et al. 2019. MHC class Ⅰ diversity predicts non-random mating in Chinese alligators (Alligator sinensis)[J]. Heredity, 122(6): 809-818. DOI:10.1038/s41437-018-0177-8 |
Klein J. 1986. The Natural history of the major histocompatibility complex[M]. New York: Wiley & Sons.
|
Kumar S, Stecher G, Li M, et al. 2018. MEGA X:molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
Lan H, Zhou T, Wan QH, et al. 2019. Genetic diversity and differentiation at structurally varying MHC haplotypes and microsatellites in bottlenecked populations of endangered crested ibis[J/OL]. Cells, 8(4): 377[2020-01-20]. https://doi.org/10.3390/cells.8040377.
|
Li L, Wang BB, Ge YF, et al. 2014. Major histocompatibility complex class Ⅱ polymorphisms in forest musk deer (Moschus berezovskii) and their probable association with purulent disease[J]. International Journal of Immunogenetics, 41(5): 401-412. DOI:10.1111/iji.12135 |
Luo M, Pan H. 2013. MHC Ⅱ DRB variation and trans-species polymorphism in the golden snub-nosed monkey (Rhinopithecus roxellana)[J]. Chinese Science Bulletin, 58(18): 2119-2127. DOI:10.1007/s11434-013-5713-6 |
Piertney SB, Oliver MK. 2006. The evolutionary ecology of the major histocompatibility complex[J]. Heredity, 96(1): 7-21. DOI:10.1038/sj.hdy.6800724 |
Rousset F. 2008. GENEPOP'007:a complete re-implementation of the GENEPOP software for Windows and Linux[J]. Molecular Ecology Resources, 8(1): 103-106. DOI:10.1111/j.1471-8286.2007.01931.x |
Sun L, Zhou T, Stone GN, et al. 2019. Seeing-good-gene-based mate choice:from genes to behavioural preferences[J]. Journal of Animal Ecology, 88(11): 1708-1719. DOI:10.1111/1365-2656.13071 |
Sunnucks P, Wilson ACC, Beheregaray LB, et al. 2000. SSCP is not so difficult:the application and utility of single-stranded conformation polymorphism in evolutionary biology and molecular ecology[J]. Molecular Ecology, 9(11): 1699-1710. DOI:10.1046/j.1365-294x.2000.01084.x |
Yao G, Zhu Y, Wan Q, et al. 2015. Major histocompatibility complex class Ⅱ genetic variation in forest musk deer (Moschus berezovskii) in China[J]. Animal Genetics, 46(5): 535-543. DOI:10.1111/age.12336 |
Zeng QQ, He K, Sun DD, et al. 2016a. Balancing selection and recombination as evolutionary forces caused population genetic variations in golden pheasant MHC class Ⅰ genes[J/OL]. BMC Evolutionary Biology, 16: 42[2020-01-20]. https://doi.org/10.1186/s12862-016-0609-0.
|
Zeng QQ, Zhong GH, He K, et al. 2016b. Molecular characterization of classical and nonclassical MHC class Ⅰ genes from the golden pheasant (Chrysolophus pictus)[J]. International Journal of Immunogenetics, 43(1): 8-17. DOI:10.1111/iji.12245 |
Zhai T, Yang H, Zhang R, et al. 2017. Effects of population bottleneck and balancing selection on the Chinese alligator are revealed by locus-specific characterization of MHC genes[J/OL]. Scientific Reports, 7: 5549[2020-01-20]. https://doi.org/10.1038/s41598-017-05640-2.
|
Zhang P, Huang K, Zhang B, et al. 2018. High polymorphism in MHC-DRB genes in golden snub-nosed monkeys reveals balancing selection in small, isolated populations[J/OL]. BMC Evolutionary Biology, 18: 29[2020-01-20]. https://doi.org/10.1186/s12862-018-1148-7.
|
Zhu Y, Grueber C, Li Y, et al. 2020. MHC-associated Baylisascaris schroederi load informs the giant panda reintroduction program[J]. International Journal for Parasitology:Parasites and Wildlife, 12: 113-120. DOI:10.1016/j.ijppaw.2020.05.010 |
Zhu Y, Liu HY, Yang HQ, et al. 2017. Factors affecting genotyping success in giant panda fecal samples[J/OL]. Peerj, 5: e3358[2020-01-20]. https://doi.org/10.7717/peerj.3358.
|
Zhu Y, Sun DD, Ge YF, et al. 2013a. Isolation and characterization of class Ⅰ MHC genes in the giant panda (Ailuropoda melanoleuca)[J]. Chinese Science Bulletin, 58(18): 2140-2147. DOI:10.1007/s11434-012-5582-4 |
Zhu Y, Wan QH, Yu B, et al. 2013b. Patterns of genetic differentiation at MHC class Ⅰ genes and microsatellites identify conservation units in the giant panda[J]. BMC Evolutionary Biology, 13(1): 227[2020-01-20]. https://doi.org/10.1186/1471-2148-13-227.
|
Zhu Y, Wan QH, Zhang HM, et al. 2019. Reproductive strategy inferred from major histocompatibility complex-based inter-individual, sperm-egg, and mother-fetus recognitions in giant pandas (Ailuropoda melanoleuca)[J]. Cells, 8(3): 257[2020-01-20]. https://doi.org/10.3390/cells8030257.
|
 2021, Vol. 40
2021, Vol. 40




