扩展功能
文章信息
- 张雪艳, 余浩洋, 孙天琳, 邱实, 蔡延森, 李静
- ZHANG Xueyan, YU Haoyang, SUN Tianlin, QIU Shi, CAI Yansen, LI Jing
- Alu家族在4种旧世界猴基因组中的特征
- Characteristics of Alu Family in Four Old World Monkey Genomes
- 四川动物, 2019, 38(6): 638-645
- Sichuan Journal of Zoology, 2019, 38(6): 638-645
- 10.11984/j.issn.1000-7083.20190159
-
文章历史
- 收稿日期: 2019-05-07
- 接受日期: 2019-07-04
转座子(transposable elements,TEs)是灵长目Primates动物基因组的重要组成,对基因功能、基因表达调控和基因组进化有重要影响(Feschotte,2008;Deininger,2011),根据序列结构特征,可以分为短散在重复序列(short interspersed elements,SINEs)、长散在重复序列(long interspersed elements,LINEs)、长末端重复序列(long terminal repeat,LTR)和DNA元件(Deininger et al., 2002;Wicker et al., 2007)。其中,Alu元件是灵长目动物特有的SINEs家族,也是灵长目基因组中进化最成功、数量最多的TEs,在人类基因组中超过100万个拷贝,占10.6%以上(Lander et al., 2001)。完整的Alu元件长约300 bp,需要借助另一种逆转座子L1编码的酶,通过逆转座作用在基因组中扩增(Dewannieux et al., 2003)。
基因组中绝大多数的Alu元件没有转座能力,只有少数source genes具有转座活性,可以进行自我复制,并插入到基因组的新位置(Deininger,2011)。这些source genes会随着宿主基因组的进化不断积累突变,从而衍生出新的Alu亚家族。根据碱基突变,可将Alu分为3个主要的亚家族:最古老的AluJ、中间的AluS和最年轻的AluY(Batzer & Deininger,2002;Han et al., 2007;Liu et al., 2009)。古老的Alu亚家族在灵长类物种分化前就存在于祖先基因组中,因此,在不同的物种中分布非常相似。而一些年轻的Alu元件在基因组中插入时间较短,尚未在基因组中固定下来,因而在不同物种间呈现出插入/缺失的状态(Deininger,2002)。研究Alu元件在不同灵长目动物基因组中的插入/缺失多态性,有助于阐明它们对灵长目基因组多样性的影响。
猴科Cercopithecidae即旧世界猴,是物种最丰富的灵长目类群,分为猕猴亚科Cercopithecinae和疣猴亚科Colobinae,它们在大约15百万年前由共同祖先分化而来(Perelman et al., 2011;Pozzi et al., 2014)。东非狒狒Papio anubis、猕猴Macaca mulatta和绿猴Chlorocebus sabaeus属于猕猴亚科,其中,东非狒狒主要分布于非洲,是狒狒属Papio中分布最广的一种;猕猴是猕猴属Macaca代表动物,是生物医学领域常用的模式动物(Han et al., 2007);绿猴在西非国家几乎都有分布,也被广泛应用于生物医学研究(Tchitchek et al., 2012)。长鼻猴Nasalis larvatus属于疣猴亚科,仅分布于亚洲东南部的加里曼丹岛,被世界自然保护联盟(IUCN)列为濒危(EN)物种(Meijaand et al., 2008)。随着高通量测序技术的迅速发展,这4种猴科动物的基因组序列都已公布,为全基因组水平的TEs研究奠定了基础。尽管这些猴科动物基因组公布时已有关于TEs的数量、频率等的分析,但它们在不同基因组之间的差异尚未揭示。本研究基于更新的Repbase数据库对东非狒狒、猕猴、绿猴和长鼻猴基因组中的TEs进行了重新注释,并比较了TEs的组成和分布特征,着重分析了灵长类特有的Alu元件在不同旧世界猴基因组中的插入/缺失多态性,旨在揭示TEs对旧世界猴基因组多样性和进化的影响。
1 材料与方法 1.1 基因组来源东非狒狒(GCF_000264685.2,2 724 327 674 bp)、猕猴(GCF_000772875.2,2 824 209 708 bp)、绿猴(GCF_000409795.2,2 503 700 750 bp)和长鼻猴(GCA_000772465.1,2 672 372 328 bp)的基因组序列下载于NCBI(http:/www.ncbi.nlm.nih.gov/)。
1.2 4种旧世界猴基因组TEs注释基因组公布时,不同研究者进行重复序列注释时所用的Repbase数据库不一致,不利于挖掘物种间的差异。因此,本研究基于更新的Repbase数据库,使用RepeatMasker(http://www.repeatmasker.org/)对东非狒狒、猕猴、绿猴和长鼻猴基因组的TEs进行分析,RepeatMasker的使用参数设置为“-e crossmatch- Pa 8-nolow species primates-s-gff”,其他为默认参数。将RepeatMasker的输出结果按照不同TEs的亚家族名称分为独立的文件,用来分析不同TEs的长度、占基因组比例、拷贝数和分歧率。
1.3 多态性Alu位点及物种特有Alu位点的鉴定通过基因组两两比较的方法,鉴定东非狒狒、猕猴、绿猴和长鼻猴基因组中存在的插入/缺失多态性的Alu逆转座子,其方法如下:
提取长度大于250 bp的Alu逆转座子及其上、下游各200 bp的侧翼序列。使用Blat将提取的序列分别比对到其他3个物种的基因组上,参数minIdentity设为95,即序列的长度和序列相似度都大于95%的序列时,认为这个位点是2个物种共有的位点。如果只有侧翼序列的相似度达到95%以上,但中间存在1段长度约300 bp的序列缺失,那么这个位点可能是潜在多态性的。
根据Blat比对结果,提取这些潜在插入/缺失多态性的位点上、下游各200 bp的侧翼序列,将侧翼序列连接起来,重新比对到目标基因组上,如果在该基因组上只能匹配到1个长度和序列相似度都大于95%的片段,则该位点为2个基因组上具有插入/缺失多态性的位点。
将基因组两两比较筛选的插入/缺失多态性的位点做韦恩图,找出在1个基因组中插入而在其他3个基因组中都缺失的位点,这样的位点即成为该物种特有的插入位点。
2 结果 2.1 4个旧世界猴基因组中TEs的基本特征东非狒狒、猕猴和绿猴基因组约47%(47.34%~ 47.96%)由TEs组成,而长鼻猴的仅36.12%(表 1)。长鼻猴各种类型的TEs都明显少于其他3个物种,这可能是由于其基因组是引导到猕猴染色体组装的,其3条染色体(21、22和23号染色体)的TEs尚未包含在内。LINEs是占比最高的TEs,在长鼻猴基因组中的占比为16.37%,在其他3个基因组中占比均超过20%;其次为SINEs和LTR元件;DNA元件占比最低。SINEs是拷贝数最丰富且平均分歧率最低的TEs。尽管长鼻猴的SINEs拷贝数最少,但其分歧率在4个基因组中却最大。
| 物种 | 项目 | SINEs | LINEs | LTR元件 | DNA元件 | 未分类 | 总数 |
| 东非狒狒 Papio anubis |
长度/Mb | 382.07 | 579.04 | 243.76 | 100.34 | 1.30 | 1 306.52 |
| 占基因组比例/% | 14.02 | 21.25 | 8.95 | 3.68 | 0.05 | 47.96 | |
| 拷贝数 | 1 665 608 | 958 592 | 489 121 | 421 280 | 7 251 | 3 541 852 | |
| 平均分歧率/% | 17.69 | 24.04 | 21.13 | 22.89 | — | — | |
| 猕猴 Macaca mulatta |
长度/Mb | 396.82 | 593.38 | 249.16 | 102.22 | 1.32 | 1 342.91 |
| 占基因组比例/% | 14.05 | 21.01 | 8.82 | 3.62 | 0.05 | 47.55 | |
| 拷贝数 | 1 713 347 | 970 759 | 497 511 | 427 630 | 7 354 | 3 616 601 | |
| 平均分歧率/% | 17.62 | 24.11 | 21.09 | 22.86 | — | — | |
| 绿猴 Chlorocebus sabaeus |
长度/Mb | 345.57 | 519.02 | 226.25 | 93.09 | 1.28 | 1 185.21 |
| 占基因组比例/% | 13.80 | 20.73 | 9.04 | 3.72 | 0.05 | 47.34 | |
| 拷贝数 | 1 523 121 | 897 164 | 458 414 | 391 329 | 8 625 | 3 278 653 | |
| 平均分歧率/% | 18.17 | 24.22 | 21.25 | 22.90 | — | — | |
| 长鼻猴 Nasalis larvatus |
长度/Mb | 251.53 | 437.54 | 194.11 | 81.10 | 1.12 | 965.40 |
| 占基因组比例/% | 9.41 | 16.37 | 7.26 | 3.03 | 0.04 | 36.12 | |
| 拷贝数 | 1 188 594 | 781 891 | 397 578 | 342 672 | 7 562 | 2 718 297 | |
| 平均分歧率/% | 19.44 | 24.30 | 21.24 | 22.97 | — | — |
4个基因组中,SINEs在分歧率为13%和30%左右存在2个峰,表明SINEs在这2个阶段存在活跃的转座活动。而LINEs、LTR和DNA元件都仅在分歧率为20%左右存在1个峰(图 1)。分歧率小于10%的TEs都来自SINEs和LINEs。此外,东非狒狒和猕猴TEs的分歧率分布非常相似,尤其是分歧率<5%的TEs数量明显多于绿猴和长鼻猴,其中主要是SINEs。
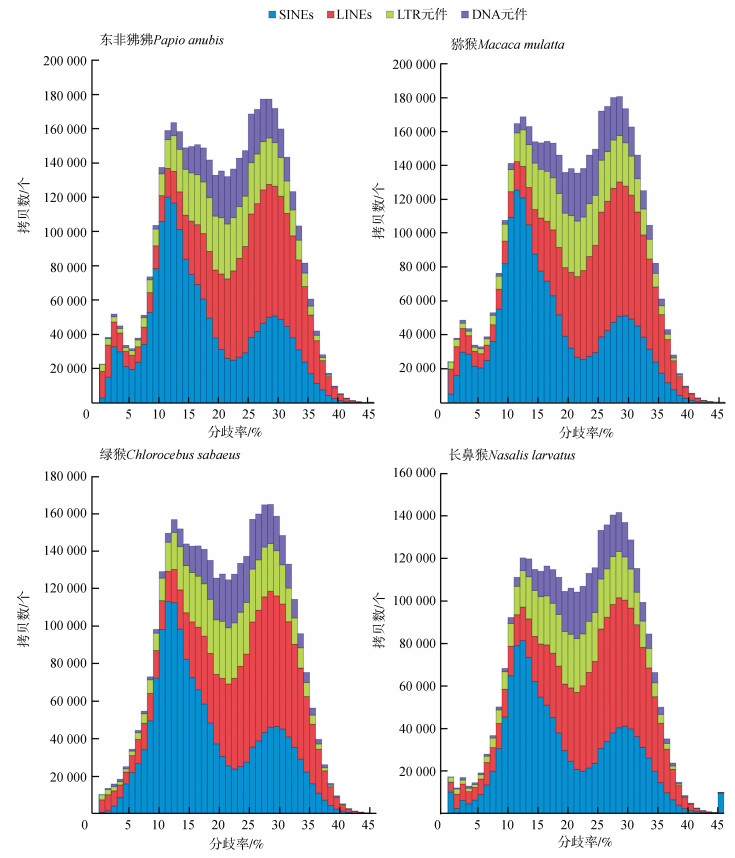
|
| 图 1 4个旧世界猴基因组中主要转座子的分歧率 Fig. 1 Divergence rates of major transposable elements in the genomes of the 4 Old World monkey species |
| |
东非狒狒、猕猴、绿猴和长鼻猴基因组中分别鉴定出1 140 275个、1 179 668个、1 037 140个和764 499个Alu元件,占总SINEs的64.32%~68.85%。去除长度≤250 bp的Alu元件后,分别保留了887 182个、936 449个、812 029个和562 850个Alu元件,其中AluS的数量远多于AluJ和AluY家族(表 2)。AluJ分为4个亚家族,其中,AluJb和AluJr较多;AluS包含17个亚家族,其中以AluSz、AluSx和AluSx1为主;AluY包含18个亚家族,其中AluY(未划分到其他特定亚家族的AluY元件)和AluYRa1最丰富。4个基因组中,猕猴的Alu元件比其他3个物种多,东非狒狒在各亚家族组成和数量上都与猕猴类似,而长鼻猴基因组AluJ、AluS和AluY家族的拷贝数明显少于其他3个物种。另外,4个物种在AluJ和AluS的组成和数量上的差异不大,而在AluY上有较大的差异。如东非狒狒和猕猴基因组中的AluY和AluYRa1数量远高于绿猴、长鼻猴,而绿猴基因组中的AluYRc0数量约为其他3个物种的2倍;长鼻猴基因组中的AluYRa2几乎为其他3个物种的3倍(图 2)。
| 元件 | 拷贝数/个 | |||
| 东非狒狒 Papio anubis |
猕猴 Macaca mulatta |
绿猴 Chlorocebus sabaeus |
长鼻猴 Nasalis larvatus |
|
| AluJ | 173 688 | 180 313 | 163 401 | 123 117 |
| AluS | 523 191 | 550 845 | 498 232 | 336 607 |
| AluY | 190 303 | 205 291 | 150 396 | 103 126 |
| 总数 | 887 182 | 936 449 | 812 029 | 562 850 |
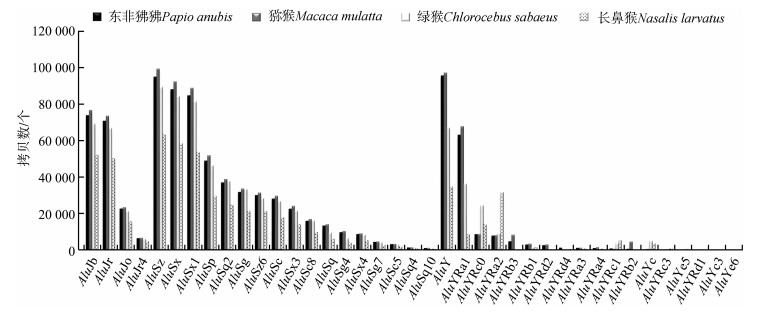
|
| 图 2 4个旧世界猴基因组中≥250 bp的AluJ、AluS和AluY家族的亚家族分布 Fig. 2 The subfamily distribution of AluJ, AluS and AluY families with length of ≥250 bp in the genomes of 4 Old World monkey species |
| |
在东非狒狒基因组插入而在猕猴基因组同源位置无Alu插入的位点共20 231个,是4个基因组间多态性位点最多的。插入长鼻猴基因组而在其他任意一个基因组中缺失的位点数目均远低于其他3个基因组之间的多态性位点。东非狒狒与猕猴2个基因组间多态性位点数量大致相当;与之不同,插入东非狒狒和猕猴基因组,而在绿猴基因组缺失的位点分别有18 371个和17 684个。插入绿猴基因组而在东非狒狒和猕猴基因组中缺失的位点数仅有7 397个和7 646个(表 3)。
| 物种 | 东非狒狒- | 猕猴- | 绿猴- | 长鼻猴- | 物种特有的Alu位点 | |||
| 总数 | AluJ | AluS | AluY | |||||
| 东非狒狒+ | — | 20 231 | 18 371 | 6 902 | 2 321 | 0 | 7 | 2 314 |
| 猕猴+ | 19 289 | — | 17 684 | 6 818 | 2 228 | 1 | 5 | 2 222 |
| 绿猴+ | 7 397 | 7 646 | — | 3 047 | 1 202 | 1 | 16 | 1 185 |
| 长鼻猴+ | 3 630 | 3 688 | 3 138 | — | 2 131 | 1 | 100 | 2 030 |
| 注:+表示在该物种同源区域有Alu插入,-表示在该物种同源区域有Alu缺失;下同 Notes:+ represents that there is an Alu insertion in the homologous region of the species,- represents the absence of Alu in the homologous region of the species;the same below | ||||||||
基于基因组间比对的结果,进一步鉴定了各物种特有的Alu插入(species specific Alu,SSA),即该元件仅插入某物种,而在其他3个基因组的同源区域都缺失的位点(图 3)。共获得了7 882个SSA位点,包括东非狒狒2 321个、猕猴2 228个、绿猴1 202个以及长鼻猴2 131个。长鼻猴基因组中的多态性位点中约85%以上都是特有的,而其他3个基因组的比例均未超过13%(图 3)。研究这些特有Alu亚家族发现,95%以上的SSA位点都属于AluY亚家族,猕猴亚科3个物种的SSA主要属于AluYRa1亚家族,而长鼻猴的主要属于AluYRa2亚家族(图 4)。猕猴中分别有309个和364个位点属于AluYRb2和AluYRb3亚家族,明显多于其他3个物种。长鼻猴和绿猴中AluYc亚家族分别有376个和182个,多于东非狒狒(10个)和猕猴(2个)(图 4)。此外,3个猕猴亚科动物中,AluS和AluJ的数量很少,而长鼻猴中有100个位点属于AluS(表 3)。
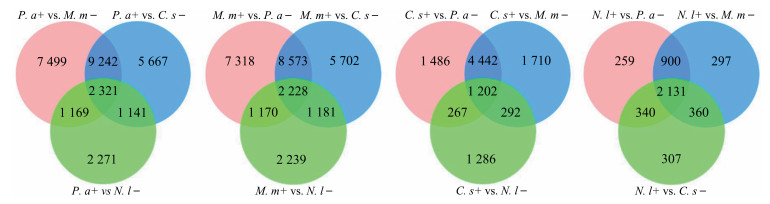
|
| 图 3 4个旧世界猴物种特有的Alu插入位点 Fig. 3 Species-specific Alu insertions in 4 Old World monkey species P.a.东非狒狒Papio anubis,M.m.猕猴Macaca mulatta,C.s.绿猴Chlorocebus sabaeus,N.l.长鼻猴Nasalis larvatus |
| |
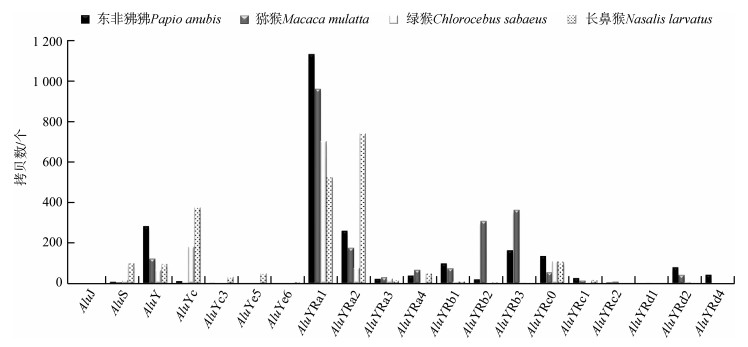
|
| 图 4 4个旧世界猴物种特有的Alu位点上的亚家族分布 Fig. 4 Subfamily distribution of the species-specific Alu insertions in 4 Old World monkey species |
| |
Han等(2007)在全基因组水平揭示了猕猴TEs的组成和进化,并鉴定了旧世界猴特有的4个AluY亚家族,分别命名为AluYRa~AluYRd,并认为它们是旧世界猴中最年轻、最活跃的TEs。虽然许多旧世界猴基因组已被报道,但是关于这些基因组在TEs组成及分布上的差异,以及TEs对旧世界猴基因组多样性的影响的研究较少。本研究对4种旧世界猴基因组中的TEs进行了分析和比较,包括1个疣猴亚科物种(长鼻猴)和3个猕猴亚科物种(东非狒狒、猕猴和绿猴),其中,东非狒狒和猕猴同属于狒狒族。结果显示,4个基因组中TEs的组成和分布与它们的进化关系吻合,东非狒狒和猕猴的TEs特征相似,二者又与绿猴、长鼻猴有较大差异,尤其是新近整合到基因组上的TEs(分歧率<10%),其数量显著多于绿猴和长鼻猴。分歧率是根据TEs序列中累积的突变位点计算其与Repbase数据库中一致序列的分化程度。一般说来,TEs上累积的突变越多,分歧率就越大,表明其插入基因组的时间越长,是更古老的TEs,反之亦然(Peng et al., 2018)。分歧率<10%的TEs都来自SINEs和LINEs,表明这两类转座子中都包含许多相对年轻的转座元件,这与已经报道的其他灵长目动物基因组中情况一致(Konkel et al., 2007;Mclain et al., 2013)。进一步分析显示,这种差异的重要来源是灵长类特有的Alu元件的差异,尤其是最年轻的AluY家族,在4个旧世界猴基因组中差异显著。东非狒狒和猕猴基因组中AluY和AluYRa1亚家族数量远高于绿猴、长鼻猴,这2个亚家族可能是狒狒族中转座最活跃的。与之不同,绿猴基因组中AluYRc0的数量约为其他物种的2倍;长鼻猴基因组中AluYRa2的数量是其他物种的3倍,这与AluYRa2亚家族在长鼻猴中的拷贝数显著多于其他3个物种的结果一致。本研究结果表明,随着灵长目动物的进化和物种分化,各Alu亚家族在不同物种中的扩增活性存在较大差异,从而累积不同的拷贝数,而正是由于Alu特定亚家族在基因组中扩增的差异,塑造了不同灵长目动物基因组的多样性。
灵长目动物特有的Alu家族随着灵长目动物的进化而进化,其扩增活性在进化历史的不同阶段不相同(Konkel et al., 2010)。AluJ的转座高峰约在灵长目动物分化早期,目前绝大部分AluJ已不具备转座活性,固定在各灵长目动物基因组中;AluS是由AluJ家族分化而来,在大约45百万年前大量扩增;AluY是最晚整合到灵长目动物基因组的,因此一些AluY在人类基因组中仍具有转座活性(Roy-Engel et al., 2008)。由于这些元件在距今较短的时间插入基因组,尚未在基因组中固定下来,因此在不同物种基因组中具有插入/缺失多态性。这也是构成灵长目动物基因组多样性的重要因素,同时这些多态性的位点也可作为遗传标记,用于揭示不同灵长目动物之间的系统发育关系(Salem et al., 2003;Ray et al., 2005)。通过4个旧世界猴基因组的两两比较,我们鉴定了大量在不同基因组间具有插入/缺失多态性的Alu位点。值得注意的是,除去组装质量不高的长鼻猴外,同为猕猴亚科的3个物种,东非狒狒或猕猴基因组插入而在绿猴基因组缺失的位点数是绿猴基因组插入而在东非狒狒或猕猴中缺失的位点数的2倍多。这一结果提示在狒狒族与其他旧世界猴分化后,Alu在基因组中的扩增活性显著高于绿猴,从而累积了更多的拷贝。我们进一步鉴定了4个旧世界猴物种特有的7 882个Alu位点,它们是不同物种分化之后整合到基因组的最年轻的元件。和预期一致,这些位点超过95%来源于AluY家族。然而我们仍然发现长鼻猴SSA中有100个AluS元件,这表明在长鼻猴所属的疣猴亚科和猕猴亚科分化时一些AluS元件仍然存在转座活性,从而在疣猴亚科动物基因组中特异地扩增。也正因为疣猴亚科的长鼻猴与其他3个猕猴亚科物种的分歧较早,因此基因组也存在更多的特有插入。这些物种特有的插入在不同灵长目动物中来源的亚家族不同,3个猕猴亚科物种SSA数量最多的都属于AluYRa1,而长鼻猴则属于AluYRa2,这表明这2个亚家族可能在不同旧世界猴基因组中仍然存在转座活性。
综上,本研究比较了4种旧世界猴TEs的组成与分布,尤其是灵长目动物特有的Alu家族的特征。并且揭示了AluY家族的转座活动对于旧世界猴基因组的进化及多样性具有重要意义。鉴定的插入/缺失多态性位点和物种特有的Alu位点也为今后开展系统进化以及种群遗传学研究提供了丰富的遗传标记。
Batzer MA, Deininger PL. 2002. Alu repeats and human genomic diversity[J]. Nature Reviews Genetics, 3(5): 370-379. DOI:10.1038/nrg798 |
Deininger PL, Batzer MA. 2002. Mammalian retroelements[J]. Genome Research, 12(10): 1455-1465. DOI:10.1101/gr.282402 |
Deininger PL. 2011. Alu elements:know the SINEs[J]. Genome Biology, 12(12): 236. DOI:10.1186/gb-2011-12-12-236 |
Dewannieux M, Esnault C, Heidmann T. 2003. LINE-mediated retrotransposition of marked Alu sequences[J]. Nature Genetics, 35(1): 41-48. DOI:10.1038/ng1223 |
Feschotte C. 2008. Transposable elements and the evolution of regulatory networks[J]. Nature Reviews Genetics, 9(5): 397-405. DOI:10.1038/nrg2337 |
Han K, Konkel MK, Xing JC, et al. 2007. Mobile DNA in Old World monkeys:a glimpse through the rhesus macaque genome[J]. Science, 316(5882): 238-240. |
Konkel MK, Walker JA, Batzer MA. 2010. LINEs and SINEs of primate evolution[J]. Evolutionary Anthropology:Issues, News, and Reviews, 19(6): 236-249. DOI:10.1002/evan.20283 |
Konkel MK, Wang J, Liang P, et al. 2007. Identification and characterization of novel polymorphic LINE-1 insertions through comparison of two human genome sequence assemblies[J]. Gene, 390(1-2): 28-38. DOI:10.1016/j.gene.2006.07.040 |
Lander ES, Linton LM, Birren B, et al. 2001. Initial sequencing and analysis of the human genome[J]. Nature, 409(6822): 860-921. DOI:10.1038/35057062 |
Liu GE, Alkan C, Jiang L, et al. 2009. Comparative analysis of Alu repeats in primate genomes[J]. Genome Research, 19(5): 876-885. DOI:10.1101/gr.083972.108 |
Mclain AT, Carman GW, Fullerton ML, et al. 2013. Analysis of western lowland gorilla (Gorilla gorilla gorilla) specific Alu repeats[J]. Mobile DNA, 4(1): 26. DOI:10.1186/1759-8753-4-26 |
Meijaard E, Nijman V, Supriatna J. 2008. Nasalis larvatus [DB/OL]. [2019-01-30]. The IUCN Red List of Threatened Species 2008: e. T14352A4434312. http://dx.doi.org/10.2305/IUCN.UK.2008.RLTS.T14352A4434312.en.
|
Peng CJ, Niu LL, Deng JB, et al. 2018. Can-SINE dynamics in the giant panda and three other Caniformia genomes[J]. Mobile DNA, 9(1): 32. DOI:10.1186/s13100-018-0137-0 |
Perelman P, Johnson WE, Roos C, et al. 2011. A molecular phylogeny of living primates[J]. PLoS Genetics, 7(3): e1001342. DOI:10.1371/journal.pgen.1001342 |
Pozzi L, Hodgson JA, Burrell AS, et al. 2014. Primate phylogenetic relationships and divergence dates inferred from complete mitochondrial genomes[J]. Molecular Phylogenetics and Evolution, 75: 165-183. DOI:10.1016/j.ympev.2014.02.023 |
Ray DA, Xing JC, Hedges DJ, et al. 2005. Alu insertion loci and platyrrhine primate phylogeny[J]. Molecular Phylogenetics and Evolution, 35(1): 117-126. DOI:10.1016/j.ympev.2004.10.023 |
Roy-Engel AM, Batzer MA, Deininger PL. 2008. Evolution of human retrosequences: Alu[M]// Langley RJ, Renno T. Encyclopedia of life sciences. Chichester: John Wiley & Sons, Ltd.
|
Salem AH, Ray DA, Xing JC, et al. 2003. Alu elements and hominid phylogenetics[J]. Proceedings of the National Academy of Sciences of the United States of America, 100(22): 12787-12791. DOI:10.1073/pnas.2133766100 |
Tchitchek N, Jacquelin B, Wincker P, et al. 2012. Expression sequence tag library derived from peripheral blood mononuclear cells of the Chlorocebus sabaeus[J]. BMC Genomics, 13: 279. DOI:10.1186/1471-2164-13-279 |
Wicker T, Sabot F, Hua-Van A, et al. 2007. A unified classification system for eukaryotic transposable elements[J]. Nature Reviews Genetics, 8(12): 973-982. DOI:10.1038/nrg2165 |
Zhou X, Wang B, Pan Q, et al. 2014. Whole-genome sequencing of the snub-nosed monkey provides insights into folivory and evolutionary history[J]. Nature Genetics, 46(12): 1303-1310. DOI:10.1038/ng.3137 |
 2019, Vol. 38
2019, Vol. 38




