扩展功能
文章信息
- 王志红, 王超, 彭锐
- WANG Zhihong, WANG Chao, PENG Rui
- 日本三角涡虫Caspase-7基因的克隆及功能研究
- Cloning and Functional Analysis of Caspase-7 Gene from Dugesia japonica
- 四川动物, 2019, 38(1): 11-19
- Sichuan Journal of Zoology, 2019, 38(1): 11-19
- 10.11984/j.issn.1000-7083.20180133
-
文章历史
- 收稿日期: 2018-04-26
- 接受日期: 2018-05-24
日本三角涡虫Dugesia japonica属于扁形动物门Platyhelminthes涡虫纲Turbellaria,因其强大的再生能力成为研究再生的最佳模式生物之一(Rink,2013;Mangel et al., 2016)。对于任何类型的创伤,涡虫几乎都能够完成修复和再生,被誉为是“切不死的动物”(Tan et al., 2012)。涡虫强大的再生能力源于其体内成体干细胞的增殖与分化(Wagner et al., 2011;DeHaan & Ebert,2015)。涡虫在再生过程中不仅有新组织的产生,创伤后剩余的旧组织(先存组织)还会通过细胞死亡和增殖的相互协调进行身体重塑,以适应身体的比例(Baguñá & Romero,1981),因此,在涡虫再生过程中,细胞凋亡可能起到了重要的作用。
细胞凋亡是细胞死亡的一种形式,用来消除机体中不需要的细胞,从而调节组织内的平衡(Jang et al., 2007)。细胞凋亡主要由Caspase介导。Caspase是一种在多细胞生物中高度保守的含半胱氨酸的天冬氨酸蛋白水解酶,其功能已被广泛研究(郭素英等,2009),但是Caspase家族在涡虫中的研究还很少。有学者从涡虫cDNA文库中鉴定出了Caspase-like gene 1、2、3,分别命名为Djclg-1、Djcl-2g、Djclg-3,并发现只有Djclg-3基因的表达量较高,而Djclg-3基因在控制涡虫体细胞数量、消除不必要的组织或者细胞,以及在组织器官重塑过程中起到了重要作用,这是第一次对涡虫凋亡相关基因的报道(Hwang et al., 2004)。Caspase-7与Caspase-3同为Caspase家族中细胞凋亡的重要执行者,执行蛋白质水解的协调程序,并且在癌症的发生和进展中起重要作用(Thomsen et al., 2013),除了参与细胞的调控外,还参与非凋亡活动的调控,如调节细胞的增殖、分化等(Park et al., 2018),但在涡虫体内的作用还未见报道。
本文以本实验室转录组测序所获得的涡虫Caspase-7基因表达序列标签为基础,通过cDNA末端快速扩增(rapid amplification of cDNA ends,RACE)技术克隆得到了该基因的mRNA全序列并分析了其结构特点。同时还研究了干扰Caspase-7基因表达对涡虫再生的影响。
1 实验与方法 1.1 实验材料日本三角涡虫由清华大学吴畏教授实验室提供。室温下以饮用水饲养,每天换水1次,并隔1天喂食猪肝1次。
1.2 实验试剂本实验中所用rTaq DNA聚合酶、RNAiso、SMARTerTM RACE cDNA Amplification Kit购于TaKaRa公司;琼脂糖凝胶回收试剂盒购于天根生化科技(北京)有限公司;实时荧光定量PCR(quantitative real-time PCR,qPCR)Master Mix购于Innovagene公司;引物合成及测序由北京六合华大基因科技有限公司完成。
1.3 实验方法 1.3.1 Caspase-7基因的克隆根据SMARTerTM RACE cDNA Amplification Kit说明书制备cDNA模板,利用SMART通用引物和Caspase-7-RACE-F1/R1引物(表 1)进行巢式PCR第一轮扩增。然后利用巢式PCR通用引物和Caspase-7-RACE-F2/R2引物(表 1)进行第二轮扩增。琼脂糖凝胶电泳检测PCR产物,并切胶回收与预期片段大小一致的条带,将测序结果拼接,与转录组测序结果比对。
| 引物名称 | 引物序列(5'-3') |
| Caspase-7-RACE-F1 | CATGTAAAGGACGGGATTTGAATC |
| Caspase-7-RACE-F2 | GTCAATAGAAACATGAGCATC |
| Caspase-7-RACE-R1 | CAATAATATGGATACTGTCGATGC |
| Caspase-7-RACE-R2 | CGGATTAAATTTATCACCTCGTTC |
| Caspase-7-qPCR-F | TTTACCCGAACGAGAAT |
| Caspase-7-qPCR-R | TGGCGTAATAGTTCAAATGG |
| β-actin-qPCR-F | TGCTCCTGAAGAACATCC |
| β-actin-qPCR-R | CAGTTGTTCGACCCGATG |
| Caspase-7-PCR-F1 | TAATACGACTCACTATAGGG ATTTAGTATCTCCGGTATTGG |
| Caspase-7-PCR-R1 | TAATACGACTCACTATAGGT GGTCGTGATGCTCATGTTTCTA |
| Caspase-7-PCR-F2 | TAATACGACTCACTATA GGTGAGCATCACGACCATA |
| Caspase-7-PCR-R2 | TAATACGACTCACTATA GGTTTATCACCTCGTTCTGG |
| Djclg-3-qPCR-F | GAAAATAAGAGTGCAGAGGCGATGG |
| Djclg-3-qPCR-R | CCGTCAAACCCAGATCAGTCCGAAC |
| PCNA-qPCR-F | ACCTATCGTGTCACTGTCTTTGACCGAAAA |
| PCNA-qPCR-R | TTCATCATCTTCGATTTTCGGAGCCAGATA |
利用MEGA 6对5'-RACE和3'-RACE结果进行拼接;利用DNAMAN将核苷酸序列翻译为氨基酸后,与其他19个物种(表 2)的Caspase-7氨基酸序列进行同源性分析;利用SWISS-MODEL网站在线预测涡虫Caspase-7蛋白三维结构;最后利用MEGA 6中的邻接法进行Caspase-7系统发育树的重建,选择氨基酸取代模型,并采用自举检验法(bootstrap test,重抽样1 000次)评估系统发育树中各节点的置信度。
| 物种 | GenBank登录号 |
| 大熊猫Ailuropoda melanoleuca | XP_011230112.1 |
| 家犬Canis lupus familiaris | XP_005637795.1 |
| 家猫Felis catus | XP_019669393.1 |
| 东方蝾螈Cynops orientalis | AFN55259.1 |
| 鲤鱼Cyprinus carpio | XP_018968723.1 |
| 青鳉Oryzias latipes | XP_023805391.1 |
| 美丽硬仆骨舌鱼Scleropages formosus | KPP76042.1 |
| 热带爪蟾Xenopus tropicalis | NM_001016299.2 |
| 水螅Hydra vulgaris | NM_001280824.1 |
| 果蝇Drosophila navojoa | XM_018113683.1 |
| 曼氏血吸虫Schistosoma mansoni | FJ905906.1 |
| 埃及血吸虫Schistosoma haematobium | XM_012944010.1 |
| 长牡蛎Crassostrea gigas | NM_001305322.1 |
| 小马岛猬Echinops telfairi | XP_004701336.1 |
| 川金丝猴Rhinopithecus roxellana | XP_010361523.1 |
| 小家鼠Mus musculus | NM_007611.2 |
| 中国仓鼠Cricetulus griseus | XM_003515184.3 |
| 马Equus caballus | XP_014588811.1 |
| 山羊Capra hircus | XP_005698554.1 |
分别提取切后再生0 h、6 h和1~3 d涡虫的RNA,以反转录后的cDNA为模板,通过qPCR方法检测Caspase-7基因在不同时间段的表达量,引物序列参见表 1。以0 h为对照,β-actin为内参。每组设置3个重复。qPCR体系:2×qPCR Master mix 5 μL,cDNA 0.5 μL,Caspase-7 -qPCR正、反向引物各0.3 μL,ddH2O 3.9 μL。qPCR反应条件:95 ℃ 10 min;95 ℃ 15 s,60 ℃退火并延伸60 s,39个循环。结果按2-ΔΔCt法进行计算分析并绘图。
1.3.4 Caspase-7 dsRNA体外转录及干扰用2对带有T7启动子的Caspase-7 PCR引物(表 1)从涡虫cDNA中扩增得到Caspase-7不同区域的DNA,并以此为模板,利用T7 RNA聚合酶在37 ℃体外转录获得干扰用的2条Caspase-7 dsRNA。选取40条长度约1 cm的涡虫,平均分为2组。一组将制备好的2条Caspase-7 dsRNA与涡虫食物混合,然后喂食涡虫,以干扰涡虫体内Caspase-7基因的表达,作为Caspase-7干扰组;另一组将DEPC水与涡虫食物混合并喂食涡虫,作为对照组。Caspase-7干扰组与对照组都隔1天喂食1次,共喂6次,具体方法参见Rouhana等(2013)。
1.3.5 Caspase-7干扰效率检测及干扰后表型观察在对涡虫进行第6次喂食dsRNA后的第4天,涡虫体内dsRNA被消化后(Rouhana et al., 2013),分别提取对照组和Caspase-7干扰组的RNA,反转录成cDNA,利用Caspase-7 qPCR引物检测各组Caspase-7基因的表达量。另外,在第6次喂食后的第3天,将涡虫分为2组:一组于涡虫咽前部位进行切割,另一组不切割,然后每天观察并拍照记录涡虫形态变化。2组涡虫在观察表型的过程中均不喂食。
1.3.6 Caspase-7干扰后Djclg-3及PCNA基因表达变化检测Caspase-7干扰组与对照组涡虫在不切割直接饥饿7 d后,提取RNA,并反转录成cDNA,分别利用Djclg-3基因、PCNA基因和β-actin qPCR引物(表 1),通过qPCR技术检测Djclg-3基因与PCNA基因的表达水平。以β-actin为内参,每组设置3个重复。结果按2-ΔΔCt法进行计算分析并绘图。
2 结果 2.1 Caspase-7基因全长克隆及序列分析对5'-RACE结果中约250 bp的片段(图 1:a中箭头所指)和3'-RACE结果中约770 bp的片段(图 1:b中箭头所指)进行琼脂糖凝胶回收并测序验证,将5'-RACE和3'-RACE所得序列进行拼接,得到涡虫Caspase-7基因全长(图 2;GenBank登录号:MH256116)。该基因全长935 bp,编码251个氨基酸,5'-UTR区长86 bp,3'-UTR区长93 bp。

|
| 图 1 涡虫Caspase-7基因RACE扩增结果 Fig. 1 RACE amplification of planarian Caspase-7 gene a. 5'-RACE,b. 3'-RACE,1~4. 4个重复组,Marker. DL2000 a. 5'-RACE, b. 3'-RACE, 1-4. 4 replicates, Marker. DL2000 |
| |

|
| 图 2 涡虫Caspase-7基因全长序列 Fig. 2 The complete sequence of planarian Caspase-7 gene 编码序列上方为Caspase-7基因结构示意图(共935 bp),其中,左右两端白色区域分别代表 5'-UTR(86 bp)和3'-UTR(93 bp)区,中间阴影部分表示编码区(CDS,756 bp),CDS区两端黑色和灰色部分分别代表ATG起始密码子和TAA终止密码子;碱基序列下方为每个密码子编码的氨基酸,*终止密码子 Upper: a schematic representation of the planarian Caspase-7 gene (935 bp in total), the white regions at the ends of 2 sides represent the 5'-UTR (left, 86 bp) and 3'-UTR (right, 93 bp) regions, respectively, the middle shadow region indicates the coding sequence (CDS, 756 bp), the black and gray portions at both ends of the CDS region represent the ATG start codon and the TAA stop codon, respectively; lower: the complete sequence of planarian Caspase-7 mRNA, amino acids encoded by each codon are shown under the nucleotide sequences, and * represents the stop codon |
| |
为进一步确认所得序列为涡虫Caspase-7基因序列,将克隆得到的日本三角涡虫Caspase-7氨基酸序列与其他19个物种进行同源比对,发现Caspase家族中2大保守而关键的结构域LSHG和QAC- (Q/R)G(Hwang et al., 2004)也存在于涡虫Caspase-7基因(图 3)中。在这2大结构域中,组氨酸和半胱氨酸是催化活性中心的重要残基。

|
| 图 3 涡虫Caspase-7与其他物种同源比对部分结果 Fig. 3 Homology comparison of planarian Caspase-7 and other species a. Caspase-7的结构特征,b.同源序列比对;黑色框线和灰色框线为Caspase家族2个保守的关键结构域 a. structural characteristics of the Caspase-7, b. homologous sequence alignment; the 2 key domains conserved in the Caspase family are shown in the black frame and gray frame, respectively |
| |
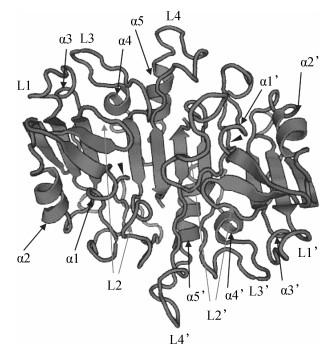
根据克隆所得的涡虫Caspase-7氨基酸序列预测得到了其三维结构(图 4),其中包含2个催化单位,每个催化单位都具有5个α螺旋和6个β折叠;涡虫Caspase-7的潜在催化位点由4个表面环(L1~L4)构成;另外,涡虫Caspase-7蛋白酶活性中心的关键残基——半胱氨酸残基(图 4:黑色三角形所指)位于L2环上。
|
| 图 4 涡虫Caspase-7蛋白三维结构的预测 Fig. 4 Predicted three-dimensional structure of planarian Caspase-7 protein α1~α5(α1'~α5')分别代表 5个α螺旋;L1~L4(L1'~L4')分别代表 4个表面环;2个黑色三角形所指为半胱氨酸残基所在位点 α1-α5 (α1'-α5') represent 5 alpha helices, respectively; L1-L4 (L1'-L4') represent 4 surface loops, respectively; two black triangles point to the cysteine site, key amino acid residue in active center |
| |
进化树显示,涡虫Caspase-7基因和同为扁形动物门的曼氏血吸虫Schistosoma mansoni及埃及血吸虫S. haematobium没有较近的亲缘关系,而与刺胞动物门Cnidaria水螅Hydra vulgaris有最近的亲缘关系(图 5),说明不同物种之间Caspase-7基因可能发生了趋同进化。

|
| 图 5 涡虫与其他物种Caspase-7基因进化树 Fig. 5 Phylogenetic analysis of Caspase-7 gene between planarian and other species |
| |
为分析Caspase-7基因是否参与涡虫细胞凋亡调控,通过qPCR检测涡虫切后再生不同时间段Caspase-7基因的表达量变化(图 6)。Caspase-7基因在涡虫切后再生6 h轻微升高,在涡虫切后再生3 d升高明显(P<0.05),其余时间点变化不明显。

|
| 图 6 涡虫切后再生不同时间段Caspase-7基因表达量变化 Fig. 6 The expression changes of Caspase-7 gene at different times during planarian regeneration * P<0.05 |
| |
为了进一步分析Caspase-7基因在涡虫中的作用,通过喂食Caspase-7 dsRNA干扰涡虫体内Caspase-7基因的表达。qPCR结果显示,喂食Caspase-7 dsRNA能成功干扰涡虫体内的Caspase-7基因的表达(图 7:a,P<0.01)。Caspase-7干扰组涡虫切后的头部片段(5/5)和尾部片段(5/5)均能完成再生,说明干扰Caspase-7并不影响涡虫的再生。但在Caspase-7干扰组涡虫切后的第11天,其头部及尾部发生溶解(5/5)(图 7:d),而此时对照组涡虫形态仍然正常(5/5)。此外,Caspase-7干扰组未切割涡虫在饥饿7 d后(图 7:c),头部和尾部也发生部分溶解(5/5),而对照组正常(5/5)。这说明干扰Caspase-7的表达对饥饿环境下的成体涡虫(再生完成后的或未切割的成虫)会造成明显的影响,导致虫体发生溶解。

|
| 图 7 干扰Caspase-7对涡虫的影响 Fig. 7 Effect of Caspase-7 interference on planarian growth a. Caspase-7干扰检测结果,b.涡虫于咽前切后再生头部第6天的表型,c.未切割涡虫饥饿第7天的表型,d.涡虫于咽前切后再生头部第11天的表型;** P<0.01 a. the Caspase-7 interference efficiency, b. the regenerative planarian heads after 6 days of amputation, c. the planarians after starvation for 7 days, d. the regenerative planarian heads after 11 days of amputation; ** P < 0.01 |
| |
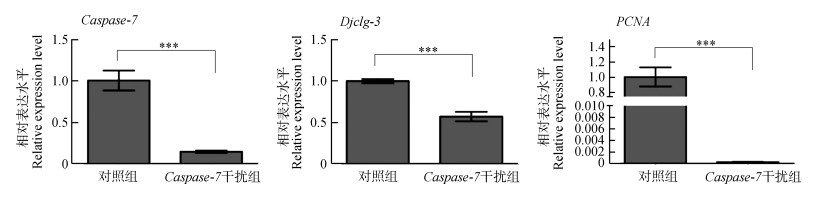
Caspase-7干扰组涡虫在饥饿环境下发生溶解时,检测细胞凋亡的另一关键基因Djclg-3以及增殖相关基因PCNA的表达变化。qPCR结果显示,成体涡虫饥饿处理7 d后,Djclg-3和PCNA基因的表达量相较于对照组显著降低(P<0.001),即细胞凋亡与增殖大幅度减少(图 8)。
|
| 图 8 Caspase-7干扰后各基因表达变化 Fig. 8 Gene expression changes after Caspase-7 interference *** P<0.001 |
| |
Caspase-7是Caspase家族的重要成员之一,在细胞凋亡过程中发挥着促凋亡作用。未激活的Caspase-7以酶原的形式存在,一旦被上游分子剪切激活后,活化的Caspase-7可以对其底物进行切割,导致细胞凋亡发生(Hill et al., 2016)。细胞凋亡的缺失会导致多种癌症的发生(Putt et al., 2006;Wolan et al., 2009;Thomsen et al., 2013)。此外,Caspase-7还在炎症反应、神经激活和细胞分化中起着重要的作用(Martinon & Tschopp,2007;Kuranaga,2012)。Caspase-7不仅参与凋亡的调控,还参与调节细胞周期、细胞增殖及分化等(Chaudhary et al., 2016;Lei et al., 2017;Park et al., 2018)。
本研究发现,在涡虫切后再生6 h和3 d,Caspase-7基因表达量升高,这2个时间点是涡虫切后凋亡发生的2个高峰期(Hwang et al., 2004),这表明Caspase-7可能在涡虫中参与细胞凋亡的调控。进一步分析发现,干扰Caspase-7基因后,涡虫再生没有受到影响,但再生完成后或未切割的成体涡虫在饥饿环境下,Caspase-7干扰组虫体会逐渐溶解直至死亡。其发生溶解时,Djclg-3与PCNA基因表达量显著降低。
细胞凋亡在涡虫组织重塑的过程中起着重要的作用(Pellettieri et al., 2010)。Caspase-3是Caspase家族中另一重要的凋亡执行者(Chai et al., 2001;Jang et al., 2007),可以被Caspase家族中凋亡起始者Caspase-8和Caspase-9所剪切,活化的Caspase-3可以降解多种细胞蛋白,并在细胞凋亡过程中负责细胞形态的改变和DNA的断裂,同时还可以调节细胞的增殖和组织器官的尺寸(Liu et al., 2017;Yosefzon et al., 2018)。已有研究表明,Djclg-3基因在涡虫再生过程中参与涡虫细胞数量的控制,对组织重塑过程有调控作用(Hwang et al., 2004)。在饥饿环境下,涡虫没有外在能量的供应,只能靠自身细胞的死亡来提供生存所需的能量和物质,因此涡虫会发生退行性生长,即涡虫躯体会不断缩小。此时,涡虫组织和器官会通过细胞死亡和增殖的协调而重塑,以适应不断缩小的身体比例(Mead & Christman,1998;Weissman,2000)。受Caspase-7基因干扰后,Djclg-3与PCNA基因的表达量显著降低,可能导致饥饿环境下涡虫的细胞凋亡与增殖减少,涡虫组织重塑难以完成,以至于虫体不断溶解并最终死亡。该结果说明,Caspase-7可能在涡虫的组织重塑中发挥着重要作用。
本文首次报道涡虫中Caspase-7基因全长并分析了其结构特点,检测了涡虫再生不同时间点的表达量变化,并对Caspase-7在涡虫中的功能进行了初步探索,为涡虫细胞凋亡的研究及细胞凋亡与再生关系的探索奠定了基础。
| 郭素英, 舒瑞阳, 彭建新, 等. 2009. 昆虫中分离与鉴定的caspase及其作用[J]. 应用昆虫学报, 46(5): 678–683. |
| Baguñá J, Romero R. 1981. Quantitative analysis of cell types during growth, degrowth and regeneration in the planarians Dugesia mediterranea and Dugesia tigrina[J]. Hydrobiologia, 84(1): 181–194. DOI:10.1007/BF00026179 |
| Chai J, Wu Q, Shiozaki E, et al. 2001. Crystal structure of a procaspase-7 zymogen: mechanisms of activation and substrate binding[J]. Cell, 107(3): 399–407. DOI:10.1016/S0092-8674(01)00544-X |
| Chaudhary S, Madhukrishna B, Adhya AK, et al. 2016. Overexpression of caspase 7 is ERα dependent to affect proliferation and cell growth in breast cancer cells by targeting p21Cip[J]. Oncogenesis, 5(4): e219. DOI:10.1038/oncsis.2016.12 |
| DeHaan RL, Ebert JD. 2015. Fundamentals of planarian regeneration[J]. Annual Review of Cell and Developmental Biology, 20(1): 725–757. |
| Hill ME, Macpherson DJ, Wu P, et al. 2016. Reprogramming Caspase-7 specificity by regio-specific mutations and selection provides alternate solutions for substrate recognition[J]. ACS Chemical Biology, 11(6): 1603–1612. DOI:10.1021/acschembio.5b00971 |
| Hwang JS, Kobayashi C, Agata K, et al. 2004. Detection of apoptosis during planarian regeneration by the expression of apoptosis-related genes and TUNEL assay[J]. Gene, 333(3): 15–25. |
| Jang M, Park BC, Lee AY, et al. 2007. Caspase-7 mediated cleavage of proteasome subunits during apoptosis[J]. Biochemical and Biophysical Research Communications, 363(2): 388–394. DOI:10.1016/j.bbrc.2007.08.183 |
| Kuranaga E. 2012. Beyond apoptosis: caspase regulatory mechanisms and functions in vivo[J]. Genes to Cells, 17(2): 83–97. DOI:10.1111/gtc.2012.17.issue-2 |
| Liu PF, Hu YC, Kang BH, et al. 2017. Expression levels of cleaved caspase-3 and caspase-3 in tumorigenesis and prognosis of oral tongue squamous cell carcinoma[J]. PLoS ONE, 12(7): e0180620. DOI:10.1371/journal.pone.0180620 |
| Mangel M, Bonsall MB, Aboobaker A. 2016. Feedback control in planarian stem cell systems[J]. BMC Systems Biology, 10(1): 1–18. |
| Martinon F, Tschopp J. 2007. Inflammatory caspases and inflammasomes: master switches of inflammation[J]. Cell Death & Differentiation, 14(1): 10–22. |
| Mead RW, Christman J. 1998. Proportion regulation in the planarian, Dugesia tigrina following regeneration of structures[J]. Hydrobiologia, 383(1-3): 105–109. |
| Park JK, Doseff AI, Schmittgen TD. 2018. MicroRNAs targeting Caspase-3 and -7 in PANC-1 cells[J]. International Journal of Molecular Sciences, 19(4): 1206. DOI:10.3390/ijms19041206 |
| Pellettieri J, Fitzgerald P, Watanabe S, et al. 2010. Cell death and tissue remodeling in planarian regeneration[J]. Developmental Biology, 338(1): 76–85. DOI:10.1016/j.ydbio.2009.09.015 |
| Putt KS, Chen GW, Pearson JM, et al. 2006. Small-molecule activation of procaspase-3 to caspase-3 as a personalized anticancer strategy[J]. Nature Chemical Biology, 2(10): 543–550. DOI:10.1038/nchembio814 |
| Rink JC. 2013. Stem cell systems and regeneration in planarian[J]. Development Genes & Evolution, 223(1-2): 67–84. |
| Rouhana L, Weiss JA, Forsthoefel DJ, et al. 2013. RNA interference by feeding in vitro synthesized double-stranded RNA to planarians: methodology and dynamics[J]. Developmental Dynamics, 242(6): 718–730. DOI:10.1002/dvdy.v242.6 |
| Tan TCJ, Rahman R, Jaber-Hijazi F, et al. 2012. Telomere maintenance and telomerase activity are differentially regulated in asexual and sexual worms[J]. Proceedings of the National Academy of Sciences of the United States of America, 109(11): 4209–4214. DOI:10.1073/pnas.1118885109 |
| Thomsen ND, Koerber JT, Wells JA. 2013. Structural snapshots reveal distinct mechanisms of procaspase-3 and -7 activation[J]. Proceedings of the National Academy of Sciences of the United States of America, 110(21): 8477–8482. DOI:10.1073/pnas.1306759110 |
| Wagner DE, Wang IE, Reddien PW. 2011. Clonogenic neoblasts are pluripotent adult stem cells that underlie planarian regeneration[J]. Science, 332(6031): 811–816. DOI:10.1126/science.1203983 |
| Weissman IL. 2000. Stem cells: units of development, units of regeneration, and units in evolution[J]. Cell, 100(1): 157–168. DOI:10.1016/S0092-8674(00)81692-X |
| Wolan DW, Zorn JA, Gray DC, et al. 2009. Small molecule activators of a proenzyme[J]. Science, 326(5954): 853–858. DOI:10.1126/science.1177585 |
| Lei B, Zhou XM, Lv DJ, et al. 2017. Apoptotic and nonapoptotic function of caspase 7 in spermatogenesis[J]. Asian Journal of Andrology, 19(1): 47–51. |
| Yosefzon Y, Soteriou D, Feldman A, et al. 2018. Caspase-3 regulates YAP-dependent cell proliferation and organ size[J]. Molecular Cell, 70(4): 573–587. DOI:10.1016/j.molcel.2018.04.019 |
 2019, Vol. 38
2019, Vol. 38




