扩展功能
文章信息
- 陈映竹, 宋旭颢, 周闯, 张文博, 陈本平, 吴少彬, 岳碧松
- CHEN Yingzhu, SONG Xuhao, ZHOU Chuang, ZHANG Wenbo, CHEN Benping, WU Shaobin, YUE Bisong
- 绿翅短脚鹎线粒体基因组测序分析
- Sequencing and Analysis of the Mitochondrial Genome of Ixos mcclellandii
- 四川动物, 2018, 37(6): 646-652
- Sichuan Journal of Zoology, 2018, 37(6): 646-652
- 10.11984/j.issn.1000-7083.20180121
-
文章历史
- 收稿日期: 2018-04-16
- 接受日期: 2018-06-15
2. 四川老君山国家级自然保护区, 四川屏山 645350;
3. 长江师范学院生命科学与技术学院, 重庆 408100
2. Sichuan Laojunshan National Nature Reserve, Pingshan, Sichuan Province 645350, China;
3. College of Life Science and Technology, Yangtze Normal University, Chongqing 408100, China
绿翅短脚鹎Ixos mcclellandii隶属于雀形目Passeriformes鹎科Pycnonotidae短脚鹎属Ixos,主要分布于印度、中国、缅甸、越南、老挝、泰国和马来半岛等地,常栖息在海拔1 000~3 000 m的阔叶林、针阔叶混交林及次生林等,尤以林缘疏林和沟谷地带较常见,主要以植物果实、种子和昆虫为食(Moyle & Marks,2006;邓青珊等,2008;丁虎林等,2013;郑光美,2017)。尽管该物种较为常见且分布较广,但与其相关的研究资料却十分缺乏。
线粒体是生物体内重要的细胞器,参与能量转换、细胞凋亡等重要生理过程(Cleland & Youle,2011;Bess et al., 2012;Min et al., 2013)。线粒体基因组以其结构简单、进化速率快、分子量小、拷贝数高、不含内含子、遗传过程中不发生重组或倒位,以及严格遵守母系遗传等特点,成为分子系统学和物种进化研究中的常用分子标记,在生物的起源演化以及种群遗传学研究中被广泛使用(Parmakelis et al., 2015;Li et al., 2017;Zheng et al., 2018)。在雀形目鸟类线粒体基因组中,控制区(control region,CR)的数量有2种情况,大多数鸟类线粒体基因组只有1个非编码控制区,如绿背山雀Parus monticolus(白洁,2012)、苍头燕雀Fringilla coelebs(Marshall et al., 2013)、鸦科Corvidae和椋鸟科Sturnidae(钱朝菊,2013)等。部分物种的线粒体基因组具有2个控制区,根据相似性又分为:1)控制区高度相似,如阔嘴鸟科Eurylaimidae(Mindell et al., 1998)、家燕Hirundo rustica(陈娟,2015)、长尾山雀属Aegithalos(Wang et al., 2015)和鹎科(Wen & Liao,2016)等;2)控制区高度异质,如百灵科Alaudidae的云雀Alauda arvensis(Elodie et al., 2011)等。本研究对绿翅短脚鹎线粒体基因组进行测序分析,为该物种的相关研究提供基础数据。
1 材料和方法 1.1 样品采集和DNA提取绿翅短脚鹎肌肉样品采自四川老君山国家级自然保护区,利用试剂盒TIANamp Genomic DNA Kit(北京天根生化科技有限公司)提取线粒体基因组DNA。
1.2 引物设计及PCR扩增线粒体基因组PCR扩增引物用Premier 5.0设计。PCR反应程序为:95 ℃预变性5 min;94 ℃变性40 s,50~60 ℃退火30 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min;4 ℃保存。PCR产物经电泳检测后,选择条带明亮、清晰单一且长度一致的送成都擎科科技有限公司测序。测序结果峰图文件首先在NCBI的GenBank数据库中进行局部序列相似性搜索(BLAST),以确定得到的测序结果为目的片段。得到的正确DNA序列数据用DNASTAR中的Editseq进行编辑处理。将拼接好的序列以Fasta格式输出,利用SeqBuilder工具完成基因注释。
2 结果 2.1 线粒体基因组结构绿翅短脚鹎线粒体基因组序列全长17 838 bp(GenBank登录号:KX640824),为一双链闭合环状DNA分子,包含37个编码基因(22个tRNA基因、13个蛋白质基因、2个rRNA基因)以及2个高度相似的控制区(CR1和CR2)(表 1)。其中,除了8个tRNA基因和ND6基因在轻链上编码外,其余都在重链上编码。该线粒体基因组A、T、G、C的含量分别是27.81%、28.45%、25.36%和18.38%。
| 基因 | 位置 | 大小/bp | 间隔区 | 起始密码子 | 终止密码子 |
| tRNA-Phe | 1~68 | 68 | 0 | ||
| 12S rRNA | 69~1 048 | 980 | -4 | ||
| tRNA-Val | 1 045~1 114 | 70 | 0 | ||
| 16S rRNA | 1 115~2 720 | 1 606 | -3 | ||
| tRNA-Leu (UUR) | 2 718~2 792 | 75 | 11 | ||
| ND1 | 2 804~3 781 | 978 | 7 | ATG | AGG |
| tRNA-Ile | 3 789~3 862 | 74 | 6 | ||
| tRNA-Gln (L) | 3 869~3 939 | 71 | -1 | ||
| tRNA-Met | 3 939~4 007 | 69 | 0 | ||
| ND2 | 4 008~5 047 | 1 040 | 0 | ATG | TA |
| tRNA-Trp | 5 048~5 117 | 70 | 1 | ||
| tRNA-Ala (L) | 5 119~5 187 | 69 | 10 | ||
| tRNA-Asn (L) | 5 198~5 270 | 73 | 0 | ||
| tRNA-Cys (L) | 5 271~5 337 | 67 | -1 | ||
| tRNA-Tyr (L) | 5 337~5 407 | 71 | 1 | ||
| COX Ⅰ | 5 409~6 956 | 1 548 | -6 | GTG | AGG |
| tRNA-Ser (UCN)(L) | 6 951~7 023 | 73 | 4 | ||
| tRNA-Asp | 7 028~7 096 | 69 | 11 | ||
| COX Ⅱ | 7 106~7 789 | 684 | 2 | ATG | TAA |
| tRNA-Lys | 7 791~7 859 | 69 | 1 | ||
| ATP8 | 7 861~8 028 | 168 | -10 | ATG | TAA |
| ATP6 | 8 019~8 702 | 684 | 7 | ATG | TAA |
| COX Ⅲ | 8 710~9 493 | 784 | 0 | ATG | T |
| tRNA-Gly | 9 494~9 562 | 69 | 0 | ||
| ND3 | 9 563~9 913 | 351 | 1 | ATA | TAA |
| tRNA-Arg | 9 915~9 984 | 70 | 1 | ||
| ND4L | 9 986~10 282 | 297 | -7 | ATG | TA |
| ND4 | 10 276~11 653 | 1 378 | 0 | ATG | T |
| tRNA-His | 11 654~11 723 | 70 | 2 | ||
| tRNA-Ser (AGY) | 11 726~11 789 | 64 | -1 | ||
| tRNA-Leu (CUN) | 11 789~11 859 | 71 | 0 | ||
| ND5 | 11 860~13 677 | 1 818 | 9 | ATG | AGA |
| Cyt-B | 13 686~14 828 | 1 143 | 3 | ATG | TAA |
| tRNA-Thr | 14 832~14 900 | 69 | -7 | ||
| CR1 | 14 901~16 017 | 1 116 | 0 | ||
| tRNA-Pro (L) | 16 018~16 087 | 70 | 16 | ||
| ND6 (L) | 16 103~16 621 | 519 | 1 | ATG | TAA |
| tRNA-Glu (L) | 16 623~16 693 | 71 | 0 | ||
| CR2 | 16 694~17 838 | 1 144 | 0 | ||
| 注:L表示该基因位于轻链;间隔区中的负数表示邻近基因之间重合的碱基数 Note:“L” indicates that the gene is located in the light chain;negative numbers in the “intergenic nucleotide” indicate the number of overlapping bases between adjacent genes | |||||
在绿翅短脚鹎线粒体基因组中,部分基因间存在重叠或间隔现象,重叠长度一般为1~10 bp,间隔1~16 bp。其中,有2个重叠的开放阅读框:ATP6和ATP8重叠了10 bp,ND4L和ND4重叠了7 bp。另外,还有一些重叠发生在蛋白质基因和相邻tRNA基因之间。除COX Ⅰ、ND3基因的起始密码子分别为GTG、ATA外,其余蛋白质基因的起始密码子均为ATG,而终止密码子则有6种,即TAA、TAG、AGG、ATA、TA和T(表 1)。13个蛋白质基因中,密码子的组成及使用具有一定偏倚,使用频率最高的密码子为CUA(217次)和ACC(205次),而GGU和GCG的使用频率最低,均为24次(表 2)。
| 氨基酸 | 密码子 | 数量 | 密码子使用相对概率 | 频率/% | 氨基酸 | 密码子 | 数量 | 密码子使用相对概率 | 频率/% | |
| Phe-F | UUU | 79 | 0.81 | 1.32 | Tyr-Y | UAU | 85 | 0.74 | 1.42 | |
| UUC | 116 | 1.19 | 1.95 | UAC | 144 | 1.26 | 2.42 | |||
| Leu-L | UUA | 90 | 0.83 | 1.51 | Stop | UAA | 148 | 1.64 | 2.48 | |
| UUG | 55 | 0.51 | 0.92 | UAG | 107 | 1.18 | 1.79 | |||
| CUU | 102 | 0.94 | 3.64 | His-H | CAU | 113 | 0.94 | 1.91 | ||
| CUC | 119 | 1.10 | 1.71 | CAC | 128 | 1.06 | 2.15 | |||
| CUA | 217 | 2.00 | 3.64 | Gln-Q | CAA | 177 | 1.28 | 2.97 | ||
| CUG | 69 | 0.63 | 1.16 | CAG | 100 | 0.72 | 1.68 | |||
| Ile-I | AUU | 104 | 0.86 | 3.47 | Asn-N | AAU | 116 | 0.90 | 1.95 | |
| AUC | 138 | 1.14 | 1.82 | AAC | 142 | 1.10 | 2.38 | |||
| Met-M | AUA | 148 | 1.35 | 2.32 | Lys-K | AAA | 133 | 1.15 | 2.23 | |
| AUG | 72 | 0.65 | 1.21 | AAG | 98 | 0.85 | 1.64 | |||
| Val-V | GUU | 30 | 0.76 | 0.51 | Asp-D | GAU | 67 | 1.02 | 1.07 | |
| GUC | 41 | 1.04 | 0.57 | GAC | 64 | 0.98 | 1.06 | |||
| GUA | 57 | 1.45 | 0.62 | Glu-E | GAA | 83 | 1.16 | 1.39 | ||
| GUG | 29 | 0.74 | 0.48 | GAG | 60 | 0.84 | 1.01 | |||
| Ser-S | UCU | 121 | 1.16 | 1.81 | Cys-C | UGU | 38 | 0.78 | 0.63 | |
| UCC | 154 | 1.48 | 2.43 | UGC | 60 | 1.22 | 1.01 | |||
| UCA | 134 | 1.29 | 1.78 | Trp-W | UGA | 85 | 1.28 | 1.42 | ||
| UCG | 54 | 0.52 | 0.61 | UGG | 48 | 0.72 | 0.81 | |||
| Pro-P | CCU | 169 | 1.11 | 2.84 | Arg-R | CGU | 37 | 0.85 | 0.62 | |
| CCC | 179 | 1.17 | 1.87 | CGC | 46 | 1.06 | 0.77 | |||
| CCA | 198 | 1.30 | 3.01 | CGA | 62 | 1.43 | 1.04 | |||
| CCG | 64 | 0.42 | 1.07 | CGG | 29 | 0.67 | 0.48 | |||
| Thr-T | ACU | 128 | 0.96 | 2.15 | Ser-S | AGU | 55 | 0.53 | 0.92 | |
| ACC | 205 | 1.53 | 3.44 | AGC | 107 | 1.03 | 1.79 | |||
| ACA | 154 | 1.15 | 2.58 | Stop | AGA | 52 | 0.57 | 0.87 | ||
| ACG | 49 | 0.37 | 0.82 | AGG | 55 | 0.61 | 0.92 | |||
| Ala-A | GCU | 55 | 0.84 | 0.92 | Gly-G | GGU | 24 | 0.56 | 0.41 | |
| GCC | 108 | 1.66 | 1.81 | GGC | 49 | 1.15 | 0.82 | |||
| GCA | 74 | 1.13 | 1.24 | GGA | 65 | 1.52 | 1.09 | |||
| GCG | 24 | 0.37 | 0.41 | GGG | 33 | 0.77 | 0.55 |
绿翅短脚鹎线粒体基因组中,12S rRNA和16S rRNA位于tRNA-Phe基因与tRNA-Leu (UUR)基因之间,被tRNA-Val基因分隔开(表 1)。tRNA基因分布在rRNA基因和蛋白质基因之间,序列长度为64~75 bp。通过tRNAscan-SE 1.21(Lowe & Chan,2016)预测22个tRNA基因,结果显示,除tRNA-Ser (AGY)基因在DHU臂上形成1个简单的环,其他21个tRNA基因的二级结构均为典型的三叶草形。
2.4 非编码控制区绿翅短脚鹎线粒体基因组的2个控制区(CR1和CR2)位于tRNA-Thr和tRNA-Phe基因之间,被tRNA-Pro、ND6和tRNA-Glu基因分隔开,长度分别为1 116 bp和1 144 bp。CR1与CR2相似度达91.6%,序列相同部分约1 000 bp,仅在两端序列之间有所不同。2个控制区均可分为3个结构域,即Domain Ⅰ、Domain Ⅱ及Domain Ⅲ(图 1)。Domain Ⅰ包含ETAS1元件和1个茎环结构,2个控制区中均未发现ETAS2元件。Domain Ⅱ中分布着6个保守序列元件,即F-box、E-box、D-box、C-box、Bird-box和B-box。Domain Ⅲ中只有1个保守元件CSB1,且上游存在1个插入片段。
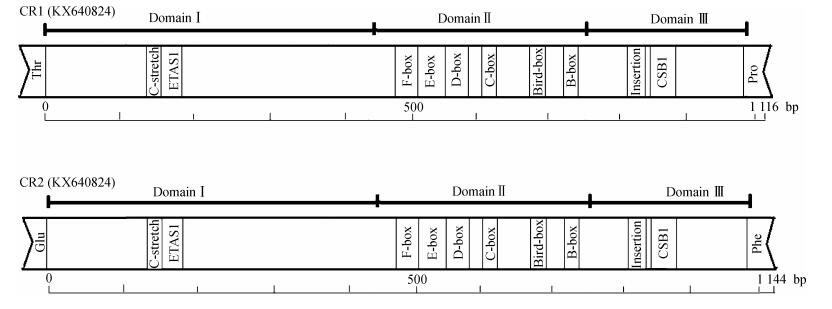
|
| 图 1 绿翅短脚鹎线粒体控制区的结构 Fig. 1 The structure of 2 control regions (CR) in Ixos mcclellandii mtDNA |
| |
本研究中测定的绿翅短脚鹎线粒体基因组与已公布的部分雀形目鸟类(Chen et al., 2015)具有类似组成及结构,即基因组为双链闭合环状的DNA分子,包含37个编码基因和2个高度相似的控制区。线粒体基因组中,13个蛋白质基因中相邻的2个基因之间、一些蛋白质基因和相邻tRNA基因之间均存在碱基的重叠,这一特征说明了线粒体基因组的紧凑和精简性(Curole & Kocher,1999;Kumar et al., 2015)。密码子第三位上的碱基变异在蛋白质基因中可以同义替换,即不会改变阅读框所编码的氨基酸(Choudhury et al., 2017)。本研究结果显示,同一个氨基酸同时有几个密码子,如编码亮氨酸的密码子有TTA、TTG、CTT、CTC、CTA和CTG等,均符合密码子简并性特点。第三位上的碱基变异在进化上的选择压力较小,碱基的偏倚性最能体现序列进化的变异特征。碱基含量中,A+T(56.26%)高于G+C(43.74%)。这一现象和多数脊椎动物线粒体基因组一致(Zhuang et al., 2013;孙利元等,2017;Zhang et al., 2018)。
3.2 控制区分析控制区是线粒体中变异最大的区域,线粒体长度的变异也主要集中在控制区,大多数表现为碱基的缺失、插入和串联重复序列的不同等,而控制区的主要功能是调控线粒体基因组的复制和转录(Mjelle et al., 2008;陈四海等,2011;Mikami et al., 2013;Tzur & Rosset,2015)。控制区作为线粒体最主要的非编码区域,其碱基偏倚是反映线粒体基因组在进化历程中变异积累的重要特征。它是整个线粒体基因组序列变异最大、碱基替换速率快、进化最快的一个区域,其序列差异可能是造成鸟类不同物种线粒体基因组序列差异的重要原因。控制区序列已经成为鸟类系统发育关系研究的一个重要分子标记(Keith et al., 2012;Schirtzinger et al., 2012;Song et al., 2014;章明等,2016)。
比较雀形目鸟类已有的线粒体基因组发现,控制区存在3种类型:1)常见于多数物种的单控制区;2)2个高度相似的控制区;3)2个异质性很高的控制区。以本课题组测得的绿翅短脚鹎、金眶鹟莺Seicercus burkii及棕腹大仙鹟Niltava davidi线粒体基因组为代表加以阐述。
绿翅短脚鹎线粒体基因组的2个控制区可分为3个结构域(Domain Ⅰ、Domain Ⅱ、Domain Ⅲ),其相似度达91.6%,二者序列相同的部分约1 000 bp,仅在2个控制区开始部位约70 bp和存在于CR2末端约90 bp的序列有所不同,其余部分完全相似。2个控制区的存在可能是有利的或具有一定的功能性,因此,在某些情况下可以在进化过程中保留而不完全退化(Eberhard et al., 2001)。值得注意的是,本研究并未在2个控制区的Domain Ⅲ中发现对线粒体基因组复制和转录起始有重要作用的LSP/HSP元件,但在画眉Garrulax canorus中则存在该结构(Chen et al., 2015)。
与绿翅短脚鹎相比,金眶鹟莺的线粒体基因组含有2个异质性很高的控制区,长度分别为1 127 bp和269 bp(未发表数据)。尽管2个控制区也位于tRNA-Thr和tRNA-Phe基因之间,且同样被tRNA-Pro、ND6和tRNA-Glu基因分隔,但控制区之间的相似度仅为16.6%。在CR1中,Domain Ⅰ分布着C-stretch、ETAS1-2及CSB1-like元件;Domain Ⅱ为中央保守区,主要分布有F-box、E-box、D-box、C-box、Bird-box和B-box元件;Domain Ⅲ主要分布有CSB1和LSP/HSP 2个保守元件。与绿翅短脚鹎线粒体基因组不同的是,在金眶鹟莺的CR2中未能找到这些保守元件。灰头鸦雀Paradoxornis gularis和云雀等物种线粒体具有与金眶鹟莺类似的控制区结构(Qian et al., 2013)。
棕腹大仙鹟线粒体基因组中只有1个控制区,位于tRNA-Thr和tRNA-Phe基因之间,长度为1 193 bp(未发表数据)。在Domain Ⅰ中,分布着CSB1-like、ETAS1-2元件;Domain Ⅱ中分布有F-box、E-box、D-box、C-box、Bird-box和B-box元件;Domain Ⅲ中,分布着1个CSB1保守元件,下游有1段poly-T序列,这种控制区结构与大多数鸟类线粒体基因组的控制区相同,如长尾山椒鸟Pericrocotus ethologue、灰背伯劳Lanius tephronotus、树麻雀Passer montanus和黑枕黄鹂Oriolus chinensis等(林立亮,2011;高瑞瑞,2013;钱朝菊,2013)。
| 白洁. 2012.大山雀和绿背山雀线粒体基因组序列测定和系统发生分析[D].西安: 陕西师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10718-1012514795.htm |
| 陈娟. 2015.鹰形目和雀形目七种鸟类的线粒体全基因组序列的测序与分析[D].芜湖: 安徽师范大学. http://www.wanfangdata.com.cn/details/detail.do?_type=degree&id=D744784 |
| 陈四海, 区又君, 李加儿. 2011. 鱼类线粒体DNA及其研究进展[J]. 生物技术通报, 3: 13–20. |
| 邓青珊, 朱琼琼, 鲁长虎. 2008. 南方红豆杉的天然更新格局及食果鸟类对其种子的传播[J]. 生态学杂志, 27: 712–717. |
| 丁虎林. 2013.浙江天童三种鹎科鸟类栖息地选择的季节变化[D].上海: 华东师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10269-1013267882.htm |
| 高瑞瑞. 2013.三种雀形目鸟类线粒体基因组测定及雀形目鸟类系统发生研究[D].西安: 陕西师范大学. |
| 林立亮. 2011.四种山雀线粒体基因组测序与雀形目线粒体谱系基因组学分析[D].西安: 陕西师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10718-1011142989.htm |
| 钱朝菊. 2013.雀形目13种鸟类线粒体全基因组序列的测定与分析[D].芜湖: 安徽师范大学. http://cdmd.cnki.com.cn/Article/CDMD-10370-1014104015.htm |
| 孙利元, 杨天燕, 孟玮, 等. 2017. 8种石首鱼类线粒体基因组特征及分子系统进化分析[J]. 海洋科学, 41: 48–54. |
| 章明, 贾晓旭, 陆俊贤, 等. 2016. 文昌鸡线粒体控制区遗传多态性及系统进化分析[J]. 安徽农业大学学报, 43: 690–693. |
| 郑光美. 2017. 中国鸟类分类与分布名录(第三版)[M]. 北京: 科学出版社. |
| Bess AS, Crocker TL, Ryde IT, et al. 2012. Mitochondrial dynamics and autophagy aid in removal of persistent mitochondrial DNA damage in Caenorhabditis elegans[J]. Nucleic Acids Research, 40: 7916–7931. DOI:10.1093/nar/gks532 |
| Chen DS, Qian CJ, Ren QQ, et al. 2015. Complete mitochondrial genome of the Chinese hwamei Garrulax canorus (Aves: Passeriformes): the first representative of the Leiothrichidae family with a duplicated control region[J]. Genetics and Molecular Research, 14: 8964–8976. DOI:10.4238/2015.August.7.5 |
| Choudhury MN, Uddin A, Chakraborty S. 2017. Gene expression, nucleotide composition and codon usage bias of genes associated with human y chromosome[J]. Genetica, 145: 295–305. DOI:10.1007/s10709-017-9965-y |
| Cleland MM, Youle RJ. 2011. Mitochondrial dynamics and apoptosis[J]. Genes & Development, 22: 1577. |
| Curole JP, Kocher TD. 1999. Mitogenomics: digging deeper with complete mitochondrial genomes[J]. Trends in Ecology & Evolution, 14(10): 394–398. |
| Eberhard JR, Wright TF, Bermingham E. 2001. Duplication and concerted evolution of the mitochondrial control region in the parrot genus Amazona[J]. Molecular Biology and Evolution, 18(7): 1330–1342. DOI:10.1093/oxfordjournals.molbev.a003917 |
| Elodie B, Fanny R, Thierry A. 2011. Microdialect and group signature in the song of the skylark Alauda arvensis[J]. Bioacoustics, 20: 219–233. DOI:10.1080/09524622.2011.9753647 |
| Keith BF, Benesh MK, Vandergon AJ, et al. 2012. Contrasting evolutionary dynamics and information content of the avian mitochondrial control region and ND2 gene[J]. PLoS ONE, 7: e46403. DOI:10.1371/journal.pone.0046403 |
| Kumar CS, Hazarika NM, Kumar S. 2015. Analysis of synonymous codon usage in the vp2 protein gene of infectious bursal disease virus[J]. Archives of Virology, 160: 2359–2366. DOI:10.1007/s00705-015-2505-0 |
| Li WX, Zhang D, Boyce K, et al. 2017. The complete mitochondrial DNA of three monozoic tapeworms in the Caryophyllidea: a mitogenomic perspective on the phylogeny of eucestodes[J]. Parasite Vector, 10: 314. DOI:10.1186/s13071-017-2245-y |
| Lowe TM, Chan PP. 2016. tRNAscan-SE on-line: integrating search and context for analysis of transfer RNA genes[J]. Nucleic Acids Research, 44: W54–W57. DOI:10.1093/nar/gkw413 |
| Marshall HD, Baker AJ, Grant AR. 2013. Complete mitochondrial genomes from four subspecies of common chaffinch (Fringilla coelebs): new inferences about mitochondrial rate heterogeneity, neutral theory, and phylogenetic relationships within the order Passeriformes[J]. Gene, 517: 37–45. DOI:10.1016/j.gene.2012.12.093 |
| Mikami E, Fuku N, Takahashi H, et al. 2013. Polymorphisms in the control region of mitochondrial DNA associated with elite Japanese athlete status[J]. Scandinavian Journal of Medicine & Science in Sports, 23: 593–599. |
| Min W, Kalyanasundaram A, Jie Z. 2013. Structural and biomechanical basis of mitochondrial movement in eukaryotic cells[J]. International Journal of Nanomedicine, 8: 4033–4042. |
| Mindell DP, Sorenson MD, Dimcheff DE. 1998. Multiple independent origins of mitochondrial gene order in birds[J]. Proceedings of the National Academy of Sciences of the USA, 95: 10693–10697. DOI:10.1073/pnas.95.18.10693 |
| Mjelle KA, Karlsen BO, Jorgensen TE, et al. 2008. Halibut mitochondrial genomes contain extensive heteroplasmic tandem repeat arrays involved in DNA recombination[J]. BMC Genomics, 9(1): 1–11. DOI:10.1186/1471-2164-9-1 |
| Moyle RG, Marks BD. 2006. Phylogenetic relationships of the bulbuls (Aves: Pycnonotidae) based on mitochondrial and nuclear DNA sequence data[J]. Molecular Phylogenetics and Evolution, 40: 687–695. DOI:10.1016/j.ympev.2006.04.015 |
| Parmakelis A, Spanos E, Papagiannakis G, et al. 2015. Mitochondrial DNA phylogeny and morphological diversity in the genus Mastus (Beck, 1837): a study in a recent (Holocene) island group (Koufonisi, south-east Crete)[J]. Biological Journal of the Linnean Society, 78: 383–399. |
| Qian C, Wang Y, Guo Z, et al. 2013. Complete mitochondrial genome of skylark, Alauda arvensis (Aves: Passeriformes): the first representative of the family Alaudidae with two extensive heteroplasmic control regions[J]. Mitochondrial DNA, 24: 246–248. DOI:10.3109/19401736.2012.752481 |
| Schirtzinger EE, Tavare ES, Gonzales LA, et al. 2012. Multiple independent origins of mitochondrial control region duplications in the order Psittaciformes[J]. Molecular Phylogenetics and Evolution, 64: 342–356. DOI:10.1016/j.ympev.2012.04.009 |
| Song XH, Huang J, Yan C, et al. 2014. The complete mitochondrial genome of Accipiter virgatus, and evolutionary history of the pseudo-control regions in Falconiformes[J]. Biochemical Systematics and Ecology, 58: 75–84. |
| Tzur S, Rosset S. 2015. Strictly conserved tri-nucleotide motif "CAT" is associated with TAS DNA protein-binding sites in human mitochondrial DNA control region[J]. Mitochondrial DNA, 28: 250–253. |
| Wang X, Yuan H, Liu N, et al. 2015. Seven complete mitochondrial genome sequences of bushtits (Passeriformes, Aegithalidae, Aegithalos): the evolution pattern in duplicated control regions[J]. Mitochondrial DNA, 26(3): 350–356. DOI:10.3109/19401736.2014.1003821 |
| Wen L, Liao F. 2016. Complete mitochondrial genome of Pycnonotus xanthorrhous (Passeriformes, Pycnonotidae) and phylogenetic consideration[J]. Biochemical Systematics and Ecology, 69: 83–90. DOI:10.1016/j.bse.2016.08.009 |
| Zhang H, Bai Y, Shi X, et al. 2018. The complete mitochondrial genomes of Tarsiger cyanurus, and phoenicurus auroreus: a phylogenetic analysis of Passeriformes[J]. Genes & Genomics, 40: 151–165. |
| Zheng BY, Cao LJ, Tang P, et al. 2018. Gene arrangement and sequence of mitochondrial genomes yield insights into the phylogeny and evolution of bees and sphecid wasps (Hymenoptera: Apoidea)[J]. Molecular Phylogenetics and Evolution, 124: 1–9. DOI:10.1016/j.ympev.2018.02.028 |
| Zhuang X, Qu M, Zhang X, et al. 2013. A comprehensive description and evolutionary analysis of 22 grouper (Perciformes, Epinephelidae) mitochondrial genomes with emphasis on two novel genome organizations[J]. PLoS ONE, 8: e73561. DOI:10.1371/journal.pone.0073561 |
 2018, Vol. 37
2018, Vol. 37




