扩展功能
文章信息
- 聂虎, 曹莎莎, 赵明朗, 杜林方
- NIE Hu, CAO Shasha, ZHAO Minglang, DU Linfang
- 红尾蚺和原矛头蝮基因组微卫星分布特征比较分析
- Comparative Analysis of Microsatellite Distributions in Genomes of Boa constrictor and Protobothrops mucrosquamatus
- 四川动物, 2017, 36(6): 639-648
- Sichuan Journal of Zoology, 2017, 36(6): 639-648
- 10.11984/j.issn.1000-7083.20170070
-
文章历史
- 收稿日期: 2017-03-08
- 接受日期: 2017-05-24
微卫星是由1~6个核苷酸为基本重复单元构成的简单串联重复序列,广泛分布于动植物基因组中,但其在基因编码区、非翻译区和内含子的分布并不随机,并且5'UTR、3'UTR和内含子、外显子微卫星的收缩或扩张可通过多种方式引起基因功能的改变,从而影响细胞功能,最终导致表型变化和疾病发生(Li et al., 2004)。基因组层面的微卫星分析有助于比较不同物种之间微卫星的分布特征、了解基因组功能,并为开发微卫星标记提供方便(李午佼等,2014;Wang et al., 2016)。
红尾蚺Boa constrictor又称红尾蟒,蚺科Boidae卵胎生无毒蛇,主要分布于中美洲、南美洲以及加勒比海附近的一些岛屿。在某些地区,红尾蚺能调节负鼠Didelphis virginiana的种群规模,防止利什曼病传播给人类(Laurie & Janalee,2009);原矛头蝮Protobothrops mucrosquamatus又称龟壳花,蝰科Viperidae原矛头蝮属Protobothrops管牙类毒蛇,广泛分布于中国大陆以及印度、孟加拉、缅甸等地,已被列入中华人民共和国国家林业局2000年8月1日发布的《国家保护的有益的或者有重要经济、科学研究价值的陆生野生动物名录》。红尾蚺和原矛头蝮高质量的全基因组测序和组装已经完成(Kajitani et al., 2014;Kerkkamp et al., 2016),这为在基因组水平上开展红尾蚺和原矛头蝮微卫星研究提供了可能。
本研究主要目的有:第一,比较有毒蛇原矛头蝮和无毒蛇红尾蚺基因组层面微卫星数量、种类和丰度的异同;第二,比较2种蛇基因组不同区域(即基因间区、内含子和外显子)微卫星的分布特征;第三,比较2种蛇基因区微卫星丰度分布的位置特征;第四,探讨含有微卫星的编码序列(coding sequence,CDS)的功能,分析含有微卫星的编码基因在2种蛇中的差异。本研究有助于加深对蚺科和蝰科基因组的认识和了解,也为后续筛选和开发大量高质量的2种蛇类微卫星标记提供方便。
1 材料和方法 1.1 数据来源原矛头蝮、人Homo sapiens和小鼠Mus musculus的基因组从NCBI Genome数据库下载,登录号分别为GCF_001527695.2、GCF_000001405.36、GCF_000001635.25。红尾蚺的基因组从http://platanus.bio.titech.ac.jp/Snake.tgz下载(Kajitani et al., 2014)。
1.2 微卫星鉴定利用MISA在红尾蚺和原矛头蝮基因组中搜索1~6个核苷酸重复类型微卫星(Thiel et al., 2003)。运行MISA时,misa.ini文件def设置为“1-12 2-7 3-5 4-4 5-4 6-4”,int设置为100,即单核苷酸重复次数不小于12次,二核苷酸重复次数不小于7次,三核苷酸重复次数不小于5次,四核甘酸、五核苷酸和六核苷酸重复次数都不小于4次,如果2个微卫星之间距离小于100 bp,则认为是1个复合型微卫星。
1.3 微卫星分类根据重复单元的序列,对微卫星进行分类。如果2个微卫星是循环排列或者反向互补,则认为这2个微卫星属于同一类。比如微卫星ACG包括了微卫星ACG、CGA、GAC、TGC、GCT和CTG(Jurka & Pethiyagoda,1995)。
1.4 微卫星在基因组中的分布通过微卫星与特定区域的位置重叠,计算出微卫星在各个区域的分布。如果微卫星的位置与编码基因,或外显子,或内含子的位置完全重叠,则认为微卫星位于编码基因,或外显子,或内含子。否则,认为微卫星位于基因间区。另外,若微卫星位于基因上游或下游500 bp,则认为微卫星位于基因上游或下游。
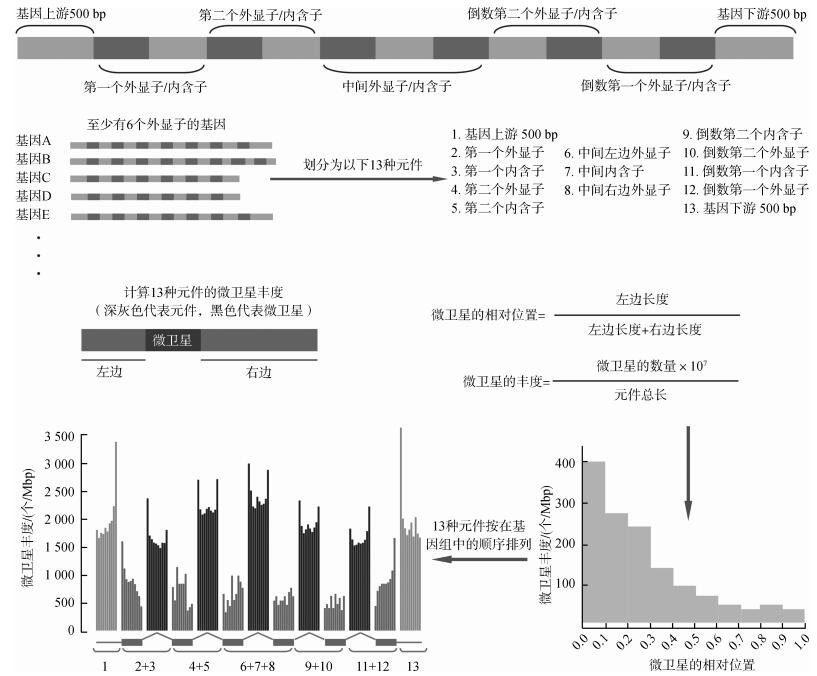
1.5 微卫星在基因组中的丰度分布为计算微卫星在基因组中的丰度分布,把基因中的外显子和内含子归为以下13种元件:基因上游500 bp、第一个外显子、第一个内含子、第二个外显子、第二个内含子、中间左边外显子、中间内含子、中间右边外显子、倒数第二个内含子、倒数第二个外显子、倒数第一个内含子、倒数第一个外显子和基因下游500 bp。微卫星在某个类型元件中的相对位置为微卫星到元件左端的距离除以元件长度与微卫星长度的差。微卫星的丰度为某个类型元件中微卫星的数量除以元件长度,单位为个/Mbp (Fujimori et al., 2003)。具体的计算过程如图 1。
|
| 图 1 微卫星丰度分布的计算方法 Fig. 1 Method to calculate the distribution of microsatellites abundance |
| |
对微卫星坐标和基因CDS的坐标进行重叠,如果某个基因CDS含有微卫星,则筛选出该CDS。将筛选出来的CDS通过BLASTx比对到NR蛋白质数据库,然后对注释出的蛋白进行GO功能分析(Conesa et al., 2005)。使用OrthoMCL对含有微卫星的CDS进行基因家族分析(Li et al., 2003)。
2 结果 2.1 红尾蚺和原矛头蝮基因组中微卫星的分布特征利用MISA搜索微卫星,在红尾蚺1.48 G全基因组序列中共搜索到398 860个微卫星,总长度为8 550 741 bp,占基因组序列总长度的0.59%。在原矛头蝮1.67 G全基因组序列中共搜索到422 364个微卫星,总长度为12 243 226 bp,占基因组序列总长度的0.73%。红尾蚺和原矛头蝮基因组中微卫星的丰度分别为275.46个/Mbp和252.33个/Mbp(表 1),两者比较相似。红尾蚺基因组中最多的5种微卫星类型为A、AC、AAAT、AG和AAT,原矛头蝮基因组中最多的5种微卫星类型为A、AAT、AC、C和AAAT(表 2),两者最常见的微卫星类型有所不同。红尾蚺基因组6种重复类型微卫星中,最丰富的是单碱基重复类型,其次是四碱基、二碱基、三碱基、五碱基和六碱基重复类型。原矛头蝮基因组6种重复类型微卫星中,最丰富的也是单碱基重复类型,其次是三碱基、四碱基、二碱基、五碱基、六碱基重复类型。2个物种单碱基重复类型最丰富的都是(A)n,红尾蚺(A)n类型占单碱基重复类型的88.86%,原矛头蝮(A)n类型占单碱基重复类型的74.37%(表 1,表 3)。红尾蚺基因组四碱基重复类型以(AAAT)n、(AAAC)n、(AATG)n和(AATG)n为主,原矛头蝮基因组三碱基重复类型以(AAT)n、(AGG)n、(AAC)n、和(ATG)n为主。2个物种六碱基重复类型丰度最低,都以(ACATAT)n为主。
| 微卫星类型 | 红尾蚺Boa constrictor | 原矛头蝮Protobothrops mucrosquamatus | |||||||
| 数量/个 | 长度/bp | 丰度/(个/Mbp) | 比例/% | 数量/个 | 长度/bp | 丰度/(个/Mbp) | 比例/% | ||
| 单核苷酸 | 127 438 | 1 768 476 | 88.01 | 31.95 | 112 325 | 1 584 938 | 67.10 | 26.59 | |
| 二核苷酸 | 64 318 | 1 298 646 | 44.42 | 16.13 | 86 241 | 2 386 200 | 51.52 | 20.42 | |
| 三核苷酸 | 55 284 | 1 195 623 | 38.18 | 13.86 | 103 517 | 4 275 309 | 61.84 | 24.51 | |
| 四核苷酸 | 120 262 | 3 101 836 | 83.05 | 30.15 | 90 822 | 2 861 504 | 54.26 | 21.50 | |
| 五核苷酸 | 25 677 | 949 820 | 17.73 | 6.44 | 26 263 | 1 015 665 | 15.69 | 6.22 | |
| 六核苷酸 | 5 881 | 236 340 | 4.06 | 1.47 | 3 196 | 119 610 | 1.91 | 0.76 | |
| 总计 | 398 860 | 8 550 741 | 275.46 | 100 | 422 364 | 12 243 226 | 252.33 | 100 | |
| 基因组序列 总长度/bp |
1 447 999 364 | 1 673 876 332 | |||||||
| 注:丰度=某类型微卫星数量/基因组大小;比例=某类型微卫星数量/微卫星总数。 Notes:Abundance=number of certain repeat microsatellites/genome size; percentage=number of certain repeat microsatellites/total number of microsatellites. |
|||||||||
| 红尾蚺 Boa constrictor |
原矛头蝮 Protobothrops mucrosquamatus |
|||
| 类型 | 个数 | 类型 | 个数 | |
| A | 113 242 | A | 83 531 | |
| AC | 37 281 | AAT | 53 161 | |
| AAAT | 36 774 | AC | 42 878 | |
| AG | 17 121 | C | 28 794 | |
| AAT | 15 617 | AAAT | 27 465 | |
| AAAC | 14 588 | AG | 27 093 | |
| C | 14 196 | AGG | 19 502 | |
| AT | 9 782 | AAGG | 13 229 | |
| AAC | 9 604 | AAC | 9 021 | |
| ATG | 8 821 | ATG | 7 918 | |
| 类型 | 红尾蚺 Boa constrictor |
原矛头蝮 Protobothrops mucrosquamatus |
| 单碱基重复 | A (113 242) 88.86% | A (83 531) 74.37% |
| C (14 196) 11.14% | C (28 794) 25.63% | |
| 二碱基重复 | AC (37 281) 57.96% | AC (42 878) 49.72% |
| AG (17 121) 26.62% | AG (27 093) 31.42% | |
| AT (9 782) 15.21% | AT (16 083) 18.65% | |
| CG (134) 0.21% | CG (187) 0.22% | |
| 三碱基重复 | AAT (15 617) 28.25% | AAT (53 161) 51.35% |
| AAC (9 604) 17.37% | AGG (19 502) 18.84% | |
| ATG (8 821) 15.96% | AAC (9 021) 8.71% | |
| AGG (8 252) 14.93% | ATG (7 918) 7.65% | |
| 四碱基重复 | AAAT (36 774) 30.58% | AAAT (27 465) 30.24% |
| AAAC (14 588) 12.13% | AAGG (13 229) 14.57% | |
| AAGG (8 693) 7.23% | AGGG (6 410) 7.06% | |
| AATG (7 882) 6.55% | AAAC (6 240) 6.87% | |
| 五碱基重复 | AAAAT (4 473) 17.42% | AATAG (10 540) 40.13% |
| AAAAC (2 763) 10.76% | AAAAT (2 050) 7.81% | |
| AAGGG (1 477) 5.75% | AAAAC (1 207) 4.60% | |
| AAATT (1 435) 5.59% | AAGAT (984) 3.75% | |
| 六碱基重复 | ACATAT (799) 13.59% | ACATAT (286) 8.95% |
| AACCCT (677) 11.51% | AAGGAG (204) 6.38% | |
| AAATAT (382) 6.50% | AACCCT (177) 5.54% | |
| AAGGAG (305) 5.19% | ATATAG (148) 4.63% | |
| 注:括号内数字表示该重复类型在基因组中出现的次数,后面的百分数表示占所在重复类型的百分比。 Notes:Number in parenthesis indicates occurrence number of the repeat and the following percentage means the percentage in the repeat. |
||
通过分析和比较微卫星在红尾蚺基因组和原矛头蝮基因组中各个区域的分布,发现基因间区微卫星数量最多、丰度最高,其次是内含子和外显子,编码区数量最少、丰度最低。另外,还发现红尾蚺基因组和原矛头蝮基因组非翻译区微卫星的丰度比编码区高(表 4)。对人和小鼠基因组中的微卫星进行鉴定和分析,发现这2种蛇基因组与这2种哺乳动物相比,编码区微卫星的数量和丰度差异很小,而在基因间区、外显子和内含子微卫星的数量和丰度差异较大(表 4)。
| 物种 | 基因区微卫星数/个[丰度/(个/Mbp)] | 基因间区 | |||
| 编码区 | 非翻译区 | 外显子 | 内含子 | ||
| 红尾蚺 Boa constrictor |
1 638 (51.60) | 1 225 (213.17) | 2 863 (76.36) | 111 031 (259.45) | 284 927 (290.09) |
| 原矛头蝮 Protobothrops mucrosquamatus |
1 512 (48.77) | 1 187 (152.92) | 2 699 (69.63) | 120 432 (243.49) | 299 174 (262.73) |
| 人类 Homo sapiens |
1 794 (47.76) | 8 210 (252.68) | 10 004 (142.81) | 426 480 (374.05) | 584 031 (302.48) |
| 小鼠 Mus musculus |
1 558 (41.02) | 8 915 (296.99) | 10 473 (154.02) | 358 091 (383.36) | 592 071 (339.30) |
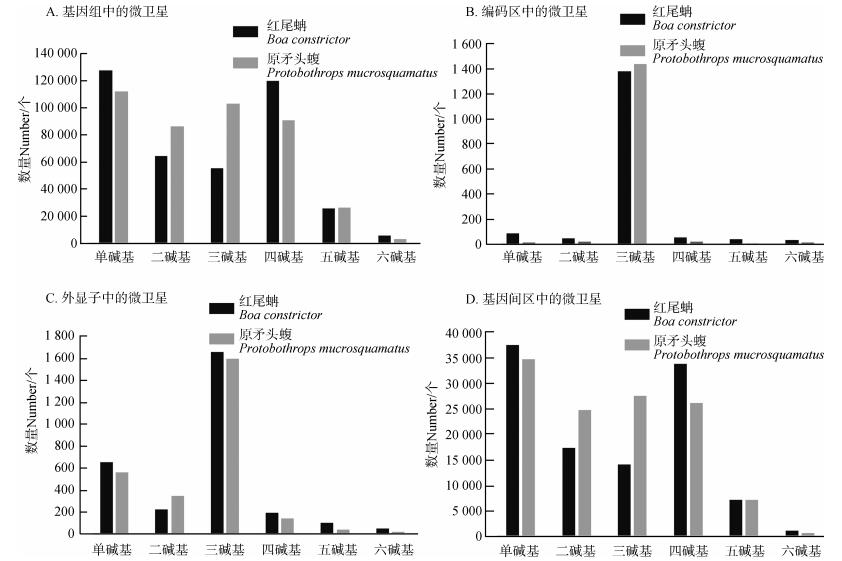
分析红尾蚺和原矛头蝮基因组编码区、外显子和内含子中微卫星的重复类型,发现两者编码区和外显子主要是三碱基重复类型,红尾蚺基因组编码区三碱基重复类型占编码区微卫星的84.07%,原矛头蝮编码区三碱基重复类型占编码区微卫星的95.11%(图 2:B)。红尾蚺和原矛头蝮基因间区中微卫星的类型主要是单碱基、四碱基、二碱基和三碱基重复类型,各种类型都不占主导优势(图 2:D),其分布和整个基因组中微卫星的分布类似(图 2:A)。比较编码区和外显子微卫星的重复类型(图 2:B,C),发现外显子单碱基重复类型比编码区多,而外显子包括编码区和非翻译区,说明非翻译区以单碱基重复类型为主。
|
| 图 2 红尾蚺和原矛头蝮基因组不同区域的微卫星类型的分布 Fig. 2 The distribution of microsatellite types in different genomic regions of Boa constrictor and Protobothrops mucrosquamatus |
| |
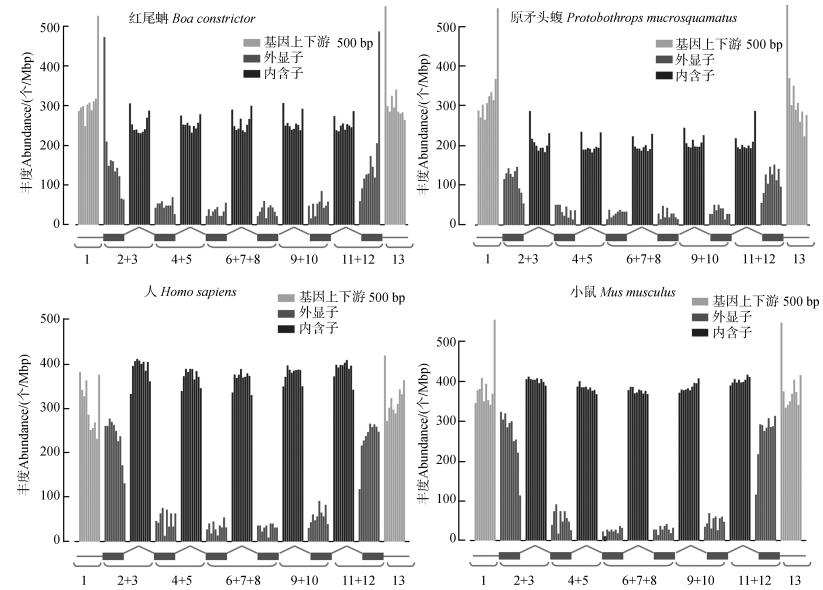
红尾蚺基因组中有1 552个CDS含有微卫星,其中只含有1个、2个、3个和4个微卫星的CDS分别有1 480个、61个、8个和3个。原矛头蝮基因组中有1 397个CDS含有微卫星,其中只含有1个、2个、3个、4个和5个微卫星的CDS分别有1 308个、69个、15个、4个和1个。红尾蚺基因组中含有4个微卫星的CDS有3个,分别来源于基因ZFP36L2、H1C1、JUND。原矛头蝮基因组中含有5个微卫星的CDS来源于基因WNK2,含有4个微卫星的CDS分别来源于基因PRDM2、H1C1、LOC107297696、SKOR2。计算每个区域微卫星的丰度,发现红尾蚺和原矛头蝮基因组中微卫星丰度分布相似(图 3)。
红尾蚺基因上游500 bp、外显子、内含子和基因下游500 bp各个区域微卫星的丰度分别为318.40个/Mbp、83.41个/Mbp、255.15个/Mbp和320.79个/Mbp。原矛头蝮基因上游500 bp、外显子、内含子和基因下游500 bp各个区域微卫星丰度分别为392.34个/Mbp、70.17个/Mbp、242.66个/Mbp和380.36个/Mbp。在转录起始位点附近的微卫星丰度最高,而且越靠近转录起始位点,微卫星丰度越高。在基因上游500 bp和下游500 bp内微卫星呈对称丰度分布,内含子微卫星丰度比外显子高,且在内含子分布较均匀,内含子5'-和3'-微卫星丰度要比内含子内部区域高。我们也计算了人和小鼠基因及其上下游微卫星的丰度分布(图 3),人基因上游500 bp、外显子、内含子和基因下游500 bp各个区域微卫星丰度分别为307.79个/Mbp、71.92个/Mbp、379.03个/Mbp,324.40个/Mbp,小鼠基因上游500 bp、外显子、内含子和基因下游500 bp各个区域微卫星丰度分别为388.44个/Mbp、115.23个/Mbp、391.02个/Mbp、386.06个/Mbp。人和小鼠内含子微卫星丰度比外显子高,这与红尾蚺和原矛头蝮基因组微卫星的丰度分布类似。4个物种的基因组中,基因的第一个外显子和最后一个外显子微卫星的丰度比内部外显子高。红尾蚺和原矛头蝮基因上下游500 bp内微卫星的丰度比内含子高,而人和小鼠基因上下游500 bp内微卫星的丰度和内含子比较接近。
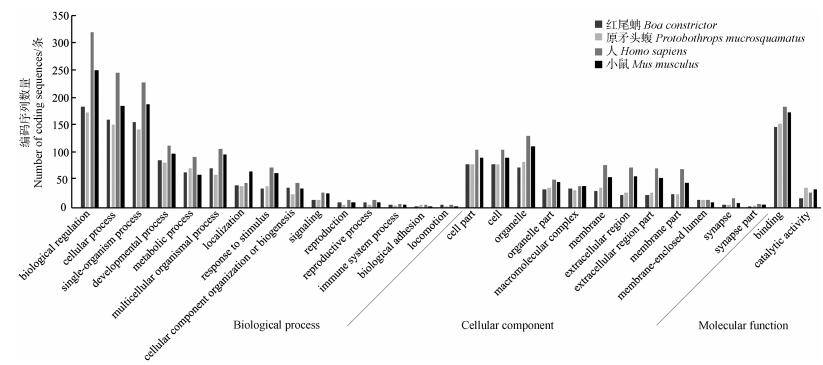
2.3 含有微卫星的编码序列的功能分析红尾蚺和原矛头蝮基因组中含有微卫星的CDS分别有1 552条和1 397条,分别来源于1 431个和1 291个蛋白编码基因。提取红尾蚺和原矛头蝮基因组中含有微卫星的CDS,使用BLASTx比对到NR数据库,其中分别有1 066 (68.69%)条和1 047 (74.95%)条能比对到NR数据库,然后对结果进行GO注释,分别有736条和773条CDS能够被GO功能归类。红尾蚺含有微卫星的CDS被分配到3 142个GO条目,原矛头蝮含有微卫星的CDS被分配到3 268个GO条目。图 4展示了红尾蚺和原矛头蝮含有微卫星的编码区的GO功能注释的比较。“Biological process”本体中,“biological regulation”和“cellular process”分配的CDS数量最多;“Cellular component”本体中,“organelle”“cell part”和“cell”分配的CDS数量最多;“Molecular function”本体中,“binding”和“catalytic activity”分配的CDS数量最多。红尾蚺和原矛头蝮基因组中分配到“biological regulation” (GO:0065007)条目的CDS最多,分别有185条和175条,占各自总数的25.14%和22.64%。相比之下,人和小鼠基因组中含有微卫星的CDS分别有1 644条和1 458条,分别来源于1 443个和1 331个编码基因,其中分别有1 320条和1 155条CDS能比对到NR数据库,分别有1 116条和954条CDS能够被GO功能归类。人和小鼠基因组中分配到“biological regulation” (GO:0065007)条目的CDS也最多,分别有321条和251条,占各自总数的28.76%和26.31%。总体来看,红尾蚺和原矛头蝮基因组含有微卫星的CDS的功能归类相似,与人和小鼠相比存在一定差异。
|
| 图 4 红尾蚺、原矛头蝮、人和小鼠基因组中含有微卫星的编码区的GO功能归类 Fig. 4 GO classifications of coding sequences with microsatellites in the genomes of Boa constrictor, Protobothrops mucrosquamatus, Homo sapiens and Mus musculus |
| |
对红尾蚺、原矛头蝮、人和小鼠4个物种含有微卫星的CDS使用OrthoMCL进行直系同源分析,一共可以归类到494个基因家族,其中红尾蚺和原矛头蝮含有微卫星的CDS可以归类到263个基因家族,人和小鼠含有微卫星的CDS可以归类到328个基因家族,并且只有3个基因家族在这4个物种之间共享。共享的3个基因家族分别为ONECUT2 (one cut homeobox 2)基因家族、LOC107401594(cyclin-dependent kinase 8)基因家族和HOXD8 (homeobox D8)基因家族。红尾蚺和原矛头蝮含有微卫星的CDS相比,两者共享155个基因家族,红尾蚺特有的基因家族有42个,原矛头蝮特有的基因家族有66个。人和小鼠含有微卫星的CDS相比,两者共享141个基因家族,人特有的基因家族有97个,小鼠特有的基因家族有90个。2个蛇类物种和2个哺乳类物种含有微卫星的CDS相比,共享的基因家族有97个,蛇类特有的基因家族有166个,哺乳类特有的基因家族有231个。
3 讨论本研究从红尾蚺(基因组大小为1.48 G,Contig N50为47 kb)和原矛头蝮(基因组大小为1.67 G,Contig N50为21 kb)全基因组中分别鉴定出398 860个和422 364个微卫星,数量的差异可能和基因组的大小、组装质量和物种基因组的特异性有关。Wang等(2016)从亚利桑那州树皮蝎Centruroides exilicauda和马氏正钳蝎Mesobuthus martensii的全基因组中分别鉴定出114 026个和211 868个微卫星,而亚利桑那州树皮蝎的基因组大小为926 Mbp (Contig N50为5 kb),马氏正钳蝎的基因组大小为925 Mbp (Contig N50为45 kb),两者基因组大小相近,而且是近源物种,但是鉴定出来的微卫星数量差距很大,说明测序的质量对基因组中微卫星的识别有较大影响。本研究选取已测序蛇类物种中Contig N50最高的2个物种来做分析,主要是为了更加全面地鉴定出全基因组中的微卫星。红尾蚺和原矛头蝮基因组中微卫星的含量比较相近,分别占基因组的0.59%和0.73%,与大型哺乳动物大熊猫Ailuropoda melanoleuca (0.64%)和北极熊Ursus maritimus (0.79%)相似(李午佼等,2014)。红尾蚺和原矛头蝮基因组中微卫星的丰度分别为275.46个/Mbp和252.33个/Mbp,与大熊猫(371.8个/Mbp)、北极熊(405.6个/Mbp)相比偏低(李午佼等,2014),与人(315.93个/Mbp)、小鼠(342.68个/Mbp)相比也偏低,这是否暗示了蛇类物种基因组中微卫星的丰度比哺乳类物种低,有待进一步确认。
红尾蚺与原矛头蝮基因组中6种重复类型微卫星所占比例的排序不一致,且最丰富的前5种微卫星也不一致。研究发现大熊猫和北极熊基因组中6种重复类型微卫星比例的排序和最丰富的前5种微卫星都表现出一致性(李午佼等,2014)。大熊猫和北极熊都是熊科Ursidae动物,相比之下,红尾蚺属于蚺科而原矛头蝮属于蝰科。说明了基因组中微卫星组成特征差异在一定程度上可以反映物种间的亲缘关系。
蛇亚目Serpentes的红尾蚺和原矛头蝮、哺乳纲Mammalia灵长目Primates的人和小鼠以及哺乳纲食肉目Carnivora的大熊猫和北极熊(李午佼等,2014)、节肢动物门Arthropoda的亚利桑那州树皮蝎和马氏正钳蝎(Wang et al., 2016)都是单碱基重复类型数量最多。但在其他物种的基因组中,如中国对虾Fenneropenaeus chinensis (高焕等,2004)、蜜蜂Apis mellifera (魏朝明等,2007)等出现了二碱基重复类型为主的情况,而酿酒酵母Saccharomyces cerevisiae (Katti et al., 2001)、粗糙脉孢菌Neurospora crassa (李成云等,2004)等基因组中占主导地位的是三碱基重复类型,说明不同物种中不同重复类型的丰度差异较大。不同物种的优势微卫星重复类型不一样,反映了不同物种基因组特征。有研究认为(A)n类型微卫星的高频出现是由高密度散在分布的逆转录转座子,如Alu和LINE,以及经加工的假基因的Poly A尾所产生的(Tóth et al., 2000)。红尾蚺中重复序列含量最高的类型为LINE,占基因组的13.03%(Yin et al., 2016),与这一推测相符。
本研究4个物种中,非翻译区(包括5'UTR和3'UTR)微卫星的丰度都比编码区高,说明微卫星在非翻译区聚集,推测其可能影响基因的转录活性。红尾蚺和原矛头蝮2个蛇类物种与人类和小鼠2个哺乳类物种相比,编码区微卫星的数量和丰度相差很小,而在基因的内含子、外显子和基因间区相差很大。这表明了蛇类与哺乳类基因中编码区微卫星的数量和丰度相差较小,可能是因为CDS在不同物种中比较保守,受到的选择压力大。
微卫星对扩张和收缩非常敏感,编码区单个单碱基重复、二碱基重复、四碱基重复和五碱基重复单元的插入或缺失都会导致移码。本研究发现,红尾蚺基因编码区中三碱基重复类型占编码区总数的84.07%,原矛头蝮基因编码区中三碱基重复类型占编码区总数的95.11%,在编码区6种重复类型中占绝对优势。原矛头蝮基因编码区三碱基重复类型比红尾蚺高,可能因为红尾蚺是一种比原矛头蝮更古老的蛇类(Reyes-Velasco et al., 2015),单碱基、二碱基、四碱基和五碱基类型在进化过程中发生插入或缺失突变,导致蛋白功能改变,从而很可能在进化过程中被淘汰;另一种可能是编码区三碱基重复类型的增加可以增加性状的多样性,有利于物种在进化过程中的适应性改变,从而在进化过程中被保留。有研究表明在对人、大猩猩Pan troglodytes、红毛猩猩Pongo pygmaeus、猕猴Macaca mulatta 4个高等哺乳动物中微卫星的比较分析,发现编码区6种重复类型的进化速度超过非编码区2倍多(Loire et al., 2013)。由此可见,编码区微卫星所受到的选择压力比非编码区大,进化速度更快。
红尾蚺和原矛头蝮2种蛇类物种基因组微卫星的丰度分布位置特征相似,并且与人和小鼠2种哺乳动物中微卫星的丰度分布位置特征也相似,都是基因上下游500 bp丰度最高,内含子次之,而外显子最低。有研究报道拟南芥Arabidopsis thaliana和水稻Oryza sativa ssp. japonica cv. Nipponbare 2种植物中微卫星的丰度沿着基因区5'到3'方向呈现出递减的趋势(Fujimori et al., 2003)。说明动物和植物基因组微卫星的丰度分布特征存在差异。有研究对42个已经测序的原核生物基因组编码区微卫星的差异和丰度进行分析,发现编码区微卫星的丰度呈“U型”分布,即基因左右末端微卫星的丰度较高,中间区域微卫星的丰度较低(Lin & Kussell,2012)。这说明真核生物和原核生物基因区微卫星的丰度分布存在差异。
对红尾蚺和原矛头蝮基因组含有微卫星的编码序列进行GO注释分析,可以看出这2个物种含有微卫星的编码区注释出的功能分类基本一致,但是与人和小鼠2种哺乳动物以及2种蝎子的结果(Wang et al., 2016)差异较大。对红尾蚺、原矛头蝮、人和小鼠4个物种中包含微卫星的CDS进行直系同源分析,发现只有3个基因家族被这4个物种共享,2个蛇类物种之间共享的基因家族比各自特有的基因家族多,2个哺乳类物种之间共享的基因家族也比各自特有的基因家族多。这说明含有微卫星的CDS的功能在不同门类间存在种系差异。微卫星的收缩或扩张为物种适应性进化过程中的遗传变异提供了丰富的原材料(Kashi & King,2006)。对群体之间、近源物种之间、种系之间基因组层面微卫星的挖掘和比较分析,将有助于进一步了解微卫星在基因组中的功能。
| 高焕, 刘萍, 孟宪红, 等. 2004. 中国对虾(Fenneropenaeus chinensis)基因组微卫星特征分析[J]. 海洋与湖沼, 35(5): 249–254. |
| 李成云, 李进斌, 周晓罡, 等. 2004. 粗糙脉孢菌基因组中的微卫星序列的组成和分布[J]. 中国农业科学, 37(6): 851–858. |
| 李午佼, 李玉芝, 杜联明, 等. 2014. 大熊猫和北极熊基因组微卫星分布特征比较分析[J]. 四川动物, 33(6): 874–878. |
| 魏朝明, 孔光耀, 廉振民, 等. 2007. 蜜蜂全基因组中微卫星的丰度及其分布[J]. 昆虫知识, 44(4): 501–504. DOI:10.7679/j.issn.2095-1353.2007.113 |
| Conesa A, Götz S, García-Gómez JM, et al. 2005. Blast2GO:a universal tool for annotation, visualization and analysis in functional genomics research[J]. Bioinformatics, 21(18): 3674–3676. DOI:10.1093/bioinformatics/bti610 |
| Fujimori S, Washio T, Higo K, et al. 2003. A novel feature of microsatellites in plants:a distribution gradient along the direction of transcription[J]. FEBS Letters, 554(1): 17–22. |
| Jurka J, Pethiyagoda C. 1995. Simple repetitive DNA sequences from primates:compilation and analysis[J]. Journal of Molecular Evolution, 40(2): 120–126. DOI:10.1007/BF00167107 |
| Kajitani R, Toshimoto K, Noguchi H, et al. 2014. Efficient de novo assembly of highly heterozygous genomes from whole-genome shotgun short reads[J]. Genome Research, 24(8): 1384–1395. DOI:10.1101/gr.170720.113 |
| Kashi Y, King DG. 2006. Simple sequence repeats as advantageous mutators in evolution[J]. Trends in Genetics, 22(5): 253–259. DOI:10.1016/j.tig.2006.03.005 |
| Katti MV, Ranjekar PK, Gupta VS. 2001. Differential distribution of simple sequence repeats in eukaryotic genome sequences[J]. Molecular Biology and Evolution, 18(7): 1161–1167. DOI:10.1093/oxfordjournals.molbev.a003903 |
| Kerkkamp HM, Kini RM, Pospelov AS, et al. 2016. Snake genome sequencing:results and future prospects[J]. Toxins, 8(12): 360–375. DOI:10.3390/toxins8120360 |
| Laurie JV, Janalee PC. 2009. Herpetology:an introduction biology of amphibians and reptiles (third edition)[M]. London: Academic Press: 551-578. |
| Li L, Stoeckert CJ, Roos DS. 2003. OrthoMCL:identification of ortholog groups for eukaryotic genomes[J]. Genome Research, 13(9): 2178–2189. DOI:10.1101/gr.1224503 |
| Li YC, Korol AB, Fahima T, et al. 2004. Microsatellites within genes:structure, function, and evolution[J]. Molecular Biology and Evolution, 21(6): 991–1007. DOI:10.1093/molbev/msh073 |
| Lin WH, Kussell E. 2012. Evolutionary pressures on simple sequence repeats in prokaryotic coding regions[J]. Nucleic Acids Research, 40(6): 2399–2413. DOI:10.1093/nar/gkr1078 |
| Loire E, Higuet D, Netter P, et al. 2013. Evolution of coding microsatellites in primate genomes[J]. Genome Biology and Evolution, 5(2): 283–295. DOI:10.1093/gbe/evt003 |
| Reyes-Velasco J, Card DC, Andrew AL, et al. 2015. Expression of venom gene homologs in diverse python tissues suggests a new model for the evolution of snake venom[J]. Molecular Biology and Evolution, 32(1): 173–183. DOI:10.1093/molbev/msu294 |
| Thiel T, Michalek W, Varshney R, et al. 2003. Exploiting EST databases for the development and characterization of gene-derived SSR-markers in barley (Hordeum vulgare L.)[J]. Theoretical and Applied Genetics, 106(3): 411–422. DOI:10.1007/s00122-002-1031-0 |
| Tóth G, Gáspári Z, Jurka J. 2000. Microsatellites in different eukaryotic genomes:survey and analysis[J]. Genome Research, 10(7): 967–981. DOI:10.1101/gr.10.7.967 |
| Wang C, Kubiak L, Du L, et al. 2016. Comparison of microsatellite distribution in genomes of Centruroides exilicauda and Mesobuthus martensii[J]. Gene, 594(1): 41–46. DOI:10.1016/j.gene.2016.08.047 |
| Yin W, Wang Z, Li Q, et al. 2016. Evolutionary trajectories of snake genes and genomes revealed by comparative analyses of five-pacer viper[J]. Nature Communications, 13107(7): 1–11. |
 2017, Vol. 36
2017, Vol. 36