扩展功能
文章信息
- 王文栋, 束梅影, 张达艳, 徐世清
- WANG Wendong, SHU Meiying, ZHANG Dayan, XU Shiqing
- 家蚕昼夜节律生物钟基因的生物信息学分析
- Bioinformatics Analysis of Circadian Rhythm Biological Clock Genes in Bombyx mori
- 四川动物, 2016, 35(2): 275-282
- Sichuan Journal of Zoology, 2016, 35(2): 275-282
- 10.11984/j.issn.1000-7083.20150316
-
文章历史
- 收稿日期: 2015-10-13
- 接受日期: 2015-12-15
2. 苏州大学医学部, 江苏苏州 215123
2. Medical College of Soochow University, Suzhou, Jiangsu Province 215123, China
昼夜节律(circadian rhythm)又称近日节律,是最普遍的生物节律现象,受遗传基因调控,是生物体生命活动以近24 h为周期的变动。无论是单细胞生物,还是高等动植物均具有昼夜节律现象。昆虫昼夜节律生物钟的分子机制在黑腹果蝇Drosophila melanogaster中有较为深入的研究,在其他昆虫中的研究相对较少(Williams & Sehgal,2001)。目前在果蝇中已确认的主要昼夜节律生物钟基因有周期蛋白基因(Period,Per)、永恒蛋白基因(Timeless,Tim1)、时钟蛋白基因(Clock,Clk)、周期循环蛋白基因(Cycle,Cyc)、旋转蛋白基因(Vrille,Vri)和隐花色素基因(Cryptochrome,Cry)等(Young,2000)。尽管不同物种中生物钟基因的结构和功能存在着某些细节差异,但它们的基本反馈环路是非常保守的(Sauman & Reppert,1996;Rubin et al.,2006;Codd et al.,2007)。
昆虫的昼夜节律生物钟基因不仅是驱动自身生理和行为昼夜节律输出的分子基础(Hall,2003),还调控了多种近年节律生物行为的时相,例如滞育和迁徙等(Saunders,2002),这些现象可成为研究昼夜节律生物钟基因的参照。目前,大红斑蝶Danaus plexippus已经成为研究昼夜节律生物钟在昆虫迁徙过程中导航作用的模式物种(Reppert,2006)。而昆 虫滞育的季节性则是研究昆虫对光周期响应的典范。
家蚕Bombyx mori是卵滞育鳞翅目昆虫,其滞育发生在胚胎初期,受亲代胚胎发育后期与胚后时期昼夜节律授时因子(温度和光照等)的诱导,是一种典型的对外界环境特征主动适应的现象。目前家蚕昼夜节律生物钟基因Cry1、Cry2、Per和Tim已经完成了克隆和鉴定(Iwai et al.,2006;王文栋等,2011),其在不同光照条件下的组织表达已经有所研究,但关于家蚕昼夜节律生物钟基因的研究还是相对较少,而家蚕的滞育是对昼夜节律授时因子(温度和光照等)响应的一种现象,这为研究家蚕昼夜节律生物钟基因提供了一个很好的参照。在家蚕中是否具有与果蝇或大红斑蝶类似的生物钟基因网络呢?对家蚕昼夜节律生物钟相关基因的克隆和鉴定,最终获得基因反馈环路是我们研究的目标。
1 材料与方法 1.1 数据资料电子克隆、基因结构及分子进化分析所使用的表达序列标签(expressed sequence tags,EST)、全基因组鸟枪法(whole genome shotgun,WGS)和蛋白序列均来自美国国家生物信息中心(National Center for Biotechnology Information,NCBI)数据库(http://www.ncbi.nlm.nih.gov/)。
1.2 电子克隆家蚕Bmvri、Bmpdp、Bmcyc、Bmtim2和Bmclk基因的电子克隆分别以柞蚕Antheraea perhyi VRI蛋白序列(登录号:AAS92609)、黑腹果蝇PDP蛋白序列(登录号:AAF20153)、黑腹果蝇CYC蛋白序列(登录号:AAF49107)、黑腹果蝇TIM2蛋白序列(登录号:AAF54908)和柞蚕CLK蛋白序列(登录号:AAR14936)作为质询序列在家蚕的ESTs数据库中进行tblastn比对,检索出与质询序列有同源性或有部分重叠的ESTs(同源长度≥100 bp,同源性在50%以上,85%以下),用DNAstar软件中的SeqMan将检出序列拼接成Contig,再用Contig为质询序列进行nblast检索和Contig的拼接和校对,直至没有新的EST 可供拼接为止。利用DNAstar软件的EditSeq寻找Contig中的开放阅读框(open reading frame,ORF),用Translation tool将它们翻译成蛋白序列。
1.3 基因结构和染色体定位分析应用NCBI提供的blastn界面,将克隆获得的家蚕Bmvri,Bmcyc,Bmtim2和Bmpdp基因的ORF序列在家蚕WGS数据库中进行比对,确定外显子、内含子区域,进一步用Sim4在线工具(Florea et al.,1998)进行验证,利用GSDS(郭安源等,2007)绘制基因结构图。基因染色体定位采用SilkDB数据库(Xia et al.,2004)提供的在线分析软件SilkMap。采用蛋白质组学服务器ExPASy(Gasteiger et al.,2003)提供的Translate tool界面将ORF转化为蛋白序列。
1.4 芯片表达谱分析将家蚕昼夜节律生物钟基因的ORF序列在BmMDB数据库(http://silkworm.swu.edu.cn/microarray/index.php)中进行blast比对,寻找到目的基因的探针序列,最终获得目的基因在5L-3d多组织(器官)中的芯片检测表达谱,统计分析荧光值作为基因表达量值,绘制柱形图。
1.5 蛋白序列分析同源性分析和多序列比对由在线工具ClustalW(Thompson et al.,1994)和Weblogo(Crooks et al.,2004)完成。蛋白翻译和基序(MOTIFS)分析分别采用蛋白质组学服务器ExPASy提供的Translate tool界面和ScanProsite分析界面。蛋白质功能域的预测由SMART在线工具(Letunic et al.,2009)完成。
1.6 分子系统进化分析应用MEGA 4.0软件(Tamura et al.,2007)对蛋白质序列数据进行分子系统进化分析,采用Bootstrap Test of Phylogeny界面中的邻接法(neighbor-joining,NJ;Saitou & Nei,1987),5 000次重复构建分子进化树,进行分子系统进化分析。
2 结果与分析 2.1 确定和克隆家蚕中可能存在的昼夜节律生物钟基因目前已经克隆和登录的家蚕昼夜节律生物钟基因有BmPer,BmTim1,Bmcry1和Bmcry2基因。利用现有的数据资源,电子克隆了家蚕中与果蝇其他昼夜节律生物钟基因(Vri,Pdp,Tim2,Cyc)同源基因的完整ORF序列(长度分别为:1 164 bp,858 bp,3 756 bp和1 986 bp),以及家蚕中果蝇Clk基因的同源基因的ORF片段(1 266 bp),根据克隆获得的Bmvri,Bmpdp,Bmtim2和Bmcyc基因的ORF序列推导它们编码产物BmVRI、BmCYC、BmTIM2和BmPDP蛋白的序列(表 1)。 可见家蚕基本具有果蝇昼夜节律生物钟基因的同源基因,这为建立家蚕的昼夜节律生物钟网络及研究其分子机制提供了序列基础。
| 基因Gene | 基因数据来源Genomic data source | 基因长度Gene length/bp | 蛋白数据来源Protein data source | 蛋白长度Protein length/AA |
| Bmvri | clone ORF | 1 164 | deduce | 387 |
| Bmpdp | clone ORF | 858 | deduce | 285 |
| Bmper | DQ393413 | 3 714 | ABF21088 | 1 113 |
| Bmtim1 | DQ392962 | 3 688 | ABD52733 | 997 |
| Bmtim2 | clone ORF | 3 756 | deduce | 1 251 |
| Bmcry1 | HM747059 | 1 753 | deduce | 536 |
| Bmcry2 | HM747060 | 2 400 | deduce | 730 |
| Bmcyc | clone ORF | 1 986 | deduce | 661 |
| Bmclk | clone fragment | 1 266 | — | — |
家蚕5龄第3天10种组织(器官)的芯片检测表达谱显示(图 1:A,B),Bmpdp基因在头和中肠表达丰度最高,在脂肪体和血淋巴中较低,在睾丸、卵巢、体壁、丝腺(前、中、后部)和马氏管中都有一定的表达。Bmtim2基因在生殖腺(睾丸、卵巢)和丝腺(前、中、后部)中表达丰度最高,在马氏管中较低,在其他组织(器官)均有一定表达。Bmcyc基因在马氏管和后部丝腺中表达丰度最高,在中肠和血淋巴中表达量很低,在其他组织(器官)均有一定表达。Bmvri基因在生殖腺、头部、中肠和丝腺(前、中、后部)表达丰度最高,在脂肪体中表达量较低,在其他组织(器官)中均有一定表达。
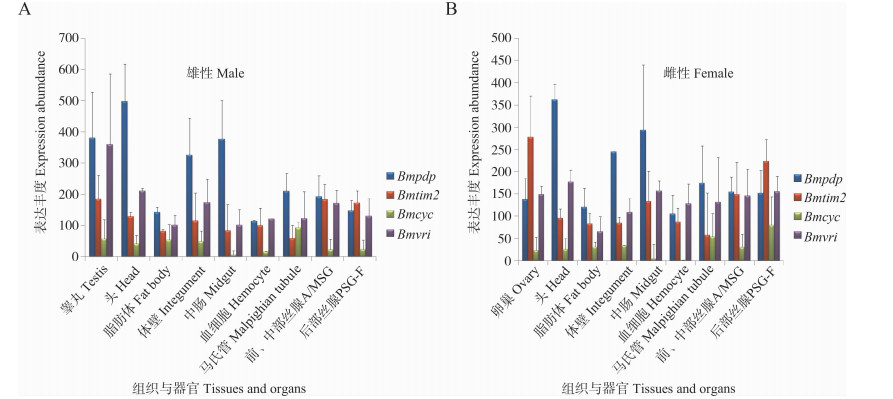
|
| 图 1 家蚕Bmpdp,Bmtim2,Bmcyc和Bmvri基因芯片表达谱 Fig. 1 Microarray expression profiles of Bmpdp, Bmtim2, Bmcyc and Bmvri genes |
染色体定位结果(图 2:A)显示,Bmpdp基因位于家蚕第1号染色体的nscaf2734上,具体位置为(114 154~122 417)nt;Bmtim2基因位于家蚕 第9号染色体的nscaf3048上,具体位置为(444 426~ 477 441)nt;Bmcyc基因位于家蚕第1号染色体的nscaf2734上,具体位置为(280 403~296 865)nt;Bmvri基因位于家蚕第27号染色体的nscaf3072上,具体位置为(2 451 469~24 527 593)nt。Bmpdp与Bmcyc基因位于同一条染色体的同一个nscaf上,只是具体位置存在差异。
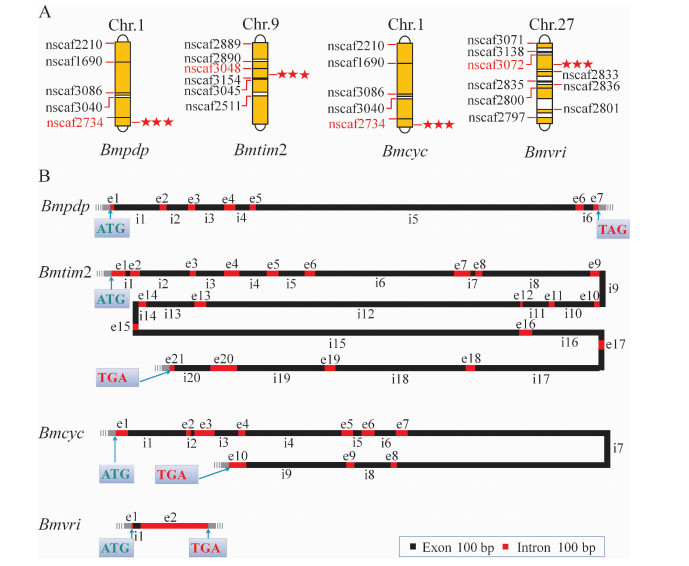
|
| 图 2 家蚕Bmpdp, Bmtim2, Bmcyc, Bmvri基因的染色体定位(A)及基因结构分析(B) Fig. 2 Chromosome locations (A) and structures (B) of Bmpdp, Bmtim2, Bmcyc and Bmvri genes |
根据电子克隆拼接Bmpdp,Bmtim2,Bmcyc,Bmvri基因的ORF序列,应用NCBI提供的blastn界面,分别在家蚕WGS全基因组数据库中进行比对分析,获得了它们的基因结构(图 2:B)。
Bmvri基因的ORF分布在WGS-BABH01026441上,具有2个外显子,1个内含子。Bmpdp基因的ORF分布在WGS-BABH01031184上,具有7个外显子,6个内含子。Bmtim2基因的ORF分布在WGS-BABH01032134上,具有21个外显子,20个内含子。Bmcyc基因具有10个外显子,9个内含子,第1~7外显子位于WGS-BABH01031197上,第8~10外显子位于WGS-BABH01031193上,第7内含子存在缺口,WGS-AADK01000483可以修补这个缺口,从而获得完整的第7内含子。这些基因的内含子/外显子边界均符合GT-AG法则。
2.4 家蚕昼夜节律生物钟蛋白的功能域及基序位点 2.4.1 VRI和PDPSMART分析结果(图 3:A-1,A-2)显示,家蚕VRI(BmVRI)与黑腹果蝇VRI(DmVRI)及哺乳动物小鼠Mus musculus的NFIL3/E4BP4(MmNFIL3/E4BP4)都具有bZIP domain和DNA binding domain,而家蚕PDP(BmPDP)与黑腹果蝇PDP(DmPDP)及小鼠的HLF(MmHLF)除具有bZIP domain和DNA binding domain以外,还具备转录活性域(TDA domain)及脯氨酸和酸性氨基酸富集区(PAR domain)。
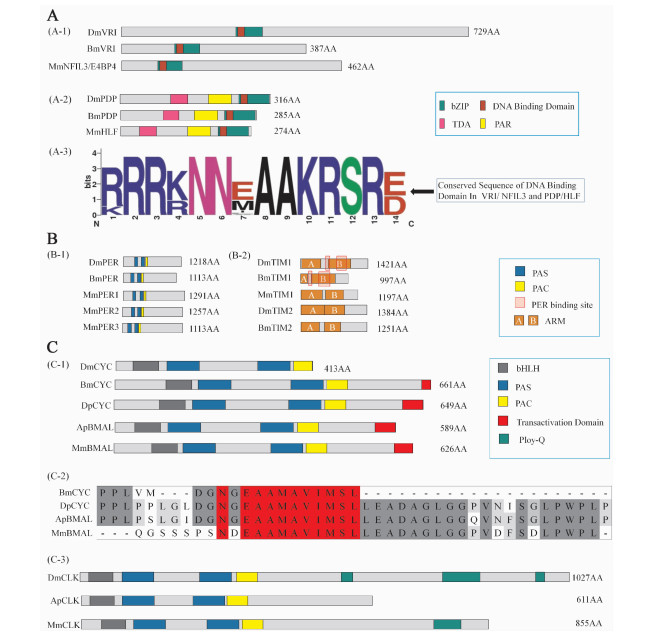
|
| 图 3 家蚕昼夜节律生物钟蛋白的功能域与基序位点 Fig. 3 Putative functional domains and motifs on Bombyx mori circadian rhythm clock proteins |
Weblogo序列比对分析结果(图 3:A-3)显示,不同物种的VRI、NFIL3/E4BP4、PDP和HLF的DNA binding domain是高度保守的,只有个别氨基酸残基有差异。家蚕BmVRI和BmPDP蛋白的DNA binding domain氨基酸残基序列非常相似,推测它们可以结合相似的DNA序列,这很可能就是Clk基因的启动子序列。
2.4.2 PER和TIM(TIM1和TIM2)结果(图 3:B-1,B-2)显示,家蚕BmPER与果蝇DmPER和小鼠的3种PER蛋白(MmPER1、MmPER2和MmPER3)一样,都具备2个PAS功能域(信号蛋白的信号传感功能域)以及1个PAC功能域(存在于PAS基序的C-端,有助于PAS结构域的折叠)。
家蚕BmTIM1和BmTIM2,果蝇DmTIM1和DmTIM2,小鼠MmTIM1都具备ARM功能域(参与蛋白的信号传导),但在家蚕BmTIM1、果蝇DmTIM1中还具备PER-binding domain,从结构上看,昆虫的TIM1可以与PER结合,而昆虫的TIM2与哺乳动物小鼠的MmTIM1相似,都没有PER-binding domain,从功能域组成上看,昆虫的TIM2与脊椎动物的TIM1相似。
PER和TIM(TIM1和TIM2)都不具备DNA结合位点,但它们都具有蛋白质互相作用的基序位点。可见PER和TIM是通过蛋白质互作实现信号传感的。
2.4.3 CYC和CLK家蚕BmCYC与果蝇的DmCYC、大红斑蝶的DpCYC、柞蚕的ApBMAL和小鼠的MmBMAL相似,都具有参与蛋白信号传导的bHLH、PAS、PAC功能域(图 3:C-1),除此之外,鳞翅目昆虫家蚕BmCYC、大红斑蝶的DpCYC、柞蚕的ApBMAL还具备与小鼠MmBMAL相似的转录激活功能域(transactivation domain),而且这个功能域很保守,都具有一段N-EAAMAVIMSL序列(图 3:C-2)。从功能域上看,鳞翅目昆虫的CYC与果蝇的CYC差异大于与脊椎动物的BMAL的差异。
在家蚕中没有电子克隆获得Clock基因,但在大红斑蝶、柞蚕中都存在Clock基因,它们编码的CLK蛋白与果蝇的DmCLK、小鼠的MmCLK相似,都具备bHLH、PAS、PAC功能域,但DmCLK和MmCLK中都有Ploy-Q,而柞蚕中没有这个结构域(图 3:C-3)。
2.5 系统发生关系 2.5.1 VRI和PDP昆虫VRI在脊椎动物中的同源蛋白为NFIL3/E4BP4,昆虫PDP在脊椎动物中的同源蛋白为HLF/TEF。分子系统发生树构建结果(图 4:A,B)显示,家蚕中的VRI和PDP分属于昆虫的VRI与PDP蛋白组群,与其在脊椎动物中的同源蛋白进化距离较远。
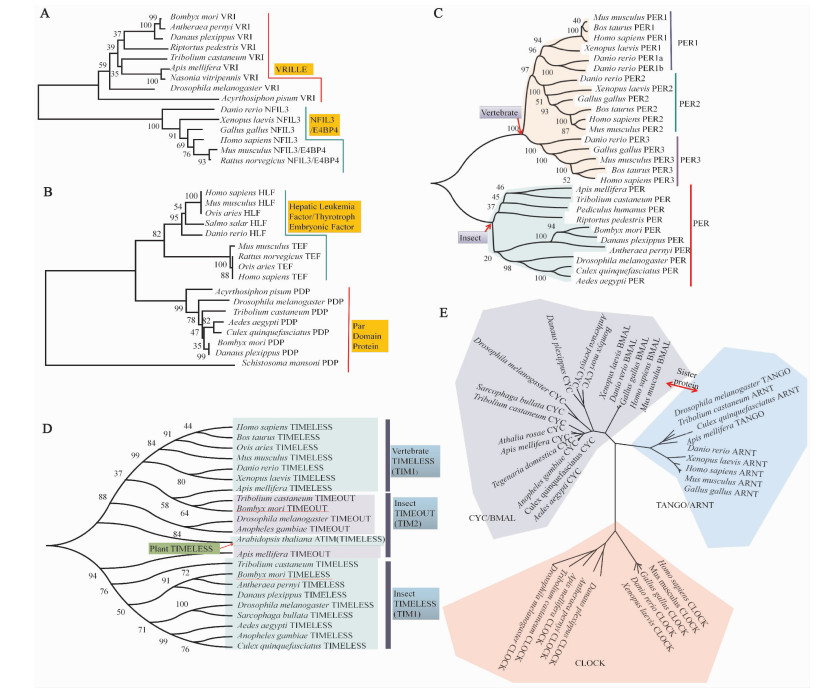
|
| 图 4 昼夜节律生物钟蛋白的进化树分析 Fig. 4 Phylogenetic trees of circadian rhythm clock proteins |
通过在NCBI数据库中检索,发现脊椎动物(人Homo sapiens、小鼠、奶牛Bos taurus、斑马鱼Danio rerio)都具有PER1、PER2和PER3蛋白,其中斑马鱼的PER1又分为PER1a和PER1b两种亚型,在原鸡Gallus gallus中只查询到了PER2和PER3,而在爪蟾Xenopus laevis中只查询到PER1和PER2。此外,在植物中没有查询到相应的PER蛋白。在昆虫(家蚕、柞蚕、大红斑蝶、黑腹果蝇、埃及伊蚊Aedes aegypti、致倦库蚊Culex quinquefasciatus、蜜蜂Apis mellifera、赤拟谷盗Tribolium castaneum、体虱、点蜂缘蝽Riptortus pedestris)中只发现1种PER。
利用MEGA 4.0软件构建进化树(图 4:C)后发现,脊椎动物的PER(PER1、PER2和PER3)聚为一大类,昆虫的PER聚为一大类,其中脊椎动物的PER1和PER2进化距离更近,同纲目昆虫物种的PER进化距离较近,符合它们的亲缘关系。
2.5.3 TIM1(TIMELESS)和TIM2(TIMEOUT)通过NCBI数据库检索,发现在昆虫(家蚕、柞蚕、大红斑蝶、黑腹果蝇、麻蝇、疟蚊、埃及伊蚊、 致倦库蚊、蜜蜂、赤拟谷盗)和脊椎动物(人、奶牛、绵羊Ovis aries、小鼠、斑马鱼、爪蟾)中都具有TIM1,此外植物拟南芥Arabidopsis thaliana也具有TIM1,称为ATIM。可见TIM1从植物到昆虫再到脊椎动物是普遍存在的。
目前关于TIM2的报道还不是很多,在NCBI数据库中没有查询到脊椎动物的TIM2,只有部分昆虫(黑腹果蝇、疟蚊、赤拟谷盗、蜜蜂)中报道具有TIM2,我们根据黑腹果蝇的TIM2蛋白序列,电子克隆了家蚕的Tim2基因,并翻译为蛋白序列用于进化分析。
分子系统发生树构建结果(图 4:D)显示,选取的蛋白数据聚为两大类:脊椎动物的TIM1与昆虫的TIM2聚为一类,昆虫的TIM1聚为一类。拟南芥的ATIM(TIM1)与昆虫的TIMEOUT进化距离较近。无论从进化距离还是功能域组成,昆虫的TIM2都与脊椎动物的TIM1相似,而蜜蜂的TIM1与脊椎动物的TIM1进化距离较近,蜜蜂的TIM2与昆虫的TIM1进化距离较近。由此可见脊椎动物的TIM1在昆虫中的真正同源蛋白是TIM2,而不是TIM1。
2.5.4 CLK和CYC(BMAL)分子系统发生树构建结果(图 4:E)显示,昆虫的CYC与脊椎动物的BMAL分属两类,脊椎动物的BMAL是CYC在脊椎动物中的同源蛋白,而在昆虫和脊椎动物中均还存在一种TANGO/ARNT蛋白,它们与CYC/BMAL进化关系较近,推测CYC/BMAL与TANGO/ARNT起源于同一种蛋白,进化产生了分歧,形成了2个大的分支。
而CLK在昆虫和脊椎动物中是普遍存在的,它们分为昆虫CLK和脊椎动物CLK两类,同纲目昆虫物种同源蛋白进化距离较近,符合它们的亲缘关系。
2.6 家蚕昼夜节律生物钟基因网络的初步整合根据家蚕中已知和克隆获得的昼夜节律生物钟基因和它们编码蛋白的功能域特性,以及果蝇(Giebultowicz,2001;Leise & Moin,2007)和大红斑蝶(Zhu et al.,2008)的昼夜节律生物钟网络分子机制,整合了家蚕的昼夜节律生物钟基因网络(图 5)。
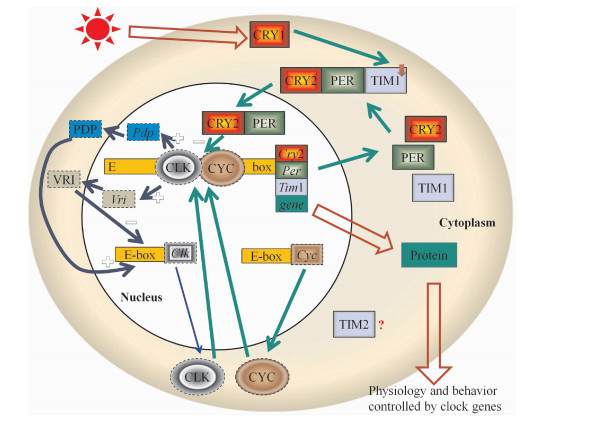
|
| 图 5 家蚕昼夜节律生物钟基因互作网络 Fig. 5 Gene interaction network of circadian rhythm clock in Bombyx mori |
在第一个环路中,BmCLK和BmCYC蛋白结合形成异二聚体,在细胞核中结合到Bmcry2、Bmper、Bmtim1基因和受生物钟基因调控和控制的某些生理和行为的基因E-box上,正向调节它们的转录。而BmCRY1、BmPER和BmTIM1在细胞浆堆积形成BmCRY1/BmPER/BmTIM1三聚体,当BmCRY1感受到光刺激后将信号传到这个三聚体,BmTIM1被降解,从而BmCRY1/BmPER异二聚体转移到细胞核,抑制BmCLK/BmCYC异二聚体的形成,从而抑制它们自身的转录,此环路主要是调节昼夜节律的周期长短和时相。
在第二个环路上,BmVRI和BmPDP分别是BmClk基因转录的阻遏物和激活物,Bmvri基因编码的BmVRI蛋白属于bzip转录因子,Bmpdp基因(PAR domain protein)编码的BmPDP蛋白含有PAR(Proline and Acidic Rich),它们的DNA结合位点高度相似,推测可结合相同的DNA序列位点,即Bmclk基因的启动子序列。BmCLK可能同时激活Bmvri和Bmpdp基因的转录。BmVRI比BmPDP堆积和消失得早,从而BmCLK/BmCYC的抑制和激活在时间上分离,BmVRI和BmPDP互相协调使得BmCLK的表达呈现昼夜振荡。第二个环路主要调节昼夜节律的幅度和特定的昼夜输出(王文栋,2011)。
3 讨论在果蝇中,VRI和PDP蛋白分别是Clk基因转录的阻遏物和激活物,它们竞争结合Clk基因的启动子序列,在家蚕中也存在这2种蛋白,并且它们具有高度相似的DNA结合功能域,这为它们竞争和结合相同的DNA序列(Clk基因的启动子)提供了序列结构基础。BmPER和BmTIM(BmTIM1和BmTIM2)都不具备DNA结合位点,但都具有蛋白质信号传导互作基序位点,而且BmTIM1上有与PER蛋白结合的位点,BmPER和BmTIM1是可以结合的,可见BmPER和BmTIM1是通过蛋白质互作实现信号传感的。BmCYC除具有蛋白质互作基序位点(bHLH、PAS、PAC)外,还具备高度保守的转录激活功能域。
昆虫的VRI、PDP、CYC和CLK在脊椎动物中的同源蛋白分别为NFIL3/E4BP4、HLF/TEF、BMAL和CLK。而在脊椎动物中PER有3种,PER1、PER2和PER3,昆虫中只有1种PER。而无论从进化距离还是功能域来看,昆虫的TIM2都与脊椎动物的TIM1相似,可见脊椎动物的TIM1在昆虫中的真正同源蛋白是TIM2,而不是TIM1。从系统进化树来看,家蚕的BmVRI、BmPDP、BmPER、BmTIM1、BmTIM2和BmCYC分属于昆虫相应的蛋白组群,同纲目昆虫物种的同源蛋白进化距离较近,符合它们的亲缘关系。
随着基因和蛋白数据库的发展和完善,昼夜节律生物钟相关基因在越来越多物种中被克隆和鉴定。家蚕的滞育是典型的对温度和光照主动适应的现象,所以进一步研究温度、光照和食物对家蚕昼夜节律生物钟基因Bmcry1,Bmcry2,Bmper,Bmtim1,Bmtim2,Bmcyc,Bmclk,Bmvri,Bmpdp等的影响,将整合出详尽的家蚕昼夜节律生物钟基因网络图,也对研究其他昆虫物种的生物钟基因有所帮助。
| 郭安源, 朱其慧, 陈新, 等. 2007. GSDS: 基因结构显示系统[J]. 遗传, 29(8): 1023-1026. |
| 王文栋, 梁辉, 朱晓苏, 等. 2011. 家蚕生物钟基因Bmcry1与Bmcry2的克隆及生物信息学分析[J]. 昆虫学报, 54(1): 9-19. |
| 王文栋. 2011. 家蚕TIME-EA4进化及昼夜节律生物钟基因的克隆、进化和反馈环路研究[D]. 苏州: 苏州大学: 80-89 . |
| Codd V, Dolezel D, Stehlik J, et al. 2007. Circadian rhythm gene regulation in the housefly Musca domestica[J]. Genetics, 177: 1539-1551. |
| Crooks GE, Hon G, Chandonia JM, et al. 2004. WebLogo: a sequence logo generator[J]. Genome Research, 14: 1188-1190. |
| Florea L, Hartzell G, Zhang Z, et al. 1998. A computer program for aligning a cDNA sequence with a genomic DNA sequence[J]. Genome Research, 8(9): 967-974. |
| Gasteiger E, Gattiker A, Hoogland C, et al. 2003. ExPASy: the proteomics server for in-depth protein knowledge and analysis[J]. Nucleic Acids Research, 31(13): 3784-3788. |
| Giebultowicz JM. 2001. Peripheral clocks and their role in circadian timing: insights from insects[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 356(1415): 1791-1799. |
| Hall JC. 2003. Genetics and molecular biology of rhythms in Drosophila and other insects[J]. Advances in Genetics, 48: 1-280. |
| Iwai S, Fukui Y, Fujiwara Y, et al. 2006. Structure and expressions of two circadian clock gene, period and timeless in the commercial silk moth, Bombyx mori[J]. Journal of Insect Physiology, 52: 625-637. |
| Leise TL, Moin EE. 2007. A mathematical model of the Drosophila circadian clock with emphasison posttranslational mechanisms[J]. Journal of Theoretical Biology, 248(1): 48-63. |
| Letunic I, Doerks T, Bork P. 2009. SMART 6: recent updates and new developments[J]. Nucleic Acids Research, 37: 229-232. |
| Reppert SM. 2006. A colorful model of the circadian clock[J]. Cell, 124: 233-236. |
| Rubin EB, Shemesh Y, Cohen M, et al. 2006. Molecular and phylogenetic analyses reveal mammalian-like clockwork in the honey bee (Apis mellifera) and shed new light on the molecular evolution of the circadian clock[J]. Genome Research, 16: 1352-1365. |
| Saitou N, Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 4: 406-425. |
| Sauman I, Reppert SM. 1996. Circadian clock neurons in the silkmoth Antheraea pernyi: novel mechanisms of period protein regulation[J]. Neuron, 17: 889-900. |
| Saunders DS. 2002. Insect clocks 3rd edition[M]. Amsterdam: Elsevier Science: 560. |
| Tamura K, Dudley J, Nei M, et al. 2007. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 24: 1596-1599. |
| Thompson JD, Higgins DG, Gibson TJ. 1994. ClustalW: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 22: 4673-4680. |
| Williams JA, Sehgal A. 2001. Molecular components of the circadian system in Drosophila[J]. Annual Review of Physiology, 63: 729-755. |
| Xia Q, Zhou Z, Lu C, et al. 2004. A draft sequence for the genome of the domesticated silkworm (Bombyx mori)[J]. Science, 306: 1937-1940. |
| Young MW. 2000. Circadian rhythms: marking time for a kingdom[J]. Science, 288: 451-453. |
| Zhu H, Sauman I, Yuan Q, et al. 2008. Cryptochromes define a novel circadian clock mechanism in monarch butterflies that may underlie sun compass navigation[J].PLoS Biology, 6: e4. DOI: 10.1371/journal.pbio.0060004. |
 2016, Vol. 35
2016, Vol. 35




