扩展功能
文章信息
- 刘旭, 刘振生, 丁由中, 王小明
- LIU Xu, LIU Zhensheng, DING Youzhong, WANG Xiaoming
- 基于粪便DNA的岩羊微卫星引物的筛选及个体识别
- Microsatellite Primers Screening and Individual Identification of Blue Sheep Based on the Fecal DNA
- 四川动物, 2015, 34(2): 169-173
- Sichuan Journal of Zoology, 2015, 34(2): 169-173
- 10.3969/j.issn.1000-7083.2015.02.002
-
文章历史
- 收稿日期:2014-11-11
- 接受日期:2015-01-04
2. 国家海洋局海岛研究中心, 福建平潭 350400;
3. 东北林业大学野生动物资源学院, 哈尔滨 150040;
4. 国家林业局野生动物保护生物学重点实验室, 哈尔滨 150040
2. Island Research Center, The State Oceanic Administration, Pingtan, Fujian Province 350400, China;
3. College of Wildlife Resources, Northeast Forest University, Harbin 150040, China;
4. Key Laboratory of Conservation Biology, State Forestry Administration, Harbin 150040, China
岩羊Pseudois nayaur是国家Ⅱ级重点保护野生动物,主要分布在青藏高原及其周边地区,是典型的高山动物,营社会集群性生活(王小明等,1998;曹丽荣等,2005)。历史上岩羊曾遍布整个宁夏(Allen,1940),而现在宁夏地区仅在贺兰山才有其分布(王香亭,1990)。贺兰山地处我国西北干旱地区,其四周被河流、沙漠、城市所包围,生境破碎化使贺兰山岩羊无法与其他分布区的岩羊产生有效的基因交流。近年来贺兰山岩羊种群增长迅速,已经从20世纪80年代1000只左右发展到现在的10 000余只(刘振生等,2004)。适时开展针对贺兰山岩羊的全面、深入的生态学研究是对其采取科学管理和保护的重要前提,因而,研究生活在这座"生态孤岛"上的岩羊种群是否是健康持续发展的种群显得尤为迫切和重要。为了更好地保护和管理贺兰山的岩羊种群,我们采用非损伤性取样方法对岩羊粪便DNA进行微卫星引物筛选,评估其遗传多样性,为进一步的保护和管理奠定基础。 1 研究地区概况
宁夏贺兰山国家级自然保护区位于贺兰山山脉东坡的中段和北段,地理坐标为38°19'~39°22'N,105°49'~106°41'E,面积1935.36 km2。贺兰山为地垒式山地,山体庞大,地势陡峻。全年干燥少雨,寒暑变化强烈,日照强,无霜期短,因其海拔高,而又具有山地气候的特点。由于贺兰山位于我国温带草原区与荒漠区的分界处,所以其植被类型比较复杂。它有标志山地所在水平地带属性的草原和荒漠,也有山地植被垂直系列中出现的针叶林和疏林草原,还有各种灌丛、草甸和落叶阔叶林等(刘振生等,2005a,2005b,2008)。 2 研究材料及方法 2.1 样品采集
于2012年5—8月在贺兰山国家级自然保护区开展岩羊粪便样品采集。通过粪便的颜色、风干程度来判断样本的新鲜度,采集较新鲜的粪便样本以保证所提取DNA模板的质量。为了避免重复采样,我们采取对主要粪便堆进行采集,不采集附近的零散粪粒,并且每一处只采集一份粪便样品。将不同地点采集的粪便样品分别装入盛有无水乙醇的采集瓶中,并带回实验室-20 ℃保存,共采集到粪便样品222份。对照样品为本实验室已有的岩羊组织样品DNA(来源于贺兰山自然死亡的岩羊个体的毛皮)。 2.2 研究方法 2.2.1 岩羊粪便DNA的提取
粪便DNA使用E.Z.N.A.TM Stool DNA Kit(Omega Bio-tek)试剂盒,参照说明书指示提取。 2.2.2 微卫星位点筛选
根据重复单元大于等于2和重复次数较多的原则,通过本实验室已有引物、查阅文献和NCBI公共数据库得到36对岩羊近缘物种(绵羊Ovis aries、牦牛Bos grunniens、家牛Bos taurus、印度水牛Bubalus bubalis、山羊Capra hircus)的微卫星位点信息和引物序列。先用岩羊组织样品提出的DNA进行试验,然后用成功扩增的位点对粪便样品中提出的DNA进行检测。 2.2.3 PCR扩增
PCR扩增采用15 μL的反应体系:DNA模板1.5 μL、Premix Ex Taq聚合酶(1.25 U)7.5 μL、牛血清白蛋白(BSA)(20 g·L-1;Takara,Otsu,Japan)0.5 μL、引物各1 μL(10 μM)、双蒸水定容至15 μL。PCR扩增程序如下:94 ℃预变性3 min;94 ℃变性30 s,退火30 s(退火温度因引物而异),72 ℃延伸45 s,35个循环;最后72 ℃延伸20 min。在使用PCR扩增仪对各引物进行扩增时,设置退火温度梯度,以寻找各对引物的最适退火温度。PCR产物经2.0%琼脂糖凝胶电泳检测,溴化乙锭(EB)染色后,在凝胶成像系统中观察扩增效果,并拍照记录。为了确保结果准确可靠,我们对每个样本均重复扩增2次以上,统计2次扩增一致的条带。 2.2.4 PCR产物的检测
采用毛细管电泳法确定位点的多态性。将初步筛选出的引物重新合成,在上游引物5’端分别用三色荧光标记羟基荧光素(FAM)、六氯荧光素(HEX)和羟基四甲基罗丹明(TAMARA)。对222份粪便DNA样品重新进行扩增。扩增产物取5 μL进行2.0%琼脂糖凝胶电泳30 min,检验PCR反应是否成功。扩增后将PCR产物按比例混合成一组,每组10~15 μL,低温送至上海美吉生物技术有限公司测定等位基因的大小。等位基因结果使用Genemapper v.4.0软件(美国应用生物系统公司)进行分析。为获取准确的基因型,本实验重复进行2次PCR扩增,若同一个样品2次重复PCR结果读取得到的数据不一致或无法确定,则再进行2次重复扩增,直至得到准确的基因型为止。 2.2.5 数据统计及分析
在测得样品的基因型数据后,用Microsatellite Tool kit程序来寻找数据中相匹配的基因型,并且判断不同的岩羊个体(Park,2001),进行种群数量评估,鉴别个体时设定以下原则:所用微卫星座位上的基因型都相同或有一个不同即为同一个个体(Bellemain et al.,2005)。然后用Gimlet 1.3.3(Valière,2002)软件分析这些选定的微卫星位点在识别无亲缘关系个体和双胞胎的概率(PI值)。等位基因数量(A)、观测杂合度(HO)和期望杂合度(HE)、Hardy-Weinberg平衡检测用GENEPOP 4.2计算。多态信息含量(PIC)用Mstools软件计算。 3 结果与分析 3.1 粪便DNA质量检测结果
使用本实验室已有的岩羊微卫星引物(IDVGA-29)检测所提取的岩羊粪便DNA质量,由图 1可见,岩羊粪便DNA成功扩增,粪便DNA扩增产物与组织DNA扩增产物大小一致(160 bp)。

|
|
图 1 位点IDVGA-29对岩羊粪便和组织DNA的扩增产物电泳结果
Fig. 1 Electrophoresis results of blue sheep fecal DNA and tissue DNA amplification products based on primer IDVGA-29
注: 1~18. 不同采样点岩羊粪便的DNA扩增产物; 19. 阴性对照; 20. 岩羊组织DNA扩增产物。 Notes: 1~18. the PCR amplifications of the blue sheep’s fecal DNA; 19. the negative control; 20. the PCR amplification of the blue sheep’s tissue DNA. |
本实验从36对微卫星引物中筛选出 9 对可以在岩羊组织及粪便中稳定扩增的微卫星引物(表 1)用于识别岩羊个体。通过毛细管电泳法测定等位基因的大小,本实验岩羊粪便中筛选到的微卫星位点大小均在100~200 bp,且多态性较好,适合用于岩羊的遗传多样性和个体识别的研究。

|
将9对引物分为3组,在上游引物5’端分别用三色荧光标记FAM、HEX和TAMARA,对222份粪便DNA样品进行个体识别。 3.3 岩羊个体识别
所有的222份岩羊粪便DNA样品在9对微卫星位点上都能稳定扩增。通过对扩增获得的基因型反复比对,结果显示,这222份岩羊样品来自于183个完全不同基因型的个体。
用Gimlet 1.3.3软件对这9对微卫星位点的联合PI值计算后发现,随着微卫星位点的增加,PI值降低,当位点数目达到9时,有亲缘关系的个体概率为0,双胞胎的概率也从一个位点的45%几乎降低为0(图 2),即9个位点识别个体的几率为99.99%。
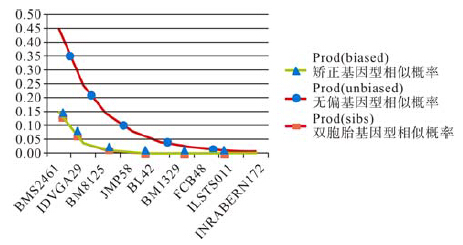
|
| 图 2 联合9个微卫星位点的PI值分布 Fig. 2 Distribution of the PI value after adding different microsatellite loci |
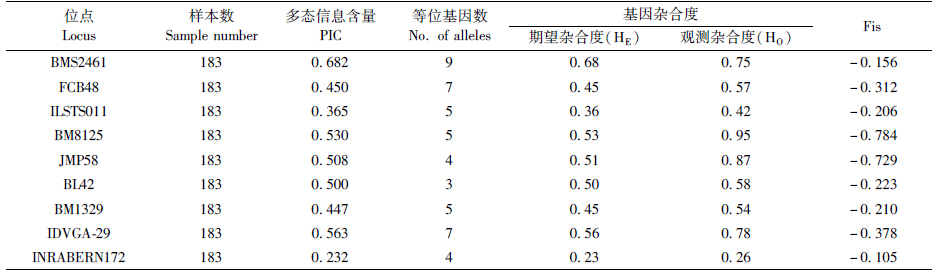
根据微卫星基因座位上的等位基因频率,计算了从岩羊粪便DNA中筛选到的9个微卫星基因座位在样品中的基因杂合度、多态信息含量、等位基因数和个体识别能力(表 2)。统计结果表明,在9个微卫星位点中,总共检测到了49个等位基因。等位基因数最少的为3个(BL42),最多的为9个(BMS2461)。各位点基因杂合度介于0.26~0.95之间,其中最大的是BM8125,最小的是INRABERN172,平均杂合度为0.48;各位点多态信息含量介于0.23~0.68之间,其中最大的是BMS2461,最小的是INRABERN172,平均多态性为0.48。
将所有采样点的岩羊个体作为一个种群用Fisher的方法检测到9个位点中除INRABERN172外都显著偏离Hardy-Weinberg平衡,P<0.001。而且,根据Hardy-Weinberg平衡假设得出的期望杂合度在所有位点都要比观测杂合度低。9个位点的Fis值均为负值。
|
1991年Moore发现了哺乳动物基因组中的保守性微卫星位点(Moore & Sergeant,1991),从而使得微卫星可以跨物种使用。我们可以利用基因库或其他文献中已有的同种或相近物种的微卫星位点来寻找相应的微卫星位点,并根据该位点来设计相应引物到目标DNA中进行扩增(洪艳云,2004)。目前国内外尚没有微卫星选择的统一标准,一般取决于等位基因数目的多少。Barker(1994)在全球家畜品种间遗传距离测定草案的建议中提出微卫星选择的标准是:每个微卫星位点的等位基因数应不少于4个,以便使每个位点的有效等位基因数大于2,从而减少遗传距离估计的标准误差。在本研究中,考虑材料的特殊性,以及是进行物种内的研究,我们选择的微卫星是等位基因数多于3个的位点。
本研究从岩羊近缘物种的36个微卫星位点中筛选到9个(BMS2461、FCB48、ILSTS011、BM1329、IDVGA-29、BL42、BM8125、JM58、INRABERN172)在岩羊DNA基因组中显示多态性的位点,9个微卫星位点在岩羊野生种群中基因杂合度介于0.26~0.95之间,平均杂合度为0.48;除INRABERN172多态性稍低外(PIC值<3),其余PIC值均属中高多态性(PIC值>3或5)。这表明9个位点多态信息含量丰富,可以应用于岩羊遗传多样性与保护研究。
由于2个不同个体的基因型完全一致的概率为10-12,因此法医上把非同卵双生的2个样品的基因型完全一致认定为同一个体或同卵双生(姜先华,1990)。而岩羊在自然条件下多为单胎,所以依据法医标准同时结合岩羊的繁殖特性把基因型完全相同的个体判定为同一个体。在用Gimlet软件对这9个微卫星位点进行联合PI值计算时发现,这9个位点对个体的识别率接近100%,即我们研究中所选的微卫星位点个数在理论上足以进行个体识别。 4.2 遗传多样性
遗传多样性是指一个种群或物种遗传变异的程度,包括等位基因多样性和杂合度两方面。等位基因多样性关系到种群的长期适应,而杂合度则对当前的适应起重要作用(Allendorf,1986)。本研究中9个位点偏离Hardy-Weinberg平衡,观测杂合度高于期望杂合度,存在杂合子过剩的情况。在濒危物种种群遗传学研究中,经常会遇到不符合Hardy-Weinberg平衡的现象(Ardren et al.,1999),其原因可能是多方面的。本研究中观测杂合度高于期望杂合度,可能是因为贺兰山岩羊是生活在特殊环境下的一个边缘种群,边缘种群在适应性进化过程中可能会导致不平衡选择,一些适应值低的位点,其纯合子容易被淘汰,或者一些位点显示杂合优势等,使得基因组中的某些位点偏离Hardy-Weinberg平衡,表现杂合过剩。另外,一些二碱基微卫星在PCR过程中容易链滑动产生结巴(stutter)带而导致错误分型(Edwards et al.,1991;Taberlet et al.,1996,1999),在对一些低质量或低浓度的DNA模板扩增时,这种现象会更严重(Morin et al.,2001;Pompanon et al.,2005)。本研究中使用的是粪便DNA,可能在一定程度上增加了某些位点的错误分型。不管怎样,本研究中9个位点在贺兰山岩羊种群中都表现杂合过剩,这是一个有趣的现象,其原因还有待于进一步研究。
| 曹丽荣, 刘振生, 王小明, 等. 2005. 贺兰山保护区冬季岩羊集群特征的初步分析[J]. 兽类学报, 25(2): 200-204. |
| 洪艳云. 2004. SSR在普氏原羚个体识别和分子保护遗传学中的应用[D]. 长沙: 湖南农业大学. |
| 姜先华. 1990. DNA指纹图在法医学鉴定中应用的研究(I)[J]. 中国法医学杂志, 5(1): 11-15. |
| 刘振生, 曹丽荣, 王小明, 等. 2004. 宁夏贺兰山岩羊种群的管理和保护[J]. 野生动物, 25(1): 56. |
| 刘振生, 曹丽荣, 王小明, 等. 2005a. 贺兰山岩羊冬季对卧息地的选择[J]. 兽类学报, 25(1): 1-8. |
| 刘振生, 王小明, 李志刚, 等. 2005b. 贺兰山岩羊冬春季取食生境的比较[J]. 动物学研究, 26(6): 580-589. |
| 刘振生, 王小明, 李志刚, 等. 2008. 贺兰山岩羊(Pseudois nayaur)夏季取食和卧息生境选择[J]. 生态学报, 28: 4277-4285. |
| 王香亭. 1990. 宁夏脊椎动物志[M]. 银川: 宁夏人民出版社: 602-603. |
| 王小明, 李明, 唐绍祥, 等. 1998. 春季岩羊种群生态学特征的初步研究[J]. 兽类学报, 18(1): 27-33. |
| Allen G. 1940. The mammals of China and Mongolia(Ⅱ)[J]. New York: The America Museum of Natural History: 1269-1274. |
| Allendorf FW. 1986. Genetic drift and the loss of alleles versus heterozygosity[J]. Zoo Biology, 5: 181-190. |
| Ardren WR, Borer S, Thrower F, et al. 1999. Inheritance of 12 microsatellite loci in Oncorhynchus mykiss[J]. Journal of Heredity, 90: 529-536. |
| Baker KH, Hoelzel AR. 2013. Evolution of population genetic structure of the British roe deer by natural and anthropogenic processes (Capreolus capreolus)[J]. Ecology and Evolution, 3(1): 89-102. |
| Barker JSF. 1994. A global protocol for determining genetic distances among domestic livestock breeds[C]. Proc 5th world Congr Genet Appl Livestock Production Science, 21: 501-508. |
| Bellemain E, Swenson JE, Tallmon O, et al. 2005. Estimating population size of elusive animals with DNA from hunter-collected feces: four methods for brown bears[J]. Conservation Biology, 19(1): 150-161. |
| Dario B, Allan FM, Jacob G, et al. 2006. Development of a linkage map and mapping of phenotypic polymorphisms in a free-living population of soay sheep (Ovis aries)[J]. Genetics, 173(3): 1521-1537. |
| da Silva Ec, McManus CM, de Paiva Guimarães MP, et al. 2014. Validation of a microsatellite panel for parentage testing of locally adapted and commercial goats in Brazil[J]. Genetics and Molecular Biology, 37(1): 54-60. |
| Edwards A, Civitello A, Hammond HA, et al. 1991. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats[J]. American Journal of Human Genetics, 49: 746-756. |
| Guérin G, Nocart M, Kemp SJ. 1994. Fifteen new synteny assignments of microsatellites to the bovine genome[J]. Animal genetics, 25(3):179-181. |
| Holly BE, Bruce RH, Jay AW, et al. 2010. Molecular genealogy tools for white-tailed deer with chronic wasting disease[J]. Canadian Journal of Veterinary Research, 74(2): 153-156. |
| Kappes SM, Keele JW, Stone RT, et al. 1997. Second-generation linkage map of the bovine genome[J]. Genome Research, 7(3):235-249. |
| Moore SS, Sergeant LL. 1991. The conservation of dinulceotide microsacellites among mammalian genomics allows use of heterologous PCR primer pairs in closely related species[J]. Genomics, 10: 654-660. |
| Morin PA, Chambers KE, Boesch C, et al. 2001. Quantitative polymerase chain reaction analysis of DNA from noninvasine samples for accurate microsatellite genotyping of wild chimpanzees (Pan troglodytes verus)[J]. Molecular Ecology, 10: 1835-1844. |
| Niu LL, Li HB, Ma YH, et al. 2012. Genetic variability and individual assignment of Chinese indigenous sheep populations (Ovis aries) using microsatellites[J]. Animal Genetics, 43(1):108-111. |
| Park SDE. 2001. Trypanotolerance in west African cattle and the population genetic effects of selection[D]. Dublin: University of Dublin. |
| Pompanon F, Bonin A, Bellemain E, et al. 2005. Genotyping errors: causes, consequences and solutions[J]. Nature Reviews Genetics, 6: 847-859. |
| Taberlet P, Griffin S, Goossens B, et al. 1996. Reliable genotyping of samples with very low DNA quantities using PCR[J]. Nucleic Acids Research, 24: 3189-3194. |
| Taberlet P, Waits LP, Luikart G. 1999. Noninvasive genetic sampling: look before you leap[J]. Trends in Ecology & Evolution, 14: 323-327. |
| Valière N. 2002. GIMLET: a computer program for analysing genetic individual identification data[J]. Molecular Ecology Notes, 2: 377-379. |
| Zhou H, Li D, Zhang Y, et al. 2007. Genetic diversity of microsatellite DNA loci of Tibetan antelope (Chiru, Pantholops hodgsonii) in Hoh Xil National Nature Reserve, Qinghai, China[J]. Journal of Genetics and Genomics, 34(7):600-607. |
 2015, Vol. 34(2)
2015, Vol. 34(2)




