扩展功能
文章信息
- 马炳存, 程建国, 罗燕, 罗茜, 周鑫, 高雪华, 王朋
- MA Bingcun, CHENG Jianguo, LUO Yan, LUO Xi, ZHOU Xin, GAO Xuehua, WANG Peng
- 林麝肠道乳酸菌及肠杆菌多态性分析及耐药性研究
- Diversity Analysis and Antimicrobial Susceptibility Test of Lactic Acid Bacteria and Enterobacteria that Isolated from Forest Musk Deer Intestine
- 四川动物, 2015, 34(1): 29-40
- Sichuan Journal of Zoology, 2015, 34(1): 29-40
- 10.3969/j.issn.1000-7083.2015.01.005
-
文章历史
- 收稿日期:2014-06-24
- 接受日期:2014-09-25
2. 四川养麝研究所, 四川都江堰 611830;
3. 四川农业大学动物医学院, 四川雅安 625000
2. Sichuan Institute of Musk Deer Breeding, Dujiangyan, Sichuan Province 611830, China;
3. College of Veterinary Medicine, Ya'an Campus, Sichuan Agricultural University, Ya'an, Sichuan Province 625000, China
林麝Moschus berezovskii属偶蹄目Artiodactyla麝科Moschidae麝属Moschus,是珍贵的野生药用资源动物,被列为国家Ⅰ级重点保护野生动物,并且被列入世界自然保护联盟濒危物种红色名录和国际贸易公约濒危物种附录Ⅱ中(Zhang,1998)。由于生态环境的破坏及过度捕杀,野麝资源濒临灭绝,麝香资源濒于枯竭(Yang et al.,2003),为了麝资源的保护及可持续利用,人工养麝已被列入《中国21世纪议程》优先项目计划,也列入了《中国中药现代化产业(四川)基地》的首批项目和西部开发项目。1958年以来,以四川养麝研究机构为代表,对麝的驯化、行为学、环境生态、饲养管理与繁殖、活体取香、麝香成分及药理等相关内容进行了较为详细的研究并取得了较大的成功,但对麝的疾病防治研究虽然从未间断却未获得突破。马炳存等: 林麝肠道乳酸菌及肠杆菌多态性分析及耐药性研究
国内对麝类的研究主要集中在野生麝类资源的保护、麝香的开发及应用方面。对麝的人工养殖技术、疾病防治等方面的研究较少。在实际生产中麝的肺化脓性疾病和肠炎性疾病一直是人工规模化养殖的技术瓶颈,严重限制了人工养麝的快速发展。因此加强对麝疾病防治方面的研究,从根本上解决困扰人工规模化养麝的疾病问题,应当是目前林麝人工养殖研究的主要方向。几十年来,林麝肠炎疾病导致幼麝死亡率居高不下。一旦林麝发生肠炎,临床上通常首先使用大剂量的广谱抗生素进行治疗,然而抗生素的过量使用又会杀死林麝肠道中的大量正常菌群,导致林麝消化系统功能紊乱。动物胃肠道中肠道菌群和肠道疾病的关系十分复杂(Mhida et al.,2004;Ojetti et al.,2009),在肠道疾病中有害和有益微生物菌群的种类和数量起重要作用,早在1980年就有临床证据(Balsari et al.,1982)表明肠道疾病患者粪便中有益微生物数量减少。因此,了解林麝肠道微生物群落组成,调查其肠道微生物群落结构及耐药情况,对指导临床用药、防止林麝养殖中肠道疾病的发生和发展具有巨大的现实意义。本研究首次分析了林麝肠道中可培养细菌的基因型及表型并且对其耐药性进行了测定,对林麝人工养殖过程中肠炎疾病的防控具有现实的指导意义。1 材料与方法 1.1 主要试剂
Man-Ragosa-Sharpe(MRS)培养基、营养琼脂(NA)培养基、水解酪蛋白(MH)培养基以及羊血培养基均购自杭州微生物试剂有限公司。2×Taq PCR MasterMix Kit购自天根生化试剂(北京)有限公司。溶菌酶购自天根生化科技(北京)有限公司。PCR反应引物(27F:5-AGAGTTTGATCCTGGCTCAG-3和1942R:5-TACGGYTACCTTGTTACGACTT-3)的合成以及16S rDNA的测序均由生工生物工程(上海)有限公司完成。脉冲场凝胶电泳(PFGE)试剂盒购自美国Bio-Rad生物公司。 1.2 样品采集及菌株分离
从四川养麝研究所养殖场中选取10头未用过抗生素的健康成年林麝,采集其新鲜粪便样品于4 ℃冻存盒中,立即送往实验室进行细菌的分离培养。样品经梯度稀释后分别涂布于含有1%CaCO3用于分离乳酸菌的MRS平板和分离肠杆菌的NA平板,每个梯度做3个重复。MRS平板37 ℃厌氧培养3 d,NA平板37 ℃有氧培养24 h后分别挑取单菌落划线纯化培养2次。将纯化的典型菌落分别进行革兰氏染色、氧化酶反应和形态学观察。将革兰氏阳性、氧化酶反应阳性、有CaCO3降解圈的杆菌或球菌初步判定为乳酸菌,革兰氏阴性、氧化酶反应阴性的杆菌判定为肠杆菌,保存菌种以备下一步实验之用。 1.3 菌种的生理生化鉴定和分子生物学鉴定1.3.1 形态学和生理生化鉴定
菌种培养16 h后观察其菌落形态,对菌种进行初步分类。革兰氏染色后显微观察细菌形态及染色性质。挑取适量培养物于载玻片上,加一滴3%的过氧化氢溶液,测定其氧化酶反应。
菌种的生理生化鉴定采用细菌微量生化反应管,按照文献推荐项目进行(Buchanan & Gibbons,1974),根据细菌微量生化反应管的说明书进行操作。 1.3.2 分子生物学鉴定
菌种的16S rDNA扩增采用菌落PCR(Colony PCR)技术(Sheu et al.,2000)完成。以16S rDNA通用引物27F和1942R在2×Taq PCR MasterMix Kit体系中扩增。PCR反应体系为:2×Taq PCR MasterMix 25 μL,20 mmol·L-1 27F引物1 μL,20 mmol·L-1 1942R引物1 μL,少许菌种菌落。PCR反应程序为:94 ℃ 5 min,94 ℃ 1 min,50 ℃ 1 min,72 ℃1.5 min,72 ℃ 10 min,4 ℃保温,30个循环。PCR产物的预期大小为1500 bp。PCR产物用含有0.005%Goldview的琼脂糖凝胶电泳检测。PCR产物由生工生物工程(上海)有限公司测序。菌种16S rDNA序列经BLAST程序比对后,选取部分同源序列构建进化树。利用MEGA 4.0软件中的邻接法,以1000次循环进行稳定性检测,构建进化树(Saitou & Nei,1987)。同时,采用最大简约算法(Felsenstein,1985)和最大似然算法(Kluge & Farris,1969)构建进化树,以确定邻接法构建的进化树的可靠性。1.4 脉冲场凝胶电泳基因型分析
本研究采用PFGE技术分析各菌株的基因型,肠杆菌和乳酸菌基因组DNA分别用限制性内切酶XbaⅠ和NotⅠ进行酶切。PFGE的操作参考Ribot等(2006)的报道。基因组DNA分别经50 U的XbaⅠ和NotⅠ 37 ℃酶切2 h后,取出消化好的胶块,切下2 mm宽度的一小段胶条贴至PFGE模具的梳子上,待其自然干燥5 min固定后倒胶。将含有胶条的琼脂糖凝胶放入已冷却至14 ℃的CHEF DRII电泳系统中(Bio-Rad,Laboratories)进行PFGE。具体程序为:起始转换时间(initial switch time)=1 s,终末转换时间(final switch time)=15 s,电泳角度(included angle)=120°,电压为6 V·cm-1,14 ℃运行20 h。
使用Quantity One 4.6.2软件对PFGE结果进行分型分析,采用平均连接聚类算法(UPGMA)来计算PFGE模型,并构建进化树。1.5 药敏实验
所有待检菌株的药敏实验按照“临床与实验室标准研究所”的步骤完成(CLSI,2009)。用生理盐水稀释待检菌株的过夜培养物至108 CFU·cm-1,均匀涂布于MH(检测肠杆菌)或MRS(检测乳酸菌)平板上,在每个平板的适当位置放置抗生素药敏纸片。37 ℃培养24 h后测定各药敏纸片的抑菌圈大小。同时以大肠杆菌(ATCC25922)作为质控菌株。
参考Charteris等(1998)的报道,选择37种抗生素药敏纸片来测定乳杆菌、明串球菌和食窦魏斯氏菌的耐药性。肠杆菌和肠球菌的药敏纸片根据CLSI的推荐选择。2 结果 2.1 形态学及生理生化鉴定
本研究共分离到38株细菌,经革兰氏染色鉴定和氧化酶实验,有22株为革兰氏阴性、氧化酶阳性、杆状,初步判定为肠杆菌;16株为革兰氏阳性、氧化酶阴性、球状,初步判定为乳酸菌。
经生理生化鉴定,22株肠杆菌中(表 1),大肠杆菌Escherichia coli 11株(编号为NA4-2、YAN3-3、NA2-3、NA5-10、NA1-2、NA2-6、NA3-4、NA1-4、NA4-8、NA2-7和NA1-8);阴沟肠杆菌Enterobacter cloacae 3株(编号为NA2-2、NA5-9和NA5-5);河生肠杆菌E. amnigenus 2株(编号为NA1-10和NA4-3);肺炎克雷伯氏菌Klebsiella pneumoniae 2株(编号为NA1-9和NA1-13);泰勒肠杆菌Enterobacter taylorae 1株(编号为NA5-2);蜂房哈夫尼菌Hafnia alvei 1株(编号为NA4-1);另有2株(编号为NA3-3和NA5-6)无明确的生理生化鉴定结果。16株乳酸菌中(部分结果见表 2),蒙氏肠球菌Enterococcus mundtii 5株(编号为YAN2-2、YAN1-4、MRS3-6、MRS3-13和YAN3-4);耐久肠球菌E. durans 4株(编号为MRS4-5、MRS3-2、MRS4-10和MRS3-3);明串球菌Leuconostoc fallax 3株(编号为MRS2-2、MRS2-5和MRS3-11);植物乳杆菌Lactobacillus plantarum 1株(编号为M.D.L1);嗜酸乳杆菌L. acidipiscis 1株(编号为M.D.L2);屎肠球菌E. faecium 1株(编号为M.D.E.S8)和食窦魏斯氏菌Weissella cibaria 1株(编号为YAN2-10)。
| 鉴定项目 Characteristics | 菌株编号Selected isolates | |||||||||||||||||||||
| NA 4 2 | NA
22 | NA 5 9 | NA
55 | NA 1 9 | NA
113 | NA 3 3 | NA
52 | NA 5 6 | NA
110 | NA 4 1 | NA
43 | YAN 3 3 | NA
23 | NA 5 10 | NA
12 | NA 2 6 | NA
34 | NA 1 4 | NA
48 | NA 2 7 | NA
18 | |
| 革兰氏染色 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 细菌形态 | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R | R |
| 接触酶 | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + |
| MR | + | - | - | - | - | - | - | - | - | - | - | - | + | + | + | + | + | + | + | + | + | + |
| 吲哚 | + | - | - | - | - | - | - | - | - | - | - | - | + | + | + | + | + | + | + | + | + | + |
| H2S | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 产气 | - | + | + | + | + | + | - | + | - | + | + | + | + | - | - | + | - | + | - | + | + | + |
| 苯丙氨酸 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 葡萄糖酸盐 | - | + | + | + | + | + | + | - | - | + | + | + | - | - | - | - | - | - | - | - | - | - |
| 柠檬酸盐 | - | + | + | + | + | + | + | + | + | + | + | + | - | - | - | - | - | - | - | - | - | - |
| 尿素 | - | - | - | - | + | + | - | - | + | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 运动性 | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 赖氨酸 | + | - | - | - | + | + | - | - | - | - | + | - | + | + | + | + | + | + | + | + | + | + |
| 鸟氨酸 | + | + | + | + | - | - | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | W |
| 棉籽糖 | - | + | + | + | + | + | - | - | W | + | - | + | - | - | - | - | - | - | - | - | - | - |
| 山梨醇 | - | W | W | W | - | - | + | W | W | W | - | - | - | - | - | W | - | - | - | W | W | + |
| 侧金盏花醇 | - | + | + | + | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - | - |
| 木糖 | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + | + |
| 注: +. 阳性,-. 阴性,R. 杆状,W. 弱反应,UI. 未鉴定到具体种。 Note: +. Positive,-. Negative,R. Rod,W. Weak reaction,UI. Unidentified isolates. | ||||||||||||||||||||||
电泳结果表明16S rDNA的PCR产物片段大小为1500 bp(图 1)。所有菌株的16S rDNA序列经BLAST程序比对后,找到与其同源的标准菌株序列。使用MEGA4.0软件对菌株的16S rDNA序列和标准株序列进行多序列比对并构建系统发育进化树(图 2)。
本研究中所有测序的16S rDNA序列均提交至GenBank数据库,收录号分别为JF690863~JF690896、M.D.L1(HM753266)、M.D.L2(HM753265)。4株菌(NA2-2、NA5-9、NA5-5和NA4-1)生理生化和分子生物学的鉴定结果不一致,综合分析生理生化和分子生物学的鉴定结果,最终将NA2-2、NA5-9和NA5-5鉴定为阴沟肠杆菌,NA4-1鉴定为河生肠杆菌。另有3株菌(NA5-2、NA3-3和NA5-6)由于生理生化鉴定结果和分子生物学鉴定结果差异太大,无法将其鉴定到具体的种。
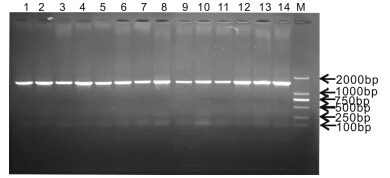 |
| 图 1 部分菌株16S rDNA PCR产物电泳检测结果 Fig. 1 Electrophoretic analysis of partial colony PCR productsM. DL2000 DNA标志物,1~14为部分分离菌株的16S rDNA PCR产物。 M. D2000 DNA marker,1~14. partial colony of PCR products. |
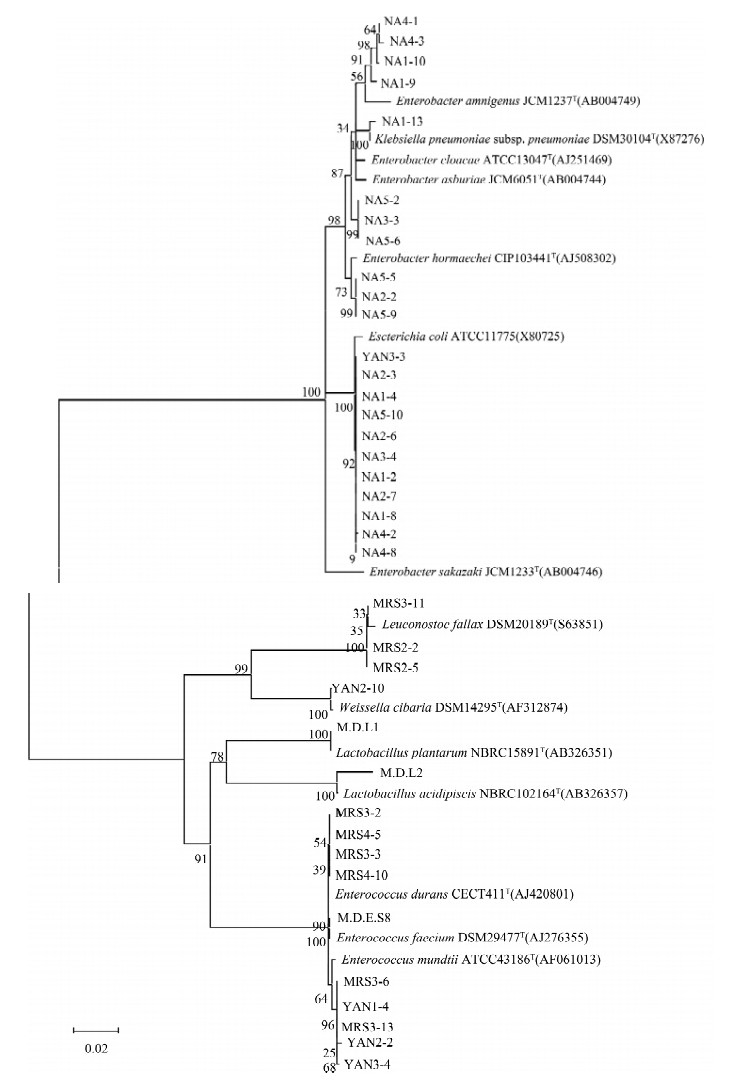 |
| 图 2 分离菌株的16S rDNA系统发育进化树 Fig. 2 The 16S rDNA phylogenetic tree of the isolated strains |
经XbaⅠ和NotⅠ酶切后,38株细菌的PFGE分型结果如图 3所示。PFGE进化树分析结果表明,22株肠杆菌共分为7个(A~G)基因型。除基因型A仅有菌株NA4-2一株菌外,其余各基因型都有多个菌株。大肠杆菌经限制性内切酶Xba Ⅰ酶切后得到了3种基因型(A、F、G),说明大肠杆菌各菌株之间基因差异较大。16株乳酸菌共分为9种基因型,其中耐久肠球菌的菌株MRS4-10、MRS3-3的基因型与其他2株耐久肠球菌MRS3-2、MRS4-5的基因型明显不同。
 |
| 图 3 38株细菌经XbaⅠ酶切(A)及Not Ⅰ酶切(B)的PFGE进化树 Fig. 3 The PFGE patterns of enterobateria(A) and LAB(B)isolates after digestion with XbaⅠ and NotⅠ,respectively |
| 鉴定项目 Item | 菌株编号Selected isolates | |||||||
| MRS3-2 | MRS4-10 | MRS3-3 | MRS3-11 | YAN2-10 | MRS3-6 | MRS3-13 | YAN3-4 | |
| 革兰氏染色 | + | + | + | + | + | + | + | + |
| 细胞形态 | C | C | C | C | C | C | C | C |
| 氧化酶活性 | - | - | - | - | - | - | - | - |
| MR | - | - | - | + | + | - | - | - |
| VP | NT | NT | NT | - | - | NT | NT | NT |
| 吲哚 | NT | NT | NT | - | - | NT | NT | NT |
| 产H2S | - | - | - | - | - | - | - | - |
| 产CO2 | - | - | - | - | - | - | - | - |
| 木糖 | - | - | - | - | + | - | - | - |
| 鼠李糖 | - | - | W | - | W | + | (d)- | (d)- |
| D-核糖 | + | + | + | W | + | + | + | + |
| 甘露糖 | + | + | + | + | W | + | + | + |
| 果糖 | + | + | + | + | + | + | + | + |
| 半乳糖 | + | + | + | (d)- | + | + | + | + |
| 蔗糖 | + | + | + | + | + | (d)- | (d)- | (d)- |
| 麦芽糖 | + | + | - | (d)- | + | - | - | - |
| 纤维二糖 | + | W | + | - | + | + | + | + |
| 乳糖 | + | + | + | - | + | + | + | + |
| 淀粉 | - | - | - | - | - | - | - | - |
| 山梨醇 | - | - | - | - | - | - | - | - |
| 七叶苷 | + | + | + | - | + | + | + | + |
| 苦杏苷 | + | + | + | - | + | + | + | + |
| 棉籽糖 | - | - | + | - | - | + | + | + |
| 阿拉伯糖 | - | - | (d)- | - | - | (d)- | (d)- | (d)- |
| 山梨糖 | - | - | - | - | - | - | - | - |
| 海藻糖 | + | + | + | - | + | + | + | + |
| 明胶 | NT | NT | NT | - | NT | NT | NT | NT |
| 生理生化结果 | E. durans | E. durans | E. durans | L. fallax | W. cibaria | E. mundtii | E. mundtii | E. mundtii |
| 16S鉴定结果 | E. durans | E. durans | E. durans | L. fallax | W. cibaria | E. mundtii | E. mundtii | E. mundtii |
| PFGE结果 | D | H | I | G | B | C | C | C |
| GenBank登录号 | JF690893 | JF690868 | JF690892 | JF690866 | JF690894 | JF690865 | JF690867 | JF690896 |
| 注: +. 阳性,-. 阴性,C. 球状,W. 弱反应,NT. 未测试,d. 与参考文献有差异。 Notes: +. Positive,-. Negative,C. Cocci,W. Weak reaction,NT. Not tested,d. Different from references. | ||||||||
肠杆菌药敏实验的操作和结果解释均参照CLSI的说明进行(表 3-1和表 3-2)。阴沟肠杆菌、肺炎克雷伯氏菌、河生肠杆菌和不能鉴定的3株菌对氨苄西林(耐药率81.8%)、阿莫西林/克拉维酸复合物(耐药率100%)以及第一代头孢类抗生素(头孢唑林和头孢噻吩,耐药率81.8%)有很高的耐药性,对其他测试的抗生素敏感。大肠杆菌药敏结果表明,除了阿莫西林/克拉维酸复合物有45.4%的敏感率外,大肠杆菌对其他所有测试的抗生素敏感。值得注意的是,除了菌株NA2-3和NA4-8对阿莫西林/克拉维酸复合物敏感外,几乎所有的肠杆菌都对其有很高的耐药性。
| 抗生素分组
Groups | 抗生素名称
Antimicrobial | 浓度/μg/片 | 抑菌圈直径Antibacterial zone diameters/mm | ||||||||||
| NA2-2 | NA5-9 | NA5-5 | NA1-9 | NA1-13 | NA3-3 | NA5-2 | NA5-6 | NA1-10 | NA4-1 | NA4-3 | |||
| 青霉素类 | 氨苄西林 | 10 U | R/60 | R/120 | I/140 | R/60 | R/100 | R/70 | R/80 | R/60 | R/60 | R/110 | I/140 |
| 哌拉西林 | 100 | S/290 | S/210 | S/290 | S/240 | S/230 | S/260 | S/270 | S/285 | S/300 | S/280 | S/250 | |
| 复合物 | 阿莫西林/克拉维酸 | 20/10 | R/60 | R/60 | R/60 | R/95 | R/95 | R/60 | R60 | R/60 | R/60 | R/53 | R/60 |
| 氨苄青/舒巴坦 | 10/10 | S/150 | S/180 | S/150 | S/180 | S/180 | R/90 | R/110 | S/160 | S/180 | S/170 | S/160 | |
| 头孢类 | 头孢曲松 | 30 | S/320 | S/280 | S/280 | S/280 | S/260 | S/235 | S/250 | S/280 | S/280 | S/215 | S/250 |
| 头孢唑林 | 30 | R/60 | R/60 | R/60 | S/250 | S/250 | R/60 | R/60 | R/60 | R/60 | R/600 | R/60 | |
| 头孢噻吩 | 30 | R/60 | R/60 | R/60 | S/260 | S/220 | R/60 | R/60 | R/60 | R/60 | R/60 | R/60 | |
| 头孢呋辛 | 30 | S/205 | S/240 | S/220 | S/190 | S/230 | I/150 | S/180 | I/175 | S/210 | S/210 | S/190 | |
| 单环内酯类 | 氨曲南 | 30 | S/340 | S/340 | S/340 | S/280 | S/260 | S/280 | S/260 | S/300 | S/320 | S/320 | S/290 |
| 氨基糖苷类 | 庆大霉素 | 10 | S/200 | S/210 | S/215 | S/210 | S/220 | S/205 | S/210 | S/215 | S/220 | S/220 | S/220 |
| 链霉素 | 10 | S/200 | S/210 | S/190 | S/190 | S/200 | S/210 | S/200 | S/190 | S/160 | S/175 | I/115 | |
| 四环素类 | 四环素 | 30 | S/190 | S/160 | S/180 | S/190 | S/220 | I/140 | S/150 | R/85 | S/170 | S/190 | S/190 |
| 氯霉素类 | 氯霉素 | 30 | S/260 | S/260 | S/280 | S/280 | S/260 | S/270 | S/280 | S/280 | S/240 | S/270 | S/250 |
| 磺胺类 | 萘啶酸 | 30 | S/240 | S/230 | S/250 | S/250 | S/230 | S/240 | S/225 | S/240 | S/260 | S/220 | S/225 |
| 磺胺异噁唑 | 300 | S/260 | S/280 | S/280 | S/230 | S/240 | S/250 | S/250 | S/275 | S/270 | S/280 | S/240 | |
| 诺氟沙星 | 10 | S/320 | S/320 | S/340 | S/290 | S/270 | S/330 | S/320 | S/340 | S/290 | S/280 | S/300 | |
| 环丙沙星 | 5 | S/340 | S/340 | S/310 | S/320 | S/310 | S/340 | S/340 | S/360 | S/340 | S/320 | S/280 | |
| 甲氧苄啶/磺胺异噁唑 | 375/125 | S/250 | S/280 | S/290 | S/200 | S/270 | S/250 | S/270 | S/280 | S/270 | S/290 | S/250 | |
| 其他类 | 美罗培南 | 10 | S/290 | S/300 | S/265 | S/320 | S/205 | S/270 | S/280 | S/290 | S/273 | S/270 | S/270 |
| 呋喃妥因 | 300 | S/200 | S/210 | S/190 | S/220 | S/183 | S/200 | S/210 | S/180 | S/213 | S/183 | S/210 | |
| 亚胺培南 | 10 | S/230 | S/300 | S/256 | S/290 | S/190 | S/240 | S/320 | S/230 | S/220 | S/190 | S/195 | |
| 青霉素类 | 氨苄西林 | 10 U | I/150 | I/160 | S/170 | S/185 | I/150 | S/190 | S/170 | S/180 | S/190 | R/130 | I/165 |
| 哌拉西林 | 100 | S/260 | S/310 | S/280 | S/300 | S/220 | S/260 | S/280 | S/260 | S/260 | S/270 | S/260 | |
| 复合物 | 阿莫西林/克拉维酸 | 20/10 | R/120 | S/190 | I/160 | I/150 | R/130 | R/60 | I/173 | S/200 | I/135 | R/130 | I/170 |
| 氨苄青/舒巴坦 | 10/10 | S/215 | S/230 | S/186 | S/240 | S/210 | S/190 | S/190 | S/220 | S/180 | S/190 | S/210 | |
| 头孢类 | 头孢曲松 | 30 | S/270 | S/320 | S/320 | S/300 | S/310 | S/280 | S/3000 | S/310 | S/280 | S/300 | S/300 |
| 头孢唑啉 | 30 | S/210 | S/260 | S/240 | S/260 | S/230 | R/100 | S/235 | S/240 | S/220 | I/164 | S/220 | |
| 头孢噻吩 | 30 | I/175 | S/200 | S/193 | S/180 | S/220 | R/60 | S/240 | S/220 | S/196 | S/200 | S/190 | |
| 头孢呋辛 | 30 | S/210 | S/260 | S/230 | S/260 | S/220 | S/200 | S/230 | S/230 | S/210 | S/220 | S/220 | |
| 单环内酯类 | 氨曲南 | 30 | S/260 | S/340 | S/320 | S/280 | S/300 | S/300 | S/300 | S/320 | S/280 | S/260 | S/380 |
| 氨基糖苷类 | 庆大霉素 | 10 | S/200 | S/190 | S/160 | S/160 | S/190 | S/160 | S/1753 | S/220 | S/200 | S/180 | S/190 |
| 链霉素 | 10 | R/110 | I/120 | I/140 | I/130 | S/160 | S/150 | I/120 | S/150 | I/120 | I/130 | I/138 | |
| 四环素类 | 四环素 | 30 | S/160 | S/195 | S/160 | S/180 | R/80 | S/153 | S/190 | S/180 | R/60 | S/150 | S/170 |
| 氯霉素类 | 氯霉素 | 30 | S/220 | S/250 | S/250 | S/250 | S/250 | S/250 | S/240 | S/220 | S/250 | I/165 | S/220 |
| 磺胺类 | 萘啶酸 | 30 | S/220 | S/255 | S/260 | S/250 | S/260 | S/210 | S/220 | S/220 | S/250 | S/230 | S/220 |
| 磺胺异噁唑 | 300 | S/240 | S/290 | S/280 | S/280 | S/290 | S/240 | S/280 | S/260 | S/260 | S/210 | S/250 | |
| 诺氟沙星 | 10 | S/320 | S/390 | S/320 | S/300 | S/320 | S/300 | S/320 | S/340 | S/380 | S/360 | S/320 | |
| 环丙沙星 | 5 | S/320 | S/360 | S/360 | S/360 | S/380 | S/300 | S/360 | S/330 | S/310 | S/320 | S/380 | |
| 甲氧苄啶/磺胺异噁唑 | 375/125 | S/280 | S/300 | S/280 | S/280 | S/315 | S/280 | S/290 | S/290 | S/280 | S/210 | S/290 | |
| 其他类 | 美罗培南 | 10 | S/260 | S/270 | S/280 | S/280 | S/245 | S/320 | S/290 | S/273 | S/310 | S/280 | S/300 |
| 呋喃妥因 | 300 | S/170 | S/210 | S/190 | S/200 | S/19.0 | R/135 | S/200 | S/213 | S/190 | S/180 | S/210 | |
| 亚胺培南 | 10 | S/200 | S/195 | S/225 | S/250 | S/225 | S/300 | S/230 | S/220 | R/80 | S/260 | S/320 | |
| 注:R 耐药,I中介,S 敏感; 下表同。 Notes: Susceptibility expressed as R(resistant),I(intermediate) and S(susceptible); the same below. | |||||||||||||
目前对除肠球菌外的乳酸菌的药敏实验操作及结果的解释并没有统一的标准,本研究参考Charteris(1998)的结果来解释乳酸菌的药敏实验结果(部分结果见表 4)。除MRS3-6对盘尼西林和氨苄西林耐药外,肠球菌对所有测试的抗生素均敏感。乳杆菌M.D.L1和M.D.L2对除链霉素外的所有氨基糖苷类抗生素和磺胺类抗生素耐药,除此之外,M.D.L1和M.D.L2还对氨曲南、万古霉素、甲硝唑和多粘菌素B耐药,对四环素、氯霉素、红霉素、林可霉素、呋喃妥因、亚安培能和美罗培南敏感。在头孢类抗生素中M.D.L1和M.D.L2对头孢克肟中介,M.D.L2对头孢西丁耐药,而M.D.L1则对其中介。除头孢克肟和头孢西丁之外,M.D.L1和M.D.L2对其他头孢类抗生素均敏感。另外,M.D.L1和M.D.L2除对盘尼西林中介外,对所有测试的青霉素类抗生素均敏感。明串球菌(MRS2-2、MRS2-5、MRS3-11)青霉素类、氨基糖苷类和磺胺类抗生素表现出较强的耐药性,对头孢类抗生素则十分敏感。对氨曲南、万古霉素、甲硝唑和多粘菌素B有很强的耐药性,而对呋喃妥因、亚安培能和美罗培南则较为敏感。食窦魏斯氏菌(YAN2-10)的药敏结果与明串球菌较为相似,但对呋喃妥因表现出很强的耐药性。
| 抗生素分组 Groups | 抗生素名称 Antimicrobial | 浓度/ μg/片 | 菌株编号Selected isolates | ||||||||
| MRS3-2 | MRS4-10 | MRS3-3 | MRS3-11 | YAN2-10 | MRS3-6 | MRS3-13 | YAN3-4 | ||||
| 青霉素类 | 盘尼西林 | 10 U | 24.3/S | 24.0/S | 23.0/S | 12.0/R | 17.0/R | 9.0/R | 25.3/S | 27.0/S | |
| 氨苄西林 | 10 | 34.0/S | 30.0/S | 33.0/S | 16.7/S | 20.0/S | 18.0/R | 32.0/S | 33.0/S | ||
| 阿莫西林 | 10 | — | — | — | 17.5/R | 17.0/R | — | — | — | ||
| 哌拉西林 | 100 | — | — | — | 12.0/R | 16.5/R | — | — | — | ||
| 头孢类 | 头孢噻吩 | 30 | — | — | — | 31.0/S | 23.0/S | — | — | — | |
| 头孢拉定 | 30 | — | — | — | 26.0/S | 16.5/I | — | — | — | ||
| 头孢唑林 | 30 | — | — | — | 35.0/S | 32.0/S | — | — | — | ||
| 头孢呋辛 | 30 | — | — | — | 32.0/S | 28.0/S | — | — | — | ||
| 头孢克洛 | 30 | — | — | — | 25.0/S | 21.5/S | — | — | — | ||
| 头孢西丁 | 30 | — | — | — | 31.0/S | 28.0/S | — | — | — | ||
| 头孢克肟 | 5 | — | — | — | 18.5/S | 15.0/I | — | — | — | ||
| 头孢唑肟 | 30 | — | — | — | 29.6/S | 27.0/S | — | — | — | ||
| 头孢噻肟 | 30 | — | — | — | 27.0/S | 24.0/S | — | — | — | ||
| 头孢他啶 | 30 | — | — | — | 19.5/S | 19.0/S | — | — | — | ||
| 头孢哌酮 | 75 | — | — | — | 25.0/S | 30.0/S | — | — | — | ||
| 头孢曲松 | 30 | — | — | — | 31.0/S | 24.0/S | — | — | — | ||
| 单环内酯类 | 氨曲南 | 30 | — | — | — | 6.0/R | 6.0/R | — | — | — | |
| 多肽类 | 万古霉素 | 30 | 22.0/S | 21.3/S | 21.0/S | 16.5/I | 6.0/R | 19.2/S | 20.0/S | 19.0/S | |
| 氨基糖苷类 | 阿米卡星 | 30 | — | — | — | 20.0/S | 6.0/R | — | — | — | |
| 庆大霉素 | 10 | — | — | — | 6.0/R | 18.3/S | — | — | — | ||
| 卡那霉素 | 30 | — | — | — | 6.0/R | 9.0/R | — | — | — | ||
| 萘替卡星 | 30 | — | — | — | 20.0/S | 20.0/S | — | — | — | ||
| 链霉素 | 10 | — | — | — | 8.5/R | 6.0/R | — | — | — | ||
| 四环素类 | 四环素 | 30 | 18.0/S | 19.5/S | 21.0/S | 14.0/R | 31.0/S | 28.0/S | 29.5/S | 32.0/S | |
| 氯霉素类 | 氯霉素 | 30 | 25.5/S | 24.5/S | 27.0/S | 24.5/S | 28.5/S | 22.0/S | 30.0/S | 30.0/S | |
| 大环内酯类 | 红霉素 | 15 | 27.5/S | 26.0/S | 25.0/S | 6.0/R | 26.0/S | 21.0/I | 21.0/S | 22.0/S | |
| 林克霉素类 | 克林霉素 | 2 | — | — | — | 19.5/S | 17.0/S | — | — | — | |
| 磺胺类 | 磺胺异噁唑 | 300 | — | — | — | 6.0/R | 6.0/R | — | — | — | |
| 甲氧苄啶 | 5 | — | — | — | 6.0/R | 6.0/R | — | — | — | ||
| 诺氟沙星 | 10 | 19.0/S | 20.0/S | 20.0/S | 15.5/R | 6.0/R | 18.0/S | 19.0/S | 22.0/S | ||
| 环丙沙星 | 5 | 21.5/S | 22.0/S | 22.0/S | 20.0/S | 11.5/R | 17.0/S | 20.5/S | 22.0/S | ||
| 萘啶酸 | 30 | — | — | — | 6.0/R | 6.0/R | — | — | — | ||
| 硝基咪唑类 | 甲硝唑 | 5 | — | — | — | 6.0/R | 6.0/R | — | — | — | |
| 多粘菌素类 | 多粘菌素B | 300 | — | — | — | 6.0/R | 6.0/R | — | — | — | |
| 其他抗生素 | 呋喃妥因 | 300 | 24.0/S | 21.6/S | 23.6/S | 23.0/S | 6.0/R | 20.0/S | 23.4/S | 24.0/S | |
| 亚胺培能 | 10 | — | — | — | 31.0/S | 18.0/S | — | — | — | ||
| 美罗培南 | 10 | — | — | — | 32.0/S | 21.6/S | — | — | — | ||
| 注: R. 耐药,I. 中介,S. 敏感,—.未测试。 Note: Susceptibility expressed as R(resistant),I(intermediate),S(susceptible) and —(not test). | |||||||||||
由于肠杆菌的广泛分布而且宿主特异性等导致的表型多样性早有报道(Chantal et al.,2011)。目前对林麝肠道菌群的多样性所知甚少。本文发现肠杆菌和肠球菌是林麝肠道中的优势菌群。但是由于在本文之前对林麝肠道菌群的报道较少,加之菌种鉴定工作的复杂,使得本文中有3株肠杆菌属的细菌不能鉴定到种的水平。生理生化鉴定和表型鉴定是实验室传统的菌种鉴定手段,但是通常这些鉴定方法费时费力,而且很多时候并不能获得准确的鉴定结果。目前细菌的分子生物学鉴定方法如16S rDNA序列分析、PFGE基因分型分析(Carol et al.,2006;Agapi et al.,2011;Liu et al.,2011)等以其快速、准确而越来越受到重视。本研究中,经16S rDNA序列分析和PFGE基因分型分析发现林麝肠道中肠杆菌的表型呈多样性分布,菌株NA4-1以及NA1-10、NA4-3被鉴定为河生肠杆菌。然而,NA4-1的表型与NA1-10和NA4-3却大不相同,NA4-1的表型与蜂房哈夫尼菌更为相近。大肠杆菌对山梨醇的发酵结果也不尽相同,菌株NA1-8能够发酵山梨醇,而NA1-2、NA4-8、NA2-7仅能弱发酵山梨醇,其他的大肠杆菌则不能发酵山梨醇。
PFGE技术以其准确性高、重复性好、条带清晰的优势,被作为分子分型技术的金标准而被广泛使用(Blaiotta et al.,2001;Gatti et al.,2003)。在PFGE中,限制性内切酶的选择对菌株分型结果有着重要的影响,本研究中选择限制性内切酶XbaⅠ和NotⅠ分别对肠杆菌和乳酸菌基因组进行酶切,结果表明这2种酶切的DNA片段可以很好地将肠杆菌和乳酸菌区分开来。经平均聚类法构建进化树后,各菌株的聚类结果与16S rDNA序列分析结果相一致。与生理生化等表型鉴定技术相比,PFGE技术可以很好地在分子水平上研究菌株基因型多态性,从而比较各菌株之间的差异。本研究中大肠杆菌和耐久肠球菌经过PFGE分析可分别分为3种基因型,而生理生化鉴定结果表明这些菌株的表型差异并不明显。乳酸菌等革兰氏阳性菌株因其细胞壁较厚,且肽聚糖连接十分紧密,导致其基因组不易释放,因此在革兰氏阳性菌株的PFGE实验中,对样品的破壁裂解就显得十分重要。本研究采用胶块包埋菌体之后用溶菌酶裂解细菌细胞壁,后经蛋白酶K消化,从而使乳酸菌基因组暴露以便于后续的酶切反应。许飞利等(2011)采用裂解后包埋的方法同样也获得了清晰的电泳条带。但在菌种较多的实验中包埋后裂解则显得更为经济。本研究中药敏实验结果表明,肠杆菌除对头孢噻吩和头孢唑啉等第一代头孢类抗生素耐药外,对其余所有测试的抗生素都较敏感。因此,当对患有肠炎类疾病的林麝进行抗生素治疗时,所使用的抗生素对林麝肠道的许多正常菌群也都有杀灭作用,这直接破坏了林麝肠道的微生态平衡,不利于林麝肠炎类疾病的治疗。Sekirov等(2008)报道用霍乱弧菌感染普通豚鼠不会发病,但经链霉素预处理过后的豚鼠再感染霍乱弧菌则会引起致命性发病,表明抗生素严重干扰和破坏了肠道中的正常菌群,直接或间接杀灭了肠道中的原籍菌群,增加了肠道病原菌入侵肠道细胞的机会。所以当林麝肠炎类疾病发生时,尤其是发病的初始阶段不建议直接采用大剂量广谱抗生素进行治疗。
本研究中,乳杆菌(M.D.L1和M.D.L2)对抑制细胞壁合成的抗生素中的氨曲南、万古霉素,抑制蛋白质合成的阿米卡星、庆大霉素、卡那霉素、奈替米星,抑制核酸合成的磺胺异噁唑、甲氧苄啶、诺氟沙星、环丙沙星、萘啶酸、甲硝唑,影响细胞膜功能的多粘菌素B表现出很高的抗性。根据以往的报道(Simpson et al.,1988;Charteris et al.,1998),这些耐药基因均为乳酸菌的固有耐药基因。与Swenson等(1990)报道不同的是,明串球菌(MRS2-5)对庆大霉素、链霉素、红霉素有很高的耐药性;对万古霉素中介;对氨苄西林、林可霉素、环丙沙星敏感。Swenson等(1990)报道所有测试的明串球菌对庆大霉素和红霉素敏感;对氨苄西林、林可霉素和环丙沙星较耐药;对万古霉素有极高的耐药性(MIC≥256 μg/mL)。这些结果的不同很可能是由药敏实验中培养基以及培养条件的不同导致,Swenson等药敏实验所使用的培养基是加有5%羊血的MH培养基,在5%的CO2中培养。然而以上这些抗生素的杀菌效果深受培养基pH和培养条件中CO2浓度的影响。肠球菌的药敏情况表现出种特异性,各肠球菌株的耐药性有很大不同。屎肠球菌对万古霉素有很高的抗性,通常被称为万古霉素抗性菌(Steven et al.,1992;Glenn et al.,1995;Schouten et al.,1999),然而本研究中所分离到的林麝肠球菌对万古霉素未表现出耐药性。
药敏分析可以看出林麝肠道微生物对目前临床常用的抗生素较为敏感,因此在使用抗生素治疗林麝肠炎疾病的同时会破坏林麝肠道的正常微生物群落,对林麝肠炎疾病的治疗有较强的副作用。而益生菌作为肠道调节菌群能够很好地调节宿主肠道微生态平衡(He et al.,2012),对肠炎疾病的防控有积极意义。因此我们认为,利用林麝肠道中的天然乳酸菌经过合理配伍,开发适应林麝肠道环境的复合微生态制剂是预防和治疗林麝肠炎疾病的一个重要方向。
| 许飞利, 杨晓光, 郭云昌, 等. 2011. 乳酸杆菌脉冲场凝胶电泳分子分型的条件优化[J]. 食品工业科技, 32(2): 178-190. |
| Agapi ID, Spiros P, George-John EN. 2011. Characterization of the Enterobacteriaceae community that developed during storage of minced beef under aerobic or modified atmosphere packaging conditions[J]. International Journal of Food Microbiology, 145(1): 77-83. |
| Balsari A, Ceccarelli A, Dubini F, et al. 1982. The fecal microbial population in the irritable bowel syndrome[J]. Microbiologica, 5(3): 185-194. |
| Blaiotta G, Simeoli GM, Andolfi R, et al. 2001. Monitoring lactic acid bacteria strains during 'Cacioricotta' cheese production by restriction endonuclease analysis and pulsed-field gel electrophoresis[J]. J Dairy Res, 68(1): 139-144. |
| Buchanan RE, Gibbons NE. 1974. Bergey's Manual of determinative bacteriology, eighth[M]. Baltimore: Williams and Wilkins: 800-820. |
| Carol I, Lee L, Michael W, et al. 2006. Identification of Enterobacter sakazakii from closely related species: The use of Artificial Neural Networks in the analysis of biochemical and 16S rDNA data[J]. BMC Microbiology, 6(2): 28. |
| Chantal LB, Catherine S. 2011. Sugar metabolism, an additional virulence factor in Enterobacteria[J]. International Journal of Medical Microbiology, 301(1): 1-6. |
| Charteris WP, Kelly PM, Morelli L, et al. 1998. Antibiotic susceptibility of potentially probiotic Lactobacillus species[J]. Journal of Food Protection, 61: 1636-1643. |
| CLSI. 2009. Performance Standards for Antimicrobial Susceptibility Testing: Nineteenth Informational Supplement[M]. CLSI document M100-S19, Wayne, PA: Clinical and Laboratory Standards Institute. |
| Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap[J]. Evolution, 39(4): 783-791. |
| Gatti M, Lazzi C, Rossetti L, et al. 2003. Biodiversity in Lactobacillus helveticus strains present in natural whey starter used for Parmigiano reggiano cheese[J]. J Appl Microbiol, 95(3): 463-470. |
| Glenn JM, David KS, Joan NH, et al. 1995. Enterococci resistant to multiple antimicrobial agents, including vancomycin[J]. Ann Intern Med, 123(4): 250-259. |
| He SX, Zhou ZG, Liu YC, et al. 2012. Do dietary betaine and the antibiotic florfenicol influence the intestinal autochthonous bacterial community in hybrid tilapia (Oreochromis niloticus ♀×O. aureus ♂)?[J]. World Journal of Microbiology Biotechnology, 28(3): 785-791. |
| Kluge AG, Farris JS. 1969. Quantitative phyletics and the evolution of the anurans[J]. Systematic Zoology, 18(1): 1-32. |
| Liu Wenjun, Bao Qiuhua, Jirimutu, et al. 2011. Isolation and identification of lactic acid bacteria from Tarag in Eastern Inner Mongolia of China by 16S rRNA sequences and DGGE analysis[J]. Microbiol Res, 167(2): 110-115. |
| Mhida YR, Rolfe VE. 2004. Host-bacterial interactions in inflammatory bowel disease[J]. Clinical Science, 107(4): 331-341. |
| Ojetti V, Gigante G, Ainora ME, et al. 2009. Microflora imbalance and gastrointestinal diseases[J]. Digestive and Liver Disease Supplements, 3(2): 35-39. |
| Ribot EM, Fair MA, Gautom R, et al. 2006. Standardization of pulsed field gel electrophoresis protocols for the subtyping of Escherichia coli O157:H7, Salmonella, and Shigella for PulseNet[J]. Foodborne Pathogens and Disease, 3(1): 59-67. |
| Saitou N, Nei M. 1987. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology Evolution, 4(4): 406-425. |
| Schouten MA, Voss A, Hoogkamp-Korstanje JAA, et al. 1999. Antimicrobial susceptibility patterns of enterococci causing infections in Europe[J]. Antimicrob Agents Ch, 43(10): 2542-2546. |
| Sekirov I, Tam NM, Jogova M, et al. 2008. Antibiotic-induced perturbations of the intestinal microbiota alter host susceptibility to enteric infection[J]. Infect Immun, 76(10): 4726-4736. |
| Sheu DS, Wang YT, Lee CY. 2000. Rapid detection of polyhydroxyalkanoate-accumulating bacteria isolated from the environment by colony PCR[J]. Microbiology, 146: 2019-2025. |
| Simpson WJ, Hammond JRM, Miller RB. 1988. Avoparcin and vancomycin: useful antibiotics for the isolation of brewery lactic acid bacteria[J]. J Appl Bacteriol, 64(4): 299-309. |
| Steven G, Jana MS, Bertha CH, et al. 1992. Antimicrobial Susceptibility Patterns of Common and Unusual Species of Enterococci Causing Infections in the United States[J]. J Clin Microbiol, 30(11): 2373-2378. |
| Swenson JM, Facklam RR, Thornsberry C. 1990. Antimicrobial Susceptibility of Vancomycin-Resistant Leuconostoc, Pediococcus, and Lactobacillus Species[J]. Antimicrob Agents Ch, 34(4): 543-549. |
| Yang QS, Meng XX, Xia L, et al. 2003. Conservation status and causes of decline of musk deer (Moschus spp.) in China[J]. Biological Conservation, 109: 333-342. |
| Zhang E. 1998. Musk deer[M].// Wemmer CM, McCarthy A, Blouch R, et al. (ed.). Deer: Status Survey and Conservation Action Plan. Gland: IUCN: 72-77." |
 2015, Vol. 34(1)
2015, Vol. 34(1)




